(2 生物医学大数据中心,中国科学院上海营养与健康研究所,上海 200032)
(3 中国科学院南京地理与湖泊研究所,湖泊与流域水安全全国重点实验室,南京 211135)
(4 兰州大学泛第三极环境中心,兰州 730000)
(5 上海交通大学自然科学研究院,上海国家应用数学中心(上海交大分中心),上海 200240)
(2 Bio-Med Big Data Center, Shanghai Institute of Nutrition and Health, Chinese Academy of Sciences, Shanghai 200032, China)
(3 Key Laboratory of Lake and Watershed Science for Water Security, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 211135, China)
(4 Center for the Pan-third Pole Environment, Lanzhou University, Lanzhou 73000, China)
(5 Shanghai National Center for Applied Mathematics (SJTU Center), Institute of Natural Sciences, Shanghai Jiao Tong University, Shanghai 200240, China)
地球水圈作为连接大气、地表与深部多圈层的关键纽带,蕴藏着极其丰富的微生物资源。从浅海到深渊、从冰川到热泉、从淡水湖泊到超盐水体,不同水圈生境的理化条件差异巨大,形成了对微生物群落的独特“环境约束”——即温度、压力、pH、营养等环境因子对微生物组成、代谢功能和进化方向的选择性作用。这种环境约束不仅塑造了微生物的适应性进化,也使得特定环境的微生物群落成为“生命记录”的载体,能够反映和追溯环境的历史变化过程。过去二十年间,受益于微生物组学研究技术方法的发展,以Earth Microbiome Project (EMP)、Tara Oceans、我国水圈微生物重大研究计划等为代表的大型研究项目,积累了超过200 Tb (Tbase)的水圈微生物组学数据。然而,水圈微生物的环境适应表现为“多因一果、多果一因”的复杂模式(多个环境因子共同作用产生相似的适应性状,而同一环境因子又可能导致不同的适应策略),这种复杂关系超越了传统分析方法的处理能力,亟需新的研究手段和高维视角。
人工智能(artificial intelligence, AI)正在带来革命性的新机遇,但要成功应用到微生物组研究并带来突破性发现,必须克服三个关键难点和挑战:(1)高质量标注的大数据集;(2)可验证的金标准的建立;(3) AI模型及其结果的可解释性。首先是要获取高质量标注的大数据集,尽管当前AI相关研究均已认识到大数据积累的重要性,但以国内外水圈相关微生物研究为例,尽管已积累超过200 Tb的组学数据,但环境参数标注不完整、采样偏差严重、数据标准不统一等问题一度制约了AI模型的训练效果,亟需建立涵盖温度、压力、pH、营养等多维度环境因子的标准化数据体系(详见章节1、2)。其次,建立可验证的金标准,则必须将AI预测结果与已有的微生物生态学理论相互印证,包括“物种皆在、环境选择”的生物地理学规律、功能冗余维持生态系统稳定性的机制,以及“多数功能无处不在”的跨生境共适应策略等理论迭代,只有在这些理论框架下得到验证的AI预测才能进一步通过实验方法加以巩固和拓展,避免AI模型产生违背生物学基本规律的“幻觉”(详见章节3、4、5)。最后是如何提升AI模型及其结果的可解释性,即,如何让深度学习的“黑箱”模型输出转变成可被生物学家理解的机理认知,如何从高维特征空间映射回具体的生物学过程,这是当前国内外研究中尚未解决的难题,需要更多跨领域的深入研究(详见章节6)。
本文系统梳理了水圈微生物组研究的数据基础和理论进展,针对AI应用的三个关键难点,首先综述了我国在典型水圈生境积累的独特数据资源,为构建和验证环境感知型AI模型提供了得天独厚的条件;随后重点阐述了微生物生态学理论的迭代和多组学新技术的发展可能为AI提供的理论指导和数据约束;最后展望AI技术如何通过序列大模型、几何深度学习、多模态数据融合等方法实现与传统理论的深度融合,推动从物种分类到功能预测、从静态描述到动态追踪、从现象观察到机制解析的研究范式转变,为理解“环境如何约束生命”和“生命如何记录环境”这两个核心科学问题提供高维视角和全新的解决方案。
1 国内外水圈相关微生物组研究计划 1.1 国际水圈相关微生物研究计划Earth Microbiome Project (EMP)作为全球性大规模微生物组研究项目,覆盖了包括海洋在内的多种环境[1],成功生成了大量16S rRNA基因序列和宏基因组数据,通过QIITA、NCBI、MG-RAST等公共数据库平台提供数据共享,支持微生物群落和功能的多组学分析,在揭示全球微生物群落分布模式方面发挥了重要作用。
Tara Oceans计划(2009?2013)是针对表层海洋的国际研究计划,由23家国家研究所支持,历时38个月,航行140 000公里,在全球210个采样站系统收集了超过35 000个海洋水体和浮游生物样本。项目生成总数据量超过54.19 Tb,大规模揭示了表层海洋中约150 000种真核生物[2],阐明了表层海洋病毒群落的分布模式及其与环境因素的关系[3],并建立了开放的数据资源平台,支持多个海洋微生物数据挖掘工作[4-6]。
Malaspina Expedition 2010由西班牙科学与技术部发起,基于海洋科考船Sarmiento de Gamboa和Hespérides进行环球科考,在2010年至2011年间采集了热带和亚热带海洋的微生物群,从表层到深海(约4 000米深度)的垂直剖面数据[7]。该项目总航行约42 000海里,通过350个采样站采集了超过70 000个样本,通过122个站点的微小浮游生物样本(0.2~3 μm)16S rRNA基因测序分析以及76个宏基因组数据集,共识别了超过15 000个扩增子分类单元和2 672个高质量的宏基因组组装基因组,总数据存储量超过1.6 Tb[8]。
GEOTRACES项目于2006年由国际海洋科学联合会发起,约35个国家参与,其生物学扩展项目bioGEOTRACES在2010?2011年间建立了480个大西洋和太平洋样本的宏基因组数据集,提供了与物理和化学测量相关的跨时空微生物群落快照[9, 10]。
国际上对于冰冻圈微生物的研究计划主要集中在南北极区域。其中,北极MOSAiC科考计划(Multidisciplinary Observatory for Study of the Arctic Climate,2019?2020)在一年漂移观测的73个样本中获得了2 407个原核和56个真核基因组信息,研究了北极区域微生物群落动态,解析了其对气候变化的响应机制[11]。欧盟Marie Sklowdowska Curie Actions资助的MicroArctic计划聚焦北极微生物对气候变暖的适应能力及其在碳循环中的反馈机制。国际上对于同样属于冰冻圈的世界“第三极”青藏高原微生物研究计划较少,相关重大研究计划是由我国主导的。
1.2 我国水圈微生物研究计划2017?2021年,在我国科技部基础资源调查项目“中国湖泊微生物多样性及资源调查”资助下,我国科学家对中国五大湖区(青藏高原湖区、云贵高原湖区、东北平原与山地湖区、蒙新湖区和东部平原湖区)的湖泊微生物多样性及其资源开展了系统调查,在物种、基因和宏基因组水平上,掌握了我国湖泊微生物的多样性分布特征、资源现状和形成机理,获得了具有重要生态功能的微生物菌种资源,构建了中国湖泊微生物物种资源库及功能基因资源库,建设了中国湖泊微生物资源信息管理系统,为我国湖泊生态系统保护、生物资源利用以及环境治理等方面提供了基本材料和基础数据。
在我国“水圈微生物驱动地球元素循环的机制”重大研究计划(以下简称水圈微生物研究计划)的支持下,我国的水圈微生物研究具有明显的地域特征,主要覆盖了我国及周边区域的四大典型水圈生境——深海、浅海、冰川和湖泊。其中,青藏高原作为“世界屋脊”和“地球第三极”,其独特的地质-地理-生态特征使其成为地球与生命演化研究的天然实验室。2022年,在水圈微生物研究计划和第二次青藏高原综合科学考察研究资助下,我国科学家发布了全球首个冰川微生物数据库——青藏高原冰川微生物基因组及基因数据集,囊括了21条冰川的85个宏基因组样品和883株可培养菌株,获得了3 241个冰川细菌和古菌基因组,其中88.3%~100%为潜在新种,具有强烈的空间和生境特异性[12]。2023年,首个全球冰川表面生态系统DNA病毒数据集(SgVG)发布,获得10 840个冰川DNA病毒基因组,将原有数据扩大了15倍[13]。此外,建立的冰川抗性基因数据集研究显示,冰川抗性基因丰度与周边国家抗生素使用量呈显著正相关,表明抗性基因可能随印度季风传播至青藏高原冰川[14]。
随着我国深海探测工程技术的发展,我国的深渊微生物研究也进入了快车道。深渊是指海平面以下超过6 000米水深的海洋区域,以马里亚纳海沟挑战者深渊为代表,最深处可达约10 909米,是地球最难企及且最少被人类探索的极端环境之一。2021年,在“水圈微生物驱动的元素循环”重大研究计划等项目支持下,由上海交通大学、中国科学院深海科学与工程研究所、华大集团等多家单位合作发起的MEER计划(溟渊计划),依托“奋斗者”号万米级载人潜水器,对马里亚纳海沟、雅浦海沟和菲律宾海盆6 000~11 000米水深区域进行了系统探索,采集了1 662份深渊沉积物样本,通过宏基因组测序和16S rRNA基因扩增子测序,构建了包含93.9 Tb核酸信息的MEER微生物数据集,涵盖7 564个物种,其中89.4%为未报道的新物种,体现了极高的分类学新颖性,展现了深渊异常繁荣的微生物生态系统[15]。
1.3 水圈微生物研究计划聚焦的重大科学问题综观国内外水圈微生物研究计划,虽然研究区域和技术手段各异,但都聚焦于解决五个相互关联的重大科学问题:一是生命的环境适应极限与进化机制,如MEER计划揭示的深渊生命对高达1 100个大气压的适应机制、青藏高原冰川微生物在极低温和强紫外线复合胁迫下的生存策略及基于抗生素抗性的互作机制,这些发现不仅拓展了生命边界认知,也为天体生物学提供了重要参考;二是微生物驱动的元素循环与气候调节,包括Tara Oceans量化的海洋初级生产力对全球碳汇的贡献、GEOTRACES追踪的微生物介导的海洋铁硅循环、青藏高原冰川微生物温室气体释放的正反馈效应、MEER聚焦的深渊碳循环作用,为深入认识全球气候变化提供了微生物维度的科学依据;三是环境变化的生物响应与生态系统稳定性,通过生态学理论框架,解释微生物群落如何在组成发生剧变时维持生态功能稳定;四是微生物资源挖掘与生物技术应用,各计划发现的大量新物种和新基因为工业催化、医药开发、环境修复提供了宝贵资源;五是方法学创新与数据标准化,推动了从采样技术到多组学分析的技术革新。这些科学问题共同构成了水圈微生物研究的核心框架,也为人工智能技术在理论创新、机制解析、功能预测等方面的应用指明了方向。
2 水圈微生物组大数据变化趋势随着组学技术和大数据分析方法的快速发展,水圈微生物组学研究进入大数据时代(图 1)。MG-RAST[16]、MGnify[17, 18]等国际化数据存储与共享平台不仅为全球科研社区提供了海量公开数据,还建立了标准化的数据格式和质控体系。我国近年来建立了国家组学数据百科全书(NODE,https://www.biosino.org/node/home)这一统一的数据存储和整理平台[19],打造开放共享的数据生态,为推动水圈微生物组学研究快速发展提供了重要平台基础。相较传统组学原始数据库(如NCBI-SRA、GSA等),NODE数据库不局限于高通量测序数据,而是支持包括宏基因组、宏转录组数据以及基于质谱技术的蛋白质组、代谢组及芯片等各种数据类型,并加以标准化汇聚和整合。同时,在微生物组标准化元数据汇交方面,NODE针对海洋、湖泊、湿地等生境以及水体、沉积物等样本设计了对应的元数据标准和控制词表,引导用户提交标准化、归一化的环境微生物元数据,为数据整合提供了高质量数据基座(图 1)。基于NODE平台的数据汇总情况,我们从数据总量变化、类型演进以及生物群落研究重点转移三个维度,对水圈微生物组大数据的发展趋势及其驱动因素进行系统论述。
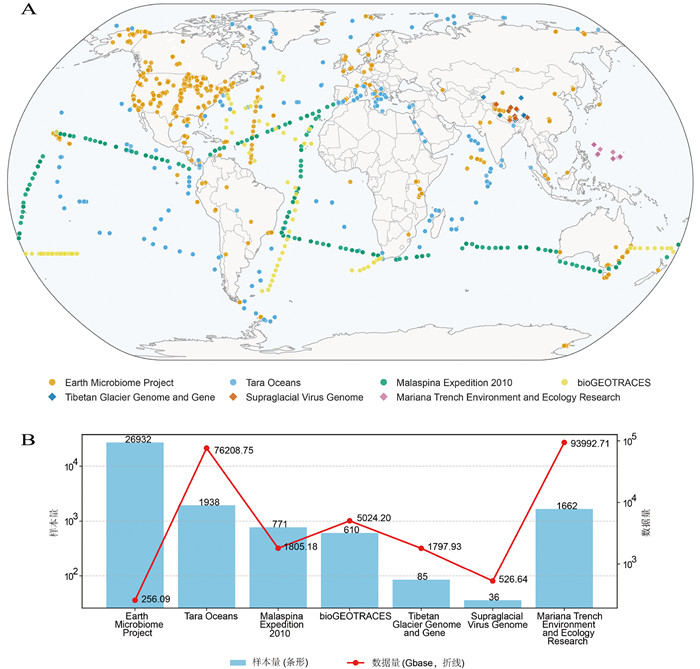
|
图 1 国内外水圈相关微生物组研究计划概览(仅统计已公开数据) |
总体来说,水圈微生物组数据量呈现出显著的指数级增长态势。以海洋微生物测序数据量为例,近年来数据量增长呈现明显的阶段性特征,四次激增主要由重大科研项目驱动:2014年Tara Oceans总项目(PRJEB402)提交数据11.7 Tb,占当年海洋样本测序数据总量(14.5 Tb)的80.7%[20];2016年Tara Oceans子项目提交数据13.8 Tb,占当年总量(21.4 Tb)的64.5%[21];2023年Tara-Pacific项目提交数据34.8 Tb,占当年总量(83.3 Tb)的41.8%[22];2025年MEER计划提交数据92 Tb,成为近十年来最大的海洋微生物数据集,也是唯一的深渊微生物大数据集[23],表明我国在海洋微生物数据积累中日趋重要的角色(图 1B)。
2.2 数据类型演进:从单一组学向多组学整合发展水圈微生物组学研究经历了从单一16S rRNA基因扩增子测序向多组学方法整合的重要转变。早期研究主要集中在16S rRNA基因扩增片段的测序上,这种方法能够有效揭示微生物的分类学信息和群落结构特征。随着测序技术进步和成本下降,宏基因组、宏转录组、蛋白质组和代谢组等多组学数据逐渐成为研究主流。多组学数据的变化趋势明显受到大型科研项目驱动:2021?2022年期间呈现从宏基因组向宏转录组转变的趋势,主要由Tara Oceans项目执行7年后由宏基因组转向宏转录组[24]以及新冠期间兴起的环境污水病毒检测需求导致[25]。这种从单一组学到多组学数据的转变使得研究者不再局限于微生物的分类学分析,而能够深入探索其功能潜力。
2.3 微生物群落研究重点的转移:从常规到极端,从物种到功能随着人类对未知生境的探索范围不断扩大,水圈微生物组学研究从常规环境向极端环境转变。深海、深渊、热液、极地等极端生境逐渐成为研究热点,这些极端环境中的微生物往往具有独特的生存策略和代谢潜能,在全球碳、氮、硫等生物地球化学循环中扮演关键角色[26],不仅拓展了我们对生命极限的认知,也为寻找新资源和生物技术应用提供了重要机遇[27]。
与此同时,研究焦点也从单纯的物种分类转向基因功能解析及其在生物地球化学循环中的作用。现代水圈微生物研究越来越重视功能基因的挖掘和代谢通路的重建,特别是与碳、氮、硫、磷等元素循环相关的关键基因。例如,深海微生物在全球碳循环中扮演关键角色,其化能自养固碳作用是深海生态系统的能量基础[27];冰川微生物通过光合作用和化能合成作用参与碳固定,同时其呼吸作用释放的CO2和CH4对全球温室气体平衡产生影响[12];热液口微生物的硫氧化和硫还原过程驱动了深海硫循环,维持了独特的化能合成生态系统[26]。通过宏基因组、宏转录组等多组学技术,研究者不仅能够识别新的功能基因,还能量化微生物介导的元素通量,评估其对全球生物地球化学循环的贡献。这种从“谁在那里”到“它们在做什么”以及“对地球系统有何影响”的研究范式转变,使得水圈微生物研究从描述性科学发展为预测性科学,为理解地球系统运行机制和应对全球环境变化提供了微生物视角的关键信息。
此外,在“One Health”理念驱动的研究框架下,环境微生物组研究将人类、动物与环境健康视为同一体系的耦合要素,不再采用“自然-人为”的二分叙述。废水处理厂、塑料污染海域与城市河流等被视作关键“哨点”生境,用于解析人类活动强度梯度下的群落结构与功能变化,追踪病原体与抗生素耐药性因子的迁移与富集,并评估化学污染与营养负荷对微生物介导的物质循环的影响[28, 29]。这一系统化视角为跨部门监测与预警网络的建设、微生物驱动的污染控制与生物修复技术开发,以及与健康相关的环境风险评估方法提供了可验证的机制基础与证据链[30]。
总体而言,水圈微生物组学数据正呈现出数量与类型并重的快速发展态势。从16S rRNA单一标记基因测序到多组学数据整合分析的技术转型,使得研究者能够在多个层次上深入探讨微生物在全球生物地球化学循环、气候调节和污染治理中的关键作用。同时,生物群落研究范围从传统海洋环境向极端环境和人为影响环境的扩展,反映了水圈微生物组学研究主题的多样化发展趋势和学科交叉融合的深化程度。
3 水圈微生物组的理论迭代:区分物种与功能随着水圈微生物组学研究的深入发展,我们对微生物群落结构与功能关系的认知正在经历深刻的理论迭代。从经典的“物种皆在,环境选择”理论到功能冗余概念的提出,再到“多数功能无处不在,而环境选择特定类群”的新认知,这些理论发展不仅丰富了我们对微生物生态学规律的理解,也为跨生境微生物组研究提供了重要的理论框架。
3.1 “物种皆在,环境选择”理论的适用边界早期在对微生物组认知有限的情况下,荷兰微生物学家Martinus Wilhelm Beijerinck在20世纪初提出了“物种皆在,环境选择”(Everything is everywhere, but the environment selects)的理论,认为微生物由于个体微小、种群数量巨大和扩散能力强能广泛分布,而特定环境条件决定哪些微生物能够繁衍生存[31]。该理论长期在微生物生物地理学研究中被广泛接受,在温带海洋、湖泊和土壤等常规生境中得到有力支撑。
然而,近年来跨生境的宏基因组学研究开始挑战这一理论的普适性。Coelho等[32]在2022年构建了包含3.03亿个物种水平基因的全球基因目录,分析来自14个主要生境的13 174个宏基因组样本,发现大多数物种水平基因都具有生境特异性,仅有少数物种水平基因在多个生境中发现。Breitbart和Rohwer[33]发现虽然环境病毒群落表现出极高的多样性(200升海水中有5 000种病毒基因型),但病毒可以在不同生物群落间移动,暗示病毒多样性在局部尺度上很高,但在全球尺度上相对有限。在极端环境中,理论的适用边界更加明显,深海深渊、热液喷口、极地冰川、超盐湖泊等极端生境中的微生物往往表现出强烈的地理隔离特征和物种的高度特化。如一项针对青藏高原冰川微生物的研究发现,88.3%~100%的微生物为潜在新种且主要分布在单一冰川,表明在极端条件下地理隔离可能超越环境选择成为塑造微生物群落结构的主导因素[12]。
3.2 功能冗余:微生物群落稳定性的重要机制研究者在微生物混合培养体系和天然环境中均发现了微生物群落组成与其代谢功能之间的脱钩现象,并提出了“功能冗余”这一概念,即在生态系统中能够执行相同生物地球化学功能的多个不同分类群或基因组共存的现象[34]。在藻类混合培养体系中,尽管藻类群落组成发生了动态变化,但藻类生物量依然维持稳态[35]。在脱氮或甲烷生产实验中也发现,尽管微生物群落组成发生了显著变化,但生物反应器的性能却保持稳定[36]。在自然环境中,尽管青藏高原盐湖沉积物中微生物群落和硫酸盐还原微生物在不同湖泊中差异巨大,但都维持着硫循环的能力[37]。
这种“功能冗余”可能是微生物系统的固有特性,对维持生态系统稳定具有重要意义[34, 38]。如果随着物种丰富度的增加,功能丰富度趋于饱和,则表明微生物群落中存在功能冗余,而功能冗余度在不同环境中表现出显著的差异性[34]。一项利用微生物模型系统探索不同环境下生物多样性与生态系统功能关系的研究表明,在一种环境中表现为功能冗余的模式,在另一种环境中可能转变为功能差异模式,这种变化源于物种角色和种间相互作用的动态调整[39]。在高寒草原生态系统中,微生物的分类多样性与其功能组成之间存在强烈的正相关性,表明该生态系统中的功能冗余度相对较低[40]。这些发现不仅丰富了我们对微生物生态学的理解,也为保护和管理生态系统提供了新的策略。
然而,近期对地中海西北沿海海洋微生物群落的宏基因组监测研究对这种“功能冗余”假说提出了挑战,该研究发现微生物群落组成的变化会改变群落的整体功能属性,表明海洋微生物多样性实际上也反映了微生物代谢的巨大多样性。这一发现并不完全排除部分功能冗余的可能性,但强调了传统功能冗余概念在海洋环境中的适用边界[41]。
3.3 物种与功能的新认知:“多数功能无处不在,而环境选择特定物种”随着跨生境微生物组学研究的深入,我们通过比较珠穆朗玛峰(强日照、低压、温度波动)和马里亚纳海沟(黑暗、高压、低温)这两种截然不同的微生物生态系统,提出新的理论模式:“多数功能无处不在,而环境选择特定类群”,即虽然不同环境中的微生物物种组成可能存在巨大差异,但其代谢功能却表现出惊人的相似性[42]。
跨生境的宏基因组测序分析发现,珠峰和马沟的微生物群落在分类组成上表现出极大差异:在属水平上,两类环境中共有微生物属仅有8个,而在种水平上只有一种(Comamonas tsuruhatensis)。然而,两种截然不同生境中微生物的代谢能力却表现出惊人共性:超过90%的代谢模块重合,特异性代谢模块分别仅为1%和8%。唯一显著差异在于CO2固定途径——珠峰主要采用光合自养型,马沟则采用化学自养型。
这种功能的保守性体现了不同生境微生物的“共适应”策略——微生物应对不同极端环境胁迫的通用应对策略。仍以马沟和珠峰微生物的对比为例,珠峰的强烈紫外线和马沟的超高压本质上都会通过产生活性氧(Reactive oxygen species, ROS)的方式对细胞造成过氧化损伤,而清除ROS的抗氧化途径在两种极端生境的微生物类群中广泛存在,它们都富集了完整的ROS清除酶系统,包括多种超氧化物歧化酶(SOD)家族——铜锌型SOD (sod1)、铁锰型SOD (sod2)和镍型SOD (sodN),以及过氧化氢酶(katE和katG)。这两个极端环境的核心ROS清除机制与其他环境中已知的途径保持一致[42]。这种“核心功能保守、辅助功能特化”的模式,揭示了极端环境微生物既有应对氧化应激的通用策略,又有针对特定环境压力源的精细调控,体现了跨生境“共适应”策略的本质。跨生境的“共适应”策略可能代表了维持生命活动的普适性功能,这一新认知提示我们微生物生态系统的核心功能可能具有较强的稳定性,在预测生态系统对环境变化的响应、评估生态风险以及制定生态保护策略时应充分考虑区分通用策略和生境特异性策略。
4 代谢功能指示环境对微生物的约束环境约束是指水圈生境中的物理、化学和生物因子对微生物施加的选择压力,这些约束因子通过自然选择机制塑造了微生物的代谢策略和功能特征。在水圈生态系统中,环境约束表现为多个层次:物理约束包括温度梯度(从极地冰川的-20 ℃到热液口的400 ℃)、压力变化(从表层到深渊的1 100个大气压)、光照分布(从透光层到永久黑暗区);化学约束涵盖pH范围(从酸性尾矿的pH 2到碱性湖泊的pH 11)、盐度差异(从淡水到饱和盐湖)、氧化还原状态(从富氧表层到缺氧深层)、营养可用性(从富营养近岸和湖泊到寡营养大洋);生物约束则包括资源竞争、代谢互补、协同共生等种间关系。这些环境约束不是孤立作用,而是形成复杂的约束网络——例如深渊的高压与低温协同限制了代谢速率,极地冰川的低温与强紫外线共同诱导氧化应激,热液口的高温与化学毒性同时挑战蛋白质稳定性。微生物通过调整基因组大小、优化代谢通路、发展特殊酶系统等策略来应对这些约束,其代谢功能组成因此成为环境约束的直接体现。
AI技术为解析环境约束与代谢功能的复杂关系提供了强大工具。传统方法难以处理“多因一果”(多个环境因子导致相同代谢响应)和“一因多果”(单一环境因子引发多种适应策略)的复杂模式,而AI能够从高维数据中自动学习环境约束的组合效应。深度神经网络可以识别哪些代谢模块组合对应特定的环境约束类型,如深海和高山冰川微生物虽处于不同环境,但都富集抗氧化通路来应对各自的氧化应激;随机森林算法能够评估不同环境因子对代谢功能的相对贡献度,区分主导约束和次要约束;图卷积网络可以构建“环境-代谢-物种”三层网络,揭示环境约束如何通过代谢重编程影响微生物群落组装和功能分配。更重要的是,AI模型能够预测未观测环境下的微生物代谢潜能,为极端环境的生物勘探、污染环境的生物修复,以及全球变化下的生态系统响应预测提供科学依据。这种从代谢功能反推环境约束、从环境约束预测代谢潜能的双向推断能力,正在成为理解和利用水圈微生物资源的关键技术。
4.1 基于代谢潜能的环境约束推断与常规环境相比,极端环境对微生物的塑造更直接地体现在代谢偏好上,因此,根据微生物代谢潜能也能够推断极端环境的关键影响。以深渊环境为例,与浅海沉积物、土壤或水体等环境中中性进化过程发挥重要作用不同,深渊微生物组主要受到均质选择(Homogeneous selection, HoS)、扩散限制(Dispersal limitation, DL)和异质选择(Heterogenous selection, HeS)过程的驱动。HoS代表存在稳定一致的环境选择压力,使得微生物群落的组成趋同以占据相似的生态位[43]。HoS在塑造深渊微生物组中的主导地位反映了极端深渊条件的广谱影响,受HoS影响的微生物类群代谢偏好表现为抗氧化(应对高压和低温诱导的ROS)以及芳香族化合物利用(应对深渊寡营养条件)。相反,DL代表微生物群落之间的物种扩散交流受到阻隔,而HeS代表不同的环境因素分别塑造了不同的微生物群落[43]。在深渊中,DL主导和HeS影响的微生物代表了多能型策略,具有较大的基因组和多样化的代谢能力,表明其采用机会主义的生存策略。DL和HeS驱动的代谢偏好暗示了除高压之外的其他环境因子对深渊微生物的影响,DL突出了微生物相互作用,而HeS指向了沉积物深度氧化还原梯度对微生物群落的塑造[15]。这种基于微生物群落特征推断环境影响的研究思路,对于难以进行直接环境测量的极端生境尤为重要,也为地球科学、地球化学过程等领域的研究提供了微生物视角的线索。
4.2 多组学综合技术解析活跃功能与环境的相互作用为深入理解微生物功能与环境的关系,区分活跃与沉默的微生物群体及其对应的代谢功能对于理解生态系统功能具有重要意义。转录组学和蛋白质组学技术为揭示微生物群落的实际活性提供了有力工具。其中,宏蛋白质组学为验证微生物群落活性提供了更为直接的方法,可同时检测原核生物和病毒的蛋白质表达,获得活跃功能及对应的微生物类群信息。早期的宏蛋白质组学研究主要采用二维凝胶电泳结合MALDI-TOF和LC-MS/MS技术,但受限于蛋白质鉴定的成功率较低,应用范围有限[44]。随着高分辨率串联质谱技术的发展,当前宏蛋白质组学在具有较高生物量的简单样本(如生物组织、肠道微生物等)中已能够实现较好的蛋白鉴定成功率,单一样品已能够获得上万个鉴定蛋白[45, 46]。然而,宏蛋白质组在水圈环境微生物样品中(尤其是极端环境样品)的应用则往往难以获得较好的蛋白鉴定成功率(一般单个样品蛋白鉴定数在几十到几百)[47-49]。这主要受到两个关键因素的制约。首先,宏蛋白质组学分析需要较大的生物量,这在水圈环境中往往难以获得:例如,深渊海水中的细胞密度(每毫升103~104个细胞)比表层海水低一到两个数量级[50],在寡营养环境如北太平洋的贫营养区域中,低氮素区域普遍存在细菌数量稀少的问题[51]。冰川融水河流(GFS)是寒冷、寡营养且不稳定的生态系统,其中的生命主要以微生物生物膜为主[52],冰川冰中的微生物生物量极低(细胞浓度范围为每毫升102~104个细胞),且通常只能获得小体积的冰样[53]。其次,可靠的宏蛋白质组学分析需要全面且精心构建的基因序列数据库来进行准确的光谱鉴定,这在具有较多未知信息的极端生态系统中构成了重大挑战。
代谢组的概念最早由Steven Oliver在1998年提出[54],其命名类比于转录组和蛋白质组。早期的代谢组研究主要集中于特定代谢物的靶向检测,方法包括分光光度学和色谱学等[55],这些研究奠定了对经典代谢通路的认知,如三羧酸循环和Calvin-Benson循环[56, 57]。进入21世纪,基于质谱(MS)的检测方法带来了巨大变革。非直接进样的气相色谱-质谱(GC-MS)[58, 59]和直接进样的高分辨率质谱[60],实现了数十至上千代谢物的检测,随后高分辨率质谱与液相色谱(LC)[61]、超高效液相色谱(UPLC)[62]联用的发展显著提升了检测的分离度和灵敏度,使代谢组学能够广泛应用于微生物组研究。然而,在环境样品中(尤其是极端环境)开展胞外代谢组学(exometabolomics)分析存在巨大挑战。其关键难点在于低生物量的极端环境样品中基质的复杂性会掩盖微生物的代谢物信号,比如在富含高浓度(毫摩尔级至摩尔级)盐和矿物质的苏打湖、酸性高盐湖、海盐晒池及深海盐卤池、深海多金属结核区等极端水圈环境中[63],复杂的本底基质会显著影响代谢物的分离与提取效率[64],并在LC-MS分析过程中强烈抑制代谢物的电离,进而造成代谢物定量性能下降,甚至产生系统性偏差[65-67]。这些因素都制约了代谢组在水圈环境微生物,尤其是极端环境微生物中的应用拓展。
综上,这些方法学挑战凸显了针对低生物量的复杂极端环境微生物样品开发高通量多组学提取和鉴定技术的迫切需求[68]。随着技术的不断发展,整合转录组学、蛋白质组学和代谢组学的综合多组学方法有望为环境微生物组研究提供更加全面和精准的技术支撑。特别是在极端环境微生物群落活性检测方面,建立低生物量、培养无关的“清洁”程序来调查微生物[53]具有重要的应用前景,这将为理解全球气候变化背景下水圈环境微生物群落的功能动态提供关键技术支撑。
5 微生物物种记录环境动态变化过程微生物群落作为环境信息的生物载体,能够记录、保存和反映其所处生境的综合特征,形成可被解读的“生命记录”。这种生命记录具有三个关键特征:首先是信息综合性,微生物群落整合了物理(温度、压力、光照)、化学(营养、pH、盐度)和生物(种间关系、食物网结构)等多维环境信息,比单一环境参数测量更全面地反映生境特征;其次是时间累积性,不同于瞬时的环境测量,微生物的基因组特征、群落演替和功能变化记录了环境条件的长期累积效应和历史变化轨迹,如青藏高原冰川微生物记录了气候变暖过程,深海沉积物微生物保存了海洋环境的演化历史;第三是反向推断能力,当直接环境测量受限时——无论是深渊的超高压、热液口的极端温度,还是冰川内部的封闭环境——都可通过解读微生物生命记录来推断环境特征,这种“从生物推环境”的策略为理解地质过程、元素循环、气候变化等提供了独特视角。
AI技术正在革新我们解读水圈微生物生命记录的能力。通过深度学习算法,可以建立微生物组成模式与环境参数之间的复杂映射关系,实现双向预测:既能从环境条件预测微生物群落结构,也能从微生物特征反推环境状态。例如,机器学习模型可以整合来自全球海洋、极地冰川、高原湖泊、深海热液等多样化水圈生境的微生物数据,学习不同环境压力下的微生物响应模式,识别跨生境的共性规律和环境特异性特征。图神经网络能够捕捉微生物互作网络与环境因子网络的耦合关系,揭示环境变化如何通过生态网络传导并被记录在群落结构中。时序深度学习模型可以从微生物群落的动态变化中重建环境演变历史,预测未来趋势。这种AI赋能的生命记录解读不仅帮助我们克服极端环境研究的技术瓶颈,还为环境监测、气候变化评估、生态系统监测提供了基于生物信息的新工具,开辟了从微生物视角理解地球系统的新途径。
5.1 基于扩增子的微生物群落研究基于前文所述微生物物种与功能的解耦关系,“环境选择特定物种”意味着具有环境特异性的微生物物种能够记录并反演所在环境的动态变化过程。自20世纪70年代Woese和Fox首次使用16S rRNA基因作为细菌和古菌的分类依据以来[69],传统微生物生态学研究主要依赖16S rRNA基因扩增子分析来推断群落组成与环境的关系,并在此基础上发展出一系列生态分析方法,如中性理论模型、群落组装分析等。基于16S rRNA基因扩增子的方法虽然具有成本低、操作简便、数据库成熟等优点,但存在根本性局限:只能提供分类信息而无法直接获得功能信息,使得生态过程分析停留在“谁在那里”的层面,无法回答“它们在做什么”。更严重的是,16S rRNA序列相似甚至完全一致的细菌在功能上可能截然不同——例如Pseudomonas fluorescens与Pseudomonas putida的16S rRNA基因序列几乎完全相同,但前者在植物根际中具有生物防治功能,而后者则以其在环境中降解污染物的能力而闻名[70];大肠杆菌的不同菌株虽然在16S rRNA序列上高度相似,但致病性株如肠出血性大肠杆菌却能够引起严重的食源性疾病[71]。这种分类与功能的脱节严重制约了我们理解微生物如何响应和记录环境变化。
5.2 基于宏基因组学的微生物生态研究传统微生物生态学分析方法严重依赖16S rRNA基因扩增子数据,无法直接建立物种分类组成与代谢功能间的联系,这一瓶颈在存在较多未知微生物信息的极端环境中尤为凸显。顺应宏基因组研究手段的快速发展,我们开发了一种基于宏基因组学种水平代表基因组(Species-level representative genomes, SRGs)的生态分析方法,它能够解析未知物种高达89.4%的深渊微生物组的生态过程及其代谢偏好。在与基于16S rRNA的方法比较验证中,两种方法在识别主要分类群的主导生态过程方面基本一致,显示出新分析方法的可靠性;并且该方法能够成功地将生态过程、代谢潜力、基因组特征和极端环境联系起来,识别出两种截然不同的深渊微生物环境适应策略——“精简型”与“多能型”,其中“精简型”微生物具有较小的基因组和特化的代谢功能以针对性地适应一类极端环境,而“多能型”微生物具有较大的基因组和多样化的代谢能力,表明其采用机会主义的极端环境生存策略[15]。因此,基于SRG的分析方法拓展了水圈微生物生态分析的深度和广度。
与传统16S rRNA基因扩增子序列变体(Amplicon sequence variants, ASV)方法相比,基于宏基因组SRG的生态分析方法具有三个显著优势:首先,基于SRG的方法能够直接将分类信息与适应性特征联系起来,在严格的组装和质量控制程序下,每个SRG代表样本中存在的一个微生物物种,其基因组可直接用于评估代谢潜力和适应机制;其次,基于SRG的方法有效避免了ASV生成过程中的多种影响因素,包括16S rRNA基因拷贝数变异、PCR扩增偏差等,提供更可靠的生态学分析结果;最后,基于SRG的方法特别适用于探索极端和未充分研究的微生物生态系统。这种将生态分析方法与微生物代谢功能相结合的方法,值得在环境微生物生态研究中进行推广。
5.3 微生物反演环境动态变化的新方法展望随着微生物组学研究的深入,基于微生物特征来反映环境特征的研究方法将是环境微生物研究的另一个重要发展方向。Tara Oceans项目在这方面提供了创新性的方法学案例,该研究开发了一种利用海洋病毒群落分布模式来追踪和表征洋流活动的新方法。研究团队建立了包含68个采样站7 193个病毒群体的全球病毒分布数据集,通过生物地理学分析算法,将病毒群落的空间分布模式与海洋物理数据进行关联分析,发现病毒群落的分布边界能够有效指示主要洋流的流向和影响范围[3]。该研究采用病毒群体整体的空间分布特征而非单一病毒类群进行分析,通过群体水平的统计方法降低数据噪声干扰,从而提高环境推断的准确性。这种方法将传统生物多样性分析与物理海洋学研究相结合,为复杂环境流动过程的监测提供了成本较低且信息丰富的替代方案。
这种通过微生物物种和功能随环境的变化来推测环境动态过程的研究思路展现出了广阔的应用前景。例如,我们在青藏高原研究中已经尝试利用抗性基因的空间分布模式来追踪印度季风的传播路径,为大气环流研究提供了新的生物学视角[14]。未来的研究方向不仅要关注微生物随大尺度地球过程的迁移模式,更要识别哪些特定微生物类群更易随环境流动而扩散,这些“指示种”往往具有特殊的生理特征或生态特性,使其能够参与大尺度的物质循环和能量流动。基于类似的方法学原理,研究人员有望开发出更多基于微生物分布的环境监测新技术,包括利用深海微生物数据表征深层洋流活动,通过土壤微生物群落变化监测地下水流动,或者基于大气微生物分布追踪污染物扩散路径等。人工智能技术的引入将极大提升这些应用的精度和效率,自动识别“环境-微生物”的对应关系,并建立不同时空尺度下的预测模型。这些新方法的开发将丰富环境科学研究手段,为环境监测、气候变化研究和生态系统管理提供更精准的技术支撑,推动微生物生态学与地球科学、环境科学等领域的深度交叉融合。
6 人工智能在水圈微生物研究中的机遇与挑战综上,水圈样品和微生物组学数据与研究技术的发展极大丰富了水圈环境的组学信息及其环境物理化学参数,但微生物的环境适应往往不由单一基因变化所主导,而是多基因、多途径协同作用的复杂结果。加之环境因子本身的多样性和“共适应”现象,微生物的环境适应往往呈现出“多因一果,多果一因”的复杂情况,因此需要更高维度的视角和分析方法,对不同环境因子下微生物环境适应的共性和特性进行有效拆解。人工智能时代的到来,为这种共性与特性的有效拆解提供了新机遇。
然而,AI手段在微生物组学研究中的挑战与其方法的原理直接相关,至少需要解决3个关键问题:(1)需要有足够训练AI模型的数据量,且有明确的数据标签;(2) AI预测结果需要准确的“金标准”来进行校验;(3)对于AI模型及其学习到的结果做到可解析。近年来,我国在极端环境微生物数据方面有深厚的积累,为解决上述三个关键难点、助力AI在水圈微生物研究中的发展提供了绝佳机遇。
6.1 极端生境微生物AI大模型构建构建行之有效的AI大模型,需要类型多样的、具有明确环境标签的、数据量足够的水圈微生物大数据库(表 1)。然而,目前在国际上绝大多数AI大模型使用的公共数据库中,微生物基因组绝大多数来自地表常规环境(如浅海、土壤等),这些常规环境中的生命应对的物理化学条件比较温和,对于实际工业生产中极端理化环境(高温、高压、极端pH)指导与应用价值有限[80]。如前文所述,我国极端环境微生物数据有多年积累,通过对深渊、高原、冰川等微生物样本的大规模采集和近200 Tb的数据总量,形成了独一无二的国家宝藏。这些极端环境特有的微生物数据不仅数据量大,差异化更大,并且具有相对完整的环境数据,且包含的极端微生物可耐受极端温度、压力、盐度、pH等多种条件。
| 表 1 可应用于水圈微生物研究的代表性深度学习框架(表征模型和功能应用模型) |
人工智能方法学的高速发展,使得数据驱动的研究模式在环境微生物学中成为可能。利用序列大模型[81]、几何深度学习[74, 82]、扩散概率生成模型[76, 83, 84]等先进技术框架能够高效地挖掘大规模数据中的复杂模式,为微生物功能分类预测、物种关系分析、基于环境变化的演化路径模拟等提供新的解决方案[79]。应用这些极端环境微生物数据进行序列、蛋白质结构、功能、代谢通路等不同层面的大模型训练,将对指导生物工程实际生产中的菌种设计有重大现实意义,为构建具有环境感知能力的AI大模型提供了独特优势(表 1)。
大规模多模态数据的共享与标准统一是为AI大模型提供高质量训练数据的前提,也是AI模型可复用与可推广的保障。INSDC (DDBJ/ENA/Gen-Bank)通过长期的三方交换与共同政策,依托SRA-BioSample-BioProject的分层与互链机制,实现原始序列与样本/项目元数据的同步归档,构成全球公共序列归档的骨干网络[85]。然而该库的样本/实验元数据依然存在大量“同名异义”“非规范表述”“单位/坐标不统一”“完整性不足”等问题,导致跨项目整合与机器学习特征构建困难;这类现象早已在对SRA/BioSample的系统评估中被明确指出(如MetaSRA指出SRA样本注释术语不统一且需本体映射;NCBI BioSample因允许自定义属性导致质量和完整性差等)[86]。我国多组学共享平台NODE采用“项目-样本-实验-运行-原始/分析数据”的层次化组织,支撑单样本多组学与同一对象多样本的数据管理与复用,平台在内容与格式上对齐国际主流标准(如SRA、TCGA、ProteomeXchange等),并在微生物组学元数据采集上参考MIxS最小信息清单以减少自由文本与异构字段带来的整合成本,提升数据的一致性与可交换性[19]。这些标准化举措不仅降低了人工智能自动解析与建模的技术门槛,也为跨数据库数据整合和知识迁移奠定了基础。
6.2 微生物实验验证反哺AI模型迭代优化AI模型预测结果必须通过实验校验才能被确认。但目前对于需要多酶联动或需要完整代谢途径的研究,AI预测结果仍缺少有效的实验校验能力,而在细胞层面上情况更甚。极端微生物来源的环境往往具有明确的极端环境因子,在人工极端环境模拟实验中可以进一步简化、明确这种环境选择压,所得到的实验结果往往体现为特定条件下生命的关键弱点和响应机制,能够为AI预测结果提供明确的实验验证,同时可进一步作为AI学习的训练数据,对AI模型进行迭代。
多组学数据集合了代谢组、基因组和蛋白质组等不同层次的生物信息,有助于进一步深入理解生物系统中不同分子层次的交互关系。AI的兴起为多组学数据的深度分析带来了革命性的机遇。这些方法通过从海量数据中自动学习特征间的潜在模式,不仅能够精准建模组学层次间的复杂非线性关系,还能实现对高维数据的有效降维和特征提取[87, 88]。深度神经网络可以端到端地对基因表达、蛋白质丰度和代谢物水平之间的动态交互关系进行捕捉,从而为生物过程提供全景式的解析能力[89, 90]。通过零样本学习和弱监督学习等方法,还能够从未标注或少量标注数据中推理出更多潜在标签,并对噪声数据进行自适应处理,从而增强数据的多样性和分析的鲁棒性[91]。
6.3 多模态数据整合助力AI模型的生物学解析针对微生物群体环境适应的“多因一果,多果一因”复杂情况,需要综合环境和微生物的多模态信息进行高维解析(图 2)。传统统计方法在处理多模态数据时能力不足,容易忽视数据间的复杂交互特征。AI方法通常以非线性变换将输入数据映射到一个隐空间,这一隐空间通常维度更高、特征表征更为全面且强大,能够挖掘生物数据间的复杂关系,揭示隐藏的模式。基于深度学习通过构建AI多模态模型,对不同组学层次的数据投射到高维隐空间并进行对齐,从而实现多模态数据的对齐和综合分析[92-94]。当前已有多种基于Transformer的蛋白质大模型被提出,用于表征蛋白质的序列、结构、进化信息等,在多种下游任务中取得了远超经验模式的预测结果[95-97]。然而,深度学习本身的黑箱性质导致这些高维表征的提取逻辑难以被直观理解。
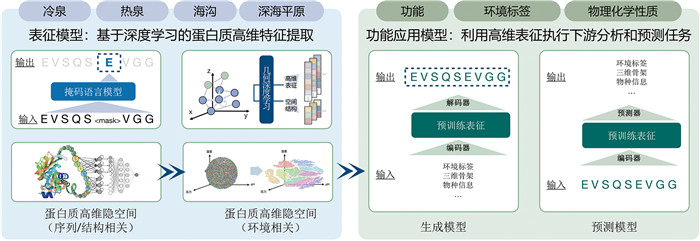
|
图 2 人工智能高维特征的提取与应用 |
我们在AI方法解析水圈微生物组跨生境的环境适应机制上已有两个方向的尝试(图 2)。(1)构建表征模型,提取不同生境微生物组及其微生物基因组在蛋白质序列和结构高维隐空间上的特征(我们称为“高维特征”)并聚类,并通过微生物数据来源的环境标签(既包括生境标签,又包括温压盐等环境因子标签)找到与环境因子最直接关联的蛋白质功能类群并与微生物基因组关联,这里我们已经发现极端环境微生物信息由于具有较明确的环境特征(如高压、高温、高盐等),可以通过引入与极端微生物环境适应相关的专家知识,比较在高维空间中自动聚集到不同簇内的蛋白质的差异与共性,能够较好地解释大模型中学到的映射规则,揭示蛋白质在物种、功能和结构等方面的异同,从而能够对微生物类群在更高维度中进行理解和分类,也为未来微生物的分离培养及资源化应用提供新的理论和实践路径。(2)构建功能应用模型,利用上述提取的高维特征执行下游分析和预测任务,将已提取的极端环境标签(尤其是温压盐等环境因子标签)、蛋白质结构的三维骨架、对应微生物的物种信息等作为输入数据对模型进行预训练,从而获得能够针对特殊需求(如提高极端条件下的稳定性)的单条蛋白质序列或全基因组序列的生成模型。反之,我们也可以通过输入新的序列,预测对应蛋白质和微生物的环境、结构和物种信息。值得一提的是,由于不同极端环境因子下的“共适应”现象的存在,这类极端微生物生成式模型也具有跨环境因子的生成能力,如我们前期通过对包含深渊耐压微生物的MEER数据集进行学习获得的AI模型,也能够生成具有较强耐碱、耐热等能力的蛋白质序列,这将大大助力极端蛋白质设计和转化应用。
总之,在人工智能时代,我们可以充分利用前期的数据积累,以连接表层和深部的典型水圈生境(深海、浅海、冰川和湖泊)为研究对象,解析跨生境的物种分布和生态驱动机制,并反演不同圈层的动力学过程(如洋流、板块运动等);通过多组学共分析,揭示水圈生境的代谢特征及组织模式;结合蛋白质语言大模型和几何深度学习等人工智能手段,提取不同环境微生物蛋白质组在序列和结构上的高维特征,解析其背后的生物学意义以认知水圈微生物的共性与特性(图 3),这将从跨生境的高维视角阐明微生物对水圈环境的适应与演化机制。
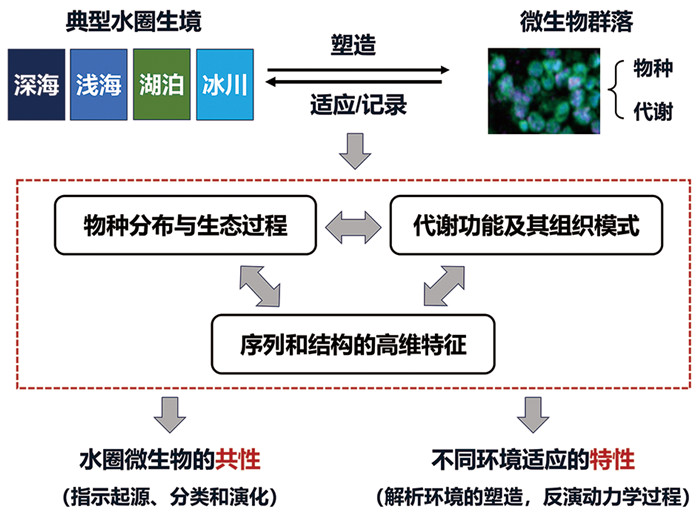
|
图 3 人工智能时代跨生境的微生物组研究策略 |
| [1] |
Thompson LR, Sanders JG, McDonald D, et al. A communal catalogue reveals Earth's multiscale microbial diversity. Nature, 2017, 551: 457-63. DOI:10.1038/nature24621 |
| [2] |
de Vargas C, Audic S, Henry N, et al. Eukaryotic plankton diversity in the sunlit ocean. Science, 2015, 348: 1261605. DOI:10.1126/science.1261605 |
| [3] |
Brum JR, Ignacio-Espinoza JC, Roux S, et al. Patterns and ecological drivers of ocean viral communities. Science, 2015, 348: 1261498. DOI:10.1126/science.1261498 |
| [4] |
Sunagawa S, Acinas SG, Bork P, et al. Tara Oceans: towards global ocean ecosystems biology. Nat Rev Microbiol, 2020, 18: 428-45. DOI:10.1038/s41579-020-0364-5 |
| [5] |
Paoli L, Ruscheweyh HJ, Forneris CC, et al. Biosynthetic potential of the global ocean microbiome. Nature, 2022, 607: 111-8. DOI:10.1038/s41586-022-04862-3 |
| [6] |
Tara Oceans - Publications[EB/OL]. [2025-07-01]. https://sunagawalab.ethz.ch/web/taraoceans.php
|
| [7] |
Duarte CM. Seafaring in the 21St Century: the Malaspina 2010 Circumnavigation Expedition. Limnol Oceanogr Bull, 2015, 24: 11-4. DOI:10.1002/lob.10008 |
| [8] |
Sánchez P, Coutinho FH, Sebastián M, et al. Marine picoplankton metagenomes and MAGs from eleven vertical profiles obtained by the Malaspina Expedition. Sci Data, 2024, 11: 154. DOI:10.1038/s41597-024-02974-1 |
| [9] |
Vance D, Little SH, de Souza GF, et al. Silicon and zinc biogeochemical cycles coupled through the Southern Ocean. Nat Geosci, 2017, 10: 202-6. DOI:10.1038/ngeo2890 |
| [10] |
GEOTRACES Project[EB/OL]. [2025-07-01]. https://www.geotraces.org
|
| [11] |
Boulton W, Salamov A, Grigoriev IV, et al. Metagenome-assembled-genomes recovered from the Arctic drift expedition MOSAiC. Sci Data, 2025, 12: 204. DOI:10.1038/s41597-025-04525-8 |
| [12] |
Liu Y, Ji M, Yu T, et al. A genome and gene catalog of glacier microbiomes. Nat Biotechnol, 2022, 40: 1341-8. DOI:10.1038/s41587-022-01367-2 |
| [13] |
Liu Y, Jiao N, Xu Zhong K, et al. Diversity and function of mountain and polar supraglacial DNA viruses. Sci Bull, 2023, 68: 2418-33. DOI:10.1016/j.scib.2023.09.007 |
| [14] |
Mao G, Ji M, Jiao N, et al. Monsoon affects the distribution of antibiotic resistome in Tibetan glaciers. Environ Pollut, 2023, 317: 120809. DOI:10.1016/j.envpol.2022.120809 |
| [15] |
Xiao X, Zhao W, Song Z, et al. Microbial ecosystems and ecological driving forces in the deepest ocean sediments. Cell, 2025, 188: 1363-77. e9. DOI:10.1016/j.cell.2024.12.036 |
| [16] |
Meyer F, Paarmann D, D'Souza M, et al. The metagenomics RAST server – a public resource for the automatic phylogenetic and functional analysis of metagenomes. BMC Bioinf, 2008, 9: 386. DOI:10.1186/1471-2105-9-386 |
| [17] |
Richardson L, Allen B, Baldi G, et al. MGnify: the microbiome sequence data analysis resource in 2023. Nucleic Acids Res, 2023, 51: D753-9. |
| [18] |
Gurbich TA, Almeida A, Beracochea M, et al. MGnify genomes: a resource for biome-specific microbial genome catalogues. J Mol Biol, 2023, 435: 168016. DOI:10.1016/j.jmb.2023.168016 |
| [19] |
凌鋆超, 曹瑞芳, 李亦学, 等. 多组学大数据共享平台研究进展. 生命科学, 2023, 35: 1553-60. |
| [20] |
Sunagawa S, Coelho LP, Chaffron S, et al. Structure and function of the global ocean microbiome. Science, 2015, 348: 1261359. DOI:10.1126/science.1261359 |
| [21] |
Carradec Q, Pelletier E, Da Silva C, et al. A global ocean atlas of eukaryotic genes. Nat Commun, 2018, 9: 373. DOI:10.1038/s41467-017-02342-1 |
| [22] |
Lombard F, Bourdin G, Pesant S, et al. Open science resources from the Tara Pacific expedition across coral reef and surface ocean ecosystems. Sci Data, 2023, 10: 324. DOI:10.1038/s41597-022-01757-w |
| [23] |
Xiao X, Wang J, Ding K. MEER: extraordinary flourishing ecosystem in the deepest ocean. Cell, 2025, 188: 1175-7. DOI:10.1016/j.cell.2024.12.037 |
| [24] |
Wutkowska M, Vader A, Logares R, et al. Linking extreme seasonality and gene expression in Arctic marine protists. Sci Rep, 2023, 13: 14627. DOI:10.1038/s41598-023-41204-3 |
| [25] |
Amman F, Markt R, Endler L, et al. Viral variant-resolved wastewater surveillance of SARS-CoV-2 at national scale. Nat Biotechnol, 2022, 40: 1814-22. DOI:10.1038/s41587-022-01387-y |
| [26] |
Dick GJ. The microbiomes of deep-sea hydrothermal vents: distributed globally, shaped locally. Nat Rev Microbiol, 2019, 17: 271-83. DOI:10.1038/s41579-019-0160-2 |
| [27] |
Zhivkoplias E, Jouffray JB, Dunshirn P, et al. Growing prominence of deep-sea life in marine bioprospecting. Nat Sustain, 2024, 7: 1027-37. DOI:10.1038/s41893-024-01392-w |
| [28] |
Wu L, Ning D, Zhang B, et al. Global diversity and biogeography of bacterial communities in wastewater treatment plants. Nat Microbiol, 2019, 4: 1183-95. DOI:10.1038/s41564-019-0426-5 |
| [29] |
Diner RE, Allard SM, Gilbert JA. Host-associated microbes mitigate the negative impacts of aquatic pollution. mSystems, 2024, 9: e00868-24. |
| [30] |
Ruan Z, Chen K, Cao W, et al. Engineering natural microbiomes toward enhanced bioremediation by microbiome modeling. Nat Commun, 2024, 15: 4694. DOI:10.1038/s41467-024-49098-z |
| [31] |
O'Malley MA. 'Everything is everywhere: but the environment selects': ubiquitous distribution and ecological determinism in microbial biogeography. Stud Hist Philos Sci Biol Biomed Sci, 2008, 39: 314-25. DOI:10.1016/j.shpsc.2008.06.005 |
| [32] |
Coelho LP, Alves R, del Río áR, et al. Towards the biogeography of prokaryotic genes. Nature, 2022, 601: 252-6. DOI:10.1038/s41586-021-04233-4 |
| [33] |
Breitbart M, Rohwer F. Here a virus, there a virus, everywhere the same virus?. Trends Microbiol, 2005, 13: 278-84. DOI:10.1016/j.tim.2005.04.003 |
| [34] |
Louca S, Polz MF, Mazel F, et al. Function and functional redundancy in microbial systems. Nat Ecol Evol, 2018, 2: 936-43. DOI:10.1038/s41559-018-0519-1 |
| [35] |
Olofsson M, Lindehoff E, Legrand C. Production stability and biomass quality in microalgal cultivation – Contribution of community dynamics. Eng Life Sci, 2019, 19: 330-40. DOI:10.1002/elsc.201900015 |
| [36] |
Sheng Y, Bibby K, Grettenberger C, et al. Geochemical and temporal influences on the enrichment of acidophilic iron-oxidizing bacterial communities. Appl Environ Microbiol, 2016, 82: 3611-21. DOI:10.1128/AEM.00917-16 |
| [37] |
Qin H, Wang S, Feng K, et al. Unraveling the diversity of sedimentary sulfate-reducing prokaryotes (SRP) across Tibetan saline lakes using epicPCR. Microbiome, 2019, 7: 71. DOI:10.1186/s40168-019-0688-4 |
| [38] |
Allison SD, Martiny JBH. Resistance, resilience, and redundancy in microbial communities. Proc Natl Acad Sci USA, 2008, 105: 11512-9. DOI:10.1073/pnas.0801925105 |
| [39] |
Fetzer I, Johst K, Sch?we R, et al. The extent of functional redundancy changes as species' roles shift in different environments. Proc Natl Acad Sci USA, 2015, 112: 14888-93. DOI:10.1073/pnas.1505587112 |
| [40] |
Fierer N, Ladau J, Clemente JC, et al. Reconstructing the microbial diversity and function of pre-agricultural tallgrass prairie soils in the United States. Science, 2013, 342: 621-4. DOI:10.1126/science.1243768 |
| [41] |
Galand PE, Pereira O, Hochart C, et al. A strong link between marine microbial community composition and function challenges the idea of functional redundancy. ISME J, 2018, 12: 2470-8. DOI:10.1038/s41396-018-0158-1 |
| [42] |
Liu Y, Zhang Z, Ji M, et al. Comparison of prokaryotes between Mount Everest and the Mariana Trench. Microbiome, 2022, 10: 215. DOI:10.1186/s40168-022-01403-y |
| [43] |
Stegen JC, Bottos EM, Jansson JK. A unified conceptual framework for prediction and control of microbiomes. Curr Opin Microbiol, 2018, 44: 20-7. DOI:10.1016/j.mib.2018.06.002 |
| [44] |
Van Den Bossche T, Armengaud J, Benndorf D, et al. The microbiologist's guide to metaproteomics. iMeta, 2025, 4: e70031. DOI:10.1002/imt2.70031 |
| [45] |
Uhlén M, Fagerberg L, Hallstr?m BM, et al. Tissue-based map of the human proteome. Science, 2015, 347: 1260419. DOI:10.1126/science.1260419 |
| [46] |
Valdés-Mas R, Leshem A, Zheng D, et al. Metagenome-informed metaproteomics of the human gut microbiome, host, and dietary exposome uncovers signatures of health and inflammatory bowel disease. Cell, 2025, 188: 1062-83. e36. DOI:10.1016/j.cell.2024.12.016 |
| [47] |
Chang Y, Fan Q, Hou J, et al. A community-supported metaproteomic pipeline for improving peptide identifications in hydrothermal vent microbiota. Brief Bioinform, 2021, 22: bbab052. DOI:10.1093/bib/bbab052 |
| [48] |
Hawley AK, Brewer HM, Norbeck AD, et al. Metaproteomics reveals differential modes of metabolic coupling among ubiquitous oxygen minimum zone microbes. Proc Natl Acad Sci USA, 2014, 111: 11395-400. DOI:10.1073/pnas.1322132111 |
| [49] |
Brum JR, Ignacio-Espinoza JC, Kim EH, et al. Illuminating structural proteins in viral "dark matter" with metaproteomics. Proc Natl Acad Sci USA, 2016, 113: 2436-41. DOI:10.1073/pnas.1525139113 |
| [50] |
Kleiner M, Thorson E, Sharp CE, et al. Assessing species biomass contributions in microbial communities via metaproteomics. Nat Commun, 2017, 8: 1558. DOI:10.1038/s41467-017-01544-x |
| [51] |
Wang DZ, Kong LF, Li YY, et al. Environmental microbial community proteomics: status, challenges and perspectives. Int J Mol Sci, 2016, 17: 1275. DOI:10.3390/ijms17081275 |
| [52] |
Ezzat L, Peter H, Bourquin M, et al. Diversity and biogeography of the bacterial microbiome in glacier-fed streams. Nature, 2025, 637: 622-30. DOI:10.1038/s41586-024-08313-z |
| [53] |
Zhong ZP, Solonenko NE, Gazitúa MC, et al. Clean low-biomass procedures and their application to ancient ice core microorganisms. Front Microbiol, 2018, 9: 1094. DOI:10.3389/fmicb.2018.01094 |
| [54] |
Oliver S. Systematic functional analysis of the yeast genome. Trends Biotechnol, 1998, 16: 373-8. DOI:10.1016/S0167-7799(98)01214-1 |
| [55] |
Fernie AR, Trethewey RN, Krotzky AJ, et al. Metabolite profiling: from diagnostics to systems biology. Nat Rev Mol Cell Biol, 2004, 5: 763-9. DOI:10.1038/nrm1451 |
| [56] |
Kornberg H. Krebs and his trinity of cycles. Nat Rev Mol Cell Biol, 2000, 1: 225-8. DOI:10.1038/35043073 |
| [57] |
Calvin M. The path of carbon in photosynthesis. Science, 1962, 135: 879-89. DOI:10.1126/science.135.3507.879 |
| [58] |
Roessner U, Luedemann A, Brust D, et al. Metabolic profiling allows comprehensive phenotyping of genetically or environmentally modified plant systems. Plant Cell, 2001, 13: 11-29. DOI:10.1105/tpc.13.1.11 |
| [59] |
Fiehn O, Kopka J, D?rmann P, et al. Metabolite profiling for plant functional genomics. Nat Biotechnol, 2000, 18: 1157-61. DOI:10.1038/81137 |
| [60] |
Allen J, Davey HM, Broadhurst D, et al. High-throughput classification of yeast mutants for functional genomics using metabolic footprinting. Nat Biotechnol, 2003, 21: 692-6. DOI:10.1038/nbt823 |
| [61] |
Perez de Souza L, Alseekh S, Scossa F, et al. Ultra-high-performance liquid chromatography high-resolution mass spectrometry variants for metabolomics research. Nat Methods, 2021, 18: 733-46. DOI:10.1038/s41592-021-01116-4 |
| [62] |
Swartz ME. UPLCTM: an introduction and review. J Liq Chromatogr Relat Technol, 2005, 28: 1253-63. DOI:10.1081/JLC-200053046 |
| [63] |
Hurst CJ. Their world: a diversity of microbial environments: Cham: Springer International Publishing, 2016.
|
| [64] |
Xu C, Couvillion SP, Sontag RL, et al. MetFish: a metabolomics pipeline for studying microbial communities in chemically extreme environments. mSystems, 2021, 6: e0105820. DOI:10.1128/mSystems.01058-20 |
| [65] |
King R, Bonfiglio R, Fernandez-Metzler C, et al. Mechanistic investigation of ionization suppression in electrospray ionization. J Am Soc Mass Spectrom, 2000, 11: 942-50. DOI:10.1016/S1044-0305(00)00163-X |
| [66] |
Sterner JL, Johnston MV, Nicol GR, et al. Signal suppression in electrospray ionization Fourier transform mass spectrometry of multi-component samples. J Mass Spectrom, 2000, 35: 385-91. DOI:10.1002/(SICI)1096-9888(200003)35:3<385::AID-JMS947>3.0.CO;2-O |
| [67] |
Bonfiglio R, King RC, Olah TV, et al. The effects of sample preparation methods on the variability of the electrospray ionization response for model drug compounds. Rapid Commun Mass Spectrom, 1999, 13: 1175-85. DOI:10.1002/(SICI)1097-0231(19990630)13:12<1175::AID-RCM639>3.0.CO;2-0 |
| [68] |
Selway CA, Eisenhofer R, Weyrich LS. Microbiome applications for pathology: challenges of low microbial biomass samples during diagnostic testing. J Pathol Clin Res, 2020, 6: 97-106. DOI:10.1002/cjp2.151 |
| [69] |
Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain: the primary kingdoms. Proc Natl Acad Sci USA, 1977, 74: 5088-90. DOI:10.1073/pnas.74.11.5088 |
| [70] |
Pereira RPA, Peplies J, Mushi D, et al. Pseudomonas-specific NGS assay provides insight into abundance and dynamics of Pseudomonas species including P. aeruginosa in a cooling tower. Front Microbiol, 2018, 9: 1958. DOI:10.3389/fmicb.2018.01958 |
| [71] |
Kolenda R, Burdukiewicz M, Schierack P. A systematic review and meta-analysis of the epidemiology of pathogenic Escherichia coli of calves and the role of calves as reservoirs for human pathogenic E. coli.. Front Cell Infect Microbiol, 2015, 5: 23. |
| [72] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-9. DOI:10.1038/s41586-021-03819-2 |
| [73] |
Nguyen E, Poli M, Durrant MG, et al. Sequence modeling and design from molecular to genome scale with Evo. Science, 2024, 386: eado9336. DOI:10.1126/science.ado9336 |
| [74] |
Zhou B, Zheng L, Wu B, et al. Protein engineering with lightweight graph denoising neural networks. J Chem Inf Model, 2024, 64: 3650-61. DOI:10.1021/acs.jcim.4c00036 |
| [75] |
Tan Y, Zheng L, Zhong B, et al. Protein representation learning with sequence information embedding: does it always lead to a better performance?[C]//Lisbon: 2024 IEEE International Conference on Bioinformatics and Biomedicine (BIBM), 2024: 233-9
|
| [76] |
Zhou B, Zheng L, Wu B, et al. A conditional protein diffusion model generates artificial programmable endonuclease sequences with enhanced activity. Cell Discov, 2024, 10: 95. |
| [77] |
Ingraham JB, Baranov M, Costello Z, et al. Illuminating protein space with a programmable generative model. Nature, 2023, 623: 1070-8. DOI:10.1038/s41586-023-06728-8 |
| [78] |
Yu T, Cui H, Li JC, et al. Enzyme function prediction using contrastive learning. Science, 2023, 379: 1358-63. DOI:10.1126/science.adf2465 |
| [79] |
Zhou B, Lv O, Wang J, et al. ODNet: opinion dynamics-inspired neural message passing for graphs and hypergraphs. Transact Mach Learn Res, 2025. |
| [80] |
Kang L, Wu B, Zhou B, et al. AI-enabled alkaline-resistant evolution of protein to apply in mass production. elife, 2025, 13: RP102788. DOI:10.7554/eLife.102788 |
| [81] |
Li M, Yang T, Ma X, et al. ProSST: protein language modeling with quantized structure and disentangled attention. Adv Neural Inform Process Syst, 2024, 37: 35700-26. |
| [82] |
Tan Y, Zhou B, Zheng L, et al. Semantical and geometrical protein encoding toward enhanced bioactivity and thermostability. elife, 2025, 13: RP98033. DOI:10.7554/eLife.98033.4 |
| [83] |
Yi K, Zhou B, Shen Y, et al. Graph denoising diffusion for inverse protein folding. Adv Neural Inform Process Syst, 2023, 36: 10238-57. |
| [84] |
Hu Y, Tan Y, Han A, et al. Secondary structure-guided novel protein sequence generation with latent graph diffusion[C]//Lisbon: 2024 IEEE International Conference on Bioinformatics and Biomedicine (BIBM), 2024: 31-41
|
| [85] |
Arita M, Karsch-Mizrachi I, Cochrane G. The international nucleotide sequence database collaboration. Nucleic Acids Res, 2021, 49: D121-4. DOI:10.1093/nar/gkaa967 |
| [86] |
Bernstein MN, Doan A, Dewey CN. MetaSRA: normalized human sample-specific metadata for the Sequence Read Archive. Bioinformatics, 2017, 33: 2914-23. DOI:10.1093/bioinformatics/btx334 |
| [87] |
Lin Z, Akin H, Rao R, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [88] |
Heinzinger M, Weissenow K, Sanchez JG, et al. Bilingual language model for protein sequence and structure. NAR Genom Bioinform, 2024, 6: lqae150. DOI:10.1093/nargab/lqae150 |
| [89] |
Camacho DM, Collins KM, Powers RK, et al. Next-generation machine learning for biological networks. Cell, 2018, 173: 1581-92. DOI:10.1016/j.cell.2018.05.015 |
| [90] |
Eraslan G, Avsec ?, Gagneur J, et al. Deep learning: new computational modelling techniques for genomics. Nat Rev Genet, 2019, 20: 389-403. |
| [91] |
Zhou Z, Zhang L, Yu Y, et al. Enhancing efficiency of protein language models with minimal wet-lab data through few-shot learning. Nat Commun, 2024, 15: 5566. DOI:10.1038/s41467-024-49798-6 |
| [92] |
Brixi G, Durrant MG, Ku J, et al. Genome modeling and design across all domains of life with Evo 2. bioRxiv, 2025. DOI:10.1101/2025.02.18.638918 |
| [93] |
Tan Y, Wang R, Wu B, et al. Retrieval-enhanced mutation mastery: augmenting zero-shot prediction of protein language model. Bioinformatics, 2025, 41: i401-9. DOI:10.1093/bioinformatics/btaf189 |
| [94] |
Hayes T, Rao R, Akin H, et al. Simulating 500 million years of evolution with a language model. Science, 2025, 387: 850-8. DOI:10.1126/science.ads0018 |
| [95] |
Tan Y, Liu C, Gao J, et al. VenusFactory: a unified platform for protein engineering data retrieval and language model fine-tuning[C]//Vienna: Proceedings of the 63rd Annual Meeting of the Association for Computational Linguistics, 2025: 230-41
|
| [96] |
Tan Y, Li M, Zhou B, et al. Simple, efficient, and scalable structure-aware adapter boosts protein language models. J Chem Inf Model, 2024, 64: 6338-49. DOI:10.1021/acs.jcim.4c00689 |
| [97] |
Li S, Tan Y, Ke S, et al. Immunogenicity prediction with dual attention enables vaccine target selection[C]//Singapore: The Thirteenth International Conference on Learning Representations, 2025
|
 2025, Vol. 37
2025, Vol. 37 

 赵维殳,上海交通大学生命科学技术学院副研究员。2017年毕业于上海交通大学生物学专业,获理学博士学位;2017?2020年在美国罗德岛大学从事博士后研究,2020年至今任职于上海交通大学,主要通过微生物学、生物信息学、计算生物学等多学科交叉手段,解析深渊、热液等极端环境条件下的微生物生命过程及其记录的地球动力学过程。以第一作者(含共同)及通讯作者在Cell、Nature Communications等高水平杂志上发表论文15篇。具有丰富出海和野外科考经验,完成了马里亚纳海沟、东南印度洋等载人深潜科考任务,在基金委重大科学考察共享航次(NORC2024-801)中担任首席助理。主持基金委重大研究计划集成项目、面上项目等,并积极推动学科建设,自主构建极端生命学科体系,获得全国青年教师教学竞赛理科组一等奖
赵维殳,上海交通大学生命科学技术学院副研究员。2017年毕业于上海交通大学生物学专业,获理学博士学位;2017?2020年在美国罗德岛大学从事博士后研究,2020年至今任职于上海交通大学,主要通过微生物学、生物信息学、计算生物学等多学科交叉手段,解析深渊、热液等极端环境条件下的微生物生命过程及其记录的地球动力学过程。以第一作者(含共同)及通讯作者在Cell、Nature Communications等高水平杂志上发表论文15篇。具有丰富出海和野外科考经验,完成了马里亚纳海沟、东南印度洋等载人深潜科考任务,在基金委重大科学考察共享航次(NORC2024-801)中担任首席助理。主持基金委重大研究计划集成项目、面上项目等,并积极推动学科建设,自主构建极端生命学科体系,获得全国青年教师教学竞赛理科组一等奖