(2 中国科学院上海生命科学信息中心,中国科学院上海营养与健康研究所,上海 200031)
(2 Shanghai Information Center for Life Sciences, Shanghai Institute of Nutrition and Health, Chinese Academy of Sciences, Shanghai 200031, China)
水圈作为地球独有的重要圈层,不仅孕育生命、支撑人类演化,还连接着大气圈、生物圈与岩石圈,承担全球物质迁移与能量转化的核心功能。广泛分布于水圈环境中的微生物在碳、氮、硫等元素的生物地球化学循环中发挥关键作用[1],是调控气候变化的重要生物因子[2-4]。为系统揭示水圈微生物在地球元素循环与生态响应中的作用机制,国家自然科学基金委在2017年启动了“水圈微生物驱动地球元素循环的机制”重大研究计划(以下简称“水圈微生物组计划”),联合生物、生态、地球化学、环境和信息等多学科力量,聚焦微生物介导的碳氮硫循环、群落形成、能量代谢等关键过程,推动观测、实验、模拟与数据平台深度融合,支撑水圈生态系统保护与“双碳”战略实施[5]。
在重大研究计划支持下,我们建设了水圈微生物组大数据平台,持续汇聚水圈微生物组计划资助产出的多组学数据,并整合国际数据库中与水体生态系统相关的公共资源,逐步形成覆盖海洋、近岸、湖泊与湿地等生态系统的统一数据体系,具备多模态、结构化和可计算等特征。
随着研究规模扩大与样本类型增多,传统以项目为单元的数据组织模式日益难以适应高通量、强关联、深挖掘的研究需求,水圈微生物组研究正加速迈向数据密集型范式。平台建设虽起步于数据标准化与共享等基础能力,但在标准体系、汇交机制与智能分析等方面的持续演进,已成为推动研究范式转型的重要支点。平台不仅重塑了数据的组织与治理方式,也深度参与知识发现过程,为水圈生态研究提供了系统化、结构化与智能化的基础设施。
下文将聚焦平台在标准制定、数据汇交、质量控制与知识挖掘等方面的建设实践,系统梳理其支撑数据密集型研究范式转型的关键机制与实施路径。
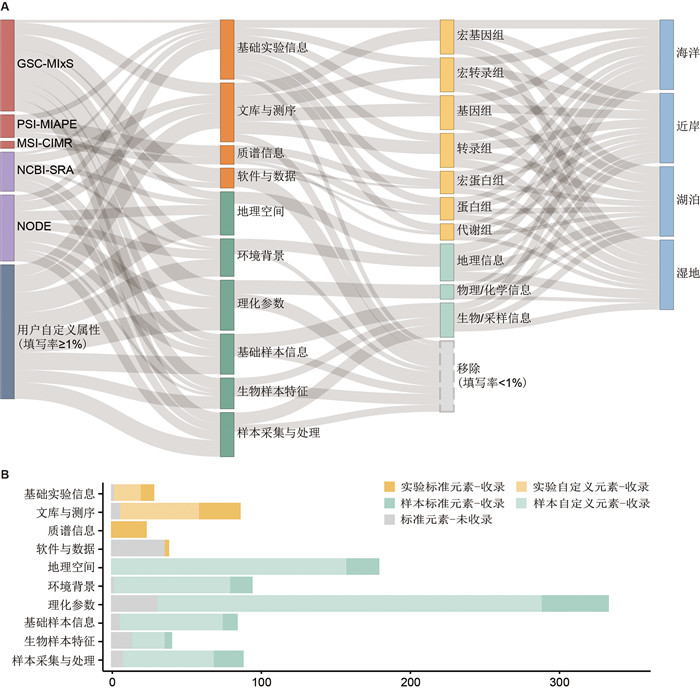
1 数据标准化描述体系构建水圈微生物组具有典型的非宿主环境特征,涵盖宏基因组、基因组、转录组等多类型数据,并伴随复杂的样本采集和生态背景信息。为实现多模态数据的整合、共享与重用,我们构建了统一的数据标准体系MASHyDEs (Microbiome Atlas/Sino-Hydros- phere Data Elements)(图 1)。标准构建首先系统引入了国际主流的数据标准与数据库框架,包括GSC (Genomic Standards Consortium)的MIxS (Minimal Information About (X) Any Sequence)[6]、HUPO-PSI (Human Proteome Organization Proteomics Standards Initiative)的MIAPE (Minimum Information about a Proteomics Experiment)[7]、MSI (Metabolomics Standards Initiative)的CIMR (Core Information For Metabolomics Reporting)[8]等国际标准,以及NCBI-SRA (https://www.ncbi.nlm.nih.gov/sra)[9]和NODE (https://www.biosino.org/node)[10]等数据库的元数据结构;在此基础上进行优化,保留实际填写率高于1%的字段,剔除104项使用频率低、表述模糊或已被技术迭代替代的内容,形成了初步的标准框架。随后,我们系统扫描现有数据库中的水圈微生物组数据,对高频自定义字段进行标准化映射,大多数字段成功对接至标准框架,仅“iron”(2.3%)和“ferrous_iron”(1.2%)未被覆盖,因此将其补充纳入,以增强对用户实际数据的兼容与表达能力。最后,根据领域专家建议,我们新增坡位指数,碳、氮、硫同位素含量等关键环境变量[11-14],以强化对地形特征、有机质来源、营养盐循环与缺氧过程等生态耦合机制的刻画,完善并最终形成MASHyDEs标准体系(图 1)。
|
(A)构建过程融合多项国际标准(MIxS、MIAPE、CIMR)与主流数据库框架(NCBI-SRA、NODE),按逻辑划分为10类元数据主题。通过用户提交数据统计,剔除104项低频字段,并补充iron与ferrous_iron两个常见自定义字段(填写率≥1%)。结合专家建议,新增4个关键环境变量以增强对微生物驱动的地球元素循环刻画能力。(B)各类元数据在不同来源中的覆盖情况,区分标准与用户自定义属性,标示是否已被实际采集。 图 1 MASHyDEs标准体系构建及数据元素结构 |
在此基础上,MASHyDEs整合了多来源的125个核心数据元素,形成了以实验、样本与环境三大维度为主干的标准结构,特别强化了水圈关键环境因子(如温度、盐度、深度等)在结构层级中的表达能力。相较于既有标准,该体系通过提升生态变量的结构优先级,使元数据在生态建模、环境推理和跨系统比较中的适用性显著增强。MASHyDEs面向实际应用需求,构建了针对海洋、近岸、湖泊和湿地四类典型生态系统的专属数据元素子集,覆盖各自核心的物理、化学与生态指标,提升了标准在实地监测与区域比较研究中的适配性与效率。在标准裁剪方面,团队通过对现有样本数据的填写率进行系统评估,剔除低使用频次元素,保留用户自定义通道,使MASHyDEs在保持完整性的同时具备良好的操作性与性价比,降低了填写成本,增强了用户友好性。
综上,MASHyDEs在结构层级、环境维度和生态适配三个层面兼容并超越既有标准,奠定了水圈多模态数据标准化、平台化管理和跨生态系统集成分析的基础,为从“数据整合”迈向“知识发现”提供了坚实保障。
2 数据汇交汇聚与质量控制在MASHyDEs标准体系的支撑下,水圈微生物组大数据平台构建了覆盖主动提交与自动汇聚的多通道数据获取体系,配套建立了面向异构组学数据的分层治理机制与质量控制流程,夯实了平台支撑数据密集型研究的基础能力。
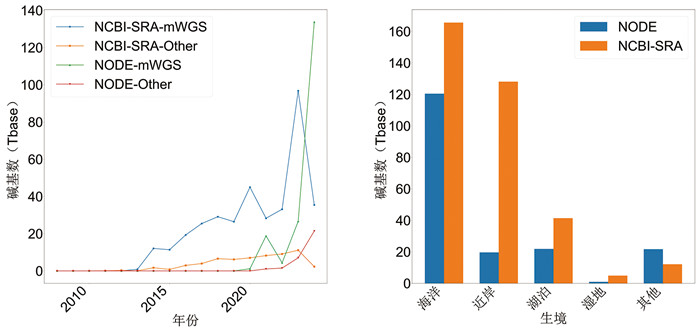
在主动提交方面,平台依托NODE构建了“系统预审+人工复核”双重机制,面向典型水体生态系统设定结构化填报模板,覆盖地理信息、生境类型、实验条件、测序策略等核心字段。截至2024年底,NODE已接收项目412项,累计样本超过34 000个,总测序数据量达到215.6 Tbase (图 2),涵盖宏基因组、扩增子、单菌基因组与转录组等多种数据类型。这一体系不仅有效提升了国内水圈微生物组数据的可重复利用水平,也丰富了生态模型构建与多组学融合分析的原始数据基础。
|
图 2 NCBI-SRA和NODE的水圈数据汇交情况 |
在自动汇聚方面,平台通过AntNest (https://www.biosino.org/antnest)系统,定期从NCBI-SRA、ENA (https://www.ebi.ac.uk/ena)[15]等国际数据库中整合公开可用的水圈样本数据,并基于MASHyDEs执行术语标准化、逻辑筛选与信息补全等自动治理流程,提升了数据在结构一致性、字段完整性与语义表达方面的整体质量。至2024年底,AntNest共整合样本近27万个,测序总量超过403 Tbase (图 2),为开展全球尺度比较研究与区域生态格局识别提供了丰富的基础数据。
在质量控制方面,平台针对主动提交(NODE)与自动汇聚(AntNest)两类数据来源,分别构建了差异化的治理策略,聚焦地理坐标、环境参数与生态分类等关键字段的标准化与补全。NODE数据采用“人工智能(AI)预治理+人工审核”模式,先通过规则引擎进行结构检测、术语比对与逻辑校验,再由工作人员进行人工确认与补全,确保信息准确性与生态学可解释性。相比之下,AntNest汇聚数据因来源分散、结构异构,平台引入大语言模型辅助推理与语义规则校验机制,自动完成术语消歧、字段补全与一致性检查,有效提升了元数据质量与结构规范性。
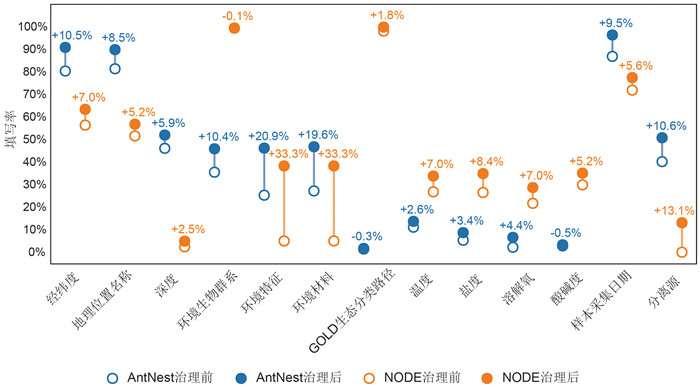
治理策略在多个核心字段上取得了实质性成效。图 3显示,无论是NODE或AntNest中的水圈样本,在治理前后关键字段的填写率都普遍上升,尤其是温度、盐度、溶解氧与酸碱度等与生态研究密切相关的核心因子。此外,环境特征和环境材料的提升特别明显(超过30%),其原因在于这2个字段的填写需要符合EnvO (Environment Ontology)规范,大多数用户对EnvO并不熟悉,极大地增加了用户填写的复杂程度,于是我们利用AI方法从样本描述等信息推断出这2个字段的值,并经确认后填写到数据记录中。
|
图 3 治理前后水圈微生物组样本关键元数据的填写率提升情况 |
与NCBI-SRA、MG-RAST等国际数据平台相比,基于NODE的水圈微生物组大数据平台的特色体现在以下方面。(1)更细致的数据标准:NCBI-SRA作为面向所有物种的测序数据归档库[9],没有对微生物特有的生境及理化因子给予特别的关注;MG-RAST聚焦于宏基因组数据的分析,虽然在宏基因组功能注释等方面非常专业[16],但是缺乏其他类型的组学数据标准;而MASHyDEs更符合特定生态研究的需求(详见前文)。(2)更方便的数据提交过程:水圈微生物组大数据平台基于MASHyDEs提供了定制版的数据汇交模板,对于每个生境类型所对应的地理信息、环境参数等字段的填写要求也比NCBI-SRA和MG-RAST等平台更为详细和规范。通过数据汇交模型,用户可以更方便地提交水圈相关的元数据,降低其他无关数据元素的干扰。(3)更高的数据填写率:NODE数据在环境信息维度上的完整性普遍优于国际数据库(图 3),特别是在EnvO与GOLD (https://gold.jgi.doe.gov)等生态系统分类标准方面。这些提升不仅由于用户提交了更多的信息,AI治理也发挥了重要作用。这些信息不仅增强了单样本的信息完整性与生态可解释性,也为后续多源数据的横向比较、因果挖掘与生态模型构建提供了坚实的数据基础。
通过构建结构统一、语义清晰、质量可控的数据资源,我们实现了对水圈微生物组元数据的标准化、精细化表示及管理,从而有望提升对后续的数据驱动研究的支撑能力。
3 分析平台与知识挖掘体系建设为支撑大规模水圈微生物组数据的分析利用,平台构建了从原始数据处理到知识发现的两级分析体系,逐步形成“流程标准化—计算自动化—结果结构化”的分析闭环。在基础分析层面,平台开发了云计算环境下的微生物组分析系统iMAC (https://www.biosino.org/iMAC),集成宏基因组、扩增子、单菌基因组等多类型数据的标准化处理流程,涵盖组装、注释、多样性计算、功能评估等核心分析环节。iMAC采用容器化封装与作业调度机制,支持大规模样本的高效并行处理,并在项目实践中实现了良好的流程稳定性与结果一致性。
在知识挖掘层面,平台进一步构建了轻量级分析环境iMAC+,包含关键微生物特征识别工具xMarkerFinder[17]与跨数据集样本匹配工具miMatch[18]。其中,xMarkerFinder支持多种机器学习算法,可自动筛选与环境因子高度关联的微生物群体,并构建预测模型,用于揭示环境驱动的微生物变化规律;miMatch则通过代谢背景信息校正样本间的差异,实现多数据集间的稳健比对,提升跨项目宏基因组研究的因果发现能力。
平台已支撑多个典型生态系统的研究任务:在湖泊[19]和红树林[20]区域,基于扩增子数据开展了微生物多样性与环境因子联动分析;在酸性矿山[21]与海陆交汇带区域[22],基于MAGs (Metagenome-Assembled Genomes)开展了物种及功能多样性研究。面向全球尺度的生态认知需求,平台还初步建立了功能导向的知识图谱构建机制,围绕次级胆汁酸[23]、甲烷代谢[24]等关键功能通路,在多组学数据基础上刻画功能基因的分布结构与多样性特征,支撑关键功能过程的全球格局重建与生态解释。这些模块不仅实现了水圈微生物组数据“从结构到机制”的知识转化,也拓展了平台从数据处理工具向智能化分析基础设施的角色边界。
分析平台体系的建立不仅实现了数据“从分析到解释”的高效转化,也推动了微生物组研究从传统生物信息分析向以AI驱动的智能分析的跃升,为高通量、多生态系统、多数据源场景下的水圈微生物组研究提供了可持续支撑。
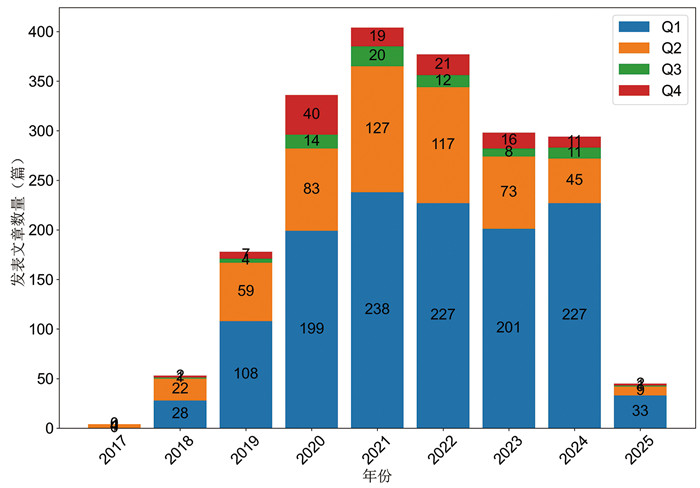
4 科研成果与共享成效分析自“水圈微生物驱动地球元素循环的机制”重大研究计划启动以来,水圈微生物组相关研究在科研产出与数据共享方面均取得了显著进展。2017–2024年,计划资助项目共发表论文1 989篇(图 4),其中已有1 009篇论文伴随数据公开,数据共享率超过50%,体现了项目在推动科研成果开放获取与数据可用性方面的积极成效。
|
图 4 水圈基金资助文献的JCR分区分布情况 |
平台在支撑数据汇交与规范管理方面逐渐发挥基础性作用,其所汇聚的数据涵盖三类来源:(1)计划资助项目产生的、尚未发表的但是已经提交到NODE的多组学原始数据;(2)已发表研究中通过NODE平台等渠道主动提交的数据;(3)通过AntNest自动汇聚的国际数据库中与水体生态相关的公开数据。基于统一的标准体系,平台对不同来源的数据进行了结构化和语义方面的统一治理,支撑了多样本整合与跨尺度分析的需求。
从共享数据库的分布变化来看,早期研究主要依赖国际数据库进行数据托管,尤其是NCBI-SRA和NCBI-GenBank,2019–2021年间托管数量快速增长,累计贡献超过300篇论文数据(表 1)。随着国内数据平台建设的推进,NODE和eLMSG (https://www.biosino.org/elmsg)[25]等平台在近年逐渐发挥更大作用。NODE在2023年的托管数量已超越ENA,eLMSG自2021年起支持水圈样本汇交,近三年提交量稳步上升。整体趋势表明,国内平台在支持环境元数据结构化表达、组学数据协同管理和项目组织管理方面已形成初步优势,研究者使用意愿不断提升。
| 表 1 专项资助的论文伴随数据共享情况 |
从全部1 989篇资助论文的文本统计结果来看,研究内容集中于微生物多样性、碳氮硫代谢、极端环境适应机制、生态系统响应及功能基因解析等方向,反映出水圈微生物组研究的典型跨学科特征(图 5)。在方法体系方面,术语如MAGs、expression、network、machine learning等频繁出现,表明高通量组学数据与智能计算方法在研究中的深入应用。同时,database、data sharing等关键词的聚集,也体现出研究者对数据平台、知识组织与成果结构化沉淀的高度关注。在部分代表性研究中,数据汇交的标准化实践已成为研究传播的一部分。例如,2025年发表于Cell杂志的深渊微生物组研究[26],所产生的1 194个样本的原始测序数据通过NODE可直接获取;拼接得到的7 564个微生物基因组在eLMSG发布,并可通过邮件联系作者获取,其中超过89%为新物种。这类成果的数据共享,不仅展示了平台对复杂数据结构的承载能力,也为推动数据复用与多源比对研究提供了实证支撑。
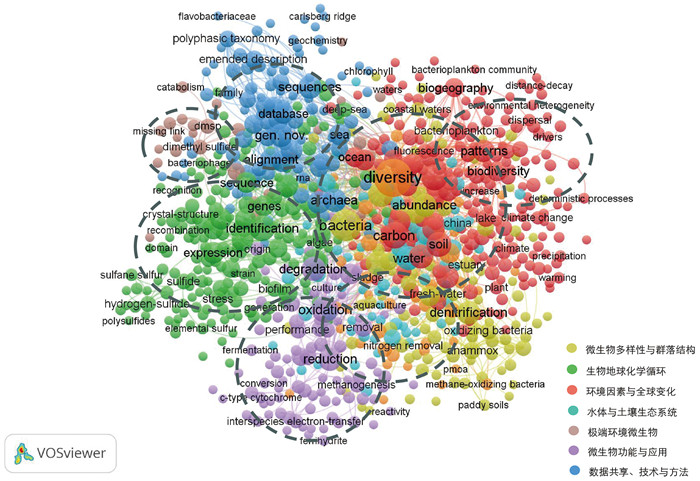
|
图 5 水圈基金资助文献关键词聚类 |
综上,水圈微生物组大数据平台作为集标准化管理、共享发布与再利用支持于一体的开放基础设施,在支撑科研数据生命周期管理、推动研究成果共享机制完善方面发挥了持续性作用。其在数据治理、资源开放与知识转化之间架起了有效桥梁,助力水圈生态系统研究范式加速向数据密集型转型。
5 总结及展望近年来,随着“水圈微生物组计划”的持续推进,融合观测、实验、模拟与数据平台的综合研究体系逐步建立,为理解水圈微生物在全球元素循环与生态响应中的作用提供了重要支撑。在这一背景下,水圈微生物组大数据平台作为配套建设的重要组成部分,围绕数据标准、质量控制、汇交机制与知识挖掘等关键环节,系统探索了适应数据密集型研究需求的建设路径。
目前,平台建设的重心仍聚焦于服务中国水圈微生物组研究的实际需求,重点支撑数据汇聚、治理规范与共享发布等任务。然而,其在多源异构数据融合、标准体系搭建和工具链开发方面所积累的经验,具备跨区域、跨学科的适应潜力。随着国际微生物组研究的开放协同格局不断发展,该平台有望逐步演化为代表中国方案的技术体系,为国际水圈生态研究提供结构化支持。同时,其体系化建设路径亦可为环境、农业、健康等领域的微生物组研究提供可借鉴的技术模式与治理机制。
在实际推进过程中,平台始终坚持“有数据,立标准;易搜索,促共享;可计算,赋能力”的建设目标,致力于构建安全可控、结构合理的数据基础设施。同时,围绕“安全管理、信息共享,技术创新、标准增值,尊重产权、高效利用”的服务理念,探索面向科研、管理与产业多元需求的协同支撑机制。通过标准化治理、智能化分析与制度化协作的耦合推进,水圈微生物组大数据平台正逐步从基础支撑向价值赋能迈进,为数据密集型研究范式的持续演化提供了坚实基础与现实支点。
致谢 感谢王寅炤、张晓华、汤凯、聂明和王建军等对MASHyDEs的指导,以及所有向NODE及eLMSG提交数据和改进意见的老师和同学。| [1] |
Falkowski PG, Fenchel T, Delong EF. The microbial engines that drive Earth's biogeochemical cycles. Science, 2008, 320: 1034-9. DOI:10.1126/science.1153213 |
| [2] |
Coelho FJ, Santos AL, Coimbra J, et al. Interactive effects of global climate change and pollution on marine microbes: the way ahead. Ecol Evol, 2013, 3: 1808-18. DOI:10.1002/ece3.565 |
| [3] |
Li Z. Marine microbial symbioses: host-microbe interaction, holobiont's adaptation to niches and global climate change. Front Microbiol, 2024, 15: 1416897. DOI:10.3389/fmicb.2024.1416897 |
| [4] |
黄力, 董海良, 全哲学, 等. 水圈微生物: 推动地球重要元素循环的隐形巨人. 微生物学报, 2020, 69: ⅰ-ⅱ. |
| [5] |
杜全生, 魏巍, 邹龙, 等. 科学基金加强水圈微生物领域基础研究. 中国科学基金, 2018, 32: 5. |
| [6] |
Yilmaz P, Kottmann R, Field D, et al. Minimum information about a marker gene sequence (MIMARKS) and minimum information about any (x) sequence (MIxS) specifications. Nat Biotechnol, 2011, 29: 415-20. DOI:10.1038/nbt.1823 |
| [7] |
Taylor CF, Paton NW, Lilley KS, et al. The minimum information about a proteomics experiment (MIAPE). Nat Biotechnol, 2007, 25: 887-93. DOI:10.1038/nbt1329 |
| [8] |
Goodacre R, Broadhurst D, Smilde AK, et al. Proposed minimum reporting standards for data analysis in metabolomics. Metabolomics, 2007, 3: 231-41. DOI:10.1007/s11306-007-0081-3 |
| [9] |
Katz K, Shutov O, Lapoint R, et al. The Sequence Read Archive: a decade more of explosive growth. Nucleic Acids Res, 2022, 50: D387-90. DOI:10.1093/nar/gkab1053 |
| [10] |
凌鋆超, 曹瑞芳, 李亦学, 等. 多组学大数据共享平台研究进展. 生命科学, 2023, 35: 1553-60. |
| [11] |
Ichino MC, Clark MR3, Drazen JC, et al. The distribution of benthic biomass in hadal trenches: a modelling approach to investigate the effect of vertical and lateral organic matter transport to the seafloor. Deep Sea Res Part Ⅰ Oceanographic Res Papers, 2015, 100: 21-33. DOI:10.1016/j.dsr.2015.01.010 |
| [12] |
Halevy I, Fike DA, Pasquier V, et al. Sedimentary parameters control the sulfur isotope composition of marine pyrite. Science, 2023, 382: 946-51. DOI:10.1126/science.adh1215 |
| [13] |
Kast ER, Stolper DA, Auderset A, et al. Nitrogen isotope evidence for expanded ocean suboxia in the early Cenozoic. Science, 2019, 364: 386-9. |
| [14] |
Omta AW, Follett CL, Lauderdale JM, et al. Carbon isotope budget indicates biological disequilibrium dominated ocean carbon storage at the Last Glacial Maximum. Nat Commun, 2024, 15: 8006. DOI:10.1038/s41467-024-52360-z |
| [15] |
Burgin J, Ahamed A, Cummins C, et al. The European Nucleotide Archive in 2022. Nucleic Acids Res, 2023, 51: D121-5. DOI:10.1093/nar/gkac1051 |
| [16] |
Meyer F, Paarmann D, D'Souza M, et al. The metagenomics RAST server - a public resource for the automatic phylogenetic and functional analysis of metagenomes. BMC Bioinformatics, 2008, 9: 386. DOI:10.1186/1471-2105-9-386 |
| [17] |
Gao W, Lin W, Li Q, et al. Identification and validation of microbial biomarkers from cross-cohort datasets using xMarkerFinder. Nat Protoc, 2024, 19: 2803-30. DOI:10.1038/s41596-024-00999-9 |
| [18] |
Liu L, Cao S, Lin W, et al. miMatch: a microbial metabolic background matching tool for mitigating host confounding in metagenomics research. Gut Microbes, 2024, 16: 2434029. DOI:10.1080/19490976.2024.2434029 |
| [19] |
Li M, Li Q, Wang S, et al. The diversity and biogeography of bacterial communities in lake sediments across different climate zones. Environ Res, 2024, 263: 120028. DOI:10.1016/j.envres.2024.120028 |
| [20] |
Du H, Pan J, Zhang C, et al. Analogous assembly mechanisms and functional guilds govern prokaryotic communities in mangrove ecosystems of China and South America. Microbiol Spectr, 2023, 11: e0157723. DOI:10.1128/spectrum.01577-23 |
| [21] |
Wang L, Liu W, Liang J, et al. Mining of novel secondary metabolite biosynthetic gene clusters from acid mine drainage. Sci Data, 2022, 9: 760. DOI:10.1038/s41597-022-01866-6 |
| [22] |
Pan S, Du H, Zheng R, et al. Supporting data for "A Holistic Genome Dataset of Bacteria and Archaea of Mangrove sediments". GigaScience Database, 2025, 14: giaf18. |
| [23] |
Yang Y, Gao W, Zhu R, et al. Systematic identification of secondary bile acid production genes in global microbiome. mSystems, 2025, 10: e0081724. DOI:10.1128/msystems.00817-24 |
| [24] |
Wang Y, Li L, Li Q, et al. MASH-Ocean 1.0: interactive platform for investigating microbial diversity, function, and biogeography with marine metagenomic data. iMeta, 2024, 3: e201. DOI:10.1002/imt2.201 |
| [25] |
National Genomics Data Center M and Partners. Database resources of the National Genomics Data Center in 2020. Nucleic Acids Res, 2020, 48: D24-33. DOI:10.1093/nar/gkz1210 |
| [26] |
Xiao X, Wang J, Ding K. MEER: extraordinary flourishing ecosystem in the deepest ocean. Cell, 2025, 188: 1175-7. DOI:10.1016/j.cell.2024.12.037 |
 2025, Vol. 37
2025, Vol. 37 

 张国庆,研究员,博士生导师。现任中国科学院上海营养与健康研究所生物医学大数据中心执行主任、上海生物医学大数据工程技术研究中心主任。主要研究方向是生物医学大数据与人工智能,包括精准医学、自然及疾病人群队列、人类表型组、环境与病原及人体微生物组等领域的数据库和知识库的研发,致力于多维生命组学数据、文献数据、健康与医疗等真实世界数据的集成与管理,以及以基于大模型的智能体为代表的数据科学关键技术研究
张国庆,研究员,博士生导师。现任中国科学院上海营养与健康研究所生物医学大数据中心执行主任、上海生物医学大数据工程技术研究中心主任。主要研究方向是生物医学大数据与人工智能,包括精准医学、自然及疾病人群队列、人类表型组、环境与病原及人体微生物组等领域的数据库和知识库的研发,致力于多维生命组学数据、文献数据、健康与医疗等真实世界数据的集成与管理,以及以基于大模型的智能体为代表的数据科学关键技术研究