(2 北京农业职业学院园艺园林学院, 北京 102442)
(3 南京农业大学国家大豆改良中心, 南京 210095)
(2 College of Horticulture, Beijing Vocational College of Agriculture, Beijing 102442, China)
(3 National Center for Soybean Improvement, Nanjing Agricultural University, Nanjing 210095, China)
大豆(Glycine max (L.) Merr.)起源于中国,拥有五千余年的栽培历史,是中国古代五大主粮作物(稻、麦、稷、菽和粟)之一,其中“菽”即指大豆,在中国农业文明中占据着举足轻重的地位。大豆作为全球重要的油料作物和蛋白质来源,贡献了全球约三分之一的植物油脂和三分之二的动物蛋白饲料[1],被广泛应用于食品、饲料及工业领域,现已成为世界范围内广泛种植的重要作物。然而,大豆在生长发育过程中面临着多种生物和非生物胁迫的严重威胁。据统计,每年因病虫害造成的大豆产量损失高达15%~30%,在重灾区甚至可能导致绝收[2]。其中,病毒病害已成为制约大豆产量和品质提升的重要因素,对全球大豆产业的可持续发展构成了重大挑战[3]。随着大豆种植规模的不断扩大,以及连作、混作和间作等多样化种植模式的推广和栽培环境(如温度、湿度和光照等)的变化,大豆病害的发生频率和种类显著增加,并常出现多种病害复合侵染的复杂局面,严重影响了大豆的安全生产和经济效益[2]。
在全球气候变化和耕作制度转型的背景下,大豆病毒病害问题日益突出。据报道,目前已知有超过100种植物病毒能够侵染大豆,其中在自然界中能够实际侵染大豆的病毒约有50种[4]。这些病毒一旦侵染大豆植株,常引发矮化、叶片卷曲、花叶、黄化和豆荚畸形等多种典型症状,严重影响大豆产量和品质[3]。值得注意的是,不同地区大豆面临的主要病毒威胁存在显著差异。例如,大豆花叶病毒(soybean mosaic virus, SMV)在全球范围内广泛分布,是威胁多国大豆生产的大豆病毒[5, 6];而菜豆荚斑驳病毒(bean pod mottle virus, BPMV)和菜豆普通花叶病毒(bean common mosaic virus, BCMV)则主要流行于美国、巴西等国家[4, 7]。此外,大豆脉坏死病毒(soybean vein necrosis virus, SVNV)主要在美国和加拿大等地被报道[8]。其他病毒如豇豆轻斑驳病毒(cowpea mild mottle virus, CpMMV)、烟草线条病毒(tobacco streak virus, TSV)、苜蓿花叶病毒(alfalfa mosaic virus, AMV)、大豆矮缩病毒(soybean dwarf virus, SbDV)、烟草环斑病毒(tobacco ringspot virus, TRSV)和黄瓜花叶病毒(cucumber mosaic virus, CMV)等也在全球多个地区被报道,对大豆生产造成了严重威胁[3, 4]。值得关注的是,大豆病毒病病原种类复杂多样,且新型病毒不断涌现。比如近年来,在我国黄淮海大豆主产区大面积暴发了一种名为大豆症青病毒(soybean stay-green associated virus, SoSGV)的病害,对我国大豆产业造成了极大的危害,重灾区甚至出现绝收[9-11]。
在我国,大豆作为关系国计民生的基础性、关键性和战略性农产品,具有不可替代的重要地位。然而,目前我国大豆产业面临着严峻的供需失衡问题。作为大豆消费大国,我国的年需求量高达1.2亿吨,居全球首位,但国内年产量仅2 000多万吨,进口依存度超80%,这使得大豆供应安全极易受到国际政治及经济形势波动的冲击。我国大豆产能不足,一方面受限于人多地少及种植面积有限;另一方面,单产水平偏低也是重要制约因素。目前我国大豆平均单产仅为美国和巴西等主要生产国的60%左右。高复种指数和重茬连作的普遍存在导致病虫害频发,成为制约大豆产量和品质提升的关键因素[12]。
综上,本文重点探讨了大豆常见的病毒病害,系统介绍了这些大豆病毒病的生物学特征及发生和危害,并梳理了当前大豆抗病毒免疫机制的最新研究进展以及基因编辑等前沿生物技术在大豆抗病毒遗传改良中的应用,以期为相关领域的研究以及大豆病毒病害的防控提供参考。
1 大豆常见病毒病害 1.1 大豆花叶病毒大豆花叶病毒(soybean mosaic virus, SMV)是在全世界范围内侵染大豆最为普遍和最具破坏性的病毒,不仅导致植株矮化、叶片花叶和畸形等典型症状,还显著影响大豆的产量和品质[3]。SMV属于马铃薯Y病毒科(Potyviridae)成员,是引发大豆花叶病的主要病原,在全球各大豆主产区均有广泛分布[13]。该病毒于1915年在美国康涅狄格州被首次报道,并于1921年由Gardner和Kendrick正式描述[14]。据记载,SMV可导致大豆减产8%~35%,极端情况下损失高达94%,其减产程度受品种、病毒株系、地理区域及侵染时期等多种因素影响[15]。SMV的症状表现因寄主基因型、病毒株系、植株感病时期和环境条件而异。大多数感病品种表现为轻度矮化、结荚数减少,荚果常见畸形、扁平、无茸毛及无籽粒等现象。叶片典型症状为三出复叶出现浅绿与深绿相间的斑驳,后期沿主脉隆起或形成疱状突起,深绿区域间可见褪绿斑,叶缘下卷或呈波浪状。部分品种的初生叶可能出现局部坏死斑,进一步发展为脉坏死、黄化及落叶;强毒株系则可引发严重矮化、系统性坏死、茎叶黄化、叶柄与茎秆坏死、顶枯,甚至植株死亡。环境温度对症状的表达具有显著影响:在18 ℃时症状最为明显,叶片皱缩加剧;24~25 ℃时症状有所减轻;而当温度超过30 ℃时,症状则基本被掩盖。此外,温度还影响潜育期,29.5 ℃时约4 d即可显症,而18.5 ℃时潜育期可延长至14 d[3, 16, 17]。
SMV病毒粒子由外壳蛋白和单链RNA组成,其基因组为正义单链RNA,长度约为10 kb,其5' 端通过共价键与病毒基因组连接蛋白(VPg)结合,3' 末端则带有多聚腺苷酸尾[6, 18]。基因组RNA包含一个大的开放阅读框(open reading frame, ORF),可翻译为一个约360 kD的多聚蛋白。此外,在P3编码区内部还存在一个由RNA聚合酶滑移产生的小ORF (pretty interesting potyvirus ORF, PIPO)。这两个多聚蛋白在病毒编码的三种蛋白酶的作用下被切割,最终产生P1、HC-Pro、P3、PIPO、6K1、CI、6K2、NIa-VPg、NIa-Pro、NIb和CP等11种具有不同功能的成熟蛋白。值得注意的是,在P3顺反子内部的小ORF与P3的N端共同编码形成融合蛋白P3N-PIPO,该蛋白在病毒的复制和系统侵染过程中发挥重要作用[19]。
SMV与大豆等寄主在长期共存过程中协同进化,形成了具有不同致病性的株系/分离物。研究发现,一些株系/分离物能够突破品种的抗性,成功侵染本应具备抗性的植株,这一现象揭示了SMV分离物间存在丰富的遗传变异和遗传多样性。根据田间采集的不同分离物在不同大豆品种上的致病性差异,世界各国对SMV分离物进行了不同的株系划分。我国利用10个对SMV反应稳定的大豆鉴别寄主,将上千个分离物划分为22个株系(SC1~SC22)[20, 21]。其中,南农1138-2大豆品种对所有分离物均表现为感病,其余鉴别寄主则对部分株系具有抗性。日本采用4个鉴别寄主将SMV分离物划分为5个株系(A~E)[22];美国利用7个鉴别寄主将其划分为7个株系(G1~G7)[23, 24]。当前报道的SMV株系数量众多,一方面反映了病毒的多样性,另一方面也反映了对病毒效应因子及其介导的抗性机制缺乏完全了解。
SMV在自然界中主要由蚜虫以非持久性(non-persistent)方式传播,目前已知至少有15个属的32种蚜虫可作为其传播媒介。其中,豌豆蚜(Acyrthosiphon pisum)、豆蚜(Aphis fabae)、大豆蚜(A. glycines)、桃蚜(Myzus persicae)和玉米蚜(Rho-palosiphum maidis)是最重要的传毒媒介[25]。和其他potyvirus一样,不同SMV分离株在蚜虫媒介上的特异性差异主要与其CP蛋白和HC-Pro蛋白的变异有关[26, 27]。此外,SMV可以通过种子传播,但其传播率因大豆品种和病毒分离株而异。在大多数品种中,SMV的种子传播率通常为0%~5%,但某些品种的传播率高达64% [27, 28],SMV种传能力受到大豆RNA沉默机制的影响,同时与SMV的P1、HCPro以及CP的变异有关[27]。在SMV流行学中,种子带毒产生的病株起着关键作用。这些病株是SMV的主要初侵染源。田间病毒扩散通常以点源聚集模式传播,随后由蚜虫介导的二次传播以中高速率向外扩展[28-31]。SMV在种子内可长期保持侵染活性,即使在丧失发芽能力的种子中仍能检测到活病毒。种子传毒率主要取决于品种特性和植株感病时期,其中开花前感染的植株种子传毒率显著较高[27, 28]。
SMV最重要的经济寄主为大豆。然而,不同SMV分离株也能侵染其他寄主,寄主范围主要限于6个科的植物,包括豆科(Fabaceae)、苋科(Amar- anthaceae)、藜科(Chenopodiaceae)、西番莲科(Pass- ifloraceae)、玄参科(Schrophulariaceae)和茄科(So-lanaceae)[25]。不同株系在不同寄主上的致病性存在显著差异,且在部分寄主中可发生潜伏侵染。此外,SMV与BPMV或AMV混合侵染时存在协同效应。在感病品种中,SMV与BPMV复合侵染可导致66%~86%的产量损失,显著高于单独SMV侵染造成的8%~25%减产。复合侵染植株表现出更为严重的症状,包括极端矮化、叶片畸形、组织坏死及叶面斑驳。同时,与单一病毒侵染相比,双重侵染植株的根瘤数量明显减少[32, 33]。
1.2 菜豆普通花叶病毒菜豆普通花叶病毒(bean common mosaic virus, BCMV)是马铃薯Y病毒属(Potyvirus)的成员之一,在全球范围内广泛分布,是严重威胁豆科作物产量和品质的重要病毒之一。BCMV最早由Stewart和Reddick从美国纽约受侵染的菜豆植株中分离获得,最初被称为“菜豆花叶相关病害”[34]。1934年,Pierce等[35]首次在菜豆品种“Corbett Refugee”中报道了对BCMV的抗性,并将该病毒正式命名为BCMV。1994年,Gunasinghe等[36]首次完成了BCMV基因组的测序。我国对BCMV的研究始于20世纪30年代,何畏冷(1936)和俞大绂(1939)最早记录了该病毒在我国的症状表现和危害情况。研究表明,BCMV在菜豆上的传播率极高,产量损失可达50%~100%[37]。由于其种子带毒率高,BCMV可随种子实现跨地区传播,目前已在欧洲、北美、南美、亚洲、大洋洲及非洲等地被检测到,除南极洲外,全球各大洲均有分布。Worrall等[38]的研究也证实,BCMV在种植豆科植物的地区普遍存在,但由于非洲部分国家缺乏有效检测,其实际危害程度尚未完全明确。近年来,随着全球贸易和种子进出口的繁荣,BCMV在世界范围内迅速传播,成为侵染豆科作物最为严重的病毒之一。
BCMV基因组为正义单链RNA,长度约为10 kb,其基因组结构和编码蛋白与SMV类似,多聚蛋白被蛋白酶切割最终产生P1、HC-Pro、P3、PIPO、6K1、CI、6K2、NIa-VPg、NIa-Pro、NIb和CP等11种具有不同功能的成熟蛋白[13]。BCMV的寄主范围极为广泛,可侵染9个科、44个属的100多种植物[39]。在自然条件下,BCMV可侵染多种豆科作物,如大豆、菜豆(Phaseolus vulgaris L.)、木豆(Cajanus cajan (L.) Millsp.)、决明(Senna tora (L.) Roxb.)、鹰嘴豆(Cicer arietinum L.)、兵豆(Lens culinaris Medic.)、蚕豆(Vicia faba L.)、豌豆(Pisum sativum L.)和香豌豆(Lathyrus odoratus L.)等。在温室条件下,通过机械摩擦接种还可侵染非豆科植物,如克利夫兰烟(Nicotiana clevelandii)和本氏烟(N. benthamiana)[40]。BCMV的传播途径多样,主要通过蚜虫以非持久性方式传播,其中豌豆蚜、甜菜蚜和桃蚜是主要的传播媒介[38]。蚜虫获毒时间极短(1 min),无潜伏期,接触植物表皮细胞仅需数秒(3~15 s)即可获得并传播病毒,传毒效率极高[41]。Donnelly等[42]研究发现,蚜虫能够通过感知被病毒侵染和健康菜豆植株释放的不同挥发性有机化合物,选择不同的寄主,这一机制有助于解释蚜虫从被侵染植株向健康植株迁移的原因,从而促进病毒在田间的快速传播。BCMV同样能够通过种传,种传的效率与BCMV株系、侵染时间以及侵染的寄主相关,在大豆上通过种传的报道较少[43]。
1.3 豇豆轻斑驳病毒豇豆轻斑驳病毒(cowpea mild mosaic virus, CpMMV)最早由Brunt等于1973年在加纳的豇豆上发现并报道[44]。此后,1982年Thounvenel等[45]在象牙海岸的大豆上首次分离到该病毒;同年Iwaki等[46]在泰国大豆上也首次报道了CpMMV的存在。2000年,CpMMV在巴西大豆上被报道,并对当地大豆产量造成了较为严重的影响[47]。2008年,Tavassoli等[48]在伊朗大豆上检测到该病毒;在我国,2009年Chang等[49]首次在台湾地区的菜豆上发现了CpMMV的侵染;2019年杨晓等[50]在海南的豇豆上发现了CpMMV;同年,Wei等[51]首次在安徽大豆上检测到该病毒;随后Wei等[52]对其在大豆上的发生进行了系统研究,发现其在河南、湖北和安徽等至少六个省份广泛分布,并且SMV与CpMMV可共同侵染大豆。这些研究表明,CpMMV已在国内外广泛发生和传播,严重威胁豆类作物的安全生产。
CpMMV是乙型线状病毒科(Betaflexiviridae)香石竹潜隐病毒属(Carlavirus)的成员。其基因组为正单链RNA,包裹于无包膜、柔性丝状的病毒颗粒中,病毒颗粒长度约为1 000 nm,直径为12~13 nm。基因组3′端带有poly(A)尾,5′端具有帽子结构,共编码六个开放阅读框(ORF)[53]。第一个ORF位于基因组5′端,编码病毒的RNA依赖性RNA聚合酶(RdRp);ORF2、ORF3和ORF4编码病毒运动蛋白TGB1、TGB2和TGB3,这三个蛋白相对分子质量分别为25.6、11.6和7.7 kDa;ORF5编码外壳蛋白(CP,32 kDa);ORF6位于基因组3′端,编码富含半胱氨酸的核酸结合蛋白。除ORF1可直接由病毒基因组翻译外,其余五个ORF均由至少两条亚基因组RNA (sgRNAs)转录[53]。在自然条件下,CpMMV可通过种子和烟粉虱(Bemisia tabaci Gennadius)以非持久性方式传播;在实验室条件下,也可通过机械接种进行传播[53, 54]。CpMMV的寄主范围广泛,包括大豆、豇豆、花生(Arachis hypogaea L.)、长豇豆(Vigna unguiculata subsp. Sesquipedalis (L.) Verdc.)和菜豆(P. vulgaris L.)等多种豆科作物。也有报道显示其可侵染茄科、葫芦科、苋科、藜科以及芝麻科等多种植物[53, 55]。此外,CpMMV还可通过种子传播,其传播效率可能受到病毒分离物、寄主植物基因型、抗病性、侵染时期及环境条件等多种因素的影响[53]。CpMMV侵染寄主后,常引起叶片斑驳、系统性斑点、畸形及褪绿等症状,严重时可导致整株坏死,显著降低作物产量和品质。
1.4 菜豆荚斑驳病毒菜豆荚斑驳病毒(bean pod mottle virus, BPMV)于1948年在美国南卡罗来纳州的普通菜豆(Phaseolus vulgaris)上被报道[56],并于1958年在阿肯色州首次发现其侵染大豆[57]。BPMV是双分体病毒科(Secoviridae)豇豆病毒属(Comovirus)的成员[58]。该病毒的寄主范围相对较窄,主要侵染豆科植物,其中大豆是最具经济重要性的寄主。受BPMV侵染的大豆通常减产3%~52%,减产程度因其品种和侵染时期而异 [59]。受侵染的大豆种子出现种皮斑驳,从而影响种子品质[60]。BPMV与SMV在田间能够发生复合侵染,并产生协同效应显著提高BPMV的病毒积累量,进而严重影响大豆的产量[61]。BPMV在美国已造成大豆产量的巨大损失,而我国目前尚无该病毒的分布记录。但随着我国大豆进口量的持续增加,BPMV随进口大豆传入的风险日益加大。目前,已有多次报道在进境大豆中检出BPMV,表明该病毒对我国大豆生产构成潜在威胁 [62, 63]。
BPMV为双分体病毒科(Comoviridae)豇豆病毒属(Comovirus)成员。该病毒具有双组分正链RNA基因组,由RNA-1和RNA-2组成,这两条RNA分别被包裹在直径为28 nm的等径病毒颗粒中[64]。通过密度梯度离心,病毒颗粒可被分为三种组分,分别为顶层(T)、中层(M)和底层(B)。T组分含有空颗粒,而M和B组分分别含有单一分子的RNA-2和RNA-1。这三种组分的蛋白组成相同,均由60个大衣壳蛋白(L-CP,41 kDa)和60个小衣壳蛋白(S-CP,22 kDa)组成。S-CP主要存在大小两种类型,即完整蛋白和C端截短型。由于这种异质性,BPMV病毒颗粒在电泳时表现为两种形式:迁移较慢和迁移较快的两种类型,每种类型都包含M和B核蛋白组分[65, 66]。BPMV的基因组RNA均带有poly(A)尾,并且每条RNA的5′端都共价连接有一个小的碱性蛋白VPg。BPMV的基因组通过合成和后续切割大型多聚蛋白前体来表达多种蛋白。BPMV的RNA-1编码五种成熟蛋白,参与病毒复制,从5′到3′依次为:蛋白酶辅因子、解旋酶、病毒基因组连接蛋白VPg、24K蛋白酶和RNA依赖性RNA聚合酶RdRp;而RNA-2则编码一种细胞间运动蛋白和两种衣壳蛋白[66, 67]。
在田间,BPMV可由多种食叶甲虫传播,包括豆叶甲(Cerotoma trifurcata, Forster)、葡萄肖叶甲(Colaspis brunnea, Fabricius)、带纹黄瓜甲虫(Diabrotica undecimpunctata howardi, Barber)、条纹斑蝥(Epicauta vittata, Fabricius)、日本丽金龟(Popillia japonica Newman)和墨西哥豆瓢虫(Epilachna varivestis, Mulsant)[66, 68]。其中,豆叶甲是一种极其高效的传毒介体;研究表明约80%取食染病大豆的豆叶甲能够将病毒传播给健康植株[66]。BPMV的初级侵染源通常有三个:受侵染的大豆种子、上一年流行后残留在越冬豆叶甲体内的病毒,以及受病毒侵染的豆科杂草。BPMV的种子侵染率非常低,并且种皮斑驳并非种子带毒的可靠指标,因为即使种子中没有病毒也可能发生斑驳[69]。BPMV可侵染多种寄主,研究发现,一种多年生豆科山蚂蝗属杂草(Desmodium canadense)在自然条件下是BPMV的自然寄主。越冬地出现的甲虫通过取食受侵染的多年生Desmodium属植物获得病毒。当大豆幼苗出土时,这些甲虫会转移到幼苗上传播病毒,随后世代的豆叶甲在生长季节可能会继续在田块内以及田块间传播该病毒[70]。
1.5 大豆症青病毒除已知病毒的危害,新发大豆病毒的暴发也为大豆生产带来了极大的挑战。近年来,在我国黄淮海大豆主产区大面积暴发了一种名为大豆“症青”的病害,重灾区甚至出现绝收,给当地农民带来了严重的经济损失,显著削弱了农民种植大豆的积极性[71]。通过小RNA高通量测序结合生物信息学分析,Cheng等[9]首次在大豆症青样品中鉴定出一类新型重组双生病毒,随后他们通过科赫法则证实了大豆症青病毒(soybean stay-green associated virus, SoSGV)侵染大豆可以造成大豆“症青”症状。通过多年多地系统性田间调查显示,SoSGV已在我国黄淮海多地大豆产区广泛分布,对大豆生产造成极大危害[72]。Cheng等[72]进一步发现不同地区的疑似“症青”样品除共有的植株持青特征外,还表现出不同的病毒症状。根据豆荚数量和鼓粒程度,这些症状可以分为三种类型:Ⅰ型表现为瘪荚明显但结荚总数正常;Ⅱ型表现为不瘪荚但结荚总数明显减少;Ⅲ型表现为不瘪荚且结荚数正常,但豆荚发育迟缓。其中,Ⅰ型为多地田间“症青”主要表现,占主导地位。目前,通过大范围田间调查,已经明确当前黄淮海地区,SoSGV造成的大豆“症青”占主导地位,因病毒侵染大豆时期以及大豆品种差异,症青表型存在不同,但是共有的特点均为荚而不实[72]。随后,Wang等[10]通过发病大豆全微生物组学也证实了双生病毒是大豆“症青”发生的“真凶”。
SoSGV的传播介体已被鉴定为一种叶蝉科昆虫——东洋网室叶蝉(Orosius orientalis, Matsumura)[72, 73]。该昆虫为多食性害虫,寄主范围极为广泛,可取食豆科、苋科、十字花科、葫芦科、锦葵科及茄科等至少60种植物[74]。该昆虫主要分布于澳大利亚、亚洲中南部及太平洋地区。在澳大利亚的一些年份,东洋网室叶蝉的种群密度是叶蝉中最高的,占比甚至达到60%,其高发期集中在初春至夏季[74]。东洋网室叶蝉是全球多种病毒和植原体的重要传播介体,能够传播芝麻、苜蓿、甜菜、水芹和紫菀等的植原体,以及烟草黄矮病毒(tobacco yellow dwarf virus)、鹰嘴豆褪绿矮缩双生病毒(chickpea chlorotic dwarf geminivirus)等多种病毒[75]。Cheng等[9, 72]研究进一步发现,SoSGV不仅能够侵染豆科作物如大豆、豌豆、鹰嘴豆和紫云英,还可侵染本氏烟、心叶烟、普通烟及曼陀罗等实验寄主,但无法侵染玉米和马铃薯。此外,尽管油菜和萝卜属于东洋网室叶蝉的寄主,但并不能被SoSGV侵染。值得注意的是,SoSGV侵染本氏烟后同样可引发“症青”表型,包括延迟衰老和瘪荚等,揭示SoSGV可能在不同寄主中通过调控共同的分子机制,导致类似的“症青”症状[9]。
SoSGV为单组分双生病毒,全长基因组序列为2 762 nt,预测编码6个蛋白,依据氨基酸同源性和在基因组上的位置,病毒链编码外壳蛋白(V1)和沉默抑制子(V2)两个蛋白,互补链编码复制相关蛋白(C1)、转录激活蛋白(C2)、复制增强蛋白(C3)和症状决定因子(C4)四个蛋白[9]。SoSGV与双生病毒科14属代表病毒种的全基因组核酸序列一致性都低于60%,目前被归为未分类的双生病毒种。对该病毒的基因组进行分段分析,推测其可能源于双生病毒科不同属间的重组,其中C1、C2、C3和C4区域与双生病毒科Begomovirus属病毒具有更高的序列同源性,V1和V2区与双生病毒科Mastrevirus属病毒序列具有更高的同源性[9]。因此,侵染大豆导致“症青”的双生病毒可能代表了一种由双生病毒科不同病毒属病毒重组产生的新病毒[9-11]。
2 大豆抗病毒研究进展抗病育种作为病害防控中最经济、最有效的手段,已成为现代农业可持续发展的重要保障。通过选育和推广具有抗病性的作物品种,可以从源头上减少病害的发生与流行,显著降低农药使用频率和生产成本,减轻环境污染和农药残留风险[76]。近年来,大豆抗病毒研究取得了显著进展,尤其是在抗SMV基因的挖掘与利用方面。这些抗性基因的发现为培育高抗性大豆品种提供了重要的遗传资源和理论基础。然而,与其他重要作物相比,大豆抗病毒基因的挖掘和鉴定仍然相对匮乏,尚需进一步加强对大豆抗病毒遗传资源的系统挖掘与功能解析,以满足大豆产业可持续发展的需求。
2.1 大豆抗病毒的遗传基础与抗性基因的鉴定与定位目前,大豆抗病毒病抗性基因鉴定主要以SMV为主,已鉴定出多个抗SMV基因位点,如Rsv1、Rsv3和Rsv4等,这3个位点赋予大豆对SMV G1~G7小种的抗性[77, 78]。此外,我国科研工作者还鉴定出其他抗性位点(Rsc基因:Rsc4、Rsc5、Rsc7、Rsc8、Rsc15和Rsc20),这些基因赋予大豆对SMV-SC (1~22)小种的抗性[79-81]。大豆抗SMV遗传位点主要分布在2号染色体、13号染色体以及14号染色体上,其中,Rsv1位于大豆第13号染色体,来源于大豆品系PI96983,能有效抵抗SMV的多个生理小种(G1~G6),产生极端抗性(extreme resistance, ER)[82, 83]。国内学者使用BPMV介导的基因沉默技术降低了抗病品种PI96983的候选基因GmZ15和GmR42的表达量,接种SC3后检测SMV含量,结果显示两个基因的沉默都能降低植株的抗病性,而且沉默GmR42对抗病能力的降低作用更为明显[84]。Zhang等[85]使用BPMV同时沉默含有Rsv1的品种L78-379中的NLR (nucleotide-binding leucine-rich repeat)类候选基因3gG2、5gG3和6gG9,然后使用基因枪接种SMV-GUS侵染性克隆质粒,不仅在接种叶上检测到了GUS活性,还在系统叶中通过RT-PCR检测到了SMV的积累。但由于候选基因之间具有较高的序列相似度,目前还没有证据表明哪一个NLR候选基因介导了对SMV的抗性以及候选基因之间的作用关系仍然不清楚。含有Rsv1的抗病品种PI96983对应的SMV的无毒基因(avirulent gene)最早被Hajimorad等[86]定位在P3蛋白编码区域,不亲和G7株系中P3的三个关键氨基酸的任意一个改变都会导致表型由致死性系统坏死减轻为系统坏死。含有Rsv1的抗病品种L78-379对G7的表型为系统坏死,Eggenberger等[87]将G7作为相对亲和互作的株系,并利用L78-379表现极端抗性的株系G2(N),将Rsv1对应的无毒基因鉴定为HC-Pro和P3。目前13号染色体抗SMV遗传位点定位区间都包含NLR类典型抗病候选基因。
Rsv3编码NBS-LRR类蛋白,位于第14号染色体(Chr14),对SMV的G5~G7等小种具有广谱抗性[88]。Ma等[89]构建了抗病品种Zaoshu18的BAC文库,发现了定位区段内的染色体结构变异,指出含有Rsv3的品种中,该定位区段的NLR类基因经历了基因间的片段交换和重组。进一步对不同品种中Rsv3定位区段内候选基因的等位基因序列分析,结果显示,该区段内5个NLR候选基因中的3个(NBS-C、NBS-D和NBS-E)在抗感品种中都出现明显的分化,并可能介导Rsv3对SMV的抗性。Rsv3介导的大豆对SMV的抗性是通过识别SMV的CI蛋白实现的[90]。当SMV的CI蛋白被Rsv3识别后,会激活大豆的抗病反应,增加胞间连丝胼胝体的积累,从而阻止病毒的扩散[91]。Seo等[91]将转录组测序用于解析Rsv3的抗病机理,通过对比抗SMV品种L29接种SMV G5H和G7H分离物后的转录组数据,发现PP2C类基因表达差异显著,其中一个PP2C基因在感病品种中被过表达后,SMV的胞间运输受到明显抑制,胼胝质的积累量上升。他们推测可能是病毒激活了脱落酸(abscisic acid, ABA)抗病通路,从而导致胼胝质的积累(图 1),进而产生了对SMV的抗性。Alazem等[92]进一步分析转录组数据,发现L29接种G5H后β-1, 3-葡聚糖酶表达量的下降可能减少了胼胝质的降解。此外,进一步分析发现接种SMV 24 h后,植物体内细胞自噬途径和RNA干扰途径相关基因以及JA途径和WRKY转录因子相关基因表达均呈现显著上调[93] (图 1)。
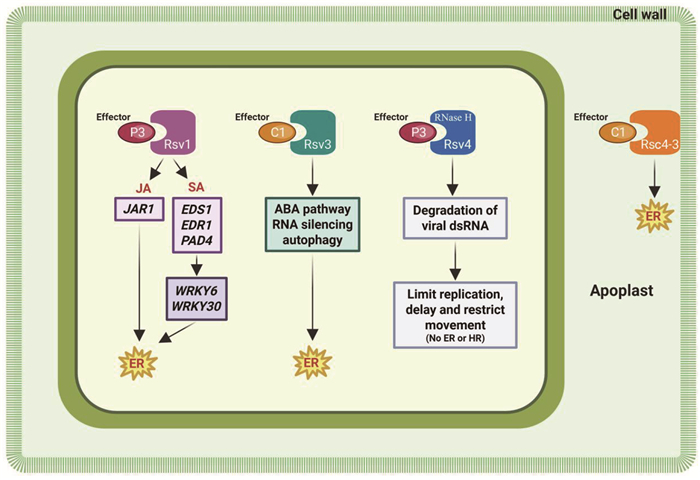
|
Rsv1能够识别SMV的效应子P3和(或)HC-Pro蛋白。当识别HC-Pro时,会诱导过敏反应,在这个过程中激活包括HSP90、EDS1、EDR1、WRKY6和WRKY30在内的多种大豆抗病相关基因,这些因子通过水杨酸(SA)和茉莉酸(JA)信号通路共同作用,赋予植物极端抗性(extreme resistance, ER)。Rsv3识别SMV的CI蛋白,会启动脱落酸(ABA)信号通路、抗病毒RNA沉默机制以及自噬过程,从而限制病毒的扩散,实现激活极端抗性。Rsv4编码一个RNase H蛋白,能够识别SMV编码的P3蛋白,靶向病毒复制复合体中的双链RNA,导致其降解,从而阻止病毒复制。Rsc4-3被鉴定为能编码一个位于细胞壁上的NB-LRR免疫蛋白,在质外体识别SMV编码的CI蛋白,诱导致死性的过敏反应,激活抗病反应。 图 1 大豆抗病基因介导的大豆对SMV的识别与抗性机制 |
我国科学家发现大豆品种大白麻对SMV-SC4株系的抗性位点Rsc4位于14号染色体。Rsc4定位区间包含Rsc4-1、Rsc4-2和Rsc4-3三个编码NLR蛋白的候选基因。Rsc4与国外定位的抗性位点Rsv3的差异在于,Rsv3定位区间除包含Rsc4-1、Rsc4-2和Rsc4-3外,还包含另外2个编码NLR蛋白的候选基因。Yin等[94]通过CRISPR/Cas9技术敲除了抗病品种大白麻中的Rsc4-3和Rsc4-1基因,使用纯合敲除的品系接种SMV SC4株系后,只有Rsc4-3敲除的大豆品系感病,进而证实了Rsc4-3是大白麻的抗SMV SC4株系抗病基因。Yin等[94]进一步从大豆品种大白麻中首次通过图位克隆法分离到Rsc4-3,其编码一个典型的NLR蛋白。有意思的是,研究者发现与以往发现的NLR类抗病基因不同,该抗病基因Rsc4-3编码的NLR蛋白在植物细胞壁上与SMV无毒因子CI特异识别并介导对SMV的抗性(图 1)。此外,他们发现棕榈酰化修饰对Rsc4-3抗SMV是必需的。值得一提的是,Rsc4-3是否和国外鉴定的Rsv3是同一个基因还需进一步确认。
Rsv4位于2号染色体,Gunduz等[95]通过遗传分析发现,Rsv4位点介导对G1~G7全部7个株系的抗性。与Rsv1、Rsv3的抗性机制不同,Rsv4介导的抗性并不依赖于典型的过敏性反应,后续研究发现抗性基因Rsv4编码了一种RNase H家族的蛋白,该编码蛋白是一种双链RNA (dsRNA)核酸酶[96]。它能够降解双链RNA,而双链RNA是病毒复制复合体中的关键成分。Rsv4对包括SMV在内的多种potyvirus具有广谱抗性,其机制在于能够将这种双链RNA酶递送到病毒复制区,从而有效破坏病毒的复制过程(图 1),Rsv4的活性位点突变不能抑制病毒增殖[96]。
Rsv5基因位于3号染色体(Chr3),是近年新发现的SMV抗性基因,能对部分SMV株系提供抗性。与已知的Rsv1、Rsv3和Rsv4等抗性基因位点不同,Rsv5能够赋予大豆对SMV部分株系的抗性(抗G1但是不抗G7),表现出独特的抗性谱[97, 98]。RSV5抗SMV的作用机制仍然不清楚。
另外,我国科学家已经鉴定了许多其他抗SMV的Rsc基因位点[99]。比如分别赋予SMV株系SC-8和SC-9抗性的抗性基因Rsc-8和Rsc-9已被定位于大豆2号染色体。Rsc-8基因位于一段200 kb的基因组区间,该区间包含17个候选基因,基于功能预测和表达模式分析,发现Glyma02g13310、Glyma02g13320、Glyma02g13400、Glyma02g13460和Glyma02g13470这5个基因可能是Rsc-8的候选基因[100]。大豆品种科丰1号的抗性基因Rsc-7被定位到大豆2号染色体上的2.65 Mb区域[101],随后被缩小到158 kb区域[102]。在该区域的15个候选基因中,一个NBS-LRR型基因Glyma02g13600、一个HSP40基因Glyma02g13520和一个丝氨酸羧肽酶型基因Glyma02g13620可能是Rsc-7的候选基因 [102]。
除了SMV,关于其他大豆抗病毒的基因报道较少。大豆矮化病毒(soybean dwarf virus, SbDV)是一种黄症病毒科(Luteoviridae)病毒,日本学者从大豆品种“Ouhou”中鉴定出抗SbDV的关键基因Rsdv1,这是目前已知的主要抗SbDV基因。研究团队利用Sat-217和Satt211标记构建了包含该位点的近等基因系(Rsdv1-NILs),温室接种与田间试验结果表明,Rsdv1-NILs对SbDV具有抗性,说明单独Rsdv1位点即可赋予抗病性。作者进一步将Rsdv1精细定位到Sat-11与Sct-13之间一个44 kb的区域。预测该区域包含6个基因,但通过序列比对发现,这些基因均与植物中已知的病毒抗性基因无显著同源性,因此推测Rsdv1可能通过一种未知机制介导对SbDV的抗性[103]。此外,花生花叶病毒(peanut mottle virus)是另外一种能够侵染大豆的potyvirus,目前已经在大豆中鉴定出两个显性抗性基因Rpv1和Rpv3,以及一个隐性抗性基因rpv2。值得一提的是Rpv1基因,由于在多个大豆品种同一位点上存在对SMV的抗性显性基因,因此推测对这两种病毒的抗性可能由同一基因控制[104]。在抗大豆黄化矮缩病毒(soybean yellow mottle mosaic virus)基因方面,虽然已有部分抗性种质资源被鉴定,但相关基因的定位和克隆仍在研究中。此外,抗大豆皱缩病毒(soybean crinkle virus)和SoSGV等的抗性基因研究尚处于起步阶段,部分抗性材料已被发现,但具体基因尚未完全被克隆和功能解析。近年来,随着更精准的测序技术、分子标记辅助选择(marker-assisted selection, MAS)、全基因组关联分析(genome-wide association study, GWAS)及抗性基因富集测序(resistance gene enrichment sequencing, RenSeq)等新技术的应用[105],越来越多的抗病毒基因被克隆和功能解析。未来,通过基因编辑等新技术,有望进一步挖掘和利用具有广谱、持久抗病毒特性的抗性基因,为大豆抗病育种提供坚实的基因资源。
2.2 RNA干扰抗大豆病毒病RNA干扰(RNA interference)是植物抗病毒的核心分子机制之一。病毒侵染大豆后,其基因组RNA或复制中间体可被植物Dicer酶(DCL)识别并切割成小干扰RNA (siRNA)。这些siRNA被装载到RNA诱导沉默复合体(RISC)中,指导RISC特异性降解病毒RNA,从而抑制病毒的复制和积累[106, 107]。研究发现,大豆中DCL、AGO等基因的表达水平与抗病毒能力密切相关[93]。通过转基因手段表达病毒特异性siRNA或人工miRNA,可显著提高大豆对多种病毒的抗性。RNA沉默的优势在于可靶向任何病毒,且抗性持久。
转基因抗病毒技术目前虽未达到商业化应用阶段,但已被证实对SMV等大豆病毒具有良好防控效果。研究人员将SMV的外壳蛋白(CP)基因及其3' 非翻译区(3' UTR)在CaMV35S启动子的驱动下导入大豆,T3代获得了两个纯合的转基因品系。这些品系即使在每株接种高达200 μg病毒的情况下,仍对同源SMV菌株表现出高度抗性。这种RNA介导的抗性对所测试的四种SMV菌株均有效,并在田间试验中表现出极高的防效[108]。为提高RNA沉默效率,研究者设计了表达SMV靶序列反向重复转录本的构建体,以持续高水平产生siRNA用于抗SMV[109]。除了SMV,Tougou等[110]构建了在CaMV 35S启动子驱动下表达SbDV CP反向重复序列的转基因大豆。结果表明,SbDV CP siRNA的产生与对SbDV的抗性密切相关,在蚜虫接种病毒的测试中该抗性可持续至少2个月。此外,抗大豆病毒RNA沉默的一个重要应用是同时靶向一种病毒的不同分离物或者同时靶向多种病毒。Yang等[111]构建了针对SMV不同分离物及其他potyvirus的NIb基因的发夹结构,转入大豆中能够产生同时对5种SMV分离物和BCMV以及西瓜花叶病毒(watermelon mosaic virus, WMV)的抗性。Zhang等[112]构建了同时表达SMV、BPMV和AMV三种病毒的150 bp短反向重复序列的转基因大豆,这些品系能积累对应三种病毒的siRNA,并对三种病毒的混合侵染表现出抗性。该研究表明,可以在同一位点聚合对同一病毒多种毒株及不同病毒的抗性。
此外,Gao等[113]发现大豆eIF4E1蛋白能够在细胞核内与SMV的VPg蛋白相互作用,并在细胞质中与SMV的NIa-Pro和NIb蛋白结合,研究人员利用RNA干扰技术获得了eIF4E基因被沉默的转基因大豆,接种SMV后通过对病毒症状和病毒含量的检测,证实这些转基因大豆对五种SMV毒株(SC3、SC7、SC15、SC18和SMV-R)、BCMV以及WMV均表现出强烈且广谱的抗性。
2.3 激素信号通路调控大豆病毒侵染植物激素在调控植物抗病毒反应中发挥重要作用[114]。水杨酸(salicylic acid, SA)是抗病毒防御的关键信号分子,能诱导PR (pathogenesis-related)基因表达,增强植物抗性。茉莉酸(jasmonic acid, JA)和乙烯(ET)也参与调控抗病毒反应[114]。近年来研究发现,激素信号通路之间存在复杂的交互调控网络,不同病毒和大豆品种对激素信号的响应存在差异。SA含量大量增加是许多不相容互作中的标志性分子。Rsv1介导的对大多数SMV毒株的早期抗性需要多个防御基因的参与,包括涉及水杨酸(SA)和茉莉酸(JA)途径的基因,并且可能还涉及特定的WRKY转录因子[85, 115]。在SMV抗性大豆品系L78-379中,沉默与SA相关的基因GmEDR1、GmEDS1和GmPAD4,以及与JA相关的基因GmJAR1,会导致其在感染SMV G2株系时表现出与易感对照品种Williams 82相似的症状[85]。在另一项研究中,双重沉默GmEDS1a/GmEDS1b或单独沉默GmPAD4会降低病原诱导的SA积累,进一步增强大豆对SMV G5株系的易感性,这表明SA在Rsv1介导的对SMV G5株系的抗性中具有重要作用[115]。此外,沉默GmHSP90会导致植株严重矮化,并减少SMV-G2的复制[85]。这表明分子伴侣HSP90对于Rsv1介导的对SMV-G2的抗性是必需的(图 1)。
值得一提的是,脱落酸ABA在Rsv3抗性基因与SMV-G5H非致病菌株之间的不相容互作中发挥关键作用[92, 93]。Shang等[116]通过转录组发现在光照下,SMV会诱导SA信号通路相关基因表达上调,可能介导了大豆SA参与抵抗SMV的侵染。Li等[117]对感染SMV的大豆抗病品系(Kefeng 1)和感病品系(NN1138-2)进行了转录组测序,通过后续分析发现抗性相关的差异表达基因主要富集于植物激素信号转导和生物节律通路。除了SA和JA这两种在植物抗病反应中已广泛报道的激素外,ABA、吲哚-3-乙酸(indole-3-acetic acid, IAA)和细胞分裂素(cytokinin)也参与了大豆对SMV的免疫应答。
2.4 大豆代谢物在大豆抗病毒中的作用大豆富含多种次生代谢产物,尤其是异黄酮、酚类化合物和皂苷等,这些物质不仅具有重要的营养和保健价值,还具有直接抗微生物及多种动物病毒的功能[118, 119]。近年来,关于大豆代谢物(如异黄酮等)在大豆抗病害中的作用研究逐渐增多,异黄酮类物质能够通过多种机制增强抗病能力:一方面,异黄酮具有较强的抗氧化活性,能够清除病毒侵染过程中产生的活性氧(reactive oxygen species, ROS),减轻细胞损伤;另一方面,异黄酮可作为信号分子,诱导防御相关基因的表达,激活植物的系统性抗性反应。此外,部分异黄酮还可直接抑制病毒的复制和扩散[120, 121]。Cheng等[122]发现大豆GmIFS1基因的多态性与抗SMV有关。Zhang等[123]采用CRISPR/Cas9介导的多基因编辑技术,同时靶向编辑了大豆的GmF3H1、GmF3H2和GmFNSII-1基因,与野生型相比,T3代纯合三突变体的叶片异黄酮含量大约增加了两倍,在接种SMV SC7株系后,这些突变体中病毒外壳蛋白的积累减少了1/3。未来,深入解析这些代谢物的分子机制及其调控网络,将为大豆抗病毒育种和绿色防控提供新的理论基础和技术途径。
2.5 其他抗病毒策略的挖掘与利用Helm等[124]通过在大豆NLR同源基因PBS1中插入一段可被SMV的NIa-Pro蛋白酶特异性切割的多肽序列,创新性地构建了人工诱饵抗病蛋白。该蛋白的工作机制在于,当SMV侵染大豆时,病毒的NIa-Pro蛋白酶能够识别并切割该人工插入的多肽片段,进而激活NLR介导的免疫反应,诱导植物产生系统性防御,抑制病毒的扩散。此策略不仅显著增强了大豆对SMV的抗性,也为分子诱饵策略(decoy engineering)在作物抗病性改良中的应用提供了新的理论基础和技术路径,展现了基因精准编辑在作物抗病性定向改良中的巨大潜力[124]。
大豆Rsv4抗病基因编码一种RNase H家族的蛋白,具有双链RNA核酸酶活性,能够降解病毒复制复合体(viral replication complex, VRC)中的关键成分——dsRNA,因此对包括SMV在内的多种potyvirus表现出广谱抗性。基于此发现,研究者将VRC中的寄主蛋白eIF(iso)4E或其他能够进入VRC的病毒相关蛋白与拟南芥的RNase RTL2进行融合表达。该融合蛋白能够靶向病毒复制的关键部位,并依赖RTL2的RNA酶活性降解病毒复制过程中产生的dsRNA,从而有效干扰病毒的正常复制。在本氏烟中表达这些融合蛋白时,TuMV、ToMV和CMV等病毒的积累量均显著下降,验证了该策略在抑制病毒扩增方面的有效性[96]。这一研究不仅深化了对寄主蛋白与病毒复制机制关系的理解,也为利用分子工具靶向病毒复制过程、提升植物抗病毒能力提供了新的思路和方法。
基因工程技术尤其是基因编辑技术的快速发展为大豆抗病育种提供了新工具和新策略,通过转入病毒抗性基因、RNAi构建或人工miRNA,可显著增强大豆对特定病毒的抗性。近年来,CRISPR-Cas9、CRISPR-SpRY等基因编辑技术在大豆上的应用,使得对大豆抗病毒基因的精准编辑成为可能 [125-127]。例如,敲除Potyvirus属病毒侵染所需的感病基因(如eIF4E、eIF(iso)4E等),阻断病毒的侵染和复制过程,获得持久广谱的抗性。
3 存在问题与展望尽管大豆病毒病害鉴定及抗病毒机制等研究取得了显著进展,但仍面临诸多挑战。首先,大豆病毒种类繁多且遗传变异速度快,导致新发病毒病害频繁出现,部分以往主要流行的病毒病害已不再是当前生产中的首要威胁。此外,单一抗性基因易被病毒突破,抗性持久性有限,难以实现长期有效的病害防控。其次,许多已鉴定的抗性基因其分子作用机制尚未被完全阐明,且抗性性状与农艺性状的协调改良存在较大难度。此外,转基因和基因编辑大豆产品主要集中于抗除草剂和抗鳞翅目昆虫等性状,针对抗病毒等其他生物性状的产业化大豆品种仍较为稀缺。展望未来,应进一步加强对大豆抗病毒分子机制的系统性研究,深入开展野生大豆及其他种质资源的抗病毒评价与利用,深入挖掘和解析新型抗病毒基因及其作用机制。在技术层面,应充分结合分子育种、基因编辑、人工智能和合成生物学等前沿技术,推动多学科交叉融合,加速培育高产、优质、广谱且环境适应能力强的抗病高产大豆新品种。同时,要强化病毒病害的监测与预警,特别是在全球气候变化和耕作制度转型的背景下,进一步加强生物安全管控和综合防控体系,为提升大豆单产水平和实现大豆产业的可持续发展提供坚实保障。
| [1] |
Graham PH, Vance CP. Legumes: importance and constraints to greater use. Plant Physiol, 2003, 131: 872-7. DOI:10.1104/pp.017004 |
| [2] |
Savary S, Willocquet L, Pethybridge SJ, et al. The global burden of pathogens and pests on major food crops. Nat Ecol Evol, 2019, 3: 430-9. DOI:10.1038/s41559-018-0793-y |
| [3] |
Hill JH, Whitham SA. Control of virus diseases in soybeans. Adv Virus Res, 2014, 90: 355-90. |
| [4] |
Elmore MG, Groves CL, Hajimorad MR, et al. Detection and discovery of plant viruses in soybean by metagenomic sequencing. Virol J, 2022, 19: 149. DOI:10.1186/s12985-022-01872-5 |
| [5] |
Stuckey RE, Ghabrial SA, Reicosky DA. Increased incidence of Phomopsis sp. in seeds from soybeans infected with bean pod mottle virus[Glycine max, Phomopsis sojae]. Plant Dis, 1982, 66: 826. DOI:10.1094/PD-66-826 |
| [6] |
Hajimorad MR, Domier LL, Tolin SA, et al. Soybean mosaic virus: a successful potyvirus with a wide distribution but restricted natural host range. Mol Plant Pathol, 2018, 19: 1563-79. DOI:10.1111/mpp.12644 |
| [7] |
Hopkins JD, Mueller AJ. Effect of bean pod mottle virus on soybean yield. J Econ Entomol, 1984, 77: 943-7. DOI:10.1093/jee/77.4.943 |
| [8] |
Anderson NR, Irizarry MD, Bloomingdale CA, et al. Effect of soybean vein necrosis on yield and seed quality of soybean. Can J Plant Pathol, 2017, 39: 334-41. DOI:10.1080/07060661.2017.1354333 |
| [9] |
Cheng R, Mei R, Yan R, et al. A new distinct geminivirus causes soybean stay-green disease. Mol Plant, 2022, 15: 927-30. DOI:10.1016/j.molp.2022.03.011 |
| [10] |
Wang X, Wang M, Wang L, et al. Whole-plant microbiome profiling reveals a novel geminivirus associated with soybean stay-green disease. Plant Biotechnol J, 2022, 20: 2159-73. DOI:10.1111/pbi.13896 |
| [11] |
He H, Li H, Wang Y, et al. Soybean stay-green associated geminivirus: a serious threat to soybean production in China. Virology, 2025, 602: 110312. DOI:10.1016/j.virol.2024.110312 |
| [12] |
叶文武, 刘万才, 王源超. 中国大豆病虫害发生现状及全程绿色防控技术研究进展. 植物保护学报, 2023, 50: 265-73. |
| [13] |
Inoue-Nagata AK, Jordan R, Kreuze J, et al. ICTV virus taxonomy profile: Potyviridae 2022. J Gen Virol, 2022, 103: 001738. |
| [14] |
Gardner M, Kendrick JB. Soybean mosaic. J Agricul Res, 1921, 22: 111-4. |
| [15] |
Ross JP. Effect of aphid-transmitted soybean mosaic virus on yields of closely related and susceptible soybean lines. Crop Sci, 1977, 17: 869-72. DOI:10.2135/cropsci1977.0011183X001700060014x |
| [16] |
Hill JH. Soybean mosaic[M]// Hartman GL, Sinclair JB, Rupe JC, eds. Compendium of soybean diseases. St. Paul, MN: APS Press, 1999
|
| [17] |
Hill JH, Bailey TB, Benner HI, et al. Soybean mosaic virus: effect of primary disease incidence on yield and seed quality. Plant Dis, 1987, 71: 237-9. DOI:10.1094/PD-71-0237 |
| [18] |
Jayaram CH, Hill JH, Miller WA. Complete nucleotide sequences of two soybean mosaic virus strains differentiated by response of soybean containing the Rsv resistance gene. J Gen Virol, 1992, 73: 2067-77. DOI:10.1099/0022-1317-73-8-2067 |
| [19] |
Chung BY, Miller WA, Atkins JF, et al. An overlapping essential gene in the Potyviridae. Proc Natl Acad Sci U S A, 2008, 105: 5897-902. DOI:10.1073/pnas.0800468105 |
| [20] |
Li K, Yang QH, Zhi HJ, et al. Identification and distribution of soybean mosaic virus strains in Southern China. Plant Dis, 2010, 94: 351-7. DOI:10.1094/PDIS-94-3-0351 |
| [21] |
Yang Y, Lin J, Zheng G, et al. Recombinant soybean mosaic virus is prevalent in Chinese soybean fields. Arch Virol, 2014, 159: 1793-6. DOI:10.1007/s00705-014-1980-z |
| [22] |
Takahashi K, Tanaka T, Wataru I, et al. Studies on virus diseases and causal viruses of soybean in Japan. Bull Tohoku Natl Agric Exp Stn, 1980, 62: 1-130. |
| [23] |
Cho EK, Goodman RM. Strains of soybean mosaic virus: classification based on virulence in resistant cultivars. Phytopathology, 1979, 69: 467-70. DOI:10.1094/Phyto-69-467 |
| [24] |
Buzzel RI, Tu JC. Inheritance of soybean resistance to soybean mosaic virus. J Hered, 1984, 75: 82. DOI:10.1093/oxfordjournals.jhered.a109878 |
| [25] |
Edwardson JR, Christie RG. Soybean mosaic virus[M]//Edwardson JR. The potyvirus group, vol. Ⅲ. Gainesville: University of Florida, 1991: 821-35
|
| [26] |
Gibbs AJ, Hajizadeh M, Ohshima K, et al. The Potyviruses: an evolutionary synthesis is emerging. Viruses, 2020, 12: 132. DOI:10.3390/v12020132 |
| [27] |
Jossey S, Hobbs HA, Domier LL. Role of soybean mosaic virus-encoded proteins in seed and aphid transmission in soybean. Phytopathology, 2013, 103: 941-8. DOI:10.1094/PHYTO-09-12-0248-R |
| [28] |
Bowers GR, Goodman RM. Strain specificity of soybean mosaic virus seed transmission in soybean. Crop Sci, 1991, 31: 1171-4. DOI:10.2135/cropsci1991.0011183X003100050019x |
| [29] |
Hill JH, Lucas BS, Benner HI, et al. Factors associated with the epidemiology of soybean mosaic virus in Iowa. Phytopathology, 1980, 70: 536-40. DOI:10.1094/Phyto-70-536 |
| [30] |
Porto MDM, Hagedorn DJ. Seed transmission of a Brazilian isolate of soybean mosaic virus. Phytopathology, 1975, 65: 713-6. DOI:10.1094/Phyto-65-713 |
| [31] |
Domier LL, Steinlage TA, Hobbs HA, et al. Similarities in seed and aphid transmission among soybean mosaic virus isolates. Plant Dis, 2007, 91: 546-50. DOI:10.1094/PDIS-91-5-0546 |
| [32] |
Calvert LA, Ghabrial SA. Enhancement by soybean mosaic virus of bean pod mottle virus titer in doubly infected soybean. Phytopathology, 1983, 73: 992-7. DOI:10.1094/Phyto-73-992 |
| [33] |
Malapi-Nelson M, Wen RH, Ownley BH, et al. Co-infection of soybean with soybean mosaic virus and alfalfa mosaic virus results in disease synergism and alteration in accumulation level of both viruses. Plant Dis, 2009, 93: 1259-64. DOI:10.1094/PDIS-93-12-1259 |
| [34] |
Stewart VB, Reddick D. Bean mosais. Phytopathology, 1917, 7: 61. |
| [35] |
Pierce WH. Viruses of the bean. Phytopathology, 1934, 24: 97-115. |
| [36] |
Gunasinghe U, Flasinski S, Nelson R, et al. Nucleotide sequence and genome organization of peanut stripe potyvirus. J Gen Virol, 1994, 75: 2519-25. DOI:10.1099/0022-1317-75-9-2519 |
| [37] |
Jordan R, Hammond J. Bean common mosaic virus and bean common mosaic necrosis virus[M]//Mahy BWJ, Van Regenmortel MHC. Encyclopedia of Virology, 3rd ed. New York: Academic Press, 2008: 288-95
|
| [38] |
Worrall EA, Wamonje FO, Mukeshimana G, et al. Bean common mosaic virus and bean common mosaic necrosis virus: relationships, biology, and prospects for control. Adv Virus Res, 2015, 93: 1-46. |
| [39] |
Morales F, Bos L. Descriptions of plant viruses: bean common mosaic virus. Dpv337[Z]. Wellesbourne UK: Association of Applied Biologists, 1988
|
| [40] |
郑红英, 陈炯, 侯明生, 等. 菜豆普通花叶病毒研究进展. 浙江农业学报, 2002, 14: 57-62. |
| [41] |
Powell G. Intracellular salivation is the aphid activity associated with inoculation of non-persistently transmitted viruses. J Gen Virol, 2005, 86: 469-72. DOI:10.1099/vir.0.80632-0 |
| [42] |
Donnelly R, Cunniffe NJ, Carr JP, et al. Pathogenic modification of plants enhances long-distance dispersal of nonpersistently transmitted viruses to new hosts. Ecology, 2019, 100: e02725. DOI:10.1002/ecy.2725 |
| [43] |
Aishwarya P, Rangaswamy KT, Basavaraju S, et al. Evaluation of the seed-borne nature of bean common mosaic virus (BCMV) in cowpea. Int J Curr Microbiol Appl Sci, 2020, 9: 239-45. DOI:10.20546/ijcmas.2020.911.028 |
| [44] |
Brunt A, Kenten RH. Cowpea mild mottle, a newly recognized virus infecting cowpeas (Vigna unguiculata) in Ghana. Ann Appl Biol, 1973, 74: 67-74. DOI:10.1111/j.1744-7348.1973.tb07723.x |
| [45] |
Thouvenel JC, Monsarrat A, Fauquet C. Isolation of cowpea mild mottle virus from diseased soybeans in the Ivory Coast. Plant Dis, 1982, 66: 336. DOI:10.1094/PD-66-336 |
| [46] |
Iwaki M. Whitefly transmission and some properties of cowpea mild mottle virus on soybean in Thailand. Plant Dis, 1982, 66: 365-8. DOI:10.1094/PD-66-365 |
| [47] |
Almeida AR, Piuga FF, Marim SR, et al. Detection and partial characterization of a carlavirus causing stem necrosis of soybean in Brazil. Fitopatologia Brasileira, 2005, 30: 191-4. DOI:10.1590/S0100-41582005000200016 |
| [48] |
Tavassoli M, Shahraeen N, Ghorbani S. Detection and some properties of cowpea mild mottle virus isolated from soybean in Iran. Pakistan J Biol Sci, 2008, 11: 2624-8. DOI:10.3923/pjbs.2008.2624.2628 |
| [49] |
Chang CA, Chien LY, Tsai CF, et al. First report of cowpea mild mottle virus in cowpea and French bean in Taiwan. Plant Dis, 2013, 97: 1001. |
| [50] |
杨晓, 张宇, 陈莎, 等. 豇豆轻斑驳病毒海南分离物全基因组序列测定及分子特征. 中国蔬菜, 2019, 6: 35-8. |
| [51] |
Wei Z, Wu G, Ye Z, et al. First report of cowpea mild mottle virus infecting soybean in China. Plant Dis, 2020, 104: 2534. |
| [52] |
Wei Z, Mao C, Jiang C, et al. Identification of a new genetic clade of cowpea mild mottle virus and characterization of its interaction with soybean mosaic virus in co-infected soybean. Front Microbiol, 2021, 12: 650773. DOI:10.3389/fmicb.2021.650773 |
| [53] |
Zanardo LG, Carvalho CM. Cowpea mild mottle virus (Carlavirus, Betaflexiviridae): a review. Trop Plant Pathol, 2017, 42: 417-30. DOI:10.1007/s40858-017-0168-y |
| [54] |
Xu M, Ai H, Yan R, et al. Natural occurrence of cowpea mild mottle virus infecting sesame (Sesamum indicum L.) in Anhui Province, China. Plant Dis, 2024, 108: 11. |
| [55] |
Muniyappa V. Transmission of cowpea mild mottle virus by Bemisia tabaci in a nonpersistent manner. Plant Dis, 1983, 67: 391-3. DOI:10.1094/PD-67-391 |
| [56] |
Zaumeyer WJ, Thomas HR. Pod mottle, a virus disease of beans. J Agric Res, 1948, 77: 81-96. |
| [57] |
Walters HJ. A virus disease complex in Arkansas. Phytopathology, 1958, 48: 346. |
| [58] |
Sanfacon H, Wellink J, Le-Gall O, et al. Secoviridae: a proposed family of plant viruses that combines the families Secquiviridae and Comoviridae, the unassigned genera Cheravirus and Sadwavirus, and the proposed genus Torradovirus. Arch Virol, 2009, 154: 899-907. DOI:10.1007/s00705-009-0367-z |
| [59] |
Ziems AD, Giesler LJ, Graef GL, et al. Response of soybean cultivars to bean pod mottle virus infection. Plant Dis, 2007, 91: 719-26. DOI:10.1094/PDIS-91-6-0719 |
| [60] |
Hobbs HA, Hartman GL, Wang Y, et al. Occurrence of seed coat mottling in soybean plants inoculated with bean pod mottle virus and soybean mosaic virus. Plant Dis, 2003, 87: 1333-6. DOI:10.1094/PDIS.2003.87.11.1333 |
| [61] |
Anjos JR, Jarlfors U, Ghabrial SA. Soybean mosaic potyvirus enhances the titer of two comoviruses in dually infected soybean plants. Phytopathology, 1992, 82: 1022-7. DOI:10.1094/Phyto-82-1022 |
| [62] |
易汪雪, 宋绍祎, 吴东妮, 等. 单管多重RT-PCR同时检测大豆种子中三种检疫性植物病毒. 植物保护, 2016, 42: 113-7. |
| [63] |
吕燕, 郭立新, 张慧丽, 等. 2009–2019年中国进口大豆中检疫性有害生物截获情况分析. 大豆科学, 2021, 40: 257-64. |
| [64] |
Giesler LJ, Ghabrial SA, Hunt TE, et al. Bean pod mottle virus: a threat to U.S. soybean production. Plant Dis, 2002, 86: 1280-9
|
| [65] |
Gu H, Ghabrial SA. The bean pod mottle virus proteinase cofactor and putative helicase are symptom severity determinants. Virology, 2005, 333: 271-83. DOI:10.1016/j.virol.2005.01.020 |
| [66] |
Gergerich RC. Bean pod mottle[M]//Hartman GL, Sinclair JB, Rupe JC, eds. Compendium of soybean diseases. 4th ed. St. Paul, MN: APS Press, 1999: 61-2
|
| [67] |
Lin J, Guo J, Finer J, et al. The bean pod mottle virus RNA2-encoded 58-kilodalton protein P58 is required in cis for RNA2 accumulation. J Virol, 2014, 88: 3213-22. DOI:10.1128/JVI.03301-13 |
| [68] |
Wickizer SL, Gergerich RC. First report of Japanese beetle (Popillia japonica) as a vector of southern bean mosaic virus and bean pod mottle virus. Plant Dis, 2007, 91: 637. |
| [69] |
Krell RK, Pedigo LP, Hill JH, et al. Potential primary inoculum sources of bean pod mottle virus in Iowa. Plant Dis, 2003, 87: 1416-22. DOI:10.1094/PDIS.2003.87.12.1416 |
| [70] |
Bradshaw JD, Rice ME, Hill JH. No-choice preference of Cerotoma trifurcata (Coleoptera: Chrysomelidae) to potential host plants of bean pod mottle virus (Comoviridae) in soybean. J Econ Entomol, 2007, 100: 808-14. DOI:10.1603/0022-0493(2007)100[808:NPOCTC]2.0.CO;2 |
| [71] |
徐彩龙, 韩天富, 吴存祥. 黄淮海夏大豆症青发生原因探讨与防治技术. 大豆科技, 2019(3): 22-8. DOI:10.3969/j.issn.1674-3547.2019.03.004 |
| [72] |
Cheng R, Yan R, Mei R, et al. Epidemiological evaluation and identification of the insect vector of soybean stay-green associated virus. Phytopathol Res, 2023, 5: 20. DOI:10.1186/s42483-023-00177-x |
| [73] |
Yin J, Hu Z, Xu S, et al. Leafhopper transmits soybean stay-green associated virus to leguminous plants. Phytopathol Res, 2023, 5: 17. DOI:10.1186/s42483-023-00172-2 |
| [74] |
Trębicki P, Harding RM, Powell KS. Anti-metabolic effects of Galanthus nivalisagglutinin and wheat germ agglutinin on nymphal stages of the common brown leafhopper using a novel artificial diet system. Entomol Exp Appl, 2009, 131: 99-105. |
| [75] |
Pei TH, Cui X, Shi SS, et al. An introductory review on the common brown leafhopper (Orosius orientalis): a new soybean pest. Oil Crop Sci, 2024, 9: 198-203. DOI:10.1016/j.ocsci.2024.07.003 |
| [76] |
邓一文, 刘裕强, 王静, 等. 农作物抗病虫研究的战略思考. 中国科学: 生命科学, 2021, 51: 1435-46. |
| [77] |
Liu JZ, Fang Y, Pang H. The current status of the soybean-soybean mosaic virus (SMV) pathosystem. Front Microbiol, 2016, 7: 1906. |
| [78] |
Khatabi B, Fajolu O, Wen RH, et al. Evaluation of North American isolates of soybean mosaic virus for gain of virulence on Rsv-genotype soybeans with special emphasis on resistance-breaking determinants on Rsv4. Mol Plant Pathol, 2012, 13: 1077-88. DOI:10.1111/j.1364-3703.2012.00817.x |
| [79] |
Wang D, Ma Y, Liu N, et al. Fine mapping and identification of the soybean RSC4 resistance candidate gene to soybean mosaic virus. Plant Breed, 2011, 130: 653-9. DOI:10.1111/j.1439-0523.2011.01888.x |
| [80] |
Wang D, Tian Z, Li K, et al. Identification and variation analysis of soybean mosaic virus strains in Shandong, Henan and Anhui provinces of China. Soybean Sci, 2013, 32: 806-9. |
| [81] |
Wang Y, Lu J, Chen S, et al. Exploration of presence/absence variation and corresponding polymorphic markers in soybean genome. J Integr Plant Biol, 2014, 56: 1009-19. DOI:10.1111/jipb.12208 |
| [82] |
Hayes A, Jeong S, Gore M, et al. Recombination within a nucleotide-binding-site/leucine-rich-repeat gene cluster produces new variants conditioning resistance to soybean mosaic virus in soybeans. Genetics, 2004, 166: 493-503. DOI:10.1534/genetics.166.1.493 |
| [83] |
Hajimorad MR, Hill JH. Rsv1-mediated resistance against soybean mosaic virus-N is hypersensitive response-independent at inoculation site, but has the potential to initiate a hypersensitive response-like mechanism. Mol Plant Microbe Interact, 2001, 14: 587-98. DOI:10.1094/MPMI.2001.14.5.587 |
| [84] |
黄赛花. 利用VIGS技术对大豆抗大豆花叶病毒候选基因的功能分析[D]. 南京: 南京农业大学, 2014
|
| [85] |
Zhang C, Grosic S, Whitham SA, et al. The requirement of multiple defense genes in soybean Rsv1–mediated extreme resistance to soybean mosaic virus. Mol Plant Microbe Interact, 2012, 25: 1307-13. DOI:10.1094/MPMI-02-12-0046-R |
| [86] |
Hajimorad MR, Eggenberger AL, Hill JH. Loss and gain of elicitor function of soybean mosaic virus G7 provoking Rsv1-mediated lethal systemic hypersensitive response maps to P3. J Virol, 2005, 79: 1215-22. DOI:10.1128/JVI.79.2.1215-1222.2005 |
| [87] |
Eggenberger A, Hajimorad M, Hill J. Gain of virulence on Rsv1-genotype soybean by an avirulent soybean mosaic virus requires concurrent mutations in both P3 and HC-Pro. Mol Plant Microbe Interact, 2008, 21: 931-6. DOI:10.1094/MPMI-21-7-0931 |
| [88] |
Suh SJ, Bowman BC, Jeong N, et al. The Rsv3 locus conferring resistance to soybean mosaic virus is associated with a cluster of coiled-coil nucleotide-binding leucine-rich repeat genes. Plant Genome, 2011, 4: 55-64. |
| [89] |
Ma FF, Wu M, Liu YN, et al. Molecular characterization of NBS-LRR genes in the soybean Rsv3 locus reveals several divergent alleles that likely confer resistance to the soybean mosaic virus. Theor Appl Genet, 2018, 131: 253-65. DOI:10.1007/s00122-017-2999-9 |
| [90] |
Zhang C, Hajimorad M, Eggenberger AL, et al. Cytoplasmic inclusion cistron of soybean mosaic virus serves as a virulence determinant on Rsv3-genotype soybean and a symptom determinant. Virology, 2009, 391: 240-8. DOI:10.1016/j.virol.2009.06.020 |
| [91] |
Seo JK, Kwon SJ, Cho WK, et al. Type 2C protein phosphatase is a key regulator of antiviral extreme resistance limiting virus spread. Sci Rep, 2014, 4: 5905. DOI:10.1038/srep05905 |
| [92] |
Alazem M, Tseng KC, Chang WC, et al. Elements involved in the Rsv3-mediated extreme resistance against an avirulent strain of soybean mosaic virus. Viruses, 2018, 10: 581. DOI:10.3390/v10110581 |
| [93] |
Alazem M, Widyasari K, Kim KH. An avirulent strain of soybean mosaic virus reverses the defensive effect of abscisic acid in a susceptible soybean cultivar. Viruses, 2019, 11: 879. DOI:10.3390/v11090879 |
| [94] |
Yin JL, Wang LQ, Jin TT, et al. A cell wall-localized NLR confers resistance to soybean mosaic virus by recognizing viral-encoded cylindrical inclusion protein. Mol Plant, 2021, 14: 1881-900. DOI:10.1016/j.molp.2021.07.013 |
| [95] |
Gunduz I, Buss GR, Chen P, et al. Genetic and phenotypic analysis of soybean mosaic virus resistance in PI 88788 soybean. Phytopathology, 2004, 94: 687-92. DOI:10.1094/PHYTO.2004.94.7.687 |
| [96] |
Ishibashi K, Saruta M, Shimizu T, et al. Soybean antiviral immunity conferred by dsRNase targets the viral replication complex. Nat Commun, 2019, 10: 4033. DOI:10.1038/s41467-019-12052-5 |
| [97] |
Klepadlo M, Chen P, Shi A, et al. Two tightly linked genes for soybean mosaic virus resistance in soybean. Crop Sci, 2017, 57: 1844-53. DOI:10.2135/cropsci2016.05.0290 |
| [98] |
Zheng C, Chen P, Gergerich R. Characterization of resistance to soybean mosaic virus in diverse soybean germplasm. Crop Sci, 2005, 45: 2503-9. DOI:10.2135/cropsci2005.0114 |
| [99] |
Widyasari K, Alazem M, Kim KH. Soybean resistance to soybean mosaic virus. Plants (Basel), 2020, 9: 219. |
| [100] |
Wang D, Ma Y, Yang Y, et al. Fine mapping and analyses of Rsc8 resistance candidate genes to soybean mosaic virus in soybean. Theor Appl Genet, 2011, 122: 555-65. DOI:10.1007/s00122-010-1469-4 |
| [101] |
Fu S, Zhan Y, Zhi H, et al. Mapping of SMV resistance gene Rsc-7 by SSR markers in soybean. Genetica, 2006, 128: 63-9. DOI:10.1007/s10709-005-5535-9 |
| [102] |
Yan H, Wang H, Cheng H, et al. Detection and fine-mapping of SC7 resistance genes via linkage and association analysis in soybean. J Integr Plant Biol, 2015, 57: 722-9. DOI:10.1111/jipb.12323 |
| [103] |
Yamashita Y, Takeuchi T, Ohnishi S, et al. Fine mapping of the major soybean dwarf virus resistance gene Rsdv1 of the soybean cultivar Wilis. Breeding Sci, 2013, 63: 417-22. DOI:10.1270/jsbbs.63.417 |
| [104] |
Gore MA, Hayes AJ, Jeong SC, et al. Mapping tightly linked genes controlling potyvirus infection at the Rsv1 and Rpv1 region in soybean. Genome, 2002, 45: 592-9. DOI:10.1139/g02-009 |
| [105] |
Jupe F, Witek K, Verweij W, et al. Resistance gene enrichment sequencing (RenSeq) enables reannotation of the NB-LRR gene family from sequenced plant genomes and rapid mapping of resistance loci in segregating populations. Plant J, 2013, 76: 530-44. DOI:10.1111/tpj.12307 |
| [106] |
Baulcombe D. RNA silencing in plants. Nature, 2004, 431: 356-63. DOI:10.1038/nature02874 |
| [107] |
Lopez-Gomollon S, Baulcombe DC. Roles of RNA silencing in viral and non-viral plant immunity and in the crosstalk between disease resistance systems. Nat Rev Mol Cell Biol, 2022, 23: 645-62. |
| [108] |
Steinlage TA, Hill JH, Nutter FW. Temporal and spatial spread of soybean mosaic virus (SMV) in soybeans transformed with the coat protein gene of SMV. Phytopathology, 2002, 92: 478-86. DOI:10.1094/PHYTO.2002.92.5.478 |
| [109] |
Gao L, Ding X, Li K, et al. Characterization of soybean mosaic virus resistance derived from inverted repeat-SMV-HC-Pro genes in multiple soybean cultivars. Theor Appl Genet, 2015, 128: 1489-505. DOI:10.1007/s00122-015-2522-0 |
| [110] |
Tougou M, Furutani N, Yamagishi N, et al. Development of resistant transgenic soybeans with inverted repeat-coat protein genes of soybean dwarf virus. Plant Cell Rep, 2006, 25: 1213-8. DOI:10.1007/s00299-006-0186-6 |
| [111] |
Yang X, Niu L, Zhang W, et al. Robust RNAi-mediated resistance to infection of seven potyvirids in soybean expressing an intron hairpin NIb RNA. Transgenic Res, 2017, 26: 665-76. DOI:10.1007/s11248-017-0041-2 |
| [112] |
Zhang X, Sato S, Ye X, et al. Robust RNAi-based resistance to mixed infection of three viruses in soybean plants expressing separate short hairpins from a single transgene. Phytopathology, 2011, 101: 1264-9. DOI:10.1094/PHYTO-02-11-0056 |
| [113] |
Gao L, Luo J, Ding X, et al. Soybean RNA interference lines silenced for eIF4E show broad potyvirus resistance. Mol Plant Pathol, 2020, 21: 303-17. DOI:10.1111/mpp.12897 |
| [114] |
Zhao S, Li Y. Current understanding of the interplays between host hormones and plant viral infections. PLoS Pathog, 2021, 17: e1009242. DOI:10.1371/journal.ppat.1009242 |
| [115] |
Wang J, Shine M, Gao QM, et al. Enhanced disease susceptibility1 mediates pathogen resistance and virulence function of a bacterial effector in soybean. Plant Physiol, 2014, 165: 1269-84. DOI:10.1104/pp.114.242495 |
| [116] |
Shang J, Zhao LP, Yang XM, et al. Soybean balanced the growth and defense in response to SMV infection under different light intensities. Front Plant Sci, 2023, 14: 1150870. DOI:10.3389/fpls.2023.1150870 |
| [117] |
Li H, Liu J, Yuan X, et al. Comparative transcriptome analysis reveals key pathways and regulatory networks in early resistance of Glycine max to soybean mosaic virus. Front Microbiol, 2023, 14: 1241076. DOI:10.3389/fmicb.2023.1241076 |
| [118] |
Graham TL, Graham MY, Subramanian S, et al. RNAi silencing of genes for elicitation or biosynthesis of 5-deoxyisoflavonoids suppresses race-specific resistance and hypersensitive cell death in Phytophthora sojae infected tissues. Plant Physiol, 2007, 144: 728-40. DOI:10.1104/pp.107.097865 |
| [119] |
Andres A, Donovan SM, Kuhlenschmidt MS. Soy isoflavone and virus infections. J Nutr Biochem, 2009, 20: 563-9. DOI:10.1016/j.jnutbio.2009.04.004 |
| [120] |
Dixon RA, Paiva NL. Stress-induced phenylpropanoid metabolism. Plant Cell, 1995, 7: 1085-97. DOI:10.2307/3870059 |
| [121] |
Ahmad N, Xu Y, Zang F, et al. The evolutionary trajectories of specialized metabolites towards antiviral defense system in plants. Mol Hortic, 2024, 4: 2. DOI:10.1186/s43897-023-00078-9 |
| [122] |
Cheng H, Yang H, Zhang D, et al. Polymorphisms of soybean isoflavone synthase and flavanone 3-hydroxylase genes are associated with soybean mosaic virus resistance. Mol Breeding, 2010, 25: 13-24. DOI:10.1007/s11032-009-9305-8 |
| [123] |
Zhang P, Du H, Wang J, et al. Multiplex CRISPR/Cas9-mediated metabolic engineering increases soya bean isoflavone content and resistance to soya bean mosaic virus. Plant Biotechnol J, 2020, 18: 1384-95. DOI:10.1111/pbi.13302 |
| [124] |
Helm M, Qi M, Sarkar S, et al. Engineering a decoy substrate in soybean to enable recognition of the soybean mosaic virus NIa protease. Mol Plant Microbe Interact, 2019, 32: 760-9. DOI:10.1094/MPMI-12-18-0324-R |
| [125] |
Chen K, Wang Y, Zhang R, et al. CRISPR/Cas genome editing and precision plant breeding in agriculture. Annu Rev Plant Biol, 2019, 70: 667-97. DOI:10.1146/annurev-arplant-050718-100049 |
| [126] |
Zhang Z, Baxter AE, Ren D, et al. Efficient engineering of human and mouse primary cells using peptide-assisted genome editing. Nat Biotechnol, 2024, 42: 305-15. DOI:10.1038/s41587-023-01756-1 |
| [127] |
Chen X, Zhong Z, Tang X, et al. Advancing PAM-less genome editing in soybean using CRISPR-SpRY. Hortic Res, 2024, 11: uhae160. DOI:10.1093/hr/uhae160 |
 2025, Vol. 37
2025, Vol. 37 

 徐毅,南京农业大学植物保护学院教授、博士生导师。国家级青年人才,农业农村部神农青年英才。2008年获南京农业大学学士学位,2013年获得浙江大学博士学位,2014—2019年在美国康奈尔大学从事博士后研究,2019年起在南京农业大学任职。主要从事大豆等重要作物病毒致害灾变机制及防控技术研究。以第一或通讯作者在Annual Review of Phytopathology、Molecular Plant、PLoS Pathogens等核心学术期刊发表多篇研究论文,相关研究论文被ESI列为高被引论文
徐毅,南京农业大学植物保护学院教授、博士生导师。国家级青年人才,农业农村部神农青年英才。2008年获南京农业大学学士学位,2013年获得浙江大学博士学位,2014—2019年在美国康奈尔大学从事博士后研究,2019年起在南京农业大学任职。主要从事大豆等重要作物病毒致害灾变机制及防控技术研究。以第一或通讯作者在Annual Review of Phytopathology、Molecular Plant、PLoS Pathogens等核心学术期刊发表多篇研究论文,相关研究论文被ESI列为高被引论文