(2 生物育种钟山实验室,南京 210014)
(3 中国农业科学院植物保护研究所/植物病虫害综合治理全国重点实验,北京 100193)
(4 广东省农业科学院水稻研究所/广东省水稻科学技术重点实验室/广东省水稻工程实验室,广州 510640)
(2 Zhongshan Biological Breeding Laboratory, Nanjing 210014, China)
(3 State Key Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural Sciences, Beijing 100193, China)
(4 Rice Research Institute of Guangdong Academy of Agricultural Sciences, Guangdong Key Laboratory of Rice Science and Technology, Guangdong Rice Engineering Laboratory, Guangzhou 510640, China)
稻瘟病是由真菌Magnaporthe oryzae侵染引起的全球性水稻主要病害,严重威胁水稻安全生产。稻瘟病在水稻整个生长周期及各个部位均可发生,根据发病时期、部位可分为苗瘟、叶瘟、叶枕瘟、秆瘟、节瘟、穗颈瘟、枝梗瘟和谷粒瘟等,其中叶瘟和穗颈瘟最为常见,且危害较大(图 1)。全球每年因稻瘟病造成的产量损失高达10%~30%,重发年份甚至绝产,颗粒无收[1]。目前在亚洲、非洲水稻种植国家,稻瘟病已成为影响当地水稻安全生产的最主要病害之一[2],印度东部每年因稻瘟病减产约30%[3],马来西亚因穗颈瘟造成的水稻产量损失达50%~70%[4]。近年来,中国稻瘟病流行愈发严重,2012—2021年间稻瘟病年均发生面积达381.5万公顷次,稻谷每年平均损失36.8万吨[5]。2014年,因水稻成熟后期低温高湿气候影响,江苏、安徽、湖北、山东等地稻瘟病发生严重,部分地区近乎绝收[6-7]。利用抗病基因培育抗病品种是控制该病害最经济、有效和环保的措施,提高稻瘟病抗性,尤其是广谱抗性,是抗病育种工作的难点和热点问题,挖掘、克隆抗病基因并解析其抗病分子机理,对培育抗病品种具有重要的意义。

|
A:苗期叶瘟;B:穗颈瘟 图 1 2023年江苏省连云港市赣榆区田间稻瘟病自然发病 |
水稻稻瘟病抗病(resistance, R)基因被育种家优先作为目标基因应用于水稻品种改良。广谱抗性(broad-spectrum resistance, BSR) 是指植物对多个不同病原菌,或对同一病原菌的不同小种表现抗病性。截至目前已鉴定到140多个稻瘟病R基因,其中38个被克隆[8-9],大部分已克隆的R基因编码核苷酸结合富亮氨酸重复结构域受体蛋白(nucleotide-binding and leucine-rich repeat receptor, NLR)[10]。NLR类R蛋白一般直接或间接识别特异的病原菌效应因子从而产生抗病反应[11-13],导致这类R基因只能特异性识别对应的无毒基因,抗谱相对较窄。目前仅Pi1、Pi2、Pi5、Pi56(t)、Pi64、Pi9、Piz-t、Pizh、Pi50、Pigm和Pijx等被报道对稻瘟菌多个生理小种表现广谱抗性[14-16],并且仅有少数NLR类R基因如Pb1、Pi64、Pigm和Pijx等被报道具有广谱穗瘟抗性[17-21]。NLR类R基因通常以串联重复形式存在于水稻基因组中,如第6、11、12号染色体上分别成簇分布了21、27和27个R基因[9, 22]。同簇内的R基因在分子功能上并不完全相同,已克隆的Pigm位点含有13个NLR基因(R1~R13),其中R4、R6 (PigmR)和R8 (PigmS)编码完整的NLR蛋白,R4无稻瘟病抗性功能,PigmR对稻瘟菌多个生理小种具有广谱抗性,而PigmS主要通过提高产量以抵消PigmR引起的产量损失,以此来实现抗性和产量的平衡[16]。
此外,与R基因不同,一些抗病调控因子(包括抗病正调控因子和感病因子)往往对同一病原菌的不同小种具有部分抗性,通常参与抗病信号通路或代谢物生物合成途径[23-24]。近年来,许多具有广谱抗性的抗病调控因子或单倍体型被鉴定 [23, 25]。抗病正调控因子IPA1 (Ideal Plant Architecture 1)编码的类Squamosa启动子结合蛋白IPA1,在稻瘟病菌侵染时被磷酸化,磷酸化的IPA1与WRKY45的启动子结合,并促进其表达,增强水稻对稻瘟病的广谱抗性[26]。感病因子Bsr-d1 (BROAD-SPECTRUM RESISTANCE DIGU 1)编码C2H2类转录因子,克隆自籼稻广谱持久抗病品种地谷,该转录因子上游受转录因子MYBS1负调控,下游正调控过氧化物酶基因的表达从而调节H2O2积累,进而影响稻瘟病广谱抗性。对全球收集的3 000份种质资源进行单倍体型分析发现,仅313份水稻材料含有bsr-d1抗病单倍体型[23]。感病因子ROD1 (RESISTANCE OF RICE TO DISEASES 1)编码的Ca2+传感器通过激活过氧化氢酶活性,加速H2O2降解,抑制免疫反应。因此,ROD1的功能缺失导致水稻对稻瘟病、纹枯病、白叶枯病等多种病害存在广谱抗性,在籼稻中普遍存在ROD1的抗病单倍体型ROD1 (SNP1A),该单倍体型可以在不影响产量的情况下增强对稻瘟病的广谱抗性,具有重要的育种应用价值[27]。当病原菌入侵时,抗病正调控因子UMP1R2115编码的蛋白酶体成熟因子通过增加水稻26S蛋白酶体的生物合成与活性,促进过氧化物酶APX8和过氧化氢酶CatB的降解,提高侵染位点H2O2的积累,从而增强水稻对多种病原菌的抵御能力。进一步研究发现来自“雅恢2115”中的UMP1R2115是一个稀有的天然等位基因,导入到感病水稻品种后,能在不影响水稻产量的同时提高稻瘟病抗性,具有重要的应用潜力[28]。
3 R基因的育种应用 3.1 单个R基因的育种应用在目前已克隆的R基因中,具有育种实效,且在生产上大面积应用的较少。近10年来应用最多的主要为位于第6号染色体的Piz基因簇和第11号染色体的Pik基因簇的广谱抗性复等位基因(表 1)[29-60]。中国水稻研究所钱前院士团队通过标记辅助选择(marker-assisted selection, MAS)将Pigm导入到寒地优良粳稻品种“空育131”“龙粳26”和“垦鉴稻6号”中,获得12个携带Pigm基因的优良导入系,其穗瘟抗性水平普遍由受体亲本的5~7级提高至0~3级[29]。福建省农业科学院王锋研究员团队将Pi9和Pi2基因导入“闽恢3301”中,改良后的“闽恢3301-Pi9”和“闽恢3301-Pi2”抗性水平显著提升[30]。安徽荃银高科种业股份有限公司张从合研究员团队以“Q211S”为受体材料,以携带Pi9基因的抗稻瘟病材料“75-1-127”为供体,通过MAS选育出5个对苗瘟、穗瘟均表现较高抗性的新两系不育系[31]。扬州大学左示敏教授团队将Pigm导入到两个高产优质粳稻“武运粳32”和“华粳8号”中,选育出了抗稻瘟病优质高产新品种“扬农粳3091”[32]。李爱宏研究员团队利用扬稻6号(又名93-11)为核心亲本,通过Pigm的定向导入和综合农艺性状筛选,育成了具有广谱抗性的核心种质资源“抗9311”[15],利用“抗9311”又培育了系列携带Pigm的抗病耐高温优质不育系,包括两系不育系“缘88S”,改良稻瘟病抗性的不育系还包括“C815S”“隆科638S”及“KT27S”等[33-35]。此外,印度科研人员将Pik基因簇的Pi1与Pi54分别导入到优质高产品种“BPT5204”中,改良系的苗瘟抗性水平均提升至0~2级[36-37]。
| 表 1 稻瘟病抗性基因的育种应用 |
由于稻瘟病菌具有高度变异特性,携带单个R基因的抗病品种一般推广应用3~5年后便极易丧失抗性而沦为感病品种,聚合多个R基因培育持久广谱抗病品种是解决该问题的有效途径。国内多个研究团队均报道了多个R基因聚合提高抗谱的应用实例[57, 61],李爱宏研究员团队发现Piz基因簇上不同复等位R基因(尤其Pigm、Piz-t)与Pik、Pita座位的R基因以及其他R基因聚合在不同背景下均表现出稳定的广谱抗性[19-20, 62-63]。但是不同R基因聚合后,并不简单地表现为单个R基因的抗谱累加,有的还存在负向效应。国际水稻研究所以“CO39”为背景构建携带不同R基因的聚合系,鉴定结果显示Piz-5和Pita的基因聚合系抗性不如单基因抗性品系[64]。由于基因间存在较强的互作,会产生多种效应[65-67],例如在籼稻“扬稻6号”和粳稻“07GY31”背景下构建的多基因聚合系对稻瘟病菌株的抗性效应可以归为重叠效应、互补效应和互作效应三种类型,其中互补效应是影响聚合系抗谱的关键因素[62-63]。聚合具有不同抗病信号通路的R基因是实现稳定广谱抗性的一种方式,李爱宏研究员团队发现Pijx与Pigm/Piz-t在系统发育树上分属于不同分支,并处于不同信号通路,Pijx与Pigm/Piz-t聚合后可产生更为持久、广谱的抗性[21]。
3.3 R基因的分子设计育种应用随着基因组学的快速发展,通过解析品种抗病产量性状遗传规律,利用组学和基因编辑技术等手段实现稻瘟病抗性与高产优质性状协同提升成为可能。中国科学院李家洋院士团队联合储成才研究员团队、卜庆云研究员团队对收集的东北稻区546份水稻资源进行了重测序,对22个农艺性状表型进行了调查和全基因组关联分析,构建了我国东北稻区分子诠释模型,并在此基础上建立了分子设计育种体系,聚合了抽穗期基因Hd1,稻瘟病抗性基因Pita、Ptr、Pib,抗倒伏基因SCM2,粒型基因GW5、GS3、GL7,产量性状位点qSB2、qSB8、qSB10等一系列分子模块,育成的“中科发5号”在高纬度稻区实现了抗病、丰产、优质、抗倒伏等优异性状的聚合[68]。李爱宏研究员团队在解析长江中游地区200余份代表性粳稻品种抗病产量优质性状遗传改良规律的基础上,发现第6号染色体Piz基因座抗病等位基因与稻米品质劣势基因的不利连锁是制约品种产量、品质、抗病性平衡的关键因素,利用基因组设计育种策略,通过核心亲本选择和关键基因连锁累赘打破,利用广谱抗病基因Piz-t和Pigm分别创制了高产优质抗病新品种“香缘99”和“金香玉1号”[69](图 2)。
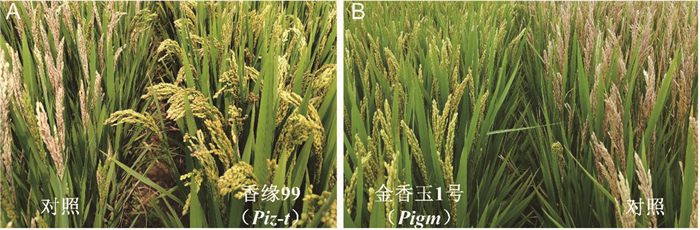
|
A: 2023年江苏省连云港市赣榆区“香缘99”田间抗病表现;B: 2020年江苏省扬州市“金香玉1号”田间抗病表现 图 2 “香缘99”与“金香玉1号”的田间抗病表现 |
与R基因相反,水稻感病因子是能够促进病原菌成功侵染,并协助病原菌与寄主植物发生亲和性反应的关键因子,感病因子的突变或下调表达可能赋予寄主非小种特异性的持久抗性[70]。因此,编辑或操纵感病因子对培育高产高抗的水稻品种具有重要应用潜力。通过规律间隔成簇短回文重复序列/规律间隔成簇短回文重复序列相关蛋白系统9 (clustered regularly interspaced short palindromic repeats/Cas9, CRISPR/Cas9)技术对“空育131”的感病因子Pi21进行定点编辑,显著增强了其稻瘟病抗性[71]。吉林省农业科学院金永梅研究员团队通过CRISPR/Cas9技术在“吉粳88”中对感病因子Bsr-d1进行编辑,创制的两个突变株系对中国东北地区的稻瘟病菌株的苗瘟抗性显著提高[72]。宁约瑟研究员团队通过CRISPR/Cas9技术同时编辑Pi21、Bsr-d1和Xa5等多个感病因子,创制了广谱抗病水稻材料[73]。此外,对RNG1和RNG3的编码区编辑,获得的编码区移码突变材料均实现了对稻瘟病和白叶枯病的广谱抗性[74]。因此,水稻感病因子的编辑为水稻抗病分子育种提供了新的广谱抗病基因和种质资源。
4 问题与挑战 4.1 苗瘟、穗瘟抗性差异的分子机理还有待研究目前已克隆的稻瘟病抗病基因绝大多数是针对苗瘟的,然而苗瘟和穗瘟的抗性很多时候表现并不一致,很多苗瘟抗性好的品种往往穗瘟抗性较差[75-76]。多组学分析表明苗瘟和穗瘟可能因发生在不同的发育阶段而有着不同的抗病分子机理[77-78]。目前抗病分子机理解析主要在苗瘟抗性,穗瘟抗性的分子机理仅有Pb1、Pijx、Pigm等少数R基因被解析[21, 79]。云南省农业科学院杨勤忠研究员团队[80]发现在水稻单基因系“IRBL9-W (Pi9)”中,相较于苗期叶片,Pi9在成株期穗部的表达量出现了明显降低,这可能是造成“IRBL9-W”成株期感穗瘟的原因。Hayashi等[17]发现Pb1抗穗瘟而感苗瘟的原因可能也是Pb1的表达水平在营养生长期显著低于生殖生长期。此外,苗瘟与穗瘟还存在不同的抗性调控途径[81-82],如OsGF14b通过茉莉酸(jasmonic acid, JA) 和水杨酸(salicylic acid, SA) 信号途径正调控穗瘟抗性而负调控苗瘟抗性[83]。
4.2 遗传背景影响R基因抗性水平育种实践表明R基因在导入不同遗传背景品种后抗性水平存在显著差异,如Pid3-I1在不同遗传背景下对来自日本的稻瘟菌株表现为部分抗性,而在“TP309”遗传背景下则对来自中国的稻瘟菌株表现出广谱抗性[84]。Pi54、Pita在籼、粳基因组中均有分布,但Pi54仅在籼稻背景下具有较好抗性水平,而Pita则在粳稻背景下具有较强抗性[85]。此外,在粳稻背景下聚合Pi2/Pi1,其苗瘟及穗瘟抗性频率分别为69.72%和46.43%,而在籼稻背景下其抗性频率则分别高达95.57%和83.33%[62-63]。其可能原因是在不同遗传背景中还存在着影响R基因抗性效应发挥的调控基因,如品种“Kanto209”背景中存在3个Pb1负调控因子,导致Pb1导入系表现为感病[86]。
5 展望大量水稻种质资源和稻瘟病菌株的重测序研究从基因组水平上揭示了稻瘟病抗性改良的遗传规律,发掘的新R基因/位点及抗性单倍型有效推进了抗稻瘟病遗传育种[87-88]。尽管稻瘟病抗病育种取得了有效进展,但由于全球气候变化以及种植结构的变革,导致稻瘟菌种群变异加剧,稻瘟病依然是制约水稻高产稳产的关键因素。稻瘟病防治目前还存在病害监测与诊断技术落后、具有育种实效的抗性基因/位点缺乏、抗病育种效率低、抗病品种培育滞后等问题,今后还需要在以下重点领域开展研究。(1)开展稻瘟菌种群变异分析,实现抗病品种合理布局。全球气候变化、种植结构调整和单一基因大面积利用往往严重影响田间稻瘟菌种群的演化。需系统采集我国主要稻区的田间稻瘟菌菌株,明确稻瘟菌种群变异规律,进而根据“基因对基因”假设,合理布局抗病品种。(2)加强稻瘟病早期监测预警,为稻瘟病预防提供依据。稻瘟菌的早期监测是防控稻瘟病的重要依据。多组学技术可用于监测侵染过程中稻瘟菌和水稻的基因表达、蛋白质变化和代谢产物的积累等。通过对多组学数据的分析与挖掘,建立稻瘟病的早期监测诊断模型,为稻瘟病预防提供依据。(3)加大抗病种质资源鉴定,挖掘、克隆新的R基因。抗病种质资源是稻瘟病抗病育种的基础,加强对已有种质资源及人为定向创制材料的抗病性鉴定,为培育广谱持久抗性水稻品种提供更多的基因资源。(4)研究稻瘟菌与水稻互作分子机制,解决不同遗传背景抗性不稳定难题。综合运用多组学技术深入研究稻瘟菌与水稻互作的分子机制。从基因组、转录组、蛋白质组和代谢组等多个层面鉴定水稻与稻瘟菌互作关键因子以及关键调控基因,为培育持久广谱抗性新品种提供理论基础。(5)创新智能设计育种新策略,创制高产优质抗病广适新品种。在常规育种基础上,结合分子设计育种和大数据分析,创新智能设计育种新策略,进而培育高产、优质、抗病等多性状协同提升的水稻新品种。
| [1] |
Devanna BN, Jain P, Solanke AU, et al. Understanding the dynamics of blast resistance in rice-Magnaporthe oryzae interactions. J Fungi, 2022, 8: 584. DOI:10.3390/jof8060584 |
| [2] |
Wang GL, Valent B. Durable resistance to rice blast. Science, 2017, 355: 906-7. DOI:10.1126/science.aam9517 |
| [3] |
Jha AK, Singh KM, Meena MS, et al. Constraints of rainfed rice production in Eastern India: an overview. SSRN Electronic J, 2012, 14: 71-84. |
| [4] |
Ashkani S, Rafii M, Shabanimofrad M, et al. Genetic analysis of resistance to rice blast: a study on the inheritance of resistance to the blast disease pathogen in an F3 population of rice. J Phytopathol, 2014, 163: 300-9. |
| [5] |
王如意, 刘杰, 冯琴, 等. 稻瘟病抗性分子机制及抗病育种策略研究进展. 植物保护, 2023, 49: 32-42. |
| [6] |
房文文, 杨军, 薛芳, 等. 山东省部分稻瘟病菌生理小种分析. 山东农业科学, 2020, 52: 111-4. |
| [7] |
陆明红, 刘万才, 朱凤, 等. 2014年稻瘟病重发原因分析与治理对策探讨. 中国植保专刊, 2015, 35: 35-9. |
| [8] |
Sahu PK, Sao R, Choudhary DK, et al. Advancement in the breeding, biotechnological and genomic tools towards development of durable genetic resistance against the rice blast disease. Plants, 2022, 11: 2386. DOI:10.3390/plants11182386 |
| [9] |
Younas MU, Wang G, Du H, et al. Approaches to reduce rice blast disease using knowledge from host resistance and pathogen pathogenicity. Int J Mol Sci, 2023, 24: 4985. DOI:10.3390/ijms24054985 |
| [10] |
Li W, Deng YW, Ning YS, et al. Exploiting broad-spectrum disease resistance in crops: from molecular dissection to breeding. Annu Rev Plant Biol, 2020, 71: 575-603. DOI:10.1146/annurev-arplant-010720-022215 |
| [11] |
McHale L, Tan X, Koehl P, et al. Plant NBS-LRR proteins: adaptable guards. Genome Biol, 2006, 7: 212. |
| [12] |
Kourelis J, Adachi H. Activation and regulation of NLR immune receptor networks. Plant Cell Physiol, 2022, 63: 1366-77. DOI:10.1093/pcp/pcac116 |
| [13] |
Ngou BPM, Ding P, Jones JDG. Thirty years of resistance: Zig-Zag through the plant immune system. Plant Cell, 2022, 34: 1447-78. DOI:10.1093/plcell/koac041 |
| [14] |
Liu ZQ, Zhu YJ, Shi HB, et al. Recent progress in rice broad-spectrum disease resistance. Int J Mol Sci, 2021, 22: 11658. DOI:10.3390/ijms222111658 |
| [15] |
Xiao N, Wu YY, Li AH. Strategy for use of rice blast resistance genes in rice molecular breeding. Rice Sci, 2020, 27: 263. DOI:10.1016/j.rsci.2020.05.003 |
| [16] |
Deng YW, Zhai KR, Xie Z, et al. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance. Science, 2017, 355: 962-5. DOI:10.1126/science.aai8898 |
| [17] |
Hayashi N, Inoue H, Kato T, et al. Durable panicle blast-resistance gene Pb1 encodes an atypical CC-NBS-LRR protein and was generated by acquiring a promoter through local genome duplication. Plant J, 2010, 64: 498-510. DOI:10.1111/j.1365-313X.2010.04348.x |
| [18] |
Ma J, Lei C, Xu X, et al. Pi64, encoding a novel CC-NBS-LRR protein, confers resistance to leaf and neck blast in rice. Mol Plant Microbe Intact, 2015, 28: 558-68. DOI:10.1094/MPMI-11-14-0367-R |
| [19] |
Wu YY, Yu L, Pan CH, et al. Development of near-isogenic lines with different alleles of Piz locus and analysis of their breeding effect under Yangdao 6 background. Mol Breeding, 2016, 36: 12. DOI:10.1007/s11032-016-0433-7 |
| [20] |
Wu YY, Chen Y, Pan CH, et al. Development and evaluation of near-isogenic lines with different blast resistance alleles at the Piz locus in japonica rice from the lower region of the Yangtze River, China. Plant Dis, 2017, 101: 1283-91. DOI:10.1094/PDIS-12-16-1855-RE |
| [21] |
Xiao N, Wu YY, Zhang XX, et al. Piix confers broad-spectrum seedling and panicle blast resistance by promoting the degradation of ATP β subunit and OsRbohC-mediated ROS burst in rice. Mol Plant, 2023, 16: 1832-46. DOI:10.1016/j.molp.2023.10.001 |
| [22] |
毛洧, 陈学伟, 王静. 水稻抗稻瘟病机制的研究进展. 中国科学: 生命科学, 2022, 52: 1495-510. |
| [23] |
Li WT, Zhu ZW, Chern M, et al. A natural allele of a transcription factor in rice confers broad-spectrum blast resistance. Cell, 2017, 170: 114-26. DOI:10.1016/j.cell.2017.06.008 |
| [24] |
Li WT, Chern M, Yin JJ, et al. Recent advances in broad-spectrum resistance to the rice blast disease. Curr Opin Plant Biol, 2019, 50: 114-20. DOI:10.1016/j.pbi.2019.03.015 |
| [25] |
Sha G, Sun P, Kong X, et al. Genome editing of a rice CDP-DAG synthase confers multipathogen resistance. Nature, 2023, 618: 1017-23. DOI:10.1038/s41586-023-06205-2 |
| [26] |
Wang J, Zhou L, Shi H, et al. A single transcription factor promotes both yield and immunity in rice. Science, 2018, 361: 1026-8. DOI:10.1126/science.aat7675 |
| [27] |
Gao M, He Y, Yin X, et al. Ca2+ sensor-mediated ROS scavenging suppresses rice immunity and is exploited by a fungal effector. Cell, 2021, 184: 5391-404. DOI:10.1016/j.cell.2021.09.009 |
| [28] |
Hu XH, Shen S, Wu JL, et al. A natural allele of proteasome maturation factor improves rice resistance to multiple pathogens. Nat Plants, 2023, 9: 228-37. DOI:10.1038/s41477-022-01327-3 |
| [29] |
田红刚, 陈红旗, 胡江, 等. 抗稻瘟病基因Pigm导入对寒地粳稻抗病性和产量性状的影响. 沈阳农业大学学报, 2016, 47: 520-6. DOI:10.3969/j.issn.1000-1700.2016.05.002 |
| [30] |
田大刚, 杨小双, 陈子强, 等. 利用分子标记辅助选择改良闽恢3301稻瘟病抗性. 南方农业学报, 2019, 50: 1665-70. DOI:10.3969/j.issn.2095-1191.2019.08.03 |
| [31] |
黄艳玲, 严志, 王慧, 等. 分子标记辅助选择定向改良不育系Q211S稻瘟病的抗性. 中国农学通报, 2018, 34: 135-40. |
| [32] |
Feng ZM, Li MY, Xu ZW, et al. Development of rice variety with durable and broad-spectrum resistance to blast disease through marker-assisted introduction of Pigm gene. Front Plant Sci, 2022, 13: 937767. DOI:10.3389/fpls.2022.937767 |
| [33] |
向聪, 任西明, 雷东阳, 等. 分子标记辅助选择改良C815S的稻瘟病抗性. 湖南农业大学学报(自然科学版), 2018, 44: 62-5. |
| [34] |
Dai XJ, He C, Zhou L, et al. Identification of a specific molecular marker for the rice blast-resistant gene Pigm and molecular breeding of thermo-sensitive genic male sterile leaf-color marker lines. Mol Breeding, 2018, 38: 72. DOI:10.1007/s11032-018-0821-2 |
| [35] |
Wang K, Fu C, Fu X, et al. Enhancing the blast resistance of an elite thermo-sensitive genic male sterile line (TGMS) Longke 638S and its derived hybrid varieties by incorporating Pigm gene. 2024. https://doi.org/10.21203/rs.3.rs-3908381/v1
|
| [36] |
Kumar SV, Srinivas-Prasad M, Rambabu R, et al. Marker-assisted introgression of Pi-1 gene conferring resistance to rice blast pathogen Pyricularia oryzae in the background of Samba Mahsuri. Int J Curr Microbiol Appl Sci, 2019, 8: 2133-46. |
| [37] |
Kumar SV, Rambabu R, Bhaskar B, et al. Introgression of durable blast resistance gene Pi-54 into indica rice cv. samba mahsuri, through marker assisted backcross breeding. Electronic J Plant Breeding, 2018, 9: 705-15. DOI:10.5958/0975-928X.2018.00084.4 |
| [38] |
Jiang J, Mou T, Yu H, et al. Molecular breeding of thermo-sensitive genic male sterile (TGMS) lines of rice for blast resistance using Pi2 gene. Rice, 2015, 8: 11. DOI:10.1186/s12284-015-0048-3 |
| [39] |
Yang D, Tang J, Yang D, et al. Improving rice blast resistance of Feng39S through molecular marker-assisted backcrossing. Rice, 2019, 12: 70. |
| [40] |
邹俊锋, 李永聪, 刘雄伦, 等. MAS育种改良籼稻恢复系'E32'及其杂交种的稻瘟病抗性. 作物研究, 2017, 31: 11-4+24. |
| [41] |
行璇, 刘雄伦, 陈海龙, 等. 分子标记辅助选择Pi9基因改良R288的稻瘟病抗性. 作物研究, 2016, 30: 487-91. |
| [42] |
廖花, 黄俊, 刘雄伦, 等. MAS育种改良籼稻恢复系R747及其杂交种稻瘟病抗性. 分子植物育种, 2019, 17: 3289-96. |
| [43] |
陈海龙, 李永聪, 刘雄伦, 等. MAS育种改良籼稻恢复系R599及其杂交种的稻瘟病抗性. 杂交水稻, 2017, 32: 61-5. |
| [44] |
李永聪, 黄俊, 廖花, 等. 分子标记辅助选择改良水稻恢复系R389及其杂交种稻瘟病抗性. 华北农学报, 2018, 33: 112-8. |
| [45] |
赖怡帆, 孙君玥, 张旭辉, 等. 分子标记辅助选择Pigm基因改良湘晚籼13号的稻瘟病抗性. 湖南农业大学学报(自然科学版), 2019, 45: 113-7. |
| [46] |
张礼霞, 王林友, 范宏环, 等. 利用Pigm基因改良粳稻保持系的稻瘟病抗性研究. 核农学报, 2017, 31: 424-31. |
| [47] |
桑世飞, 王君怡, 周静, 等. 稻瘟病抗性基因Pi-kh标记开发及在抗病育种中的应用. 分子植物育种, 2022, 20: 1588-96. |
| [48] |
Ellur RK, Khanna A, Yadav A, et al. Improvement of Basmati rice varieties for resistance to blast and bacterial blight diseases using marker assisted backcross breeding. Plant Sci, 2016, 242: 330-41. |
| [49] |
孙富, 唐梅, 何聪, 等. 分子标记辅助聚合基因Pi1和Pi2改良红色特种籼稻稻瘟病抗性研究. 湖北农业科学, 2018, 57: 23-7. |
| [50] |
田红刚, 陈红旗, 胡江, 等. 分子标记转育3个基因改良空育131稻瘟病抗性. 核农学报, 2016, 30: 2096-103. |
| [51] |
白玉路, 王平, 张琼, 等. 水稻骨干恢复系川恢907抗稻瘟病基因聚合与精准改良. 西南农业学报, 2019, 32: 947-51. |
| [52] |
Khanna A, Sharma V, Ellur RK, et al. Marker assisted pyramiding of major blast resistance genes Pi9 and Pita in the genetic background of an elite Basmati rice variety, Pusa Basmati 1. Indian J Genet Plant Breeding, 2015, 75: 417-25. |
| [53] |
张菊萍, 郝明, 曾盖, 等. MAS聚合Pi9和Pi49位点改良水稻两系不育系创5S稻瘟病抗性. 分子植物育种, 2018, 16: 7372-9. |
| [54] |
朱永生, 蔡秋华, 官华忠, 等. 多基因聚合改良杂交稻恢复系福恢676的稻瘟病抗性. 科学通报, 2023, 68: 2812-23. |
| [55] |
刘凯, 宛柏杰, 赵绍路, 等. 利用分子标记辅助选择聚合水稻Pi-ta, Pi-b和Pi-9基因. 西南农业学报, 2021, 34: 926-31. |
| [56] |
Liu Y, Zhang F, Luo X, et al. Molecular breeding of a novel PTGMS line of WDR for broad-spectrum resistance to blast using Pi9, Pi5, and Pi54 genes. Rice, 2021, 14: 96. |
| [57] |
Peng P, Jiang H, Luo L, et al. Pyramiding of multiple genes to improve rice blast resistance of photo-thermo sensitive male sterile line, without yield penalty in hybrid rice production. Plants, 2023, 12: 1389. |
| [58] |
Tanweer FA, Rafii MY, Sijam K, et al. Introgression of blast resistance genes (putative Pi-b and Pi-kh) into elite rice cultivar MR219 through marker-assisted selection. Front Plant Sci, 2015, 6: 1002. |
| [59] |
Khan GH, Shikari AB, Vaishnavi R, et al. Marker-assisted introgression of three dominant blast resistance genes into an aromatic rice cultivar Mushk Budji. Sci Rep, 2018, 8: 4091. |
| [60] |
Xiao WM, Luo LX, Wang H, et al. Pyramiding of Pi46 and Pita to improve blast resistance and to evaluate the resistance effect of the two R genes. J Integr Agr, 2016, 15: 2290-8. |
| [61] |
He Z, Xin Y, Wang C, et al. Genomics-assisted improvement of super high-yield hybrid rice variety "super 1000" for resistance to bacterial blight and blast diseases. Front Plant Sci, 2022, 13: 881244. |
| [62] |
Wu YY, Xiao N, Chen Y, et al. Comprehensive evaluation of resistance effects of pyramiding lines with different broad-spectrum resistance genes against Magnaporthe oryzae in rice (Oryza sativa L.). Rice, 2019, 12-11. |
| [63] |
吴云雨, 肖宁, 余玲, 等. 长江下游粳稻稻瘟病广谱抗性基因组合模式分析. 中国农业科学, 2021, 54: 1881-93. |
| [64] |
Hittalmani S, Parco A, Mew TV, et al. Fine mapping and DNA marker-assisted pyramiding of the three major genes for blast resistance in rice. Theor Appl Genet, 2000, 100: 1121-8. |
| [65] |
Chaipanya C, Telebanco-Yanoria MJ, Quime B, et al. Dissection of broad-spectrum resistance of the Thai rice variety Jao Hom Nin conferred by two resistance genes against rice blast. Rice, 2017, 10: 18. |
| [66] |
Chen X, Jia Y, Jia MH, et al. Functional interactions between major rice blast resistance genes, Pi-ta and Pi-b, and minor blast resistance quantitative trait loci. Phytopathology, 2018, 108: 1095-103. |
| [67] |
Divya B, Biswas A, Robin S, et al. Gene interactions and genetics of blast resistance and yield attributes in rice (Oryza sativa L.). J Genet, 2014, 93: 415-24. |
| [68] |
Chen Z, Bu QY, Liu GF, et al. Genomic decoding of breeding history to guide breeding-by-design in rice. Nat Sci Rev, 2023, 10: nwad029. |
| [69] |
Xiao N, Pan CH, Li YH, et al. Genomic insight into balancing high yield, good quality, and blast resistance of japonica rice. Genome Biol, 2021, 22: 283. |
| [70] |
Van Schie CC, Takken FL. Susceptibility genes 101: how to be a good host. Annu Rev Phytopathol, 2014, 52: 551-81. |
| [71] |
张会军. 水稻Pi21和OsBadh2基因编辑改良空育131的稻瘟病抗性及香味品质[D]. 武汉: 华中农业大学, 2016
|
| [72] |
Zhang Y, Lin XF, Li L, et al. CRISPR/Cas9-mediated knockout of Bsr-d1enhances the blast resistance of rice in Northeast China. Plant Cell Rep, 2024, 43: 100. |
| [73] |
Tao H, Shi X, He F, et al. Engineering broad-spectrum disease-resistant rice by editing multiple susceptibility genes. J Integr Plant Biol, 2021, 63: 1639-48. |
| [74] |
Xu Y, Bai L, Liu M, et al. Identification of two novel rice S genes through combination of association and transcription analyses with gene-editing technology. Plant Biotechnol J, 2023, 21: 1628-41. |
| [75] |
Ishihara T, Hayano-Saito Y, Oide S, et al. Quantitative trait locus analysis of resistance to panicle blast in the rice cultivar Miyazakimochi. Rice, 2014, 7: 2. |
| [76] |
Manojkumar HB, Deepak CA, Harinikumar KM, et al. Molecular profiling of blast resistance genes and evaluation of leaf and neck blast disease reaction in rice. J Genet, 2020, 99: 52. |
| [77] |
Kumar V, Jain P, Venkadesan S, et al. Understanding rice-Magnaporthe oryzae interaction in resistant and susceptible cultivars of rice under panicle blast infection using a time-course transcriptome analysis. Genes, 2021, 12: 301. |
| [78] |
Yan SJ, Liu Q, Naake T, et al. OsGF14b modulates defense signaling pathways in rice panicle blast response. Crop J, 2021, 9: 725-38. |
| [79] |
Inoue H, Hayashi N, Matsushita A, et al. Blast resistance of CC-NB-LRR protein Pb1 is mediated by WRKY45 through protein-protein interaction. Proc Natl Acad Sci U S A, 2013, 110: 9577-82. |
| [80] |
刘树芳, 董丽英, 李迅东, 等. 持有Pi9基因的水稻单基因系IRBL9-W对稻瘟病菌苗期和成株期抗性差异. 中国水稻科学, 2021, 35: 303-10. |
| [81] |
Dong J, Zhou L, Feng A, et al. The OsOXO2, OsOXO3 and OsOXO4 positively regulate panicle blast resistance in rice. Rice, 2021, 14: 51. |
| [82] |
Yu Y, Ma L, Wang X, et al. Genome-wide association study identifies a rice panicle blast resistance gene, Pb2, encoding NLR protein. Int J Mol Sci, 2022, 23: 5668. |
| [83] |
Liu Q, Yang J, Zhang S, et al. OsGF14b positively regulates panicle blast resistance, but negatively regulates leaf blast resistance in rice. Mol Plant Microbe Interact, 2016, 29: 46-55. |
| [84] |
Xu X, Lv Q, Shang J, et al. Excavation of Pid3 orthologs with differential resistance spectra to Magnaporthe oryzae in rice resource. PLoS One, 2014, 9: e93275. |
| [85] |
Wu YY, Xiao N, Yu L, et al. Combination patterns of major R genes determine the level of resistance to the M. oryzae in rice (Oryza sativa L.). PLoS One, 2015, 10: e0126130. |
| [86] |
Inoue H, Nakamura M, Mizubayashi T, et al. Panicle blast 1 (Pb1) resistance is dependent on at least four QTLs in the rice genome. Rice, 2017, 10: 36. |
| [87] |
Wang W, Mauleon R, Hu Z, et al. Genomic variation in 3, 010 diverse accessions of Asian cultivated rice. Nature, 2018, 557: 43-9. |
| [88] |
Li Z, Yang J, Ji X, et al. First telomere-to-telomere gapless assembly of the rice blast fungus Pyricularia oryzae. Sci Data, 2024, 11: 380. |
 2025, Vol. 37
2025, Vol. 37 

 宁约瑟,研究员,博士生导师。现任国家农业生物安全科学中心副主任、中国农业科学院植物保护研究所植保生物技术中心主任,作物病原生物功能基因组研究创新团队首席科学家。兼任中国植物病理学会副秘书长和中国农学会理事。担任SCI期刊Journal of Integrative Plant Biology等杂志编辑。国家自然科学基金优秀青年基金获得者,入选农业农村部“神农青年英才”和“杰出青年农业科学家”,获得“卫志明青年创新奖”和中国植物病理学会青年植物病理学家奖。主要从事水稻与稻瘟菌互作和水稻抗病性遗传学研究。系统解析稻瘟菌效应蛋白与水稻靶标蛋白的互作机制,创制植物中首个E3泛素连接酶鉴定体系,揭示水稻核心E3泛素连接酶介导的抗稻瘟病新机制, 为创制稻瘟病综合防控策略奠定了重要基础。以第一/通讯作者(含共同)在Developmental Cell、Science Advances、Genome Biology、Cell Reports和Science Bulletin等杂志发表高水平SCI论文38篇。总被引频次4 400余次,H指数37。多次受邀在国际植物与微生物互作大会和国际稻瘟病大会等国际会议上做特邀报告
宁约瑟,研究员,博士生导师。现任国家农业生物安全科学中心副主任、中国农业科学院植物保护研究所植保生物技术中心主任,作物病原生物功能基因组研究创新团队首席科学家。兼任中国植物病理学会副秘书长和中国农学会理事。担任SCI期刊Journal of Integrative Plant Biology等杂志编辑。国家自然科学基金优秀青年基金获得者,入选农业农村部“神农青年英才”和“杰出青年农业科学家”,获得“卫志明青年创新奖”和中国植物病理学会青年植物病理学家奖。主要从事水稻与稻瘟菌互作和水稻抗病性遗传学研究。系统解析稻瘟菌效应蛋白与水稻靶标蛋白的互作机制,创制植物中首个E3泛素连接酶鉴定体系,揭示水稻核心E3泛素连接酶介导的抗稻瘟病新机制, 为创制稻瘟病综合防控策略奠定了重要基础。以第一/通讯作者(含共同)在Developmental Cell、Science Advances、Genome Biology、Cell Reports和Science Bulletin等杂志发表高水平SCI论文38篇。总被引频次4 400余次,H指数37。多次受邀在国际植物与微生物互作大会和国际稻瘟病大会等国际会议上做特邀报告