小麦(Triticum aestivum L.)是全球重要的主食作物,为人类提供大约18%的能量和19%的蛋白质(http://www.fao.org/faostat/en/#data/FBS)。全球有88%的小麦生产区域受到条锈病的威胁[1],导致每年数十亿美元的经济损失[2]。小麦条锈病在我国早有记载,北魏时期贾思勰在《齐民要术》辨谷章中提到“春多雨,麦脚着土而黄,名黄疸瘟”,即指小麦条锈病。除南极洲外,全球各大洲的60多个国家均有条锈病的报道。小麦条锈病在发病初期会沿小麦叶脉形成椭圆形鲜黄色的夏孢子堆,因此得名“条锈病”(stripe rust或者yellow rust),病原菌为条形柄锈菌(Puccinia striiformis f. sp. tritici, Pst)。条锈菌主要在气候凉爽湿润的小麦种植区发生,主要侵染禾本科植物,如普通小麦(Triticum aestivum)、硬粒小麦(T. turgidum var. durum)、栽培型粒小麦(T. dicoccum Schrank)、野生粒小麦(T. dicoccoides Körn)和小黑麦(Triticosecale、Pst)[3]。此外,条锈菌还可以感染某些栽培大麦(Hordeum vulgare)和黑麦(Secale cereale),但这两种作物通常不会引起严重的流行病,因此它们也被称为中间宿主[3, 4]。
小麦条锈菌在其生命周期中经历五种不同的孢子阶段:担孢子、夏孢子、性孢子、锈孢子和冬孢子,分别对应小麦条锈菌的五个时期,并涉及从无性繁殖到有性繁殖阶段的转换[5]。生命周期的起始阶段为性孢子期(pycnial stage),单核的性孢子萌发,并接触携带不同遗传物质的单核受体菌丝,细胞质首先融合,随后核融合形成双核菌丝体,从而实现了条锈菌基因重组,是条锈菌快速进化和新变异(毒力)小种不断出现的基础。在夏孢子期,夏孢子堆突破侵染小麦的叶片,夏孢子以风为媒介播至数千公里以外的地区,如加拿大西部地区条锈菌组成类似美国西部,而加拿大东部地区条锈菌组成类似美国中东部,因此超强的跨区域传播能力是造成条锈病大流行的主要原因[5]。
条锈病大流行常造成巨大危害,可导致感病品种减产10%~70%,甚至绝收[6]。新中国成立以来,我国先后发生过五次全国性、极其严重的小麦条锈病大流行,分别发生在1950、1964、1990、2002和2017年,小麦总发病面积近5.5亿公顷,总产量损失高达1 380万吨。最近一次条锈病大流行发生在2019年,发病面积高达200万公顷,由于杀菌剂的使用,没有造成显著的损失[7]。因此,防控条锈病对于稳定粮食安全至关重要,是稳定小麦产量的一个关键措施。
相对于化学防治,种植携带小麦抗病基因的品种是降低条锈病损失的更经济有效和环境友好的策略。在条锈病的影响下,易感品种的产量可以下降55%~64%,而抗性品种的产量仅下降11%[8]。自2000年遭到来自东非的PstS1条锈小种入侵以来,加拿大小麦生产饱受条锈病困扰;原条锈病高发区阿尔伯塔省从2019年起提高高抗和中抗小麦品种的种植面积,快速阻止了条锈病蔓延,减少了病害损失,维持了小麦产量[9]。上述结果表明,通过选择性地进行植物遗传育种改良提高小麦对条锈病的抗性,可以稳定产量,降低化控成本和污染,助力可持续增产,以满足人类未来对小麦需求的增长,因此抗条锈病基因的克隆和机制解析具有重要意义。
2 小麦抗条锈病基因的克隆与研究进展 2.1 小麦抗条锈病的研究回顾小麦抗条锈病基因以Yr (yellow rust resistance)命名,目前已有86个被命名,主要分为全生育期抗病性(all-stage resistance, ASR)和成株期抗病性(adult-plant resistance, APR)两大类[10]。ASR常表现出小种专化抗性,在条锈菌的侵染下,快速(24小时)产生免疫反应或超敏反应(HR);在遗传上一般表现为质量遗传性状,由单基因控制,常编码植物免疫受体NLR蛋白;由于NLR蛋白主要识别条锈菌的特征分子,因此ASR可被条锈菌快速进化而克服,即使聚合多个ASR也会因压力过大而加速条锈菌的进化和选择,如欧洲2015年发现的条锈菌新毒力小种Triticale2015 (PstS13)克服了Yr3、Yr17、Yr25、Yr27和Yr32等基因的抗病性[11]。与ASR不同的是,APR的遗传特性通常为数量遗传位点,由多基因控制;目前克隆的APR基因多编码蛋白激酶和转运蛋白,对不同条锈菌小种具有广谱抗性,但抗病较慢而且只能呈现部分抗性,自2013年起受到广泛关注(图 1)。在对当前流行的生理性小种的抗性评估中,国内外153份小麦品种以APR抗性为主,因此育种实践表明APR抗病基因的育种价值更高[12]。然而APR在育种中也有局限性,比如仅在成株期发挥作用、具有高温依赖性和仅能产生部分抗性等。综合ASR和APR两类抗病基因优点,聚合多个APR辅以ASR,多抗病基因协同作用,是培育抗病品种的优先方案[13]。
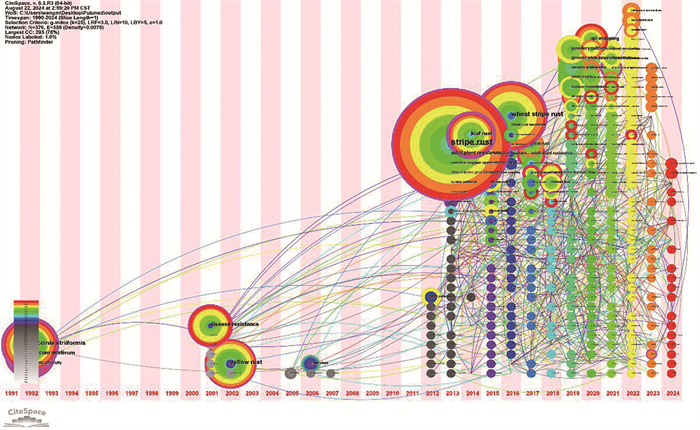
|
图 1 小麦条锈病研究的热点和趋势 |
抗条锈病研究和育种是不同阶段科学家的持续追求,从2013年开始抗条锈病研究迅速发展,小麦基因组测序完成以来呈现出多点突破、成果井喷的趋势(图 1)。目前已经命名了超过80个抗条锈病基因,还有300多个潜在的抗条锈病位点,来源包括小麦近缘物种和六倍体普通小麦等,其中大部分属于ASR抗性基因[14]。
目前有11个抗条锈病基因被克隆,大多数编码NLR蛋白(图 2)。Yr5a、Yr5b和Yr7是通过Mut-RenSeq方法克隆的基因,编码的蛋白具有NLR和锌指结构域(BED)融合的特殊形式,对世界范围内的菌种具有广泛抗性[15]。Yr9和Yr10分别来自于黑麦(Secale cereale L.)和普通小麦,其中Yr10编码的蛋白具有保守的CC-NBS-LRR结构域,属于ASR抗性,对中国现有的流行小种CYR33和CYR34都呈现抗病表型[16-18]。Yr27是利用超长测序技术组装特殊抗病材料基因组而克隆的一个抗病基因,编码一个典型的NLR免疫相关蛋白,不但抗条锈病而且抗叶锈病(与叶锈病抗性基因Lr13是等位基因),Yr27研究体现了基因组测序技术发展对作物抗病研究的推动作用[19]。YrAS2388是通过图位组图手段克隆的NLR基因,来源于普通小麦D基因组的供体粗山羊草(Aegilops tauschii L.)的抗条锈病基因,该基因含有重复的3ʹ未翻译区,是发挥抗病功能的关键;此外,同源YrAet672基因具有类似的重复3ʹ未翻译区,两者都可以归属为Yr28基因的不同单倍型[20, 21]。
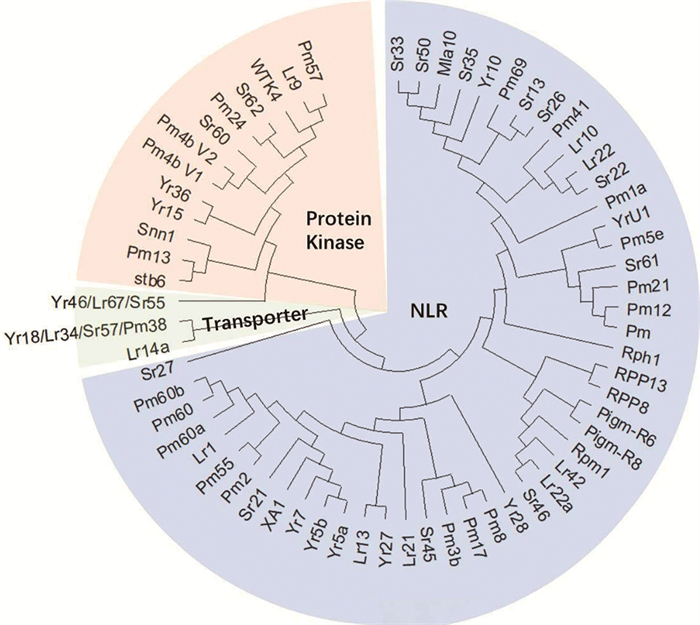
|
图 2 小麦和水稻抗病基因的系统发育 |
小麦抗条锈病基因还可以编码非NLR类型蛋白,如蛋白激酶和转运蛋白,部分非NLR蛋白的作用机制和调控网络得到了深入的研究。Yr36是来源于野生二粒小麦(Triticum turgidum L. subsp. Dicoccoides)的成株抗病基因,编码蛋白包括N端的丝氨酸/苏氨酸激酶结构域和C端的START结构域,因此又被命名为WHEAT KINASE START 1 (WKS1)。WKS1可以磷酸化一系列靶蛋白促进活性氧(ROS)积累:加速PsbO降解,使裸露的光合系统释放单氧离子;抑制类玉米黄质环氧化酶,促进超氧离子积累并转化成过氧化氢(H2O2);抑制类囊体抗坏血酸过氧化物酶(tAPX),减少H2O2分解;H2O2积累加速细胞死亡,崩溃的叶绿体脂类不完全降解产生茉莉酸(jasmonates, JA)形成正反馈,引发局部坏死,产生抗病表型[22-26]。Yr15编码一个具有串联的蛋白激酶结构域(wheat tandem kinase, WTK)的抗病蛋白,该基因来源于野生二粒小麦(wild emmer wheat, Triticum turgidum L. var. dicoccoides),是一个广谱抗条锈病基因,Yr15及一系列编码WTK的基因(如Lr9)的克隆证明了此类蛋白广泛参与小麦抗病[27, 28]。Yr18是首个被克隆的具有一因多效的基因,对白粉病、叶锈病等均有一定的抗性(Yr18/Lr34/Sr57/Pm38),编码一个ABC转运蛋白,曾被报道具有脱落酸(abscisic acid, ABA)运输能力,近期发现也可以运输芥子醇,促进木质素积累,增加细胞壁强度[29-31]。Yr46编码一个突变的己糖类转运蛋白,能与正常己糖类转运蛋白形成异源多聚体,抑制糖类转运功能,不利于病菌能量获取,对叶锈病和秆锈病也具有显著抗性(Yr46/Lr67/Sr55)。
2.3 小麦抗条锈病基因的育种潜力值得指出的是,Yr18、Yr36和Yr46属于典型的APR基因,从20世纪50年代以来已经在墨西哥的小麦品种中表现出持续的抗性,广谱抗病,抗性持久,育种价值高;但是,具有慢锈和只能产生部分抗性的缺点[32]。随着条锈菌进化和新毒力小种的不断出现,部分被广泛种植并携带抗病NLR类单一ASR基因的小麦品种抗病效果逐渐降低,甚至丧失抗性,很可能加速条锈病的蔓延和大规模的爆发[33]。由于Yr18、Yr36和Yr46的作用机制不同,具有功能叠加的可能,因此聚合多个APR或者与ASR抗性基因组合将产生持久、完全的抗性,如Yr18+Yr28+Yr36或Yr18+Yr36组合在苗期就显示出足够的条锈病抗性,且抗病效果较单一抗性基因有显著提高[34]。上述抗病基因的克隆、抗病机制解析和育种组合探索为未来小麦抗病基础研究和抗病育种提供了关键信息和不断优化的基础。
3 未来小麦耐条锈病与高产协同改良的瓶颈和策略随着小麦遗传和抗病研究的不断突破,数百个抗条锈病遗传位点被定位并且一系列重要的抗病基因得以克隆,极大地推动了小麦抗病基础研究。但是,从基因克隆到抗病育种应用还需要克服一些瓶颈。
3.1 抗病与产量的平衡制约与克服植物抗病过程涉及细胞内大量生命过程的变化,如ROS爆发等,这些变化往往负调控正常生长过程,导致生长-抗病的平衡制约(growth-defense trade-off)现象[35]。RPM1是一个已知的NLR抗病膜蛋白,可特异性地识别丁香假单胞菌(Pseudomonas syringae L.)的效应因子Avrpm1和AvrB,激活下游信号触发超敏反应,并导致宿主植物局部坏死抗病;但是,在无病原菌的条件下,携带RPM1基因的植物较对照产量降低9%[36]。水稻(Oryza sativa L.) PigmR基因编码的NLR蛋白对稻瘟病产生广谱抗性,但其持续表达会降低水稻的产量;同源蛋白PigmS可与PigmR形成二聚体抑制抗病功能,病菌入侵时叶片中PigmS高度甲基化,释放对PigmR的抑制作用,因此PigmR/PigmS组合可以达到抗病高产协同改良的效果[37]。小麦MLO (mildew resistance locus O)是一个感病基因,敲除MLO提高小麦抗白粉病能力,但产量显著降低;如果敲除范围扩大到304 kb,破坏一个负调控产量的糖转运蛋白(tonoplast monosaccharide transporter 3, TaTMT3B),可实现抗病不减产的目标[38, 39]。因此,深入研究抗病基因的作用机制,制定相应策略,通过表观遗传、精准激活以及多基因协同作用等手段,有望克服抗病基因对产量的负影响。
3.2 气候变化带来的挑战和应对策略全球气候变化对条锈菌的高温耐受性和小麦抗病产生了显著的影响:越冬期温度的提升有助于条锈菌安全越冬,提高来年菌源的病菌基数;极端气候导致的倒春寒可能造成低温环境,适合条锈菌侵入宿主。对法国不同地区的条锈菌的调查发现,南部条锈菌种对温暖的地中海气候更加适应,在野外相对于未经过选择的北部菌种具有明显的竞争优势,表明不同气候可以加速条锈菌的进化和筛选,使小麦抗条锈病面临新的挑战[40]。降雨不均衡对小麦抗条锈病和高产带来不确定因素,充分水供应(降雨或者生育期内灌溉)可以显著提高小麦产量,但是增加条锈病爆发的风险;相反,每次灌溉用水量降低30%,不但可以提高蛋白质含量,而且降低条锈病发病程度,产量降低仅为6%。上述对灌溉条件的研究从耕作制度方面走出了产量、抗性和品质平衡的关键一步[41]。在小麦基础研究方面,深入研究低温下限制高温APR基因发挥功能的机制,如表观遗传、基因转录、可变剪切、蛋白质翻译、折叠成熟、亚细胞定位、翻译后修饰和协同作用因子等不同层次的调控,探索打破低温限制小麦APR基因和光合效率不高的策略,有望为适应未来气候变化的高产抗病育种提供理论参考和重要的遗传材料。
3.3 抗病基因来源的拓宽和有效利用创造和利用种质资源是小麦抗病育种的基础,第一步是识别和探索优越的种质,包括地方种、祖先种和野生近源种等。地方品种是一种能够耐受当地生物和非生物胁迫,并在低投入农业系统中保持中等产量水平的传统作物品种,虽然现代育成品种经历了广泛的基因组区域的正向选择,但地方品种仍然包含可以在未来育种中利用的优异等位基因 [42-44]。小麦二倍体祖先种Ae. Tauschii (DD)的种群含有许多与叶部病害和害虫相关的基因,通过整合祖先种创制的“川麦42”品种主导四川盆地小麦种植超过十年,因此对祖先性状变异的遗传和生理基础的深入研究有助于准确指导未来的育种策略[45]。小麦野生近源种如Aegilopsis、Elymus、Lolium、Dasypyrum、Thinopyrum和Triticum等在开发高适应性的小麦系中具有显著潜力[46]。李振声院士团队将小麦与长穗偃麦草进行远缘杂交,将草的抗病基因转移给小麦,开创了小麦与偃麦草远缘杂交育种新领域,并育成高产、抗病、优质的“小偃”系列品种。
3.4 感病基因的发现和耐病种质创新小麦和条锈菌的互作发生在多个层次,小麦NLR等抗病蛋白识别条锈菌特征分子起始抗病反应,病菌也可以通过效应子调控小麦抗病反应。一方面,病菌通过进化可以逃逸NLR等抗病蛋白的识别;另一方面,小麦被条锈菌“劫持”的蛋白如果突变,不显著影响产量,但会显著影响抗病性。康振生院士和王晓杰教授的研究团队发现条锈菌分泌效应子PsSpg1,可与小麦蛋白激酶TaPsIPK1结合,增强激酶活性并进入细胞核,磷酸化调控TaCBF1d转录活性抑制抗性反应;通过基因编辑敲除感病基因TaPsIPK1,可以提高小麦抗病能力但不影响产量[47]。条锈菌诱导表达的基因涉及但不限于:负调控ROS爆发和细胞壁强化的蛋白,以及促进糖类、氨基酸和脂类合成并向条锈菌吸器转运的代谢酶或者相关元件;以上均有望作为自然变异、人工变异和基因编辑的优先考虑靶点,通过靶向引入变异,解除条锈菌对小麦(“殖民地”)的控制,提高条锈病耐受性,在无病原菌时不显著影响小麦产量和品质。我们预测,感病基因的缺失不能通过病菌突变而被克服,因此有可能比NLR类蛋白的抗病性更加持久,因此育种潜力值得期待。
3.5 新兴育种技术的引入和种质资源创新新技术的引入可以有效地推动小麦抗条锈病基础研究和育种实践(图 3)。小麦-条锈菌互作系统目前主要使用叶片组织进行分析,单细胞测序技术的引入有望从细胞水平解析上述互作系统,活体成像系统如显示茉莉酸信号的GFP成像系统揭示了在细胞水平小麦对条锈菌的响应和相关代谢变化[48]。CRISPR/Cas9通过创造遗传变异,彻底改变了小麦改良和功能基因分析。许多重要性状受到单核苷酸多态性(SNPs)和“弱”或“数量”等位基因的影响,而碱基编辑器、质粒编辑器和调节基因的精细调控这样的工具允许在短时间内精确操纵小麦基因组,累积与多抗性、高质量和产量相关的有利等位基因,有助于设计未来小麦[49-53]。泛基因组分析不但可以为小麦抗病基因的克隆提供参照,而且与病理学基础研究和大数据分析有机整合在一起,可以为未来小麦智能抗病育种提供“导航”。基因组编辑的最新进展为单倍体诱导相关基因的定向修饰提供了全新机会,如PLA1和CENH3等,完善单倍体选择方法(如结合花青素或荧光基因)和发展高通量表型平台对于提高单倍体技术在小麦育种中的效率至关重要,通过双单倍体(DH)育种,有望显著加速小麦抗病基因挖掘和育种过程[54-56]。
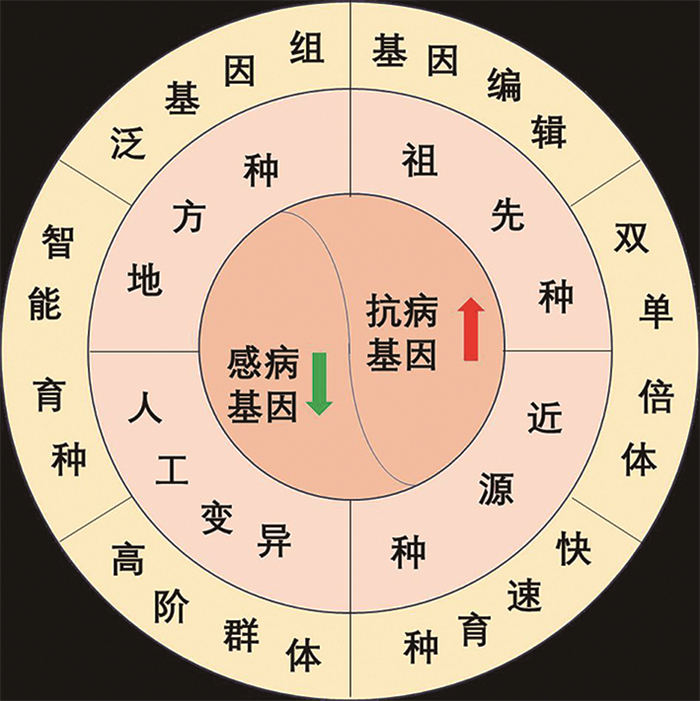
|
图 3 未来小麦抗条锈病研究和育种的策略 |
为满足人类社会日益增长的小麦需求,提高小麦抗病、产量和气候适应能力至关重要。尽管小麦抗条锈病育种面临诸多挑战,但小麦遗传学的快速发展和新技术的不断涌现将有力推动小麦抗条锈病研究。扩大小麦抗病基因库(包括地方种、祖先种和近源品种)是发现优良育种基因的首选,人工变异的创制和利用是加速自然进化过程的新手段,上述材料和来源于其他物种的信息丰富了小麦抗条锈病的武器库。加强泛组学水平的基因组和高阶杂交群体的创制和遗传研究将有助于挖掘基因组编辑和分子育种的基因和位点,为小麦抗病育种提供参考“导航图”。因此,传统育种方法与基因组编辑工具、合成生物学、双单倍体育种和人工智能等先进技术有效地整合,将为小麦抗条锈病研究和新品种创制奠定基础。
| [1] |
Beddow JM, Pardey PG, Chai Y, et al. Research investment implications of shifts in the global geography of wheat stripe rust. Nat Plants, 2015, 1: 15132. DOI:10.1038/nplants.2015.132 |
| [2] |
Ali S, Gladieux P, Leconte M, et al. Origin, migration routes and worldwide population genetic structure of the wheat yellow rust pathogen Puccinia striiformis f. sp. tritici. PLoS Pathog, 2014, 10: e1003903. DOI:10.1371/journal.ppat.1003903 |
| [3] |
Chen W, Wellings C, Chen X, et al. Wheat stripe (yellow) rust caused by Puccinia striiformis f. sp. tritici. Mol Plant Pathol, 2014, 15: 433-46. DOI:10.1111/mpp.12116 |
| [4] |
Dawson AM, Bettgenhaeuser J, Gardiner M, et al. The development of quick, robust, quantitative phenotypic assays for describing the host-nonhost landscape to stripe rust. Front Plant Sci, 2015, 6: 876. |
| [5] |
Schwessinger B. Fundamental wheat stripe rust research in the 21st century. New Phytol, 2017, 213: 1625-31. DOI:10.1111/nph.14159 |
| [6] |
Chen X. Pathogens which threaten food security: Puccinia striiformis, the wheat stripe rust pathogen. Food Secur, 2020, 12: 239-51. DOI:10.1007/s12571-020-01016-z |
| [7] |
Zhao J, Kang Z. Fighting wheat rusts in China: a look back and into the future. Phytopathol Res, 2023, 5: 6. DOI:10.1186/s42483-023-00159-z |
| [8] |
Zhou X, Fang T, Li K, et al. Yield losses associated with different levels of stripe rust resistance of commercial wheat cultivars in China. Phytopathology, 2022, 112: 1244-54. DOI:10.1094/PHYTO-07-21-0286-R |
| [9] |
Lidwell-Durnin J, Lapthorn A. The threat to global food security from wheat rust: Ethical and historical issues in fighting crop diseases and preserving genetic diversity. Glob Food Secur, 2020, 26: 100446. DOI:10.1016/j.gfs.2020.100446 |
| [10] |
Wellings C. Global status of stripe rust: a review of historical and current threats. Euphytica, 2011, 179: 129-41. DOI:10.1007/s10681-011-0360-y |
| [11] |
Riella V, Rodriguez-Algaba J, García R, et al. New races with wider virulence indicate rapid evolution of Puccinia striiformis f. sp. tritici in the Southern cone of America. Plant Dis, 2024, 108: 2454-61. DOI:10.1094/PDIS-02-24-0320-RE |
| [12] |
周警卫, 叶博伟, 张朋飞, 等. 国内外153份小麦种质条锈病抗性鉴定与评价. 中国农业科学, 2024, 57: 18-33. DOI:10.3864/j.issn.0578-1752.2024.01.003 |
| [13] |
Chen X, Coram T, Huang X, et al. Understanding molecular mechanisms of durable and non-durable resistance to stripe rust in wheat using a transcriptomics approach. Curr Genomics, 2013, 14: 111-26. DOI:10.2174/1389202911314020004 |
| [14] |
Tong J, Zhao C, Liu D, et al. Genome-wide atlas of rust resistance loci in wheat. Theor Appl Genet, 2024, 137: 179. DOI:10.1007/s00122-024-04689-8 |
| [15] |
Marchal C, Zhang J, Zhang P, et al. BED-domain-containing immune receptors confer diverse resistance spectra to yellow rust. Nat Plants, 2018, 4: 662-8. DOI:10.1038/s41477-018-0236-4 |
| [16] |
Yuan C, Wu J, Yan B, et al. Remapping of the stripe rust resistance gene Yr10 in common wheat. Theor Appl Genet, 2018, 131: 1253-62. DOI:10.1007/s00122-018-3075-9 |
| [17] |
Dibley K, Jost M, McIntosh R, et al. The wheat stripe rust resistance gene YrNAM is Yr10. Nat Commun, 2024, 15: 3291. DOI:10.1038/s41467-024-47513-z |
| [18] |
Ni F, Zheng Y, Liu X, et al. Sequencing trait-associated mutations to clone wheat rust-resistance gene YrNAM. Nat Commun, 2023, 14: 4353. DOI:10.1038/s41467-023-39993-2 |
| [19] |
Athiyannan N, Abrouk M, Boshoff WHP, et al. Long-read genome sequencing of bread wheat facilitates disease resistance gene cloning. Nat Genet, 2022, 54: 227-31. DOI:10.1038/s41588-022-01022-1 |
| [20] |
Zhang C, Huang L, Zhang H, et al. An ancestral NB-LRR with duplicated 3'UTRs confers stripe rust resistance in wheat and barley. Nat Commun, 2019, 10: 4023. DOI:10.1038/s41467-019-11872-9 |
| [21] |
Athiyannan N, Zhang P, McIntosh R, et al. Haplotype variants of the stripe rust resistance gene Yr28 in Aegilops tauschii. Theor Appl Genet, 2022, 135: 4327-36. DOI:10.1007/s00122-022-04221-w |
| [22] |
Chang CY, Yang SX, Zhang MQ, et al. Suppression of ZEAXANTHIN EPOXIDASE 1 restricts stripe rust growth in wheat. Plant Commun, 2023, 4: 100608. DOI:10.1016/j.xplc.2023.100608 |
| [23] |
Gou JY, Li K, Wu K, et al. Wheat stripe rust resistance protein WKS1 reduces the ability of the thylakoid-associated ascorbate peroxidase to detoxify reactive oxygen species. Plant Cell, 2015, 27: 1755-70. DOI:10.1105/tpc.114.134296 |
| [24] |
Wang S, Li QP, Wang J, et al. YR36/WKS1-mediated phosphorylation of PsbO, an extrinsic member of photosystem Ⅱ, inhibits photosynthesis and confers stripe rust resistance in wheat. Mol Plant, 2019, 12: 1639-50. DOI:10.1016/j.molp.2019.10.005 |
| [25] |
Yan Y, Li XM, Chen Y, et al. Phosphorylation of KAT-2B by WKS1/Yr36 redirects the lipid flux to jasmonates to enhance resistance against wheat stripe rust. J Genet Genomics, 2023, 50: 872-82. DOI:10.1016/j.jgg.2023.08.009 |
| [26] |
Fu D, Uauy C, Distelfeld A, et al. A kinase-START gene confers temperature-dependent resistance to wheat stripe rust. Science, 2009, 323: 1357-60. DOI:10.1126/science.1166289 |
| [27] |
Klymiuk V, Yaniv E, Huang L, et al. Cloning of the wheat Yr15 resistance gene sheds light on the plant tandem kinase-pseudokinase family. Nat Commun, 2018, 9: 3735. DOI:10.1038/s41467-018-06138-9 |
| [28] |
Wang Y, Abrouk M, Gourdoupis S, et al. An unusual tandem kinase fusion protein confers leaf rust resistance in wheat. Nat Genet, 2023, 55: 914-20. DOI:10.1038/s41588-023-01401-2 |
| [29] |
Schnippenkoetter W, Lo C, Liu G, et al. The wheat Lr34 multipathogen resistance gene confers resistance to anthracnose and rust in sorghum. Plant Biotechnol J, 2017, 15: 1387-96. DOI:10.1111/pbi.12723 |
| [30] |
Zhang Y, Chen G, Zang Y, et al. Lr34/Yr18/Sr57/Pm38 confers broad-spectrum resistance to fungal diseases via sinapyl alcohol transport for cell wall lignification in wheat. Plant Commun, 2024, 5: 101077. DOI:10.1016/j.xplc.2024.101077 |
| [31] |
Krattinger SG, Kang J, Bräunlich S, et al. Abscisic acid is a substrate of the ABC transporter encoded by the durable wheat disease resistance gene Lr34. New Phytol, 2019, 223: 853-66. DOI:10.1111/nph.15815 |
| [32] |
Huerta-Espino J, Singh R, Crespo-Herrera LA, et al. Adult plant slow rusting genes confer high levels of resistance to rusts in bread wheat cultivars from Mexico. Front Plant Sci, 2020, 11: 824. DOI:10.3389/fpls.2020.00824 |
| [33] |
康振生, 王晓杰, 赵杰, 等. 小麦条锈菌致病性及其变异研究进展. 中国农业科学, 2015, 48: 3439-53. DOI:10.3864/j.issn.0578-1752.2015.17.011 |
| [34] |
Wang F, Zhang M, Hu Y, et al. Pyramiding of adult-plant resistance genes enhances all-stage resistance to wheat stripe rust. Plant Dis, 2023, 107: 879-85. DOI:10.1094/PDIS-07-22-1716-RE |
| [35] |
Derbyshire MC, Newman TE, Thomas WJW, et al. The complex relationship between disease resistance and yield in crops. Plant Biotechnol J, 2024, 22: 2612-23. DOI:10.1111/pbi.14373 |
| [36] |
Tian D, Traw MB, Chen JQ, et al. Fitness costs of R-gene-mediated resistance in Arabidopsis thaliana. Nature, 2003, 423: 74-7. DOI:10.1038/nature01588 |
| [37] |
Deng Y, Zhai K, Xie Z, et al. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance. Science, 2017, 355: 962-5. DOI:10.1126/science.aai8898 |
| [38] |
Li S, Lin D, Zhang Y, et al. Genome-edited powdery mildew resistance in wheat without growth penalties. Nature, 2022, 602: 455-60. DOI:10.1038/s41586-022-04395-9 |
| [39] |
Wang Y, Cheng X, Shan Q, et al. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nat Biotechnol, 2014, 32: 947-51. DOI:10.1038/nbt.2969 |
| [40] |
Mboup M, Bahri B, Leconte M, et al. Genetic structure and local adaptation of European wheat yellow rust populations: the role of temperature-specific adaptation. Evol Appl, 2012, 5: 341-52. DOI:10.1111/j.1752-4571.2011.00228.x |
| [41] |
Alghawry A, Yazar A, Unlu M, et al. Irrigation rationalization boosts wheat (Triticum aestivum L.) yield and reduces rust incidence under arid conditions. Biomed Res Int, 2021, 2021: 5535399. DOI:10.1155/2021/5535399 |
| [42] |
Sukumaran S, Dreisigacker S, Lopes M, et al. Genome-wide association study for grain yield and related traits in an elite spring wheat population grown in temperate irrigated environments. Theor Appl Genet, 2015, 128: 353-63. DOI:10.1007/s00122-014-2435-3 |
| [43] |
Zeven AC. Landraces: a review of definitions and classifications. Euphytica, 1998, 104: 127-39. DOI:10.1023/A:1018683119237 |
| [44] |
Niu J, Ma S, Zheng S, et al. Whole-genome sequencing of diverse wheat accessions uncovers genetic changes during modern breeding in China and the United States. Plant Cell, 2023, 35: 4199-216. DOI:10.1093/plcell/koad229 |
| [45] |
Gaurav K, Arora S, Silva P, et al. Population genomic analysis of Aegilops tauschii identifies targets for bread wheat improvement. Nat Biotechnol, 2022, 40: 422-31. DOI:10.1038/s41587-021-01058-4 |
| [46] |
Ceoloni C, Forte P, Kuzmanović L, et al. Cytogenetic mapping of a major locus for resistance to Fusarium head blight and crown rot of wheat on Thinopyrum elongatum 7EL and its pyramiding with valuable genes from a Th. ponticum homoeologous arm onto bread wheat 7DL. Theor Appl Genet, 2017, 130: 2005-24. DOI:10.1007/s00122-017-2939-8 |
| [47] |
Wang N, Tang C, Fan X, et al. Inactivation of a wheat protein kinase gene confers broad-spectrum resistance to rust fungi. Cell, 2022, 185: 2961-74. DOI:10.1016/j.cell.2022.06.027 |
| [48] |
Li XM, Wang CY, Guo YT, et al. Hormone-response transcriptional landscapes of wheat seedlings and the development of a JA fluorescent reporter. Plant Cell Environ, 2022, 45: 3604-10. DOI:10.1111/pce.14443 |
| [49] |
Uauy C, Wulff BBH, Dubcovsky J. Combining traditional mutagenesis with new high-throughput sequencing and genome editing to reveal hidden variation in polyploid wheat. Annu Rev Genet, 2017, 51: 435-54. DOI:10.1146/annurev-genet-120116-024533 |
| [50] |
Li S, Zhang C, Li J, et al. Present and future prospects for wheat improvement through genome editing and advanced technologies. Plant Commun, 2021, 2: 100211. DOI:10.1016/j.xplc.2021.100211 |
| [51] |
Zhou X, Zhao Y, Ni P, et al. CRISPR-mediated acceleration of wheat improvement: advances and perspectives. J Genet Genom, 2023, 50: 815-34. DOI:10.1016/j.jgg.2023.09.007 |
| [52] |
Gao C. Genome engineering for crop improvement and future agriculture. Cell, 2021, 184: 1621-35. DOI:10.1016/j.cell.2021.01.005 |
| [53] |
Awan MJA, Pervaiz K, Rasheed A, et al. Genome edited wheat-current advances for the second green revolution. Biotechnol Adv, 2022, 60: 108006. DOI:10.1016/j.biotechadv.2022.108006 |
| [54] |
Qi X, Guo S, Zhong Y, et al. Establishment of an efficient haploid identification system by engineering anthocyanin accumulation in the wheat embryo. Plant Commun, 2023, 4: 100568. DOI:10.1016/j.xplc.2023.100568 |
| [55] |
Qu Y, Fernie AR, Liu J, et al. Doubled haploid technology and synthetic apomixis: recent advances and applications in future crop breeding. Mol Plant, 2024, 17: 1005-18. DOI:10.1016/j.molp.2024.06.005 |
| [56] |
Eliby S, Bekkuzhina S, Kishchenko O, et al. Developments and prospects for doubled haploid wheat. Biotechnol Adv, 2022, 60: 108007. DOI:10.1016/j.biotechadv.2022.108007 |
 2025, Vol. 37
2025, Vol. 37 

 缑金营,中国农业大学教授、博士研究生导师,国家万人计划科技领军人才和海外高层次人才引进计划入选者。2001年获得复旦大学学士学位,2007年获得中国科学院上海生命科学研究院上海植物生理生态研究所博士学位,2007—2012年在美国布鲁克海文国家实验室从事博士后研究,2012—2014年在美国加利福尼亚大学戴维斯分校任客座助理项目科学家,2013—2022年在复旦大学工作,2022年起在中国农业大学工作。主要研究小麦分子遗传和代谢,聚焦小麦高光合效率和抗病的基因克隆与分子机制解析。近五年来,主持“十四五”重点研发计划和国家自然科学基金委国际合作重点项目等科研项目,在Nature Plants、Nature Communications和Molecular Plant等期刊发表多篇研究论文
缑金营,中国农业大学教授、博士研究生导师,国家万人计划科技领军人才和海外高层次人才引进计划入选者。2001年获得复旦大学学士学位,2007年获得中国科学院上海生命科学研究院上海植物生理生态研究所博士学位,2007—2012年在美国布鲁克海文国家实验室从事博士后研究,2012—2014年在美国加利福尼亚大学戴维斯分校任客座助理项目科学家,2013—2022年在复旦大学工作,2022年起在中国农业大学工作。主要研究小麦分子遗传和代谢,聚焦小麦高光合效率和抗病的基因克隆与分子机制解析。近五年来,主持“十四五”重点研发计划和国家自然科学基金委国际合作重点项目等科研项目,在Nature Plants、Nature Communications和Molecular Plant等期刊发表多篇研究论文