(2 云南省中科灵长类生物医学重点实验室, 昆明 650000)
(2 Yunnan Key Laboratory of Primate Biomedical Research, Kunming 650000, China)
人胚胎学要解决的基本科学问题有两个:(1)我们每个人如何从胚胎发育而来,这属于生命发生的问题;(2)为什么有些胚胎不能发育为健康的个体,这属于生命健康的问题。只有揭示了第一个问题,才可能解决第二个问题。得益于辅助生殖技术,当前领域内对人着床前胚胎发育已经非常了解,但对着床后早期胚胎发育的认识,至今尚处于起步阶段。临床上,大约40%~60%的胚胎会因为着床失败、早期妊娠丢失(early pregnancy loss)和流产导致发育失败[1-3]。此外,一些先天性疾病的病因源自着床后早期胚胎发育异常(例如神经管和心血管发育异常)[4]。据统计,我国胎儿出生缺陷率为5.6%左右[5]。随着生育年龄普遍推迟和高龄产妇比例上升,出生缺陷率可能进一步增加。但妊娠过程中不育(胚胎着床失败、早期妊娠丢失和流产)和胎儿出生缺陷背后的胚胎异常发育机制,至今仍然知之甚少。提高对人着床后早期胚胎发育基本原理的认识和揭示胚胎异常发育的分子机制,将为诊疗不育和胎儿出生缺陷提供科学依据,从而改善辅助生殖技术,提高人口生育率,降低胎儿出生缺陷率。因此,深入研究人着床后早期胚胎发育,对人口生殖健康具有重要战略意义。
2 人着床后早期胚胎关键发育事件 2.1 着床随着第二次细胞谱系分离,即上胚层(epiblast, EPI)和下胚层(hypoblast, HB)分离,晚期囊胚通过靠近EPI的极性滋养层与子宫内膜接触并启动着床。在此过程中,子宫内膜与极性滋养层之间的相互作用至关重要。子宫内膜必须转变为蜕膜才能接纳胚胎着床[6]。蜕膜细胞分泌的WNT抑制剂DKK1[7, 8]可能作为关键母体信号,诱导极性滋养层分化为具有浸润能力的合胞体滋养层细胞[9];同时,抑制WNT信号活性还能促进绒毛外滋养层细胞特化[10, 11]。合胞体滋养层穿过子宫内膜上皮并与部分基质细胞发生融合[12],基质细胞能够促进滋养层细胞存活、增殖和合体化[13]。随后,子宫内膜来源的Notch信号诱导绒毛外滋养层细胞特化并迁移[14]。最新研究表明,子宫内膜自然杀伤细胞分泌的细胞因子也促进绒毛外滋养层细胞特化[15]。值得注意的是,EPI对极性滋养层的成熟和功能也具有关键作用,其缺失会导致胚胎定位、黏附和侵入失败[16]。综上,EPI与滋养层互作以及滋养层与子宫内膜互作对于胚胎着床至关重要。当前,对胚胎着床动态,即定位、黏附和侵入的发育过程及其背后的细胞和分子机制还所知甚少。胚胎着床和滋养层发育(包括从起始的滋养外胚层到细胞滋养层、从细胞滋养层到合胞体滋养层和绒毛外滋养层)过程中,精确的胚胎和胚外谱系互作以及母胎互作原理还有待进一步探索。
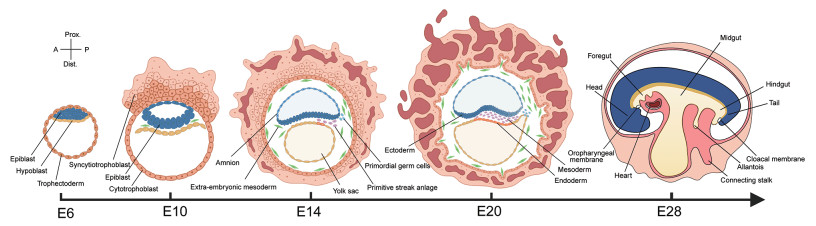
2.2 羊膜囊形成、原肠运动和器官发生随着着床,人胚胎按照精确的时空顺序并遵循既定的组织形态发生和细胞谱系特化,在母体子宫中开启一场精密有序的发育旅程(图 1)。其中,羊膜囊形成、原肠运动和器官发生都与EPI发育相关,是着床后胚胎发育的里程碑事件(图 1)[17]。
|
图 1 人着床后早期胚胎发育模式图(用BioRender绘制) |
胚胎着床后,EPI逐渐发生细胞极化并形成腔状结构,第一次实现人胚胎发育的背腹轴对称打破[18]。靠近极性滋养外胚层一端(背侧)的EPI下调多能性标志物(如SOX2、NANOG和OCT4)并上调羊膜标志物(如TFAP2A、TFAP2C和GATA3)的表达,形态上从柱状上皮转换为鳞状上皮,即形成羊膜;而靠近下胚层一端(腹侧)的EPI继续维持柱状上皮形态和多能性标志物表达[19-21]。鳞状羊膜上皮和柱状多能性EPI及它们围成的羊膜腔,共同构成双极羊膜囊结构(图 1)[18]。
双极羊膜囊形成后是原肠运动,起始于胚胎发育的卡内基分期(Carnegie stage, CS) 6~7阶段,大概受精后第14~15天。人胚胎原肠运动起始于部分EPI细胞的上皮间质转换及伴随而来的前后对称打破——前端为EPI,随着发育将下调NANOG并上调SOX2的表达,将来特化为外胚层(神经和表皮);后端为原条,下调SOX2上调T (也称Brachyury)的表达,将来发育为中内胚层(图 1)[22]。已故著名发育学家路易斯·沃伯特曾说过:“人一生中最重要的时刻不是出生、结婚和死亡,而是原肠运动”。[23]原肠运动作为人胚胎发育过程中最关键的生物学事件之一,为胚胎体轴形成、三胚层特化和器官发生奠定基础[24-27]。已有研究表明,BMP信号能够诱导羊膜发生,而羊膜又可作为信号中心,通过分泌BMP和WNT信号进一步诱导原肠运动[24-28]。前端EPI因受到前端脏壁内胚层的信号调节而拮抗羊膜细胞诱导的原条命运[29, 30]。在原肠运动过程中,细胞命运决定、分选、迁移重排和自我组织等重要发育事件背后的精确谱系互作和信号调控机制,仍需进一步探索。
当胚胎进入CS9阶段(大概受精后第21天),三胚层(外胚层、中胚层和内胚层)特化完成,胚胎发育从原肠运动进入器官发生阶段[31]。随后,椭圆形胚胎逐渐拉长,向内折叠形成细长的管状结构。目前,人胚胎器官发生的研究仍处于初级阶段,神经管闭合、体节发生、肠管形成及心血管系统发育等事件的细胞和分子机制,尚待进一步阐明。
2.3 胚外中胚层和原始生殖细胞特化人胚外中胚层(extraembryonic mesoderm, ExM)能发育形成血岛和脐带,是胎盘绒毛(基质细胞、内皮细胞、红细胞和Hofbauer细胞)、羊膜和卵黄囊的重要组成部分,并分泌丰富的细胞外基质。人ExM不仅在胎盘、羊膜、卵黄囊、脐带和造血系统的发育过程中发挥关键作用,还为胚外体腔的扩展提供结构支撑[32-35]。在啮齿类中,ExM起源自CS6的原肠细胞,但在包括人在内的灵长类中,ExM被特化自CS5的原肠前阶段(受精后11~12天)[33, 36, 37]。理论上,人ExM有三种可能的起源:细胞滋养层(cytotrophoblast, CTB)、HB和EPI[32, 33]。人多能干细胞(human pluripotent stem cells, hPSCs)的体外分化实验也表明,人ExM细胞能够通过CTB[38]、HB[39]和EPI[40, 41]的体外对应物特化而来。然而,由于缺乏组织学证据,且ExM与CTB之间存在基底膜屏障,CTB作为人ExM的发育起源被认为可能性小[33, 42]。综上,人ExM的发育起源目前尚无定论,但领域内更倾向于HB和(或) EPI起源[33, 43]。此外,关于人ExM的发育,以下关键科学问题仍有待深入探索:(1)羊膜外侧ExM在羊膜特化、结构形成及其功能维持中如何发挥作用;(2)胎盘绒毛中的ExM特化为基质细胞、内皮细胞、红细胞及Hofbauer细胞的发育动态及分子机制,以及这些衍生细胞与滋养层细胞如何协同调控胎盘的结构形成与生理功能;(3) ExM与胚外内胚层如何协同作用驱动卵黄囊形态发生,特别是血岛微环境的建立、第一波造血细胞的发育动态及其迁移路径;(4) ExM如何发育为体蒂和尿囊并最终形成胎儿脐带。
人原始生殖细胞(primordial germ cells, PGCs)是精子和卵子的前体细胞,其产生和特化对于人类生命的繁衍和进化起着决定性作用。小鼠和猪的PGCs都起源于原肠胚后端EPI[44-46]。不同的是,食蟹猴PGCs似乎起源于原肠前的新生羊膜[47]。当前,有关人PGCs的发育起源尚未确定,下面章节讨论人PGCs的发育起源相关研究进展。
3 人着床后早期胚胎发育研究进展人着床后早期胚胎位于母体子宫,尺寸较小(数百微米到几毫米),不易观察和研究。长期以来,关于人着床后早期胚胎的发育知识,主要来自体内胚胎的组织学样本[48-50]和动物胚胎研究[34, 51-53]。然而,仅通过体内胚胎的组织学观察,很难在时空上解析人着床后早期胚胎发育过程中的分子事件和信号调控机制。尽管啮齿类胚胎研究为理解人胚胎发育事件提供了线索,但随着研究的深入,啮齿类与人胚胎在形态和分子水平上的差异,使得通过研究啮齿类来探究人胚胎发育原理变得困难。即使和人同属于灵长类的猴胚胎,也与人胚胎发育存在一些重要差异。因此,人着床后早期胚胎发育的分子特征和信号调控机制,长期处于空白状态。近年来,随着单细胞转录组测序、空间转录组测序和胚胎延长培养技术的快速发展,人着床后早期胚胎发育的神秘面纱逐渐被揭开。
原肠运动和早期器官发生,是人着床后早期胚胎发育过程中最重要和最复杂的发育阶段。2021年,Tyser等[54]通过单细胞转录组测序,首次绘制CS7 (16~19天)人体内原肠胚的单细胞转录图谱,揭示这一发育阶段的EPI、PGCs、内胚层、非神经外胚层、中胚层、胚外中胚层和造血等谱系的转录特征。有关人PGCs的发育起源仍待解决,最近开发的羊膜囊胚体和着床后胚胎模型的新生羊膜和EPI位置,都有零星PGCs的分布[24, 29]。因此,基于类胚胎研究,依然不能确定人PGCs的发育起源。最新研究通过hPSCs体外分化产生PGCs和羊膜细胞,发现PGCs和新生羊膜可能共同起源于EPI样细胞衍生的TFAP2A阳性祖细胞,但这一发现是否适用于胚胎还需进一步探索[55, 56]。人流产胚胎的组织学数据显示,在受精后24天的人胚胎中,PGCs位于卵黄囊内胚层的后端区域[57]。通过分析人CS7 (16~19天)体内胚胎的单细胞转录组数据,在原条细胞亚群中发现了少量PGCs[54],暗示PGCs与原条细胞的转录特征比较接近。最新的三项研究通过体内胚胎的空间转录组和免疫荧光染色分析,发现PGCs在CS7和CS8阶段分布于胚盘后端的体蒂位置,并与EPI、羊膜和原肠细胞在转录水平上相似[30, 58];而到CS9阶段,PGCs出现在尿囊和主动脉-性腺-中肾(aorta-gonad-mesonephros, AGM)区,表明PGCs似乎从之前的体蒂位置经尿囊迁移至AGM区[31, 59]。在后续发育中,人PGCs可能遵循与小鼠相似的迁移轨迹:在扩张后肠的引导下向生殖嵴迁移并定植其中,最终与生殖嵴共同发育为性腺(睾丸或卵巢)[60]。综上,近期研究明确了人PGCs的转录特征及其在不同发育阶段的位置分布,推测其可能经历从胚盘后端羊膜和EPI的交汇处到体蒂、尿囊,再到AGM区的迁移过程。然而,人PGCs和新生羊膜是否共同起源于胚盘后端EPI衍生的TFAP2A阳性祖细胞,仍需进一步研究证实。
2023年,王晓群/吴倩/王拥军/Oscar Marín团队(CS10~20)[61]和施威扬/鲍志戎团队(CS12~ 16)[62]分别收集了体内更晚发育阶段的人胚胎,进一步绘制了人胚胎从原肠运动晚期到器官发生阶段的空间转录图谱。最近,于乐谦/郭靖涛/肖振宇/魏育蕾/王晓琰团队通过利用连续横断面高分辨空间转录组切片,结合深度学习算法进行三维对齐,形成了完整胚胎中不同细胞类型与基因表达的三维空间分布点云图,数字3D重构了首个体内完整的早期(CS7)[30]、中期(CS8)[58]和晚期(CS9)[31]人原肠胚模型;研究揭示了人原肠胚中不同细胞类型在胚胎中的空间位置分布[30, 58],还暗示了中胚层细胞可能在尚未迁出原条时就已确定细胞命运,并据此迁移到相应位置;同时,胚胎尾部的体蒂可能发挥信号组织者的角色,同中部的脊索一起构成原肠胚发育的双重信号源[58]。最新研究通过CS9胚胎的空间转录组测序数据发现:神经中胚层祖细胞(neuromesodermal progenitors, NMPs)可能不仅主导脊髓形成,还可能深度参与后脑后部区域的形成;两群NMPs并非随机混合而是存在清晰的背腹分层模式;作为造血干细胞和生殖细胞发源地,胚胎尾部的主动脉-性腺-中肾区域在CS9阶段开始形成[31]。
上述研究通过揭示关键发育事件、细胞谱系组成、转录特征以及空间组织结构等,极大促进了领域内对人胚胎从原肠运动到器官发生阶段的理解,为后续干细胞、胚胎模型和类器官的开发和鉴定提供了宝贵的参考标准。
尽管体内胚胎数据是体外模型的金标准,但由于伦理和技术限制,受精后28天前的体内胚胎材料很难获取[63]。围着床期体内胚胎的发育过程和单细胞转录组数据至今仍是空白。因此,特殊情况下获取的宝贵和稀缺的体内胚胎材料,只适合用于提供参考标准,不适合用于解析着床后早期胚胎发育的时空动态、信号调控和分子机制。2016年,人胚胎体外延长培养技术的诞生打破了体内胚胎研究面临的局限,使科学家能够实时显微观察围着床期胚胎的发育动态,解析EPI极化,羊膜腔、卵黄囊、双层胚盘、前后轴和基底膜形成,滋养层发育,多能状态转换,胚外中胚层和原条特化等关键发育事件,并绘制该阶段胚胎发育进程的转录图谱和DNA甲基化图谱[19-21, 64-66]。在此基础上,进一步鉴定了不同细胞谱系的发育动态和分子特征,揭示了FGF、WNT、BMP和Nodal信号在不同细胞谱系特化中的功能,并解析了异倍体胚胎异常发育的分子机制[26, 29, 67, 68]。
4 人胚胎模型研究进展用胚胎作为研究材料,是揭示人早期发育奥秘的基础,但面临诸如伦理关切、资源稀缺和技术挑战等限制,因此无法用其进行深入和系统的发育机制和功能研究。此外,供体年龄、职业、遗传背景和健康状况等因素还会造成胚胎的异质性。胚胎模型是利用一种或多种干细胞在体外构建的,能够在分子特征、细胞谱系组成及组织结构等方面模拟人胚胎发育过程中关键事件的细胞培养物。干细胞通常包含全能干细胞、多能干细胞、滋养层干细胞和胚外内胚层干细胞。干细胞来源的胚胎模型,可以大规模制备,均质性和可控性好,且容易进行基因编辑和谱系示踪等操作。如果能实现胚胎模型在形态结构、分子特征和发育潜能上与人胚胎高度相似,胚胎模型将与体内自然发育和体外延长培养的人胚胎形成互补,并完美地破解当前人早期胚胎发育研究过程中所面临的瓶颈问题。因此,对于人着床后早期胚胎发育而言,胚胎模型具有广阔的应用前景。
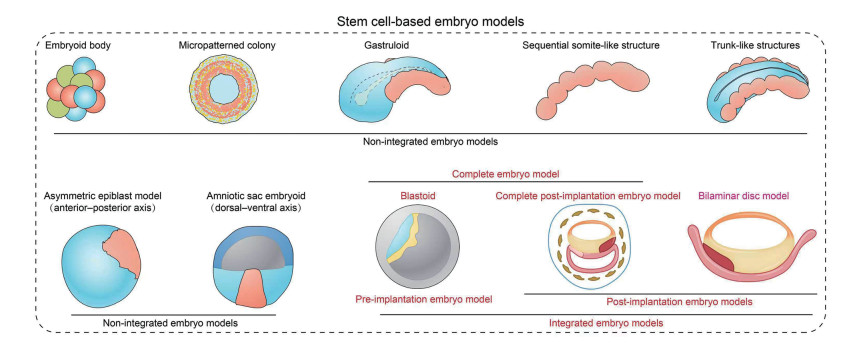
近年来,人胚胎模型研究取得了快速发展,为领域内深入研究胚胎发育从着床到早期器官发生阶段的“黑匣子”提供了可能。根据其细胞谱系组成,人胚胎模型分为两种类型:不含胚外组织的非整合胚胎模型和含胚外组织的整合胚胎模型[69-70](图 2)。非整合胚胎模型包括拟胚体[71]、图案集落(也称2D原肠体)[72]、原肠体[73, 74]、连续体节样结构[75, 76]、躯干样结构[77, 78]、不对称EPI模型(前后对称打破)[22, 79]和羊膜囊胚体(背腹对称打破)[24, 80, 81](图 2)。上述非整合胚胎模型模拟人着床后胚胎EPI的部分发育事件和组织结构,不含滋养外胚层和胚外内胚层等胚外组织。最近四年左右时间,整合胚胎模型研究取得了快速发展。与非整合胚胎模型不同,整合胚胎模型包含全部或部分胚外组织(图 2)。根据对应胚胎的发育阶段以及所含胚外组织的类型,整合胚胎模型又可细分为三种类型:类囊胚[13, 16, 82-93]、完全着床后胚胎模型[94]和双胚盘模型[29, 95-101](图 2)。类囊胚对应着床前囊胚,完全着床后胚胎模型对应围着床胚胎,两者均包含所有胚胎和胚外细胞谱系,属于完全胚胎模型,具有模拟完整胚胎发育的潜能。而双胚盘模型则包含除滋养外胚层外的所有胚胎和胚外组织,能够模拟除胎盘组织外的胚胎和胚外组织发育。当前,领域内已用胚胎模型模拟了人着床后早期胚胎发育的大部分关键事件,包括胚胎着床和母胎互作[12, 13, 16, 102];EPI极化[20, 103];多能状态转换[104];前后和背腹模式、羊膜腔、卵黄囊、双层胚盘和三层胚盘形成,以及胚外中胚层、原条、原始生殖细胞、造血系统甚至三胚层特化等[22, 24, 29, 79-81, 91, 94-101]。和常规类器官技术相比,胚胎模型中的谱系发育时序与自然胚胎更为接近,因此,胚胎模型除了模拟发育事件外,未来可能还是制备人造器官的理想平台[105]。此外,人胚胎模型制备容易实现规模化,且均质性和可控性好,未来在药物研发和毒性测试方面应用前景广阔。
5 伦理关切体外培养的人胚胎和胚胎模型对研究人着床后早期胚胎发育具有重要的科学和临床意义。然而,人胚胎发育研究具有伦理敏感性,不同于一般科学研究。1973年,澳大利亚莫纳什大学的Carl Wood团队首次实现了世界上第一例试管婴儿的受孕[106]。1978年,英国的Steptoe和Edwards让世界上第一例试管婴儿(Louise Brown)健康出生,首次实现体外受精(in vitro fertilization, IVF)胚胎的成功分娩[107]。IVF技术的出现和发展,一直伴随生物伦理讨论。公众的关切主要集中在IVF胚胎是否和人一样拥有生命权的道德地位,主要有三种代表性观点:(1)胚胎从受精开始就具有了道德地位;(2)胚胎在受精后和出生前的一段时间内应获得一定的道德地位;(3)出生前的胚胎或胎儿都不具有道德地位。
胚胎从何时起应该拥有“人”的道德地位?关于这个问题,至今没有统一的答案。胚胎获得生命权的时间节点,取决于所在国家或地区的历史文化和宗教信仰。以德国和奥地利为代表的一些国家认为受精卵就具有生命权;瑞士认为受精后7天的胚胎具有生命权;以英国为代表的一些国家认为受精后14天的胚胎具有生命权;在以色列,这个时间节点则是胚胎受精后第40天。当前,以英国为代表的“14天限制”最具影响力,成为主要的国际共识。1979年,美国的健康、教育和福利机构的道德咨询委员会出台一份详细报告[108],谨慎提出支持人胚胎研究。同时,该报告建议胚胎在体外培养不能超过14天或者原条出现阶段。1982年,英国组建了人受精和胚胎咨询委员会(沃诺克委员会),该委员会主要围绕IVF胚胎的发育而展开讨论。胚胎体外研究的“14天限制”提议作为委员会建议的一部分获得通过。该委员会最终将讨论的结果整理为一份报告在1984年发表,即著名的“沃诺克报告”[109]。1990年,英国将胚胎体外研究的“14天限制”列入英国受精与胚胎法案并一直沿用至今。“14天限制”的提出主要来源于“同卵双生”的经验主张。这种观点认为:当胚胎发育至受精后第14天,进入原肠运动阶段,便不再具备“同卵双生”的潜能,此时的胚胎就成为了一个独特个体及潜在的“人”,理应获得生命权。但是,有两个观点直接反驳“14天限制”的理论依据。其中一种观点认为,在原条形成后一周,即胚胎受精后21天仍然存在产生连体双胎的可能[110];而另一种观点认为,同卵双生发生在胚胎受精后的第一次卵裂[111]。不难看出,“14天限制”的提出,是基于一种经验主张。
随着人胚胎体外延长培养技术的进步,“14天限制”的合理性受到越来越多的质疑。2021年5月,代表国际共识的ISSCR综合了各国科学家、伦理学家以及法学家的建议后,发布了新版《干细胞研究与临床转化指南》,该指南认为:最近几年,人着床后胚胎体外培养技术取得了长足进步,利用体外延长培养的人胚胎深入研究人着床后早期胚胎发育,对于增强对不孕症、体外受精、着床后早期胚胎的妊娠失败和发育障碍的理解与干预至关重要。因此,ISSCR解除了人胚胎体外研究的“14天限制”,并鼓励有关实验室团队向所在国家或地区的监管机构申请对14天后的人胚胎发育进行体外研究[112]。关于人胚胎体外培养和研究,若取消“14天限制”,应重新科学评估并确定新的合理时间限制。
随着技术不断完善,人胚胎模型的仿真性也在逐渐提高。其中,类囊胚与真实囊胚在形态特征、细胞组成和转录水平上相似。此外,完全着床后胚胎模型在细胞组成和形态特征方面也能模拟自然胚胎的围着床期发育。最近,科学界和伦理界对人胚胎模型面临的伦理问题进行了广泛讨论[113-119]。一个基本共识是,目前人的胚胎模型与自然胚胎还相差较远,但在未来,随着技术进步,胚胎模型在细胞组成、分子特征和组织结构上与自然胚胎的相似度必然逐步提高。因此可以设想,在未来的某个时间节点,胚胎模型的生理功能将接近或等价于自然胚胎。所以,领域内必须尽快制定“人的胚胎模型等价于自然胚胎”这个临界点的评价标准,即当胚胎模型具备哪些特征后可以将其视为自然胚胎,从而以自然胚胎的伦理标准对胚胎模型进行规范研究。
制定人胚胎和胚胎模型研究的伦理规制,须纳入以下两方面考虑。(1)有益性。相关研究的开展,须有益于对人胚胎发育原理的理解或有益于人类生殖健康。(2)明确的临界点。胚胎或完全胚胎模型发育到什么阶段才等价于生命?不同国家和地区、不同历史文化和宗教信仰都可能使答案多样化。我们应该以发育生物学提供的科学理论为基础,积极召集全球不同领域对胚胎研究感兴趣的代表人士对胚胎研究的伦理规制进行广泛探讨并总结归纳,争取找到大众能够接受的折中方案。在胚胎发育过程中,心跳和痛觉是体现生命特征的两个里程碑事件,都可以作为将胚胎或完全胚胎模型界定为“生命”的临界点。原始心管通常在CS12阶段(大概受精后25~28天)形成并开始跳动。痛觉的产生源于神经系统的发育和完善,并依赖功能性神经网络的建立。一般认为,人胚胎在受精后22天左右开始出现最早的神经系统,但这时的神经系统由一团神经上皮干细胞组成,尚未形成功能性神经连接[63, 120]。有报道认为,直到人胚胎发育第42天,才开始出现第一个神经元细胞[121]。也就是说,受精后42天前的人胚胎尚未形成功能性神经细胞,不可能产生痛觉。有研究推测,胎儿可能最早在受精后12周才能感知痛觉[122]。因此,心跳先于痛觉出现,将心跳出现的CS12阶段(大概受精后25~28天)作为胚胎或完全胚胎模型具有同等道德地位的临界点较为合适。
6 伦理规制建议人胚胎体外延长培养技术以及基于干细胞的胚胎模型发展迅速,然而相应的法律规范文件较少,现行文件多为“指导原则”“技术规范”,且制定时间相对久远,其中的内容不能完全指导现在的研究。现在被许多科学家、伦理学家和政策制定者广泛接受的“14天规则”,虽然有助于维持公众对胚胎研究的信任,同时使科学家能够调查人类发育的最初阶段,但该规则制定受限于当时的科技水平,过去虽完全满足研究需要,但是目前技术发展迅猛,已经影响有利于解析人类生命起源和人类健康的科学研究。此外,辅助生殖技术的瓶颈、早孕失败的原因和许多人类疾病的发育起源等生命健康问题尚未阐明,而这些问题的回答都亟需科学技术的发展,以及制定新的伦理规制来指导科学研究。
综上,我们对人胚胎和胚胎模型的研究和应用提出如下伦理规制建议。
(1) 相关研究必须严格遵循国际干细胞协会2021年颁布的《干细胞研究与临床转化指南》、原科技部和卫生部2003年颁布的《人胚胎干细胞研究伦理指导原则》、国家卫生健康委员会2016年颁布的《涉及人的生物医学研究伦理审查办法》以及科技部2025年颁布的《人源类器官研究伦理指引》。相关研究须经医学伦理委员会批准后才可开展,并在医学伦理委员会的严格监督下实施。
(2) 使用人胚胎应遵循“最少数量”原则:在必须使用人胚胎进行研究的情况下,尽量减少人胚胎的使用数量。通过优化实验设计和提高实验效率,确保在达到科学目标的同时,使用最少数量的人胚胎。建立全国胚胎数据平台,统一编号,实现去向可追溯;引入“数据信托”模式,防止商业滥用。
(3) 重新审慎地校准人胚胎体外研究的时间限制:建议我国有关监管机构结合当前人胚胎研究的前沿发展,积极反思“14天限制”的性质并重新评估其利弊,根据科学研究环境和公众态度重新合理地校准人胚胎体外研究的时间限制。比如,英国国家生育监管机构——人类受精与胚胎学管理局(HFEA)——建议政府将人类胚胎在体外培养的时限从14天延长至28天。HFEA的这一声明已经在全球引起广泛影响(详情参见:go.nature.com/ 42j3pzp)。我们要快速响应国际环境变化,积极制定相应措施,以免导致在胚胎方面的政策或方针落后于其他国家。考虑到延长“14天规则”的高敏感性,我们建议采取发育阶段与时间节点结合限制的替代性方法。人胚胎体外延长培养的最长时间不能超过受精后28天或卡内基12 (CS12)阶段或心跳出现。选择CS12阶段作为截止时间,既能满足对原肠运动、器官发生的理解,并且人胚胎的神经环路还没有形成,不会产生痛觉。考虑到人胚胎的发育进程存在个体差异,只要符合上述三个条件中的任何一条,都必须立即终止研究。比如,特定胚胎如果开始出现心跳,即使没有发育到受精后28天或CS12阶段,也须立即终止研究。此外,在开展研究之前,需要配套延长“14天规则”的伦理监管措施。
(4) 制定符合我们国家科学需求和伦理规范的胚胎模型的伦理规则:遵守我国《人源类器官研究伦理指引》中相关条文提到的规定,即不得以生殖为目的,禁止将胚胎模型移植到动物或者人体子宫;根据研究目的,尽量使用胚胎模型代替人胚胎,非必要不使用人胚胎;尽量使用非完全胚胎模型代替完全胚胎模型,非必要不使用完全胚胎模型;对于完全胚胎模型,需要广泛考虑科学需求和伦理关切,广泛调查公众对人胚胎模型研究的态度,进而寻找符合我们国家科学需求和伦理规范的胚胎模型的研究时间节点,如可以考虑完全胚胎模型在体外培养的时间到人胚胎发育的CS12阶段或者心跳出现。考虑到不同胚胎模型的制备时间和人胚胎发育时间并不完全等同,需要以发育阶段或心跳而非发育时间作为终止研究的约束条件。
(5) 建议监管和研究机构、伦理学家以及科学和教育工作者加强公众教育,让公众充分了解和认识“人胚胎研究”面临的伦理关切及所能获得的潜在益处,并就“人胚胎研究”的时限问题和公众进行充分的沟通和讨论。比如,借助短视频、公众号等手段积极宣传,与大众积极地交流和讨论,做到研究透明,有助于让公众放心。此外,还应该努力促进决策者、科学家、公众、患者、胚胎捐献者和资助者之间就“人胚胎研究”的时限问题开展广泛的对话。
| [1] |
Jarvis GE. Early embryo mortality in natural human reproduction: what the data say. F1000Res, 2016, 5: 2765. DOI:10.12688/f1000research.8937.1 |
| [2] |
Norwitz ER, Schust DJ, Fisher SJ. Implantation and the survival of early pregnancy. N Engl J Med, 2001, 345: 1400-8. DOI:10.1056/NEJMra000763 |
| [3] |
Macklon NS, Geraedts JP, Fauser BC. Conception to ongoing pregnancy: the 'black box' of early pregnancy loss. Hum Reprod Update, 2002, 8: 333-43. DOI:10.1093/humupd/8.4.333 |
| [4] |
Bianco-Miotto T, Craig JM, Gasser YP, et al. Epigenetics and DOHaD: from basics to birth and beyond. J Dev Orig Health Dis, 2017, 8: 513-9. DOI:10.1017/S2040174417000733 |
| [5] |
吕红平. 出生缺陷防治: 提高人口素质的重要基础工程. 人口与计划生育, 2018(09): 24-7. |
| [6] |
Gellersen B, Brosens JJ. Cyclic decidualization of the human endometrium in reproductive health and failure. Endocr Rev, 2014, 35: 851-905. DOI:10.1210/er.2014-1045 |
| [7] |
Garcia-Alonso L, Handfield LF, Roberts K, et al. Mapping the temporal and spatial dynamics of the human endometrium in vivo and in vitro. Nat Genet, 2021, 53: 1698-711. DOI:10.1038/s41588-021-00972-2 |
| [8] |
Wang W, Vilella F, Alama P, et al. Single-cell transcriptomic atlas of the human endometrium during the menstrual cycle. Nat Med, 2020, 26: 1644-53. DOI:10.1038/s41591-020-1040-z |
| [9] |
Ruane PT, Garner T, Parsons L, et al. Trophectoderm differentiation to invasive syncytiotrophoblast is promoted by endometrial epithelial cells during human embryo implantation. Hum Reprod, 2022, 37: 777-92. DOI:10.1093/humrep/deac008 |
| [10] |
Shukla V, Moreno-Irusta A, Varberg KM, et al. NOTUM-mediated WNT silencing drives extravillous trophoblast cell lineage development. Proc Natl Acad Sci U S A, 2024, 121: e2403003121. DOI:10.1073/pnas.2403003121 |
| [11] |
Gu B, Ferreira LMR, Herrera S, et al. The TEA domain transcription factors TEAD1 and TEAD3 and WNT signaling determine HLA-G expression in human extravillous trophoblasts. Proc Natl Acad Sci U S A, 2025, 122: e2425339122. DOI:10.1073/pnas.2425339122 |
| [12] |
Shibata S, Endo S, Nagai LAE, et al. Modeling embryo-endometrial interface recapitulating human embryo implantation. Sci Adv, 2024, 10: eadi4819. |
| [13] |
Yu L, Logsdon D, Pinzon-Arteaga CA, et al. Large-scale production of human blastoids amenable to modeling blastocyst development and maternal-fetal cross talk. Cell Stem Cell, 2023, 30: 1246-61. e9. DOI:10.1016/j.stem.2023.08.002 |
| [14] |
Haider S, Meinhardt G, Saleh L, et al. Notch1 controls development of the extravillous trophoblast lineage in the human placenta. Proc Natl Acad Sci U S A, 2016, 113: E7710-9. |
| [15] |
Li Q, Sharkey A, Sheridan M, et al. Human uterine natural killer cells regulate differentiation of extravillous trophoblast early in pregnancy. Cell Stem Cell, 2024, 31: 181-95. e9. DOI:10.1016/j.stem.2023.12.013 |
| [16] |
Kagawa H, Javali A, Khoei HH, et al. Human blastoids model blastocyst development and implantation. Nature, 2022, 601: 600-5. |
| [17] |
艾宗勇, 张成庭, 牛宝华, 等. 人胚胎早期发育与干细胞. 合成生物学, 2024, 5: 1-19. |
| [18] |
Fu J, Warmflash A, Lutolf MP. Stem-cell-based embryo models for fundamental research and translation. Nat Mater, 2021, 20: 132-44. DOI:10.1038/s41563-020-00829-9 |
| [19] |
Deglincerti A, Croft GF, Pietila LN, et al. Self-organization of the in vitro attached human embryo. Nature, 2016, 533: 251-4. DOI:10.1038/nature17948 |
| [20] |
Shahbazi MN, Jedrusik A, Vuoristo S, et al. Self-organization of the human embryo in the absence of maternal tissues. Nat Cell Biol, 2016, 18: 700-8. DOI:10.1038/ncb3347 |
| [21] |
Xiang L, Yin Y, Zheng Y, et al. A developmental landscape of 3D-cultured human pre-gastrulation embryos. Nature, 2020, 577: 537-42. |
| [22] |
Simunovic M, Metzger JJ, Etoc F, et al. A 3D model of a human epiblast reveals BMP4-driven symmetry breaking. Nat Cell Biol, 2019, 21: 900-10. DOI:10.1038/s41556-019-0349-7 |
| [23] |
Wolpert L, Vicente C. An interview with Lewis Wolpert. Development, 2015, 142: 2547-8. DOI:10.1242/dev.127373 |
| [24] |
Zheng Y, Xue X, Shao Y, et al. Controlled modelling of human epiblast and amnion development using stem cells. Nature, 2019, 573: 421-425. DOI:10.1038/s41586-019-1535-2 |
| [25] |
Yang R, Goedel A, Kang Y, et al. Amnion signals are essential for mesoderm formation in primates. Nat Commun, 2021, 12: 5126. DOI:10.1038/s41467-021-25186-2 |
| [26] |
Mole MA, Coorens THH, Shahbazi MN, et al. A single cell characterisation of human embryogenesis identifies pluripotency transitions and putative anterior hypoblast centre. Nat Commun, 2021, 12: 3679. DOI:10.1038/s41467-021-23758-w |
| [27] |
Martyn I, Kanno TY, Ruzo A, et al. Self-organization of a human organizer by combined Wnt and Nodal signalling. Nature, 2018, 558: 132-5. DOI:10.1038/s41586-018-0150-y |
| [28] |
Zheng Y, Yan RZ, Sun S, et al. Single-cell analysis of embryoids reveals lineage diversification roadmaps of early human development. Cell Stem Cell, 2022, 29: 1402-19. e8. DOI:10.1016/j.stem.2022.08.009 |
| [29] |
Ai Z, Niu B, Yin Y, et al. Dissecting peri-implantation development using cultured human embryos and embryo-like assembloids. Cell Res, 2023, 33: 661-78. DOI:10.1038/s41422-023-00846-8 |
| [30] |
Cui L, Lin S, Yang X, et al. Spatial transcriptomic characterization of a Carnegie stage 7 human embryo. Nat Cell Biol, 2025, 27: 360-9. DOI:10.1038/s41556-024-01597-3 |
| [31] |
Yuan Y, Wang X, Yan X, et al. 3D reconstruction of a human Carnegie stage 9 embryo provides a snapshot of early body plan formation. Cell Stem Cell, 2025, 32: 1006-24. e5. DOI:10.1016/j.stem.2025.04.007 |
| [32] |
Boroviak T, Nichols J. Primate embryogenesis predicts the hallmarks of human naive pluripotency. Development, 2017, 144: 175-86. DOI:10.1242/dev.145177 |
| [33] |
Ross C, Boroviak TE. Origin and function of the yolk sac in primate embryogenesis. Nat Commun, 2020, 11: 3760. DOI:10.1038/s41467-020-17575-w |
| [34] |
Nakamura T, Okamoto I, Sasaki K, et al. A developmental coordinate of pluripotency among mice, monkeys and humans. Nature, 2016, 537: 57-62. DOI:10.1038/nature19096 |
| [35] |
Luckett WP. Origin and differentiation of the yolk sac and extraembryonic mesoderm in presomite human and rhesus monkey embryos. Am J Anat, 1978, 152: 59-97. DOI:10.1002/aja.1001520106 |
| [36] |
Bianchi DW, Wilkins-Haug LE, Enders AC, et al. Origin of extraembryonic mesoderm in experimental animals: relevance to chorionic mosaicism in humans. Am J Med Genet, 1993, 46: 542-50. DOI:10.1002/ajmg.1320460517 |
| [37] |
Cui G, Feng S, Yan Y, et al. Spatial molecular anatomy of germ layers in the gastrulating cynomolgus monkey embryo. Cell Rep, 2022, 40: 111285. DOI:10.1016/j.celrep.2022.111285 |
| [38] |
Liu Z, Tan Y, Flynn WF, et al. HAND1, partially mediated through ape-specific LTR binding, is essential for human extra-embryonic mesenchyme derivation from iPSCs. Cell Rep, 2025, 44: 115568. DOI:10.1016/j.celrep.2025.115568 |
| [39] |
Farkas K, Ferretti E. Derivation of human extraembryonic mesoderm-like cells from primitive endoderm. Int J Mol Sci, 2023, 24: 11366. DOI:10.3390/ijms241411366 |
| [40] |
Pham TXA, Panda A, Kagawa H, et al. Modeling human extraembryonic mesoderm cells using naive pluripotent stem cells. Cell Stem Cell, 2022, 29: 1346-65. e10. DOI:10.1016/j.stem.2022.08.001 |
| [41] |
Wang SL, Shi GH, Duan K, et al. Extraembryonic mesoderm cells derived from human embryonic stem cells rely on Wnt pathway activation. Cell Prolif, 2024, 58: e13761. |
| [42] |
Hertig AT, Rock J. Two human ova of the pre-villous stage, having a developmental age of about 8 and 9 days respectively. Contrib Embryol, 1949, 33: 169-86. |
| [43] |
Ai Z, Yin Y, Niu B, et al. Deconstructing human peri-implantation embryogenesis based on embryos and embryoids. Biol Reprod, 2022, 107: 212-25. DOI:10.1093/biolre/ioac096 |
| [44] |
Saitou M, Barton SC, Surani MA. A molecular programme for the specification of germ cell fate in mice. Nature, 2002, 418: 293-300. DOI:10.1038/nature00927 |
| [45] |
Ohinata Y, Payer B, O'Carroll D, et al. Blimp1 is a critical determinant of the germ cell lineage in mice. Nature, 2005, 436: 207-13. DOI:10.1038/nature03813 |
| [46] |
Kobayashi T, Zhang H, Tang WWC, et al. Principles of early human development and germ cell program from conserved model systems. Nature, 2017, 546: 416-20. DOI:10.1038/nature22812 |
| [47] |
Sasaki K, Nakamura T, Okamoto I, et al. The germ cell fate of cynomolgus monkeys is specified in the nascent amnion. Dev Cell, 2016, 39: 169-85. DOI:10.1016/j.devcel.2016.09.007 |
| [48] |
Hertig AT, Rock J, Adams EC. A description of 34 human ova within the first 17 days of development. Am J Anat, 1956, 98: 435-93. DOI:10.1002/aja.1000980306 |
| [49] |
Hertig AT. On the development of the amnion and exoccelomic membrane in the previllous human ovum. Yale J Biol Med, 1945, 18: 107-15. |
| [50] |
Hertig AT. Angiogenesis in the early human chorion and in the primary placenta of the Macque monkey. Contrib Embryol, 1935, 25: 37-82. |
| [51] |
Artzt K. Mammalian developmental genetics in the twentieth century. Genetics, 2012, 192: 1151-63. DOI:10.1534/genetics.112.146191 |
| [52] |
Bergmann S, Penfold CA, Slatery E, et al. Spatial profiling of early primate gastrulation in utero. Nature, 2022, 609: 136-43. DOI:10.1038/s41586-022-04953-1 |
| [53] |
Zhai J, Guo J, Wan H, et al. Primate gastrulation and early organogenesis at single-cell resolution. Nature, 2022, 612: 732-8. DOI:10.1038/s41586-022-05526-y |
| [54] |
Tyser RCV, Mahammadov E, Nakanoh S, et al. Single-cell transcriptomic characterization of a gastrulating human embryo. Nature, 2021, 600: 285-9. DOI:10.1038/s41586-021-04158-y |
| [55] |
Chen D, Sun N, Hou L, et al. Human primordial germ cells are specified from lineage-primed progenitors. Cell Rep, 2019, 29: 4568-82. e5. DOI:10.1016/j.celrep.2019.11.083 |
| [56] |
Castillo-Venzor A, Penfold CA, Morgan MD, et al. Origin and segregation of the human germline. Life Sci Alliance, 2023, 6: e202201706. DOI:10.26508/lsa.202201706 |
| [57] |
Witschi E. Migration of the germ cells of human embryos from the yolk sac to the primitive gonadal fold. Contrib Embryol, 1948, 32: 67-80. |
| [58] |
Xiao Z, Cui L, Yuan Y, et al. 3D reconstruction of a gastrulating human embryo. Cell, 2024, 187: 2855-74. e19. DOI:10.1016/j.cell.2024.03.041 |
| [59] |
Gomes Fernandes M, Bialecka M, Salvatori DCF, et al. Characterization of migratory primordial germ cells in the aorta-gonad-mesonephros of a 4.5-week-old human embryo: a toolbox to evaluate in vitro early gametogenesis. Mol Hum Reprod, 2018, 24: 233-43. DOI:10.1093/molehr/gay011 |
| [60] |
Hara K, Kanai-Azuma M, Uemura M, et al. Evidence for crucial role of hindgut expansion in directing proper migration of primordial germ cells in mouse early embryogenesis. Dev Biol, 2009, 330: 427-39. DOI:10.1016/j.ydbio.2009.04.012 |
| [61] |
Zeng B, Liu Z, Lu Y, et al. The single-cell and spatial transcriptional landscape of human gastrulation and early brain development. Cell Stem Cell, 2023, 30: 851-66. e7. DOI:10.1016/j.stem.2023.04.016 |
| [62] |
Xu Y, Zhang T, Zhou Q, et al. A single-cell transcriptome atlas profiles early organogenesis in human embryos. Nat Cell Biol, 2023, 25: 604-15. DOI:10.1038/s41556-023-01108-w |
| [63] |
Hurlbut JB, Hyun I, Levine AD, et al. Revisiting the Warnock rule. Nat Biotechnol, 2017, 35: 1029-42. DOI:10.1038/nbt.4015 |
| [64] |
Zhou F, Wang R, Yuan P, et al. Reconstituting the transcriptome and DNA methylome landscapes of human implantation. Nature, 2019, 572: 660-4. DOI:10.1038/s41586-019-1500-0 |
| [65] |
Lv B, An Q, Zeng Q, et al. Single-cell RNA sequencing reveals regulatory mechanism for trophoblast cell-fate divergence in human peri-implantation conceptuses. PLoS Biol, 2019, 17: e3000187. DOI:10.1371/journal.pbio.3000187 |
| [66] |
West RC, Ming H, Logsdon DM, et al. Dynamics of trophoblast differentiation in peri-implantation-stage human embryos. Proc Natl Acad Sci U S A, 2019, 116: 22635-44. DOI:10.1073/pnas.1911362116 |
| [67] |
Shahbazi MN, Wang T, Tao X, et al. Developmental potential of aneuploid human embryos cultured beyond implantation. Nat Commun, 2020, 11: 3987. DOI:10.1038/s41467-020-17764-7 |
| [68] |
Chen X, Yu H, Yin Y, et al. Abnormal lineage differentiation of peri-implantation aneuploid embryos revealed by single-cell RNA sequencing. Clin Transl Med, 2025, 15: e70326. DOI:10.1002/ctm2.70326 |
| [69] |
Zernicka-Goetz M. The evolution of embryo models. Nat Methods, 2023, 20: 1844-8. DOI:10.1038/s41592-023-02077-6 |
| [70] |
Lovell-Badge R, Anthony E, Barker RA, et al. ISSCR guidelines for stem cell research and clinical translation: the 2021 update. Stem Cell Rep, 2021, 16: 1398-408. DOI:10.1016/j.stemcr.2021.05.012 |
| [71] |
Itskovitz-Eldor J, Schuldine, M, Karsenti D, et al. Differentiation of human embryonic stem cells into embryoid bodies compromising the three embryonic germ layers. Mol Med, 2000, 6: 88-95. DOI:10.1007/BF03401776 |
| [72] |
Warmflash A, Sorre B, Etoc F, et al. A method to recapitulate early embryonic spatial patterning in human embryonic stem cells. Nat Methods, 2014, 11: 847-54. DOI:10.1038/nmeth.3016 |
| [73] |
Moris N, Anlas K, van den Brink SC, et al. An in vitro model of early anteroposterior organization during human development. Nature, 2020, 582: 410-5. DOI:10.1038/s41586-020-2383-9 |
| [74] |
Hamazaki N, Yang W, Kubo CA, et al. Retinoic acid induces human gastruloids with posterior embryo-like structures. Nat Cell Biol, 2024, 26: 1790-803. DOI:10.1038/s41556-024-01487-8 |
| [75] |
Yamanaka Y, Hamidi S, Yoshioka-Kobayashi K, et al. Reconstituting human somitogenesis in vitro. Nature, 2023, 614: 509-20. DOI:10.1038/s41586-022-05649-2 |
| [76] |
Miao Y, Djeffal Y, De Simone A, et al. Reconstruction and deconstruction of human somitogenesis in vitro. Nature, 2022, 614: 500-8. |
| [77] |
Gribaudo S, Robert R, van Sambeek B, et al. Self-organizing models of human trunk organogenesis recapitulate spinal cord and spine co-morphogenesis. Nat Biotechnol, 2024, 42: 1243-53. DOI:10.1038/s41587-023-01956-9 |
| [78] |
Rito T, Libby ARG, Demuth M, et al. Timely TGFβ signalling inhibition induces notochord. Nature, 2025, 637: 673-82. DOI:10.1038/s41586-024-08332-w |
| [79] |
Simunovic M, Siggia ED, Brivanlou AH. In vitro attachment and symmetry breaking of a human embryo model assembled from primed embryonic stem cells. Cell Stem Cell, 2022, 29: 962-72. e4. DOI:10.1016/j.stem.2022.05.001 |
| [80] |
Shao Y, Taniguchi K, Gurdziel K, et al. Self-organized amniogenesis by human pluripotent stem cells in a biomimetic implantation-like niche. Nat Mater, 2017, 16: 419-25. DOI:10.1038/nmat4829 |
| [81] |
Shao Y, Taniguchi K, Townshend RF, et al. A pluripotent stem cell-based model for post-implantation human amniotic sac development. Nat Commun, 2017, 8: 208. DOI:10.1038/s41467-017-00236-w |
| [82] |
Yanagida A, Spindlow D, Nichols J, et al. Naive stem cell blastocyst model captures human embryo lineage segregation. Cell Stem Cell, 2021, 28: 1016-22. e4. DOI:10.1016/j.stem.2021.04.031 |
| [83] |
Yu L, Wei Y, Duan J, et al. Blastocyst-like structures generated from human pluripotent stem cells. Nature, 2021, 591: 620-6. DOI:10.1038/s41586-021-03356-y |
| [84] |
Sozen B, Jorgensen V, Weatherbee BAT, et al. Reconstructing aspects of human embryogenesis with pluripotent stem cells. Nat Commun, 2021, 12: 5550. DOI:10.1038/s41467-021-25853-4 |
| [85] |
Fan Y, Min Z, Alsolami S, et al. Generation of human blastocyst-like structures from pluripotent stem cells. Cell Discov, 2021, 7: 81. |
| [86] |
Liu X, Tan JP, Schroder J, et al. Modelling human blastocysts by reprogramming fibroblasts into iBlastoids. Nature, 2021, 591: 627-32. |
| [87] |
Tu Z, Bi Y, Zhu X, et al. Modeling human pregastrulation development by 3D culture of blastoids generated from primed-to-naive transitioning intermediates. Protein Cell, 2023, 14: 337-49. |
| [88] |
Yu X, Liang S, Chen M, et al. Recapitulating early human development with 8C-like cells. Cell Rep, 2022, 39: 110994. |
| [89] |
Mazid MA, Ward C, Luo Z, et al. Rolling back human pluripotent stem cells to an eight-cell embryo-like stage. Nature, 2022, 605: 315-24. |
| [90] |
Guo M, Wu J, Chen C, et al. Self-renewing human naïve pluripotent stem cells dedifferentiate in 3D culture and form blastoids spontaneously. Nat Commun, 2024, 15: 668. |
| [91] |
Karvas RM, Zemke JE, Ali SS, et al. 3D-cultured blastoids model human embryogenesis from pre-implantation to early gastrulation stages. Cell Stem Cell, 2023, 30: 1148-65. e7. |
| [92] |
De Santis R, Rice E, Croft G, et al. The emergence of human gastrulation upon in vitro attachment. Stem Cell Rep, 2024, 19: 41-53. |
| [93] |
Alsolami S, Chandrasekaran AP, Jin Y, et al. DMSO derives trophectoderm and clonal blastoid from single human pluripotent stem cell. bioRxiv, 2024. DOI:10.1101/2023.12.31.573770 |
| [94] |
Oldak B, Wildschutz E, Bondarenko V, et al. Complete human day 14 post-implantation embryo models from naive ES cells. Nature, 2023, 622: 562-73. |
| [95] |
Pedroza M, Gassaloglu SI, Dias N, et al. Self-patterning of human stem cells into post-implantation lineages. Nature, 2023, 622: 574-83. |
| [96] |
Weatherbee BAT, Gantner CW, Iwamoto-Stohl LK, et al. Pluripotent stem cell-derived model of the post-implantation human embryo. Nature, 2023, 622: 584-93. |
| [97] |
Hislop J, Song Q, Keshavarz FK, et al. Modeling post-implantation human development to yolk sac blood emergence. Nature, 2023, 626: 367-76. |
| [98] |
Yuan G, Wang J, Liu Z, et al. Establishment of a novel non-integrated human pluripotent stem cell-based gastruloid model. bioRxiv, 2023. DOI:10.1101/2023.06.28.546720 |
| [99] |
Liu L, Oura S, Markham Z, et al. Modeling post-implantation stages of human development into early organogenesis with stem-cell-derived peri-gastruloids. Cell, 2023, 186: 3776-92. e16. |
| [100] |
Okubo T, Rivron N, Kabata M, et al. Hypoblast from human pluripotent stem cells regulates epiblast development. Nature, 2024, 626: 357-66. |
| [101] |
Sun S, Zheng Y, Kim YS, et al. A transgene-free, human peri-gastrulation embryo model with trilaminar embryonic disc-, amnion- and yolk sac-like structures. bioRxiv, 2024. DOI:10.1101/2024.08.05.606556 |
| [102] |
Rawlings TM, Makwana K, Taylor DM, et al. Modelling the impact of decidual senescence on embryo implantation in human endometrial assembloids. Elife, 2021, 10: e69603. |
| [103] |
Taniguchi K, Shao Y, Townshend RF, et al. Lumen formation is an intrinsic property of isolated human pluripotent stem cells. Stem Cell Rep, 2015, 5: 954-62. |
| [104] |
Shahbazi MN, Scialdone A, Skorupska N, et al. Pluripotent state transitions coordinate morphogenesis in mouse and human embryos. Nature, 2017, 552: 239-43. |
| [105] |
Wu J, Fu J. Toward developing human organs via embryo models and chimeras. Cell, 2024, 187: 3194-219. |
| [106] |
Wood C, Kretzer DD, Dennis P, et al. Transfer of a human zygote. Lancet, 1973, 302: 728-9. |
| [107] |
Steptoe PC, Edwards RG. Birth after the reimplantation of a human embryo. Lancet, 1978, 2: 366. |
| [108] |
Board EA, Health DO. HEW support of research involving human in vitro fertilization and embryo transfer. Biol Reprod, 1979, 21: 763. |
| [109] |
Listed N. Warnock-report of the committee of inquiry into human fertilisation and embryology. Ir Nurs News, 1985, 5: 7-8. |
| [110] |
Koch R. Conjoined twins and the biological account of personal identity. Monist, 2006, 89: 351-70. |
| [111] |
McNamara HC, Kane SC, Craig JM, et al. A review of the mechanisms and evidence for typical and atypical twinning. Am J Obstet Gynecol, 2016, 214: 172-91. |
| [112] |
Hyun I, Bredenoord AL, Briscoe J, et al. Human embryo research beyond the primitive streak. Science, 2021, 371: 998-1000. |
| [113] |
Rivron NC, Martinez Arias A, Pera MF, et al. An ethical framework for human embryology with embryo models. Cell, 2023, 186: 3548-57. |
| [114] |
De Miguel Beriain I, Rueda J, Villalba A. Re-defining the human embryo: a legal perspective on the creation of embryos in research. EMBO Rep, 2024, 25: 467-70. |
| [115] |
de Graeff N, De Proost L, Munsie M. 'Ceci n'est pas un embryon?' The ethics of human embryo model research. Nat Methods, 2023, 20: 1863-7. |
| [116] |
Rivron NC, Martinez-Arias A, Sermon K, et al. Changing the public perception of human embryology. Nat Cell Biol, 2023, 25: 1717-9. |
| [117] |
Rossant J, Fu J. Why researchers should use human embryo models with caution. Nature, 2023, 622: 454-6. |
| [118] |
Ball P. What is an embryo? Scientists say definition needs to change. Nature, 2023, 620: 928-9. |
| [119] |
Mallapaty S. Human embryo models are getting more realistic - raising ethical questions. Nature, 2024, 633: 268-71. |
| [120] |
Blackshaw BP, Rodger D. Why we should not extend the 14-day rule. J Med Ethics, 2021, 47: 712-4. |
| [121] |
Lovell-Badge R. Stem-cell guidelines: why it was time for an update. Nature, 2021, 593: 479. |
| [122] |
Derbyshire SW, Bockmann JC. Reconsidering fetal pain. J Med Ethics, 2020, 46: 3-6. |
 2025, Vol. 37
2025, Vol. 37 

 李天晴,昆明理工大学灵长类转化医学研究院二级教授,博士生导师,教育部国家高层次人才特聘教授,国务院政府特殊津贴获得者,国家重点研发计划首席科学家。2005年毕业于中国科学院昆明动物研究所,获得博士学位;2006年10月至2011年11月,在美国Stowers Institute for Medical Research从事博士后研究;2012年任职于云南省灵长类生物医学重点实验室研究员;2014年任职于昆明理工大学灵长类转化医学研究院,主要从事干细胞多能性、干细胞定向分化和标准化培养、早期胚胎发育、类器官构建以及干细胞治疗方面的研究。主持国家重点研发计划、国家自然科学基金重点项目以及省部级科研项目等10余项。以通讯作者在Nature、Cell Stem Cell、Cell Research、Science Advances、Advanced Science、Nature Communications等国际著名期刊发表研究论文40余篇,申请或获得专利授权12项,获云南省创新团队负责人、云岭学者、中青年学术技术带头人、有突出贡献优秀专业技术人员等称号
李天晴,昆明理工大学灵长类转化医学研究院二级教授,博士生导师,教育部国家高层次人才特聘教授,国务院政府特殊津贴获得者,国家重点研发计划首席科学家。2005年毕业于中国科学院昆明动物研究所,获得博士学位;2006年10月至2011年11月,在美国Stowers Institute for Medical Research从事博士后研究;2012年任职于云南省灵长类生物医学重点实验室研究员;2014年任职于昆明理工大学灵长类转化医学研究院,主要从事干细胞多能性、干细胞定向分化和标准化培养、早期胚胎发育、类器官构建以及干细胞治疗方面的研究。主持国家重点研发计划、国家自然科学基金重点项目以及省部级科研项目等10余项。以通讯作者在Nature、Cell Stem Cell、Cell Research、Science Advances、Advanced Science、Nature Communications等国际著名期刊发表研究论文40余篇,申请或获得专利授权12项,获云南省创新团队负责人、云岭学者、中青年学术技术带头人、有突出贡献优秀专业技术人员等称号