(2 中国科学院杭州医学研究所,杭州 310022)
(2 Hangzhou Institute of Medicine, Chinese Academy of Sciences, Hangzhou 310022, China)
合成生物学[1](synthetic biology)是2000年出现的一门以工程设计为理念,利用基因编辑技术、计算机模拟技术、生物工程技术以及化学合成技术等对生物体进行有目的性的设计、改造,甚至是重新合成的新兴交叉融合性学科。医学合成生物学是利用合成生物学的手段,合成具有特定生物学功能的元件,实现疾病的精准诊疗。医学合成生物学的核心之一是挖掘和开发更多的分子工具,以实现临床上的检测与治疗。
“质粒”这一概念最早是在1952年由美国生物学家Joshua Lederberg提出[2],他将“质粒”定义为任何染色体外的遗传决定因子,即能够独立于细胞的主要染色体之外存在,并且能够自我复制的一类遗传物质。这类遗传物质通常是指共价、闭合、环状的双链DNA分子,能够在细胞分裂过程中传递给后代细胞,并表达其所携带的遗传信息。1973年,Cohen等[3]通过体外连接不同质粒的限制性内切酶片段首次合成了功能性细菌质粒。双链质粒主要由转录单元和细菌骨架两部分组成[4]:转录单元由调控元件组成,如启动子、目标基因、终止子、多克隆位点(MCS)等;细菌骨架则由抗生素抗性基因、复制起点(Ori)和未甲基化的CpG基序等元件组成。双链质粒的合成需要保留细菌相关的冗余序列,然而这些冗余序列会引起哺乳动物的特异性识别和定点清除,造成高的免疫原性。已发现三类针对细胞内病原体衍生核酸的先天免疫传感器:位于体内的跨膜Toll样受体(TLR),可检测内溶酶体腔内的各种微生物DNA和RNA、细胞质RNA传感器以及细胞质DNA传感器[5]。环鸟苷酸-腺苷酸合酶(cGAS)是一种胞浆内的DNA传感器,能够识别胞浆内的双链DNA (dsDNA)和某些单链DNA (ssDNA),是尤为重要的双链DNA内源免疫通路[6]。整体而言,传统双链质粒在合成生物学中的应用存在以下4点不足:强免疫原性、高细胞毒性、多基因随机整合和少调控元件。可以通过将常用环状双链DNA质粒变为环状单链DNA (CssDNA)质粒,并去除冗余序列来解决应用上的不足。与环状双链DNA质粒相比,CssDNA不容易引起免疫系统的反应,具有更低的免疫原性,能够更安全地实现药物递送及基因编辑;CssDNA的独特结构具有较好的稳定性,且其结构具有可编程性,在构建分子机器和纳米级DNA方面具有优势。因此,CssDNA在医学合成生物学上具有重要的研究价值。
CssDNA在形状、特性和认知上与环状质粒DNA存在差异。1964年,Freifelder等[7]首次在电子显微镜下发现噬菌体φX174的基因组结构是由CssDNA组成。随后,1977年,科学家报道了一种菜豆金色黄斑病毒(golden yellow mosaic virus, GYMV),初步研究表明其核酸可能为ssDNA[8]。同年,关于两种植物病毒——玉米条纹病毒(Maize streak virus)和木薯潜伏病毒(Cassava latent virus)的研究结果发现这两种病毒含有CssDNA[9]。1982年,新发现了一种含有共价闭合的CssDNA的猪源病毒——猪圆环病毒(porcine circovirus, PCV),这是首个被发现的感染CssDNA的动物病毒[10]。与双链DNA质粒相比,CssDNA的研究并不彻底,最初是研究其复制与转录,随后发现CssDNA在复制过程中会有整合与碱基突变,并参与免疫反应[11]。随着Phi29 DNA聚合酶、滚环扩增(RCA)以及高通量测序技术的应用和发展,新型CssDNA的发现呈现爆发式增长[12-15]。同时,基于CssDNA病毒的系统研究表明,这类遗传物质通常具有编码复制相关蛋白(replication-associated protein, Rep)的特征性结构,因此被归类为CRESS DNA病毒(circular Rep-encoding single-stranded DNA viruses)[13, 16, 17]。
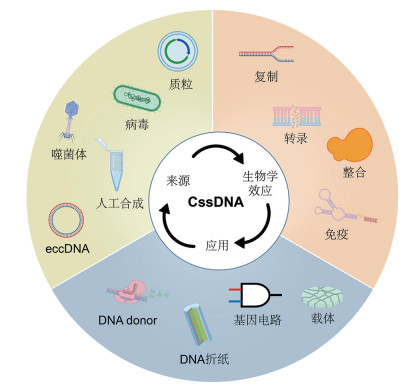
为了更好理解CssDNA在医学合成生物学上的应用,本文首先介绍CssDNA的天然来源、人工合成长CssDNA的方法;接着介绍CssDNA的生物学效应,总结CssDNA在细胞内和无细胞体系中的蛋白表达机制;最后总结CssDNA在医学合成生物学上的具体应用,展望未来的研究方向,如图 1。
|
图 1 CssDNA的研究及应用汇总图 |
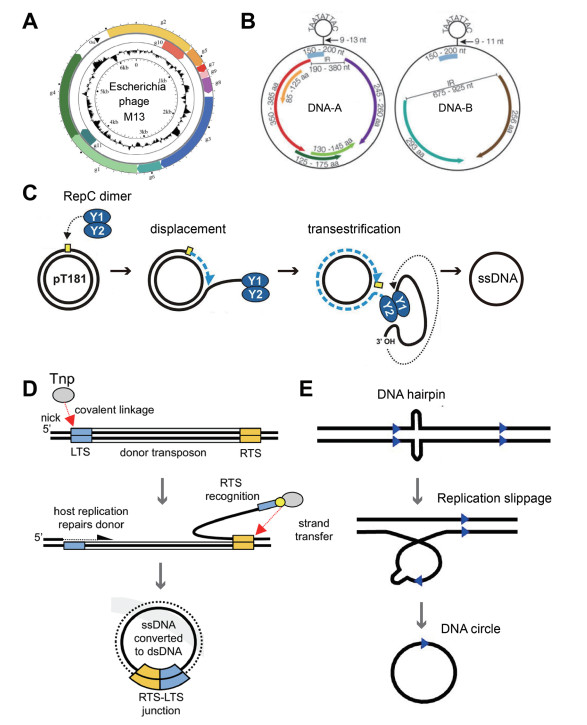
目前,在国际病毒分类委员会(ICTV)建立的13个单链DNA (ssDNA)病毒家族中,有11个存在于环状基因组中。CssDNA病毒中有4种是最受关注和研究的,即Inoviridae、Geminiviridae、Circoviridae、Anelloviridae[11]。Inoviridae家族是一类无包膜的柔性丝状噬菌体,具有5.5~10.6 kb的超螺旋、环状、正义、单链DNA基因组,编码7~15种蛋白质[18]。其中最具有代表性的是Escherichia phage M13 (图 2A),因M13mp18表现出能够增强复制率而成为组装DNA origami (DNA折纸)的初始来源[19, 20]。Geminiviridae (双生病毒科)具有高度多样化的宿主范围,能够感染单子叶植物和双子叶植物。与单链RNA病毒相比较,双生病毒的高突变率使之成为主要的植物病原体。此外,双生病毒的特殊之处是其中一些病毒包含二分基因组,即DNA-A和DNA-B[21, 22] (图 2B)。Circoviridae (圆环病毒)具有环状排列的单链DNA基因组,该基因组在正义链上编码保守的复制酶蛋白。该病毒广泛分布在哺乳动物、鱼类、鸟类,甚至昆虫中。而在猪中,已被鉴定出4种不同的PCV,即PCV1~PCV4[23]。Anelloviridae是具有小的CssDNA基因组的病毒家族,是人类病毒组的常见组成部分,其中Torque teno病毒(TTV)是一种普遍存在的小型病毒,具有高度多样化的单链负链DNA基因组和广泛的宿主范围[24, 25]。
|
(A) M13基因组[18]。(B)双生病毒基因组[13]。(C)基于质粒pT181的PCR模型[42]。(D) Helitron转座模型。红色箭头表示裂解或链转移反应。黄色圆圈表示共价键[39]。(E) eccDNA形成方式示例[41]。 图 2 CssDNA的来源 |
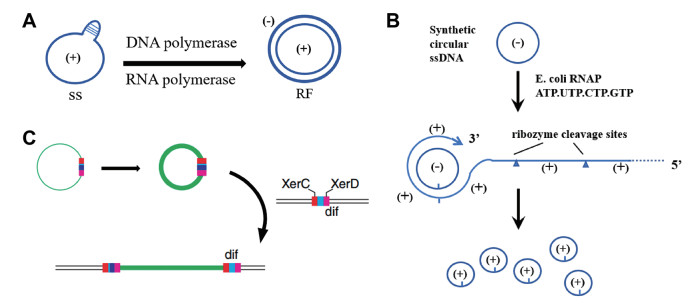
质粒作为分子生物学中最为常用的工具,其复制机制主要分为3种:θ复制、链置换复制和滚环复制,而CssDNA会以中间体的形式在滚环复制(rolling-circle replication, RCR)质粒形成过程中出现。根据对pT181质粒家族的研究可以对质粒RCR模型进行总结[26, 27] (图 2C)。RCR质粒编码Rep蛋白,Rep蛋白用作起始复制,它与位于质粒双链复制起点(DSO)内的结合序列特异性相互作用[28]。然后,Rep蛋白在切口序列处裂解DSO,并通过其活性位点的酪氨酸残基与5' 磷酸盐形成共价结合。前导链复制的开始是通过DNA聚合酶Ⅲ在位于缺口位点的游离3' -OH末端的延伸合成,此过程一直持续到前导链完全置换。在一系列的切割/重新连接过程后,圆形前导链被释放。最后,DNA末端通过DNA连接酶连接在一起,所得到的dsDNA通过DNA旋转酶转化为超螺旋结构[11],在此过程中,CssDNA则是作为质粒形成的中间体。
1.2.2 作为转座子和染色体外环状DNA (eccDNA)的中间体RCR起始蛋白可分为:Rep蛋白、Mob蛋白和转座酶[29-33]。转座因子(TE)分为原核插入序列(IS),即IS200/IS605和IS91/ISCR,以及真核Helitrons[34]。TE编码转座酶,能够催化转座反应。IS200/IS605是一类特殊的细菌插入序列,它能够利用CssDNA中间体和转座酶TnpA (转座酶TnpA属于HUH核酸内切酶)的催化作用。Ton-Hoang团队对IS200/IS605进行深入研究,并提出了一种“Peel-and-Paste”的机制来描述其转座过程[35, 36]。del Pilar Garcillán-Barcia等[37]已经证明TnpA和phiX174噬菌体A蛋白的功能相似性,并且观察到CssDNA中间产物的存在。Dyda团队报道了含有HUH核酸酶的Helitron转座酶仅在ssDNA上进行切割和链转移,并要求转座子供体位点为dsDNA形式,转座过程会产生CssDNA,随后会进行双链转化[38, 39] (图 2D)。Shibata等[40]最先发现eccDNA中存在CssDNA,并提出了一种生成小染色体外环形DNA (eccDNA)的环单链中间体模型(图 2E)。随后,Paulsen等[41]报道了关于eccDNA的三种可能机制,这些机制都需要利用CssDNA作为中间体来完成。
1.3 人工合成长CssDNA除了天然存在的CssDNA之外,还存在人工合成的CssDNA。人工合成的CssDNA可以根据其大小分为两类:长CssDNA和短CssDNA。长CssDNA主要源于噬菌体M13mp18,大小为1 000~50 000 nt (核苷酸);短CssDNA的大小为10~1 000 nt,此类CssDNA可以通过化学连接或是酶介导的方式合成。
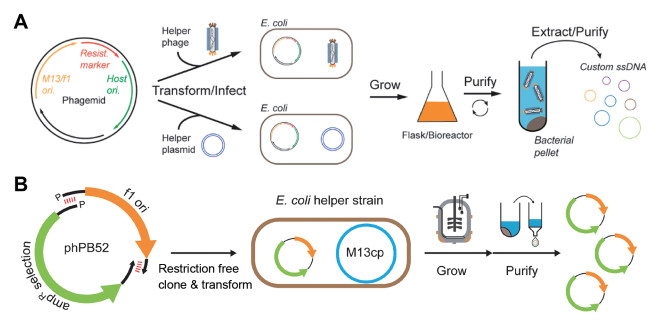
传统合成CssDNA的方法是利用T4连接酶将线性单链DNA的互补末端连接,形成环状结构[43-45]。虽然利用T4连接酶能够高效环化<100 nt的短线性单链,但对于长的线性单链的环化效率显著下降,且携带有目的蛋白基因载体的双链质粒的整体长度通常超过500 nt,而目前利用固相合成技术制备超过500 nt的线性单链也存在困难。目前,合成长CssDNA主要利用噬菌体和大肠杆菌作为宿主来实现(图 3A)。传统的摇瓶发酵方法能够生产出1~14 mg/L的纯化ssDNA,利用M13噬菌体可以实现大规模生产,通过进一步优化高细胞密度补料分批发酵条件,能够将ssDNA的产量提高至590 mg/L[46, 47]。但由于基因组包含CssDNA复制、包装和构建载体时所需要的基因和调控序列,会限制所合成CssDNA的多样性和大小[48]。噬菌体包含两个复制起点(ori),即dsDNA ori和ssDNA ori。前者是复制质粒的来源,用于噬菌体的扩增,而源自f1或者M13噬菌体的ssDNA ori负责CssDNA噬菌体的复制。为了能够合成任意的CssDNA序列,引入了辅助质粒系统,而最常用的辅助质粒是M13KO17[49]。将噬菌粒和辅助质粒共转染于大肠杆菌的感受态细胞中,辅助质粒能够编码并表达一系列对于噬菌体颗粒组装和释放必需的病毒蛋白,将生成的CssDNA包装到噬菌体中[50]。研究人员通过优化,消除了辅助质粒对CssDNA的污染[50, 51]。为了合成更长的CssDNA,LaBean团队将M13噬菌粒pBluescriptKS (-)插入到另一个常用的噬菌体λ中,生成了一种新型杂交噬菌体——λM13,这种噬菌体能够用于合成长度为51 466 nt的CssDNA[52]。为了有效避免质粒污染和提高纯化效率,Shepherd等[53]设计了一个新系统,包含phPB52和phPB84的两个微型噬菌体基因组;该基因组都包含一个f1单链ori,并没有双链ori,这能够有效避免dsDNA污染(图 3B)。
2 CssDNA的生物学效应CssDNA在细胞内具有许多功能,本文主要讨论CssDNA复制、转录、整合和免疫原性四个方面的内容。CssDNA将环状单链转化为环状双链的过程被称为“复制”,将自身(CssDNA、质粒、转座子)作为最终产物的整个过程被称为“扩增”[11]。因原核系统较为简单,已经在所得到的CssDNA中对M13/丝状噬菌体的复制和扩增过程进行了广泛的研究。
2.1 复制正义CssDNA M13基因组被分为以下模块:复制模块(gⅡ、gV和gX)、结构模块(gVⅡ、gⅠX、gVⅢ、gⅢ和gVⅠ)以及组装/挤出模块(gⅠ、gⅠV和gXⅠ)[18]。M13基因组的复制过程包括以下三个步骤:首先,正义链(+)CssDNA易位到细菌细胞质中,然后与宿主RNA聚合酶结合生成一个简短的引物[54]。宿主DNA聚合酶Ⅲ合成正义链(+)的互补链[55],将CssDNA转化为双链共价闭合DNA,称为复制形式(replicative form, RF)。在第二步中,mRNA由RF DNA和宿主RNA聚合酶转录,继续使用宿主系统翻译产生M13蛋白,主要是pⅡ。pⅡ蛋白具有核酸内切酶活性,并通过在CssDNA ori处切割dsDNA启动RCR机制来产生新(+)链[56]。宿主DNA聚合酶同样在这一步中用于合成正义链(+)CssDNA,同时pⅡ起到终止和环化的作用。生成的正义链(+)CssDNA随后进行RF的初始转化步骤。在第二步中,每个细胞的RF分子与足够高浓度的gV一起累积,这会导致gV同型二聚体的形成。而这些二聚体对ssDNA具有亲和力,可以有效包被新生成的正义链(+)CssDNA分子,从而抑制其转化为RF[57](图 4A)。RCR是DNA复制特征最全面的模型之一,在原核生物各种形式的DNA复制和扩增中起着至关重要的作用。它们具有相似的模型和机制,但在细节上有所不同。例如,它们的复制模板各不相同,包括CssDNA病毒基因组、RCR质粒和RCR转座子;此外,复制起始蛋白也不同,包括Rep、Mob和转座酶;并且它们之间涉及的生物过程也不同,包括噬菌体基因组和细菌质粒DNA复制、共轭转移和转座。然而,由于其系统的复杂性,真核生物中仍然存在许多未解决的问题,其中涉及更多的调控蛋白和生化过程[18]。
2.2 转录当CssDNA转化为RF时,转录过程即开始。噬菌体的转录仅利用两个操纵子来完成,即gⅡ-gV-gVⅡ-gⅠX-gVⅢ和gⅢ-gVⅠ-gⅠ-gⅠV,具有多个嵌套启动子和强终止子,会导致产生许多重叠的转录本[58, 59]。CssDNA还可以用于转录非编码RNA,包括microRNA、siRNA和长链非编码RNA (lncRNA)。根据报道,小尺寸的CssDNA能够在没有启动子的情况下直接转录形成RNA,Kool团队合成的特定长度的环状单链DNA核苷酸能够通过大肠杆菌RNA聚合酶进行体外转录,高效产生长重复RNA序列(图 4B),并且转录效率很高[60, 61]。随后,Ryan团队将环化后的DNA在体外和体内通过RNAP Ⅲ进行转录,并提出环状拓扑结构对高效转录有着重要影响,环化可以避免模板受到外切酶的降解,从而实现高效转录[62]。环化DNA的转录产物可以不被加帽或是5' 单磷酸化,表明可以通过设计模板避免一些RIG-I识别,能够在不触发免疫反应的情况下激活RNAP Ⅲ的先天免疫活性[62]。
2.3 整合和免疫原性Krupovic等[63]对CssDNA病毒的整合机制进行了较完整的总结。丝状噬菌体(Inoviridae)至少使用三种主要的整合机制。其中一些噬菌体编码丝氨酸或酪氨酸重组酶超家族的整合酶,而另一些则利用IS3、IS30、或IS110/IS492家族的DDE转座酶,而一些丝状噬菌体会劫持宿主XerCD重组机制(图 4C)。但是,对于真核生物CssDNA的整合机制却仍不太清楚[63-67]。
PCV2和双生病毒感染会导致宿主细胞持续感染,导致亚临床症状和免疫抑制。免疫反应主要受宿主和病毒蛋白之间的相互作用调节,而CssDNA在免疫调节中也能够发挥作用[11, 68]。PCV2的DNA基因组中具有特定的发夹结构基序(寡脱氧核糖核苷酸)[69],其能够抑制IFN-α活性,且抑制IL-6、IL-10和IL-12p40的mRNA表达。IFN-α和IFN-γ还可以增强PCV2的复制[70, 71]。Gu等[72]报道了PCV2 Rep启动子区域存在干扰素刺激反应元件(ISRE),还发现IFN-α通过IRSE诱导Rep启动子增强。
目前,人工合成的CssDNA的免疫原性也受到了广泛的关注。Lucas等[73]通过血液学检测、血浆细胞因子/趋化因子分析以及脾脏单核细胞分析等,对DNA折纸载体包括M13mp18进行了安全性评估(图 5A),结果显示CssDNA没有引起明显的毒性或不良免疫反应。
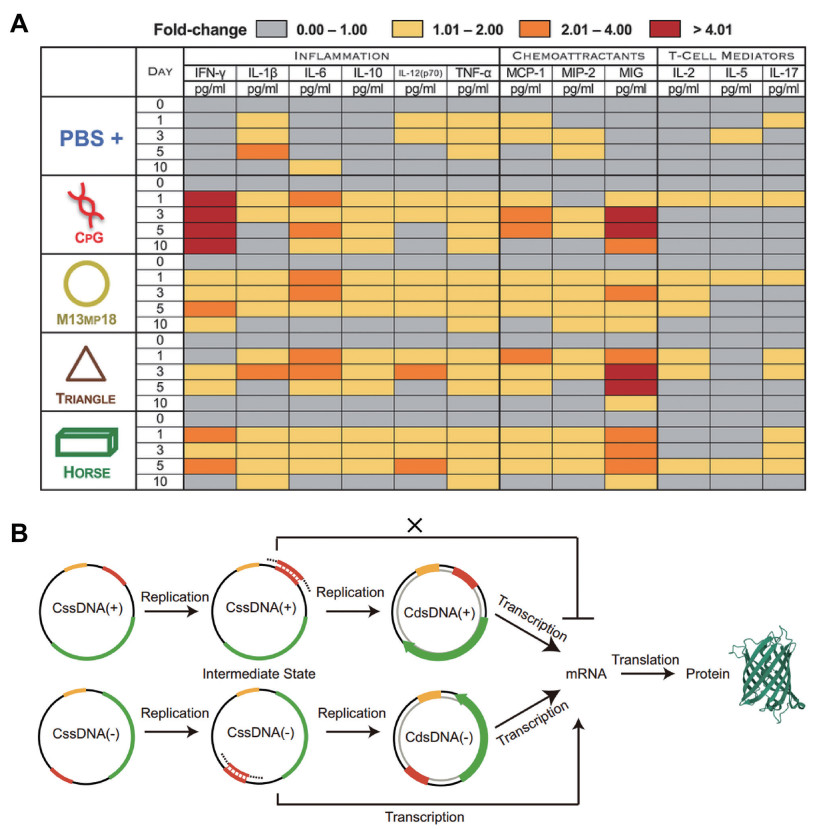
|
(A)体内CssDNA的血浆细胞因子评估。PBS+:PBS处理的对照;CpG:CpG-寡脱氧核苷酸;M13MP18:M13mp18噬菌体DNA;Triangle:三角DNA折纸;Horse:马DNA折纸[73]。(B) CssDNA(+)和CssDNA(−)载体的不同蛋白表达途径示意图[75]。 图 5 CssDNA的免疫原性、蛋白质表达机制 |
传统质粒的一个最为常用也是最为重要的功能就是作为外源蛋白表达的基因载体,CssDNA又被称作单链质粒,研究CssDNA是否具备传统质粒的载体功能尤为重要。最近,我们报道了一种基于CssDNA的基因表达调控方法,以CssDNA作为表达载体,在哺乳细胞中能够实现可切换的基因表达调控[74]。CssDNA通过DNA复制由单链转化为双链形式,实验证明以CssDNA作为载体可在多达22种细胞系以及小鼠模型中编码表达不同蛋白质的普适性。通过部分互补DNA“阻断”链形成发夹结构,阻碍单链DNA的复制过程,从而能够抑制基因表达,再通过添加触发链,利用Toehold介导的链置换,能够重新激活被抑制的基因表达。因此,我们也提出了一种基于CssDNA的哺乳细胞中基因表达的可切换调节策略。
为了进一步探索CssDNA的蛋白表达机制,我们利用无细胞蛋白质表达(cell-free protein expression, CFE)系统进行了一系列的反应。通过设计一种CssDNA含有一个T7启动子区域和一个目的蛋白的编码区域,其中正义链(CssDNA(+))包含目的蛋白表达盒的编码序列,反义链(CssDNA(-))则以相反的方向包含目的蛋白表达盒的模板序列。不同于传统载体直接转录或翻译的表达路径,CssDNA有自己特有的表达方式,且CssDNA(+)的表达路径不同于CssDNA(-)。对CssDNA(+)来说,必须通过复制过程将单链DNA转化为完整的双链DNA,才能作为转录模板用于后续的蛋白表达;而对于CssDNA(-)来说,并非所有的CssDNA(-)都需要转变为完整的双链(图 5B),当复制的中间产物含有以双链形式存在的T7启动子序列时,也可直接进行转录从而实现蛋白质的表达。这些发现不仅为合成生物学提供了新的工具和方法,还为构建复杂的基因调控网络和逻辑门奠定了基础。我们还通过调控dNTPs、酶抑制剂和互补链,成功实现了基因表达的精确控制,展示了CssDNA在无细胞系统中的巨大潜力[75]。
3 CssDNA在生物医学中的应用 3.1 CssDNA的内源免疫与长效表达传统双链质粒因其双链结构会被cGAS识别,从而引起cGAS-STING通路的免疫调节[76],此外,传统双链质粒合成所必需的细菌相关冗余序列会导致哺乳细胞的特异性识别和定点清除。双链结构和冗余序列是造成传统双链质粒具有强免疫原性的主要原因。Lucas等[73]对M13mp18等用作DNA折纸载体的材料进行了免疫原性和毒性的测试,结果表明,CssDNA在高剂量下表现出一定的免疫原性,但整体上是安全的,没有明显的毒性作用。我们最近开发了一种新型叶酸修饰的脂质纳米颗粒(folate-modified lipid nanoparticles, FA-LNPs)系统,用来递送表达IL-12的CssDNA,它可以在细胞内长效表达(图 6A),以治疗4T1三阴性乳腺癌模型[77]。实验数据显示,该系统能够有效激活抗癌免疫反应,同时在高剂量下具有良好的安全性,没有明显的毒副作用,且能减缓肿瘤生长,显著延长动物模型的生存期,并预防肿瘤复发。此外,研究发现,CssDNA在体内表达时暂未整合到宿主基因组中。与传统的双链质粒DNA不同,CssDNA在细胞内以游离态形式存在,不会引起基因组的不稳定。传统的双链质粒DNA存在未甲基化的CpG基序和细菌骨架序列,较容易被先天免疫系统识别,导致加速被清除[6]。相比之下,CssDNA的免疫原性较弱,且整合风险低,降低了潜在的基因组不稳定性风险。CssDNA作为新型的基因表达载体,能够实现活体内的长效表达,有潜力成为新的蛋白替代疗法,如用于治疗苯丙酮尿症和血友病等。
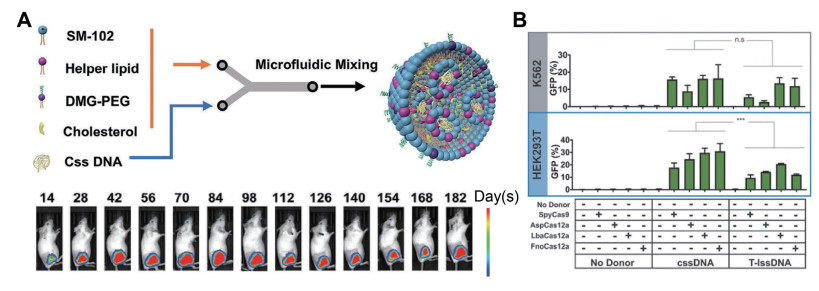
|
(A) Css DNA-LNPs的表征和肿瘤内治疗。LNPs的合成路线。注射S-SOPC LNP-Css Luciferase后相应时间点采集的小鼠体内发光图像[74]。(B) cssDNA和T-lssDNA的HDR效率表征。用不同的Cas效应物处理TLR-MCV1细胞后cssDNA和T-lssDNA介导的HDR效率比较[83]。 图 6 CssDNA的应用 |
环状单链DNA因其环状结构,不具有末端,相较于线性单链,更能抵抗核酸外切酶的降解,从而提高生物稳定性,这表明环状单链DNA在疾病诊断和治疗方面有着巨大潜力,尤其是在提高检测灵敏度和实现精准药物递送以及基因编辑等方面[78]。传统的非病毒基因靶向编辑方法主要通过使用寡核苷酸、质粒或是线性dsDNA作为供体模板来实现目标位点的DNA插入。CRISPR/Cas9技术的发展为基因功能研究和基因治疗提供了有力支持,但高效的同源定向修复(HDR)仍存在技术难题,尤其是针对一些特定的细胞,例如人类诱导多能干细胞(iPSCs)。目前最常用的供体为双链DNA供体[79],但这类供体在多种类型细胞中HDR效率低,并且存在脱靶整合风险。而单链DNA (ssDNA)供体能实现高效的HDR,且几乎不存在脱靶整合[80-84](图 6B)。目前,利用化学合成法和体外酶法生产线性单链DNA(lssDNA)的成本高、可扩展性低,且无法合成超过5 kb的长分子。Xie等[84]通过高度工程化的噬菌粒系统生产小型CssDNA,并且探讨了CssDNA作为高效基因编辑的供体模板的可能性,对比了CssDNA、dsDNA、lssDNA和腺相关病毒6 (AAV6)作为供体在实现同源定向修复(HDR)介导的基因组编辑方面的效率和安全性。Iyer等[83]通过超感染宿主细胞并且纯化噬菌体颗粒中的单链DNA,然后反向转录(RT)先生成lssDNA,再使用过程性逆转录酶生成CssDNA。该噬菌体系统能够生产长达22 kb的CssDNA,且纯度超过95%。CssDNA作为HDR供体模板在多种类型细胞中的效率显著优于lssDNA和双链质粒供体。
3.3 基于CssDNA的可调控基因电路我们使用CssDNA作为细胞外蛋白表达系统中的可编程基因载体进行基因表达,通过添加DNA合成成分(如dNTPs)、DNA聚合酶抑制剂(如aphidicolin)以及不同序列和长度的互补链,来促进或抑制CssDNA的表达水平。我们使用CssDNA (以CssDNA(-)为主)作为逻辑元件,并设计构建了基因CssDNA的双输入逻辑门,包括OR、INHIBIT、NOR、NAND和AND逻辑门(图 7A),展现了CssDNA作为细胞外蛋白表达系统中可编程性载体在基因调控方面的应用潜力[75]。
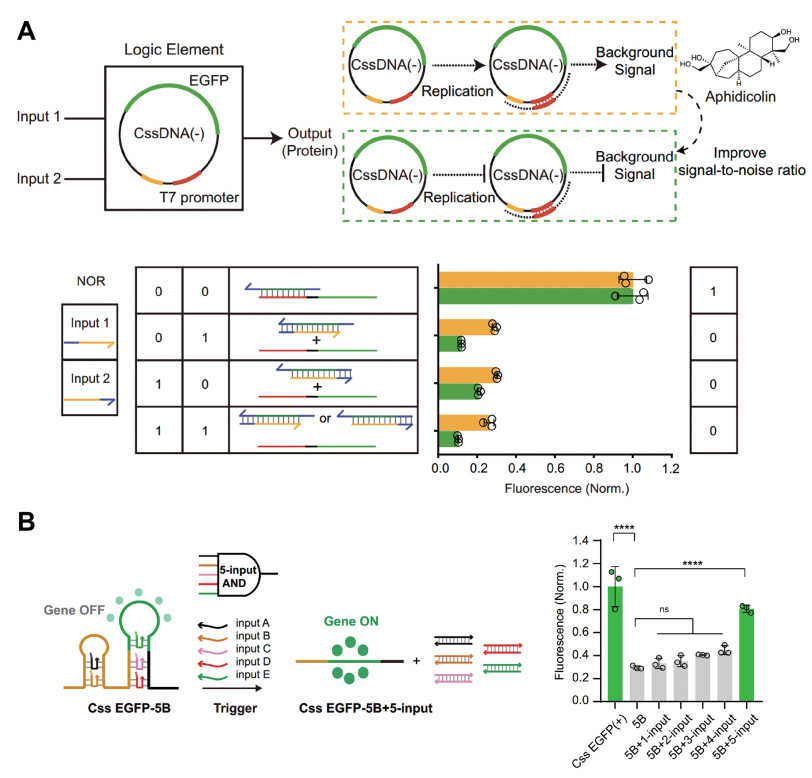
|
(A)使用CssDNA作为逻辑元件构建逻辑门。使用CssDNA(−) 作为逻辑元件,蛋白质作为输出,构建双输入逻辑门(上图)。NOR逻辑门示意图及荧光信号图(下图)[75]。(B)通过TMSD反应(5输入与门)添加5条单阻断链和5条立足点开关触发链而形成的环状单链基因表达调节器的示意图(左图)。环状单链基因表达调节剂(5输入与门)的荧光强度(右图)[74]。 图 7 CssDNA在基因电路上的应用 |
DNA折纸技术是一种利用DNA分子间碱基互补配对原则而自组装来构建纳米级复杂结构的方法,将长的支架链(ssDNA、CssDNA)用多根短的、序列定制化的DNA寡核苷酸链折叠成定制形状[85, 86]。由于DNA折纸纳米结构具有优越的可编程性[87]、生物相容性以及体内稳定性,已被用于精准药物递送和疾病治疗等领域[88],可以有效地递送小分子[89]、siRNA[90]、原核基因[91]以及蛋白质[92, 93]。Hendrik团队利用具有基因编码序列的CssDNA作为支架链形成折纸结构,并在结构中引入多个SV40核定位序列(DTS),成功在哺乳细胞中实现入核,显著提高了基因表达效率[94]。另外,他们通过优化折纸设计,并引入多种功能序列和结构(Kozak序列、嵌合内含子、WPRE、TR等),显著提高了基因表达效率,且通过折纸模块组装展现了多个基因的可控共同递送和表达能力[95]。丁宝全团队利用两条互补DNA链作为支架,构建具有基因编码序列的折纸结构,实现了脂质包覆的DNA折纸在肿瘤细胞中的高效基因表达和抗肿瘤有效性[96]。我们利用CssDNA作为可切换载体,通过脂质体(liposome)递送成功在哺乳动物细胞内实现基因表达调控。CssDNA能够折叠成复杂的纳米结构,这些结构能够通过Toehold介导的链置换反应实现对基因表达的可编程抑制和再激活(图 7B);同时还讨论了使用CssDNA构建复杂的基因电路的可能性[74]。这些成果不仅拓宽了分子机器和合成基因电路的调控研究方向,同时也为基因诊断和治疗的应用奠定了基础。
4 总结与展望随着核酸治疗领域的迅速发展,开发新的核酸载体成为研究的焦点。CssDNA是一种古老的核酸形式,因其低免疫原性、低细胞毒性、低整合风险等优势而展现出巨大的应用潜力。在本文中,我们对CssDNA的来源、细胞功能、蛋白质表达机制和生物医学应用进行了介绍。CssDNA作为新兴的医学合成生物学的分子工具,自身具有相对稳定的环形结构,抗核酸外切酶的能力强,可利用辅助质粒系统合成,不存在细菌骨架序列;相较于传统双链质粒,CssDNA不易造成沉默和清除,具有较低的细胞毒性[73]。CssDNA作为新兴的基因载体,利用一种新型叶酸修饰的脂质纳米颗粒(FA-LNPs)系统递送用于表达IL-12的CssDNA,能够在小鼠内实现长效表达,并可用于治疗4T1三阴性乳腺癌[77]。CssDNA可作为一种高效的同源定向修复(HDR)供体模板,在多种类型的细胞和基因组位点上都具有出色的表现[79-81, 84, 97],且CssDNA能够支持超大型转基因的整合,具有广泛的适用性、高特异性和低细胞毒性。CssDNA可以作为基因表达的载体[74],凭借单链形式具有可寻址性和可编程性,在无细胞系统或者哺乳细胞中均可实现精确的基因表达调控[75],通过设计特定的CssDNA序列,有望创建新的遗传回路,实现特定的生物功能或代谢路径的调节。整体而言,环状单链DNA在医学合成生物学中具有巨大潜力,未来针对环状单链DNA的进一步研究需要融合理学的结构与修饰、工学的底盘元件挖掘与优化、生物学的合成与改造以及医学的内源免疫和基因治疗等多学科知识和技术,有望把环状单链DNA发展成为医学合成生物学领域重要的分子工具之一。
| [1] |
Ausländer S, Ausländer D, Fussenegger M. Synthetic biology-the synthesis of biology. Angew Chem Int Ed Engl, 2017, 56: 6396-19. |
| [2] |
Lederberg J. Cell genetics and hereditary symbiosis. Physiol Rev, 1952, 32: 403-30. |
| [3] |
Cohen SN, Chang ACY, Boyer HW, et al. Construction of biologically functional bacterial plasmids in vitro. Proc Natl Acad Sci U S A, 1973, 70: 3240-4. |
| [4] |
Jechlinger W. Optimization and delivery of plasmid DNA for vaccination. Expert Rev Vaccines, 2006, 5: 803-25. |
| [5] |
Tan XJ, Sun LJ, Chen JQ, et al. Detection of microbial infections through innate immune sensing of nucleic acids. Ann Rev Microbiol, 2018, 72: 447-78. |
| [6] |
Mulia GE, Picanço-Castro V, Stavrou EF, et al. Advances in the development and the applications of nonviral, episomal vectors for gene therapy. Hum Gene Ther, 2021, 32: 1076-95. |
| [7] |
Freifelder D, Kleinschmidt AK, Sinsheimer RL. Electron microscopy of single-stranded DNA: circularity of DNA of bacteriophage φX174. Science, 1964, 146: 254-5. |
| [8] |
Goodman RM. Infectious DNA from a whitefly-transmitted virus of Phaseolus vulgaris. Nature, 1977, 266: 54-5. |
| [9] |
Harrison BD, Barker H, Bock KR, et al. Plant viruses with circular single-stranded DNA. Nature, 1977, 270: 760-2. |
| [10] |
Tischer I, Gelderblom H, Vettermann W, et al. A very small porcine virus with circular single-stranded DNA. Nature, 1982, 295: 64-6. |
| [11] |
Cao XS, Tang LL, Song J. Circular single-stranded DNA: discovery, biological effects, and applications. ACS Synth Biol, 2024, 13: 1038-58. |
| [12] |
Rosario K, Duffy S, Breitbart M. Diverse circovirus-like genome architectures revealed by environmental metagenomics. J Gen Virol, 2009, 90: 2418-24. |
| [13] |
Rosario K, Duffy S, Breitbart M. A field guide to eukaryotic circular single-stranded DNA viruses: insights gained from metagenomics. Arch Virol, 2012, 157: 1851-71. |
| [14] |
Lin LL, Kapoor A, Slikas B, et al. Multiple diverse circoviruses infect farm animals and are commonly found in human and chimpanzee feces. J Virol, 2010, 84: 1674-82. |
| [15] |
Dean FB, Nelson JR, Giesler TL, et al. Rapid amplification of plasmid and phage DNA using phi29 DNA polymerase and multiply-primed rolling circle amplification. Genome Res, 2001, 11: 1095-9. |
| [16] |
Zhao LL, Rosario K, Breitbart M, et al. Chapter three-eukaryotic circular Rep-encoding single-stranded DNA (CRESS DNA) viruses: ubiquitous viruses with small genomes and a diverse host range. Adv Virus Res, 2019, 103: 71-133. |
| [17] |
Krupovic M, Varsani A, Kazlauskas D, et al. Cressdnaviricota: a virus phylum unifying seven families of Rep-encoding viruses with single-stranded, circular DNA genomes. J Virol, 2020, 94: e00582-20. |
| [18] |
Knezevic P, Adriaenssens EM, Consortium IR. ICTV virus taxonomy profile: Inoviridae. J Gen Virol, 2021, 102: 001614. |
| [19] |
Rothemund PWK. Folding DNA to create nanoscale shapes and patterns. Nature, 2006, 440: 297-302. DOI:10.3969/j.issn.0253-9608.2006.05.012 |
| [20] |
Yanisch-Perron C, Vieira J, Messing J. Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mpl8 and pUC19 vectors. Gene, 1985, 33: 103-19. |
| [21] |
Jones RAC, Naidu RA. Global dimensions of plant virus diseases: current status and future perspectives. Ann Rev Virol, 2019, 6: 387-409. |
| [22] |
Bonnamy M, Blanc S, Michalakis Y. Replication mechanisms of circular ssDNA plant viruses and their potential implication in viral gene expression regulation. mBio, 2023, 14: e01692-23. |
| [23] |
Opriessnig T, Karuppannan AK, Castro AMMG, et al. Porcine circoviruses: current status, knowledge gaps and challenges. Virus Res, 2020, 286: 198044. |
| [24] |
Taylor LJ, Keeler EL, Bushman FD, et al. The enigmatic roles of Anelloviridae and Redondoviridae in humans. Curr Opin in Virol, 2022, 55: 101248. |
| [25] |
Webb B, Rakibuzzaman AGM, Ramamoorthy S. Torque teno viruses in health and disease. Virus Res, 2020, 285: 198013. |
| [26] |
Garcillán-Barcia MP, Pluta R, Lorenzo-Díaz F, et al. The facts and family secrets of plasmids that replicate via the rolling-circle mechanism. Microbiol Mol Biol Rev, 2022, 86. |
| [27] |
Solar Gd, Giraldo R, Ruiz-Echevarría MJ, et al. Replication and control of circular bacterial plasmids. Microbiol Mol Biol Rev, 1998, 62: 434-64. |
| [28] |
Khan SA. DNA-protein interactions during the initiation and termination of plasmid pT181 rolling-circle replication. Prog Nucleic Acid Res Mol Biol, 2003, 75: 113-37. |
| [29] |
Ronning DR, Guynet C, Ton-Hoang B, et al. Active site sharing and subterminal hairpin recognition in a new class of DNA transposases. Mol Cell, 2005, 20: 143-54. |
| [30] |
Messing SAJ, Ton-Hoang B, Hickman AB, et al. The processing of repetitive extragenic palindromes: the structure of a repetitive extragenic palindrome bound to its associated nuclease. Nucleic Acids Resh, 2012, 40: 9964-79. |
| [31] |
Datta S, Larkin C, Schildbach JF. Structural insights into single-stranded DNA binding and cleavage by F factor TraI. Structure, 2003, 11: 1369-79. |
| [32] |
Hickman AB, Ronning DR, Kotin RM, et al. Structural unity among viral origin binding proteins: crystal structure of the nuclease domain of adeno-associated virus Rep. Molecular Cell, 2002, 10: 327-37. |
| [33] |
Campos-Olivas R, Louis JM, Clérot D, et al. The structure of a replication initiator unites diverse aspects of nucleic acid metabolism. Proc Natl Acad Sci U S A, 2002, 99: 10310-5. |
| [34] |
Craig RJ. Replitrons: a major group of eukaryotic transposons encoding HUH endonuclease. Proc Natl Acad Sci, 2023, 120. |
| [35] |
Guynet C, Hickman AB, Barabas O, et al. In vitro reconstitution of a single-stranded transposition mechanism of IS608. Mol Cell, 2008, 29: 302-12. |
| [36] |
He S, Corneloup A, Guynet C, et al. The IS200/IS605 family and "Peel and Paste"single-strand transposition mechanism. Microbiol Spectr, 2015, 3. DOI:10.1128/microbiolspec.mdna3-0039-2014 |
| [37] |
Del Pilar Garcillán-Barcia M, Bernales I, Mendiola MV, et al. Single-stranded DNA intermediates in IS91 rolling-circle transposition. Mol Microbiol, 2001, 39: 494-502. |
| [38] |
Grabundzija I, Hickman AB, Dyda F. Helraiser intermediates provide insight into the mechanism of eukaryotic replicative transposition. Nat Commun, 2018, 9: 1278. |
| [39] |
Kosek D, Grabundzija I, Lei HT, et al. The large bat Helitron DNA transposase forms a compact monomeric assembly that buries and protects its covalently bound 5'-transposon end. Mol Cell, 2021, 81: 4271-86.e4. |
| [40] |
Shibata Y, Kumar P, Layer R, et al. Extrachromosomal microDNAs and chromosomal microdeletions in normal tissues. Science, 2012, 336: 82-6. |
| [41] |
Paulsen T, Kumar P, Koseoglu MM, et al. Discoveries of extrachromosomal circles of DNA in normal and tumor cells. Trends Genet, 2018, 34: 270-8. |
| [42] |
Wawrzyniak P, Płucienniczak G, Bartosik D. The different faces of rolling-circle replication and its multifunctional initiator proteins. Front Microbiol, 2017, 8: 2353. |
| [43] |
Tomkinson AE, Vijayakumar S, Pascal JM, et al. DNA ligases: structure, reaction mechanism, and function. Chem Rev, 2006, 106: 687-99. |
| [44] |
Doherty AJ, Dafforn TR. Nick recognition by DNA ligases. J Mol Biol, 2000, 296: 43-56. |
| [45] |
Rossi R, Montecucco A, Ciarrocchi G, et al. Functional characterization of the T4 DNA ligase: a new insight into the mechanism of action. Nucleic Acids Res, 1997, 25: 2106-13. |
| [46] |
Kick B, Praetorius F, Dietz H, et al. Efficient production of single-stranded phage DNA as SCaffolds for DNA origami. Nano Lett, 2015, 15: 4672-6. |
| [47] |
Kick B, Hensler S, Praetorius F, et al. Specific growth rate and multiplicity of infection affect high-cell-density fermentation with bacteriophage M13 for ssDNA production. Biotechnol Bioeng, 2017, 114: 777-84. |
| [48] |
Bush J, Singh S, Vargas M, et al. Synthesis of DNA origami scaffolds: current and emerging strategies. Molecules, 2020, 25: 3386. |
| [49] |
Vieira J, Messing J. Production of single-stranded plasmid DNA. Methods Enzymol, 1987, 153: 3-11. |
| [50] |
Brown S, Majikes J, Martínez A, et al. An easy-to-prepare mini-scaffold for DNA origami. Nanoscale, 2015, 7: 16621-4. |
| [51] |
Chasteen L, Ayriss J, Pavlik P, et al. Eliminating helper phage from phage display. Nucleic Acids Res, 2006, 34e145. |
| [52] |
Marchi AN, Saaem I, Vogen BN, et al. Toward larger DNA origami. Nano Lett, 2014, 14: 5740-7. |
| [53] |
Shepherd TR, Du RR, Huang H, et al. Bioproduction of pure, kilobase-scale single-stranded DNA. Sci Rep, 2019, 9: 6121. |
| [54] |
Higashitani N, Higashitani A, Guan Z-W, et al. Recognition mechanisms of the minus-strand origin of phage f1 by RNA polymerase. Genes Cells, 1996, 1: 829-41. |
| [55] |
Geider K, Kornberg A. Conversion of the M13 viral single strand to the double-stranded replicative forms by purified proteins. J Biol Chem, 1974, 249: 3999-4005. |
| [56] |
Meyer TF, Geider K, Kurz C, et al. Cleavage site of bacteriophage fd gene II-protein in the origin of viral strand replication. Nature, 1979, 278: 365-7. |
| [57] |
Lerner TJ, Model P. The "steady state" of coliphage f1:DNA synthesis late in infection. Virology, 1981, 115: 282-94. |
| [58] |
La Farina M, Model P. Transcription in bacteriophage f1-infected Escherichia coli: messenger populations in the infected cell. J Mol Biol, 1983, 164: 377-93. |
| [59] |
Smits MA, Jansen J, Konings RNH, et al. Initiation and termination signals for transcription in bacteriophage M13. Nucleic Acids Res, 1984, 12: 4071-81. |
| [60] |
Daubendiek SL, Kool ET. Generation of catalytic RNAs by rolling transcription of synthetic DNA nanocircles. Nat Biotechnol, 1997, 15: 273-7. |
| [61] |
Diegelman AM, Kool ET. Generation of circular RNAs and trans-cleaving catalytic RNAs by rolling transcription of circular DNA oligonucleotides encoding hairpin ribozymes. Nucleic Acids Res, 1998, 26: 3235-41. |
| [62] |
Seidl CI, Lama L, Ryan K. Circularized synthetic oligodeoxynucleotides serve as promoterless RNA polymerase III templates for small RNA generation in human cells. Nucleic Acids Res, 2012, 41: 2552-64. |
| [63] |
Krupovic M, Forterre P. Single-stranded DNA viruses employ a variety of mechanisms for integration into host genomes. Ann N Y Acad Sci, 2015, 1341: 41-53. |
| [64] |
Kawai M, Uchiyama I, Kobayashi I. Genome comparison in silico in neisseria suggests integration of filamentous bacteriophages by their own transposase. DNA Res, 2005, 12: 389-401. |
| [65] |
Grindley NDF, Whiteson KL, Rice PA. Mechanisms of site-specific recombination. Ann Rev Biochem, 2006, 75: 567-605. |
| [66] |
Das B, Martínez E, Midonet C, et al. Integrative mobile elements exploiting Xer recombination. Trends Microbiol, 2013, 21: 23-30. |
| [67] |
Van Duyne GD, Rutherford K. Large serine recombinase domain structure and attachment site binding. Crit Rev Biochem Mol Biol, 2013, 48: 476-91. |
| [68] |
Niu G, Chen S, Li X, et al. Advances in crosstalk between porcine circoviruses and host. Viruses, 2022, 14: 1419. |
| [69] |
Wikström FH, Fossum C, Fuxler L, et al. Cytokine induction by immunostimulatory DNA in porcine PBMC is impaired by a hairpin forming sequence motif from the genome of Porcine Circovirus type 2 (PCV2). Vet Immunol Immunopathol, 2011, 139: 156-66. |
| [70] |
Ramamoorthy S, Huang FF, Huang YW, et al. Interferon-mediated enhancement of in vitro replication of porcine circovirus type 2 is influenced by an interferon-stimulated response element in the PCV2 genome. Virus Res, 2009, 145: 236-43. |
| [71] |
Mutthi P, Theerawatanasirikul S, Roytrakul S, et al. Interferon gamma induces cellular protein alteration and increases replication of porcine circovirus type 2 in PK-15 cells. Arch Virol, 2018, 163: 2947-57. |
| [72] |
Gu JY, Zhang Y, Lian X, et al. Functional analysis of the interferon-stimulated response element of porcine circovirus type 2 and its role during viral replication in vitro and in vivo. Virol J, 2012, 9: 152. |
| [73] |
Lucas CR, Halley PD, Chowdury AA, et al. DNA origami nanostructures elicit dose-dependent immunogenicity and are nontoxic up to high doses in vivo. Small, 2022, 18: 2108063. |
| [74] |
Tang LL, Tian ZJ, Cheng J, et al. Circular single-stranded DNA as switchable vector for gene expression in mammalian cells. Nat Commun, 2023, 14: 6665. |
| [75] |
Tian ZJ, Shao DD, Tang LL, et al. Circular single-stranded DNA as a programmable vector for gene regulation in cell-free protein expression systems. Nat Commun, 2024, 15: 4635. |
| [76] |
Motwani M, Pesiridis S, Fitzgerald KA. DNA sensing by the cGAS-STING pathway in health and disease. Nat Rev Genet, 2019, 20: 657-74. |
| [77] |
Zhang YJ, Cao XS, Hu G, et al. Targeted delivery of circular single-stranded DNA encoding IL-12 for the treatment of triple-negative breast cancer. Adv Healthc Mater, 2024, 13: 2401376. |
| [78] |
Shen TT, Zhang Y, Mei L, et al. Single-stranded circular DNA theranostics. Theranostics, 2022, 12: 35-47. |
| [79] |
Zhang JP, Li XL, Li GH, et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol, 2017, 18: 35. |
| [80] |
Quadros RM, Miura H, Harms DW, et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biol, 2017, 18: 92. |
| [81] |
Codner GF, Mianné J, Caulder A, et al. Application of long single-stranded DNA donors in genome editing: generation and validation of mouse mutants. BMC Biol, 2018, 16: 70. |
| [82] |
Hao M, Wang ZG, Qiao HY, et al. Dynamic genome editing using in vivo synthesized donor ssDNA in Escherichia coli. Cells, 2020, 9: 467. |
| [83] |
Iyer S, Mir A, Vega-Badillo J, et al. Efficient homology-directed repair with circular single-stranded DNA donors. CRISPR J, 2022, 5: 685-701. |
| [84] |
Xie KQ, Starzyk J, Majumdar I, et al. Efficient non-viral immune cell engineering using circular single-stranded DNA-mediated genomic integration. Nat Biotechnol, 2024, DOI: 10.1038/s41587-024-02504-9
|
| [85] |
Dietz H, Douglas SM, Shih WM. Folding DNA into twisted and curved nanoscale shapes. Science, 2009, 325: 725-30. |
| [86] |
Douglas SM, Dietz H, Liedl T, et al. Self-assembly of DNA into nanoscale three-dimensional shapes. Nature, 2009, 459: 414-8. |
| [87] |
Yu L, Cheng J, Wang DF, et al. Stress in DNA gridiron facilitates the formation of two-dimensional crystalline structures. J Am Chem Soc, 2022, 144: 9747-52. |
| [88] |
Zhang YM, Tian XC, Wang ZJ, et al. Advanced applications of DNA nanostructures dominated by DNA origami in antitumor drug delivery. Front Mol Biosci, 2023, 10: 1239952. |
| [89] |
Li J, Fan CH, Pei H, et al. Smart drug delivery nanocarriers with self-assembled DNA nanostructures. Adv Mater, 2013, 25: 4386-96. |
| [90] |
Wang ZR, Song LL, Liu Q, et al. A tubular DNA nanodevice as a siRNA/chemo-drug co-delivery vehicle for combined cancer therapy. Angew Chem Int Ed Engl, 2021, 60: 2594-8. |
| [91] |
Jiao K, Zhu B, Guo L, et al. Programming switchable transcription of topologically constrained DNA. J Am Cheml Soc, 2020, 142: 10739-46. |
| [92] |
Li SP, Jiang Q, Liu SL, et al. A DNA nanorobot functions as a cancer therapeutic in response to a molecular trigger in vivo. Nat Biotechnol, 2018, 36: 258-64. |
| [93] |
Zhao S, Duan FY, Liu SL, et al. Efficient intracellular delivery of RNase A using DNA origami carriers. ACS Appl Mater Interfaces, 2019, 11: 11112-8. |
| [94] |
Liedl A, Grießing J, Kretzmann JA, et al. Active nuclear import of mammalian cell-expressible DNA origami. J Am Chem Soc, 2023, 145: 4946-50. |
| [95] |
Kretzmann JA, Liedl A, Monferrer A, et al. Gene-encoding DNA origami for mammalian cell expression. Nat Commun, 2023, 14: 1017. |
| [96] |
Wu XH, Yang CP, Wang H, et al. Genetically encoded DNA origami for gene therapy in vivo. J Am Chem Soc, 2023, 145: 9343-53. |
 2025, Vol. 37
2025, Vol. 37 

 唐林林,中国科学院杭州医学研究所博士后,主要的研究方向是单链DNA拓扑学调控与基因电路开发,以及功能核酸适配体创新设计与生物医学应用。目前已发表文章10余篇,其中包括Nature Communications、JACS、Small Methods等国际顶级学术期刊。;
唐林林,中国科学院杭州医学研究所博士后,主要的研究方向是单链DNA拓扑学调控与基因电路开发,以及功能核酸适配体创新设计与生物医学应用。目前已发表文章10余篇,其中包括Nature Communications、JACS、Small Methods等国际顶级学术期刊。; 宋杰,中国科学院杭州医学所研究员, 上海交通大学兼职教授,国家海外高层次人才引进青年项目获得者,国家自然科学基金优秀青年基金获得者。课题组现在主要的研究方向是DNA纳米技术与分子通信及信息存储、智能载药系统及基因治疗,以及智能诊疗仪器的开发与应用。课题组已发表文章80余篇,其中包括Science、Nature Communications、JACS等国际顶级学术期刊,详情见主页
宋杰,中国科学院杭州医学所研究员, 上海交通大学兼职教授,国家海外高层次人才引进青年项目获得者,国家自然科学基金优秀青年基金获得者。课题组现在主要的研究方向是DNA纳米技术与分子通信及信息存储、智能载药系统及基因治疗,以及智能诊疗仪器的开发与应用。课题组已发表文章80余篇,其中包括Science、Nature Communications、JACS等国际顶级学术期刊,详情见主页