(2 上海科技大学先进医用材料与医疗器械全国重点实验室,上海 201210)
(3 上海临床研究中心,上海 201210)
(2 State Key Laboratory of Advanced Medical Materials and Devices, ShanghaiTech University, Shanghai 201210, China)
(3 Shanghai Clinical Research and Trial Center, Shanghai 201210, China)
无细胞蛋白质合成(cell-free protein synthesis, CFPS)系统是在不使用完整活细胞的情况下,在体外环境中激活转录、翻译过程,实现体外蛋白质高效合成的技术[1]。它无需完整的活细胞,对细胞的依赖性低,蛋白质合成反应可在试管中进行,既适用于小规模的生物过程原型设计,也适用于大规模的生物工程,具有灵活、高效、可控性强的特点[2]。在过去的70多年里,随着分子生物学领域新技术的不断涌现,CFPS系统经历了快速和多元化的发展历程[3],逐步发展为强大的蛋白质工程平台。从功能基因组学到生产治疗性重组蛋白,CFPS系统不断改进,展现出极大的应用潜能[4]。
生物医学领域,特别在药物研发、疫苗制备、疾病诊断、个性化医疗、组织工程与再生医学等方面,是CFPS系统应用最热门的领域之一[5]。近年来,CFPS系统已成功应用于重组蛋白药物生产、高通量药物筛选、疫苗设计等方面,为解决生物医学领域中的难题提供了新的思路,推动了生物医药研究的进步[6]。本文将基于对CFPS系统的原理、制备方法及优点的介绍,重点针对CFPS系统在生物医学领域的应用展开综述(图 1),并探讨CFPS系统在生物医学研究中存在的问题和所面临的挑战,对其发展前景做出展望,以期能为生物医学领域的发展提供一定的技术支撑和借鉴。
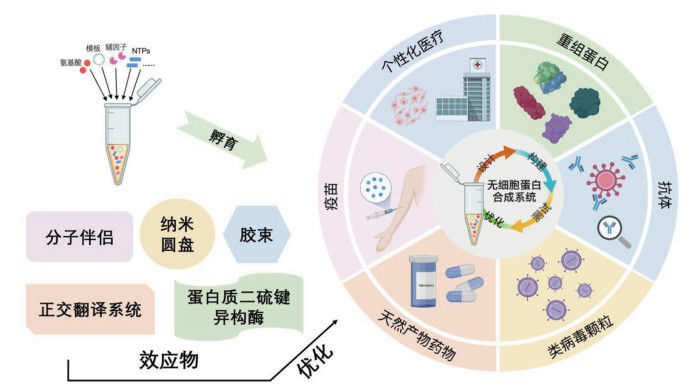
|
图 1 无细胞蛋白质合成系统优化及其在生物医学中的应用示例 |
传统的CFPS系统主要由细胞提取物、反应混合物(包含底物和能量等物质)以及携带遗传信息的DNA模板等三个部分组成。理论上,任何生物都可以作为CFPS的底盘生物。常见的CFPS细胞提取物的底盘生物有大肠杆菌[7]、酵母菌[8]、链霉菌[9]、小麦胚芽[10]、兔网织红细胞[11]、昆虫细胞[12]、哺乳动物细胞(如HeLa细胞[13])等。其中,基于大肠杆菌提取物的CFPS系统制备成本较低、操作简单、产量较高,是目前最完善、也最常用的底盘之一[14]。CFPS系统通过裂解底盘细胞获得粗细胞提取物,然后进行一系列离心、洗涤过程,去除细胞碎片及内源DNA和mRNA,保留基因转录、翻译、蛋白质折叠等所必需的元素(如RNA聚合酶、氨基酰-tRNA合成酶、核糖体以及翻译相关因子等),来模拟体内蛋白质合成环境,从而可以以外源添加的DNA或mRNA为模板,实现目标蛋白质的合成[4, 15]。CFPS系统利用底盘细胞本身的翻译机制,绕过了细胞膜的约束,有助于蛋白质折叠、难以表达的蛋白质合成以及蛋白质高通量生产。因此,与体内蛋白质表达方法相比,CFPS体外平台在蛋白质表达及药物发现的应用中具有独特的优势(表 1)。
| 表 1 体外和体内蛋白质合成系统在蛋白表达及药物发现应用中的比较 |
蛋白质疗法是一种利用蛋白质或其衍生物来治疗疾病的方法,具有靶向性强、副作用相对较小的优势,可用于治疗糖尿病、癌症、自身免疫性疾病、感染性疾病和其他疾病。蛋白质疗法主要有单克隆抗体、重组药物蛋白、疫苗和酶替代疗法等几种形式。使用CFPS系统生产治疗性蛋白可以很好地避免因化学合成或基于细胞的蛋白质合成策略而产生的难以合成大分子多肽或蛋白质、蛋白质降解、模板DNA丢失、难以高通量表达和筛选等问题,从而作为一种替代方法,在蛋白质药物生产过程中发挥越来越重要的作用[30, 31]。
2.2 抗体1986年,FDA批准了第一个治疗性单克隆抗体,开创了单克隆抗体用于疾病治疗的先河,从此抗体药物的研发备受关注。如今抗体药物的巨大潜力被挖掘出来,每年FDA批准的新药中抗体药物约占1/5。发展至今,抗体已成为医药市场上最畅销的药物种类之一,带来的经济效益逐年升高,预计到2025年将产生3 000亿美元的市场价值[32]。随着抗体及抗体片段药物的崛起,CFPS系统被认为是抗体工业的“变革性力量”,是快速生产各种抗体的有效手段[4, 33]。由于缺失恒定区,抗体对于细胞适应性的减弱导致较多抗体片段在细胞或细菌中表达效率低下。CFPS系统在表达细胞耐受性差或在细胞环境中表达困难的样品上显示出独特的优势。除了能有效合成在细胞中表达有困难的蛋白,CFPS系统还可避免细胞环境中复杂因素的干扰,可以更好地解释抗体片段表达不佳的原因。通过提供直观的实验结果,CFPS系统在指导目标基因序列的改造和优化方面也展现出无可比拟的优势。
现代免疫测定法通常是抗体或分析物与报告分子相连,从而将分析物浓度转换为信号强度[34]。Gaussia princeps luciferase荧光素酶(GLuc)是一种光发射效率极高的分泌型荧光素酶,通常用于生物发光成像和实时监测肿瘤发展情况,能够提供非常灵敏的信号输出[35]。Patel等[36]利用CFPS系统分别合成GLuc和抗体片段(scFvs),将GLuc和scFvs进行化学偶联,生产用于检测肿瘤细胞的GLuc-scFvs生物共轭物,证明了CFPS系统生产复杂的含有二硫键的蛋白质和用于检测特殊细胞的抗体片段共轭物的可行性。近年来,纳米抗体(nanobodies, Nbs),也称VHH抗体片段,是抗体研究的焦点。Nbs具有源于天然抗体基因的优势,这些基因经过了数百万年的进化,因此Nbs稳定性高、可溶性好且可严格保持单体形式,在应用过程中不易形成聚集体或二聚体[37-39]。2021年,Chen等[40]构建无细胞纳米抗体工程平台(CeVICA)用于合成SARS-CoV-2中和纳米抗体,该纳米抗体能够与SARS-CoV-2特异性结合并阻断其感染和传播,CeVICA为体外快速合成不同的Nbs提供了综合解决方案。
抗体-药物偶联物(ADC)是一种前沿的靶向化疗药物,与传统化疗和单克隆抗体疗法相比,ADC表现出更高的治疗指数和更强的疗效。由于使用天然氨基酸将药物与相关蛋白质偶联时会出现异质性以及偶联物稳定性差的情况,因此有学者提出一种位点特异性解决方案,助力药物的研发,即在CFPS系统中使用优化的非天然氨基酸(noncanonical amino acids, ncAA)来生产ADC[41]。Zimmerman等[41]对pAMF进行优化,利用pAMF的位点特异性将化疗细胞毒素与肿瘤抗原结合IgG偶联,在SKBR3细胞系中进行细胞毒性实验,证明生产的ADC没有细胞毒性,说明在制备过程中过量使用的ADC接头在偶联后已被有效清除。
2.3 重组蛋白使用异源宿主生产重组蛋白时可能会因细胞毒性或蛋白在表达过程中形成包涵体,使得下游蛋白质纯化和表征过程难以进行。与复杂的活细胞系统相比,CFPS系统环境是高度可控的,不受细胞内组分、细胞承受能力和生长代谢情况的影响,避免了细胞生长和蛋白质生产之间的竞争,所有能量都可用来生产目标蛋白质,能够实现重组蛋白的快速表达和筛选。基于大肠杆菌的CFPS系统可以有效地避免与包涵体形成相关的问题,提高重组蛋白的溶解度[42]。如使用CFPS系统表达癌症治疗药物onconase时,能以可溶的活性形式快速表达[43]。
酶是一类特殊的蛋白质治疗剂,目前所批准用于临床的酶都是与抗体不同的蛋白质。有研究者报道了一种基于CFPS系统生产沙雷肽酶(serratio-peptidase)的新方法,通过合理选择细胞提取物和优化参数,实现具有催化活性的沙雷肽酶的可溶性表达,证明CFPS是合成“难以表达”的治疗蛋白的高效平台[44]。此外,在合成治疗性激素方面,Zemella等[45]使用昆虫细胞提取物成功地在CFPS系统中合成了人促红细胞生成素(hEPO),该蛋白主要用于治疗由特定慢性疾病引起的贫血,商品化的重组hEPO在糖型上存在批次间的差异,而CFPS技术能够有效避免这一差异的产生。
2.4 类病毒颗粒类病毒颗粒(virus-like particle, VLP)是指由病毒的衣壳蛋白、核心蛋白或包膜蛋白通过自组装形成的颗粒物质,它能模拟真实病毒的结构,但不含传染性遗传物质,无生物安全隐患[46]。作为纳米颗粒载体,VLP在生物技术领域,如疫苗、药物输送、成像剂、生物催化剂等多方面的应用中得到了广泛的研究。由于结构上与原病毒相似,即使在无佐剂的情况下,低剂量就可引起强烈的免疫反应,可以降低疫苗成本[47]。通过体内方式生产VLP,往往会导致产物在结构和成分上的差别,也面临重组途径复杂、产量低的生产技术限制。与之相比,无细胞环境允许直接访问和控制VLP生产过程中的转录、翻译和组装,更有助于VLP的优化生产[48]。
目前,已有许多关于使用CFPS系统生产VLP的报道。Bundy等[49]开发出一种使用基于大肠杆菌的CFPS系统合成乙型肝炎病毒核心抗原VLP的方法,并证明在不损失蛋白质产量和活性的前提下可以将发酵设备从15 mL反应体积扩展到1 L反应体积。Ludgate等[50]实现了乙型肝炎病毒核心蛋白在基于哺乳动物(兔网织红细胞)的无细胞体系中的组装。Wang等[51]开发了一种基于酵母的适用于人乳头瘤病毒58 (HPV58) L1 mRNA体外翻译的CFPS系统,并证明VLP是在酵母无细胞系统中由HPV58 L1衣壳蛋白组装而成的。Spice等[48]使用基于巴斯德毕赤酵母的无细胞系统,开发出一种快速生产和部分纯化乙型肝炎病毒核心抗原的方法,这为新型疫苗生产纯化提供了新的思路,缓解了目前VLP制造中结构异质性的挑战。CFPS系统可以在几小时内快速生产出VLP,反应体系可以从微升级到百升级[21, 49]。作为开放灵活的系统,在CFPS反应中加入ncAA可以将功能分子连接到VLP表面,使其生产后无需拆卸和重新组装或进行化学修饰[52]。研究发现,表面尖峰顶端的多重负电荷有利于VLP的组装,可引入ncAA以实现疫苗抗原和免疫反应刺激分子的附着[53]。从病原体中提取的VLP已经在疫苗开发方面取得了重大进展,VLP保留了原病毒大量的重要抗原表位,可诱导较强的免疫反应[54]。与其他生物合成平台相比,CFPS系统表达的VLP产量高,数据显示:在基于大肠杆菌的CFPS平台中,4 h可合成并组装约600 mg/L的人类诺如病毒VLP[18];在基于HeLa细胞的CFPS系统中,24 h内合成了1.0×106 pfu/µL的脑心肌炎病毒VLP [55]。2021年,研究人员首次使用CFPS系统生产串联核心乙型肝炎核心抗原VLP,证明CFPS可用于生产更为复杂的VLP自组装产品,该策略也将用于改进基于VLP的疫苗设计,以提高产品质量[56]。
2.5 疫苗CFPS系统使用从细胞中制备的粗细胞提取物,保留了原生细胞的转录和翻译机制,无需完整的活细胞就能快速生产蛋白质。此外,该系统操纵简便、灵活开放,可用于按需生物制造,为快速生产个性化疫苗提供了平台,也为应对疫苗研发中的现有挑战提供了新策略,是一种具有前景的疫苗生产工具[56-58]。由于CFPS系统在生产过程中不需要刺激性化学物质来灭活病原体,并且不含危险的毒性物质,因此生产出的疫苗更安全,而CFPS较短的生产时间使其在个性化疫苗领域应用前景可观。CFPS可以解决当前疫苗的局限性,并扩展现有疫苗的生产技术。CFPS反应的设置非常灵活,在系统中可以加入ncAA,这些ncAA可在特定位点合成修饰蛋白,以提高蛋白质的功能性[29, 59]。
Kanter等[60]开发了一种用于生产B细胞淋巴瘤疫苗的无细胞系统,用于高效生产生物活性粒细胞-巨噬细胞集落刺激因子(GM-CSF),并确定了GM-CSF与Ig单链可变片段(scFv)结合的最佳基因顺序,证明GM-CSF-scFv融合蛋白能够保护小鼠免受肿瘤攻击,这表明无细胞表达系统生产特异性疫苗的可行性。Lu等[57]报道了一种基于大肠杆菌CFPS系统和简易复性方案,快速制备流感病毒A/California/05/2009 (H1N1)血凝素(hemagglutinin, HA)颈部结构域正确折叠蛋白的方法,为生产复杂多聚体蛋白单个结构域提供了新思路。Huang等[61]以CFPS为平台生产适合在牙龈卟啉单胞菌诱发的小鼠口腔骨质流失模型中进行疗效测试的疫苗靶点,为牙周病疫苗的开发提供了研究思路。随着冻干技术的发展,冻干无细胞体系的活性可以维持长达一年之久,使用时加水即可激活,使CFPS体系的运输和储存成本大大降低,生产疫苗的成本也随之降低[62]。在需要快速反应和大量疫苗的情况下,使用CFPS系统来生产疫苗有可能为应对大流行病和流行病威胁提供一种快速、可扩展和具有成本效益的手段。此外,作为对抗传染病的有效策略,目前已研发出基于CFPS系统的VLP疫苗[63]。无细胞技术已应用到流感疫苗Inflexal和甲型肝炎疫苗Epaxal两款商业VLP疫苗的生产中,CFPS生产疫苗产业化指日可待[64, 65]。
2.6 疾病诊断无细胞系统避免了利用活细胞生产蛋白质时易受活体细胞代谢调控限制的问题,同时也可以避免利用哺乳动物生产蛋白质时的生物危害和伦理问题。基于无细胞系统的疾病诊断可从生物传感器出发,将无细胞体系冷冻干燥到纸张和其他多孔基质材料中,降低存储、运输成本,提高便携性,适用于诊断和现场检测。该种纸基系统加水即可激活,可在室温下存储,分发过程中不需要冷链,在临床医学、全球健康、工业、研究和教育等多个领域展现出广阔的应用前景[66]。
Pardee等[66, 67]设计了纸基无细胞基因合成网络,用于生产诊断寨卡病毒和埃博拉病毒的高特异性生物传感器,大幅降低了开发成本和生产周期。Vezeau等[68]基于无细胞系统设计了针对人体生物标志物的蛋白质结合核糖开关,成功将蛋白质结合的RNA适配体转化为高效的核糖开关传感器。如单体C反应蛋白和白细胞介素-32γ蛋白,这两者因其血清可及性,常作为心血管疾病及炎症反应的生物标志物,来验证该平台在医学诊断中的应用潜力。而RNA适配体可通过设计来识别和结合多种靶蛋白,为低成本、多样化的蛋白质诊断检测的发展提供了一条创新的可靠途径。
核酸检测在感染性疾病及遗传性疾病的早期诊断中具有重要作用,有助于公共卫生应对疫情暴发并提升疾病控制能力。近年来,基于CFPS的核酸检测在诺如病毒检测中备受关注[62, 69]。CFPS检测方法结合了冻干的CFPS与“脚趾(toehold)”开关核糖调节器,利用诺如病毒结合的合成抗体从样本中捕获和富集诺如病毒颗粒,通过等温扩增生成大量诺如病毒RNA;以RNA作为CFPS反应模板,生成能裂解显色底物的β-半乳糖苷酶,进而引发由黄色到紫色的颜色变化,以检测样本中是否存在诺如病毒[69]。该方法能够检测到最低浓度为270 zmol/L的诺如病毒,提供了一种快速、灵敏、低成本的检测方法,也为其他病原体的检测设计提供了参考。此外,基于CFPS的核酸检测还可用于监测肠道微生物群和宿主生物标志物[70, 71]。随着合成生物学的发展,基于CFPS的核酸检测有望变得更加便携和低成本,使其适用于资源有限地区的现场快速检测。
2.7 个性化医疗随着人工智能(artificial intelligence, AI)的迅速发展,在AI辅助药物设计的关键环节中,大数据模型的成熟高度依赖于海量的高质量数据用于模型训练,因此,快速、高通量和自动化的数据积累方案成为必然。CFPS系统凭借操作简便、条件可控、高通量等优势,能够很好地满足AI辅助药物设计中的早期大量筛选需求。AI与CFPS系统的创新结合,为个性化精准医疗的发展开辟了全新路径,例如定制化药物的快速筛选与合成,为患者提供高度精准、量身定制的治疗方案[72]。
随着个性化医疗的不断发展与深化,人工定制细胞用于疾病诊断和治疗已成为可能,能够针对不同疾病类型及个人的多种特定需求提供解决方案。无细胞系统和脂质体的结合研究为构建人造细胞奠定了基础,传统的细胞治疗往往存在细胞致瘤性和免疫原性等风险,而基于无细胞系统开发的人造定制细胞可以有效规避这些潜在风险[73-75]。Adamala等[76]将无细胞系统与脂质体相结合,成功合成了人造类细胞Synells。Aufinger等[77]利用无细胞系统表达了基于凝胶的人工细胞器,为在细胞器水平上编程人造细胞系统提供了新的可能性。虽然人工定制细胞应用于疾病治疗尚有一段距离,但上述研究为利用无细胞系统合成人造类细胞提供了重要参考。
此外,有研究人员提出了个性化治疗疫苗的设计理念,而CFPS技术的快速性和简便性使其能够为患者快速、经济地生产和评估定制疫苗[60, 78]。在疾病诊断方面,通过对患者基因组的分析,基于CFPS的核酸检测可以帮助医生制定个性化的治疗方案,尤其是在肿瘤学、药物选择和剂量调整方面。总的来说,无细胞系统的加入有助于使医疗更加精准、有效,能够显著提高治疗效果。
2.8 天然产物药物研发天然产物是生物医药的重要来源之一[79],其广泛分布于各种生物体(如细菌、真菌和植物)中[80]。传统的天然产物药物筛选方法通常是从原始的生物体中提取纯化或者在实验室环境中异源培养,再分离提取有效成分,这种低通量发酵和筛选的方法限制了该领域的发展[81],亟需新方法来发现新颖天然产物,满足人类对天然产物药物的需求。其中,非核糖体肽(nonribosomal peptides, NRPs)是一类小分子肽类天然产物,因其复杂的结构特征而具有多种生物活性,有着巨大的药用潜力[81]。缬氨霉素(valinomycin)作为一种来源于链霉菌的非核糖体肽类天然产物,具有多重生物活性,如抗真菌、抗病毒、杀虫和抗肿瘤等[3]。Zhuang等[82]利用基于大肠杆菌的CFPS系统成功表达了来源于链霉菌的缬氨霉素合成酶Vlm1 (370 kDa)和Vlm2 (284 kDa),证明了大肠杆菌CFPS系统可以合成像缬氨霉素合成酶这样分子量巨大的非核糖体肽合成酶(nonribosomal peptide synthetase, NRPS);同时,首次实现了缬氨霉素在大肠杆菌CFPS系统中体外异源从头生物合成;通过CFPS系统与无细胞代谢工程(cell-free metabolic engineering, CFME)结合,缬氨霉素产量最终可以达到30 mg/L,为天然产物的研究和工程化提供了一条新途径。核糖体合成和翻译后修饰肽(ribosomally synthesized and post-translationally modified peptides, RiPPs)是另一类重要天然产物,具有多种化学结构和强大的生物活性。由于缺乏表征和生物合成的异源体内表达系统,目前被挖掘到的RiPPs基因簇仍然只是少数。Liu等[83]报道了一种基于无细胞基因表达系统的统一生物催化(UniBioCat)系统,该系统能够在体外构建RiPPs生物合成途径,共表达RiPPs合成中所需的前体肽和修饰酶,并最终合成成熟的RiPPs,这大大简化了该类化合物体内异源表达合成所需的步骤与过程,为研究RiPPs类天然产物提供了高效、快速的方法与平台。
3 结语与展望无细胞生物合成技术作为新兴的多学科交叉技术,是极具应用前景的合成生物学手段,正迅速成为生物医药研究中的重要工具,可应用于药物研发和疾病诊断的各个阶段,如多肽类药物高通量筛选、药物体外合成、疾病预防、体外诊断等。CFPS体系已被成功用于合成包括药物蛋白和药用天然产物等在内的不同类型的目标产物(代表性产物如表 2所示)[84-105]。CFPS系统的开放性赋予其高度的灵活性,使其能够根据不同的研究需求和应用场景,自由选择基因表达的模板。此外,整个反应过程精确可控,在反应的各个阶段可对底物、产物进行优化,在蛋白质合成的各个层面实现可控化,从而实现蛋白质合成的个性化、精确化,更有助于其在生物医学领域的应用。
| 表 2 CFPS系统合成的代表性药物蛋白等目标产物 |
尽管CFPS系统具有高可控、高通量和灵活性高等优点,在实现大规模生产时,如何确保产量、成本效益和一致性仍然是亟待解决的关键问题。目前CFPS系统在小规模实验中能够实现蛋白质的有效合成,但在扩大生产规模时,因反应条件、底物供应、反应体系等多方面的因素,反应难以像小规模实验那样被精准调控,很难维持同样的高产水平,生产成本也进一步增加。此外,质量监管规范的建立也是一项重要挑战。未来CFPS系统有望成为蛋白质生产的重要标准平台,在生物医药领域的应用逐渐向临床转化,相关质量标准和监管法规也需紧跟步伐,确保产品在不同批次间的一致性,以保证所生产蛋白质产品的安全性和有效性。重要的是,应用于CFPS系统的大部分细胞提取物成分复杂,尚未完全解析,一些细胞提取物成分引发的副反应可能会影响合成目标蛋白的活性,因此蛋白活性调控也是重要的研究方向之一。随着不同底盘生物细胞提取物的开发和相关技术的不断进步,CFPS将推动生物医学和生物技术的进一步发展,有望在更多领域的研究和应用中实现重要突破。
| [1] |
Shin J, Jardine P, Noireaux V. Genome replication, synthesis, and assembly of the bacteriophage T7 in a single cell-free reaction. ACS Synth Biol, 2012, 1: 408-13. |
| [2] |
Liu WQ, Zhang L, Chen M, et al. Cell-free protein synthesis: recent advances in bacterial extract sources and expanded applications. Biochem Eng J, 2019, 141: 182-9. |
| [3] |
Huang S, Liu Y, Liu WQ, et al. The nonribosomal peptide valinomycin: from discovery to bioactivity and biosynthesis. Microorganisms, 2021, 9: 780. |
| [4] |
Dondapati SK, Stech M, Zemella A, et al. Cell-free protein synthesis: a promising option for future drug development. BioDrugs, 2020, 34: 327-48. |
| [5] |
Maharjan A, Park JH. Cell-free protein synthesis system: a new frontier for sustainable biotechnology-based products. Biotechnol Appl Biochem, 2023, 70: 2136-49. |
| [6] |
Chiba CH, Knirsch MC, Azzoni AR, et al. Cell-free protein synthesis: advances on production process for biopharmaceuticals and immunobiological products. Biotechniques, 2021, 70: 126-33. |
| [7] |
Kwon YC, Jewett MC. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Sci Rep, 2015, 5: 8663. |
| [8] |
Hodgman CE, Jewett MC. Optimized extract preparation methods and reaction conditions for improved yeast cell-free protein synthesis. Biotechnol Bioeng, 2013, 110: 2643-54. |
| [9] |
Xu H, Yang C, Tian X, et al. Regulatory part engineering for high-yield protein synthesis in an all-Streptomyces-based cell-free expression system. ACS Synth Biol, 2022, 11: 570-8. |
| [10] |
Takai K, Sawasaki T, Endo Y. Practical cell-free protein synthesis system using purified wheat embryos. Nat Protoc, 2010, 5: 227-38. |
| [11] |
Schweet R, Lamfrom H, Allen E. The synthesis of hemoglobin in a cell-free system. Proc Natl Acad Sci U S A, 1958, 44: 1029-35. |
| [12] |
Ezure T, Suzuki T, Higashide S, et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog, 2006, 22: 1570-7. |
| [13] |
Mikami S, Masutani M, Sonenberg N, et al. An efficient mammalian cell-free translation system supplemented with translation factors. Protein Expr Purif, 2006, 46: 348-57. |
| [14] |
Yue K, Chen J, Li Y, et al. Advancing synthetic biology through cell-free protein synthesis. Comput Struct Biotechnol J, 2023, 21: 2899-908. |
| [15] |
Gregorio NE, Levine MZ, Oza JP. A user' s guide to cell-free protein synthesis. Methods Protoc, 2019, 2: 24. |
| [16] |
Zemella A, Thoring L, Hoffmeister C, et al. Cell-free protein synthesis: pros and cons of prokaryotic and eukaryotic systems. Chembiochem, 2015, 16: 2420-31. |
| [17] |
Quast RB, Sonnabend A, Stech M, et al. High-yield cell-free synthesis of human EGFR by IRES-mediated protein translation in a continuous exchange cell-free reaction format. Sci Rep, 2016, 26: 30399. |
| [18] |
Sheng J, Lei S, Yuan L, et al. Cell-free protein synthesis of norovirus virus-like particles. RSC Adv, 2017, 7: 28837-40. |
| [19] |
Merk H, Gless C, Maertens B, et al. Cell-free synthesis of functional and endotoxin-free antibody Fab fragments by translocation into microsomes. Biotechniques, 2012, 53: 153-60. |
| [20] |
Tinafar A, Jaenes K, Pardee K. Synthetic biology goes cell-free. BMC Biol, 2019, 17: 64. |
| [21] |
Zawada JF, Yin G, Steiner AR, et al. Microscale to manufacturing scale-up of cell-free cytokine production——a new approach for shortening protein production development timelines. Biotechnol Bioeng, 2011, 108: 1570-8. |
| [22] |
Georgi V, Georgi L, Blechert M, et al. On-chip automation of cell-free protein synthesis: new opportunities due to a novel reaction mode. Lab Chip, 2016, 16: 269-81. |
| [23] |
Lu Y. Cell-free synthetic biology: engineering in an open world. Synth Syst Biotechnol, 2017, 2: 23-7. |
| [24] |
Orth JHC, Schorch B, Boundy S, et al. Cell-free synthesis and characterization of a novel cytotoxic pierisin-like protein from the cabbage butterfly Pieris rapae. Toxicon, 2011, 57: 199-207. |
| [25] |
Gurramkonda C, Rao A, Borhani S, et al. Improving the recombinant human erythropoietin glycosylation using microsome supplementation in CHO cell-free system. Biotechnol Bioeng, 2018, 115: 1253-64. |
| [26] |
Dondapati SK, Pietruschka G, Thoring L, et al. Cell-free synthesis of human toll-like receptor 9 (TLR9): optimization of synthesis conditions and functional analysis. PLoS One, 2019, 14: e0215897. |
| [27] |
Thoring L, Wüstenhagen DA, Borowiak M, et al. Cell-free systems based on CHO cell lysates: optimization strategies, synthesis of " difficult-to-express" proteins and future perspectives. PLoS One, 2016, 11: e0163670. |
| [28] |
Quast RB, Mrusek D, Hoffmeister C, et al. Cotranslational incorporation of non-standard amino acids using cell-free protein synthesis. FEBS Lett, 2015, 589: 1703-12. |
| [29] |
Martin RW, Des Soye BJ, Kwon YC, et al. Cell-free protein synthesis from genomically recoded bacteria enables multisite incorporation of noncanonical amino acids. Nat Commun, 2018, 9: 1203. |
| [30] |
Papasarantos I, Klimentzou P, Koutrafouri V, et al. Solid-phase synthesis of a biotin derivative and its application to the development of anti-biotin antibodies. Appl Biochem Biotechnol, 2010, 162: 221-32. |
| [31] |
Yanagisawa T, Takahashi M, Mukai T, et al. Multiple site-specific installations of Nε-monomethyl-L-lysine into histone proteins by cell-based and cell-free protein synthesis. Chembiochem, 2014, 15: 1830-8. |
| [32] |
Lu RM, Hwang YC, Liu IJ, et al. Development of therapeutic antibodies for the treatment of diseases. J Biomed Sci, 2020, 27: 1. |
| [33] |
Hunt AC, Vogeli B, Hassan AO, et al. A rapid cell-free expression and screening platform for antibody discovery. Nat Commun, 2023, 14: 3897. |
| [34] |
Kobayashi N and Oyama H. Antibody engineering toward high-sensitivity high-throughput immunosensing of small molecules. Analyst, 2011, 136: 642-51. |
| [35] |
Venisnik KM, Olafsen T, Gambhir SS, et al. Fusion of Gaussia luciferase to an engineered anti-carcinoembryonic antigen (CEA) antibody for in vivo optical imaging. Mol Imaging Biol, 2007, 9: 267-77. |
| [36] |
Patel KG, Ng PP, Kuo CC, et al. Cell-free production of Gaussia princeps luciferase——antibody fragment bioconjugates for ex vivo detection of tumor cells. Biochem Biophys Res Commun, 2009, 390: 971-6. |
| [37] |
Muyldermans S. Nanobodies: natural single-domain antibodies. Annu Rev Biochem, 2013, 82: 775-97. |
| [38] |
Muyldermans S. A guide to: generation and design of nanobodies. FEBS J, 2021, 288: 2084-102. |
| [39] |
Flajnik MF, Deschacht N, Muyldermans S. A case of convergence: why did a simple alternative to canonical antibodies arise in sharks and camels? PLoS Biol, 2011, 9: e1001120
|
| [40] |
Chen X, Gentili M, Hacohen N, et al. A cell-free nanobody engineering platform rapidly generates SARS-CoV-2 neutralizing nanobodies. Nat Commun, 2021, 12: 5506. |
| [41] |
Zimmerman ES, Heibeck TH, Gill A, et al. Production of site-specific antibody-drug conjugates using optimized non-natural amino acids in a cell-free expression system. Bioconjug Chem, 2014, 25: 351-61. |
| [42] |
Nirenberg MW, Matthaei JH. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci U S A, 1961, 47: 1588-602
|
| [43] |
Salehi AS, Smith MT, Bennett AM, et al. Cell-free protein synthesis of a cytotoxic cancer therapeutic: onconase production and a just-add-water cell-free system. Biotechnol J, 2016, 11: 274-81. |
| [44] |
Meng Y, Yang M, Liu W, et al. Cell-free expression of a therapeutic protein serratiopeptidase. Molecules, 2023, 28: 3132. |
| [45] |
Zemella A, Thoring L, Hoffmeister C, et al. Cell-free protein synthesis as a novel tool for directed glycoengine-ering of active erythropoietin. Sci Rep, 2018, 8: 8514. |
| [46] |
Guo J, Zhou A, Sun X, et al. Immunogenicity of a virus-like-particle vaccine containing multiple antigenic epitopes of Toxoplasma gondii against acute and chronic toxoplasmosis in mice. Front Immunol, 2019, 10: 592. |
| [47] |
Bachmann MF, Rohrer UH, Kundig TM, et al. The influence of antigen organization on B cell responsiveness. Science, 1993, 262: 1448-51. |
| [48] |
Spice AJ, Aw R, Bracewell DG, et al. Synthesis and assembly of hepatitis B virus-like particles in a Pichia pastoris cell-free system. Front Bioeng Biotechnol, 2020, 8: 72. |
| [49] |
Bundy BC, Franciszkowicz MJ, Swartz JR. Escherichia coli-based cell-free synthesis of virus-like particles. Biotechnol Bioeng, 2008, 100: 28-37. |
| [50] |
Ludgate L, Liu K, Luckenbaugh L, et al. Cell-free hepatitis B virus capsid assembly dependent on the core protein C-terminal domain and regulated by phosphorylation. J Virol, 2016, 90: 5830-44. |
| [51] |
Wang X, Liu J, Zheng Y, et al. An optimized yeast cell-free system: sufficient for translation of human papillomavirus 58 L1 mRNA and assembly of virus-like particles. J Biosci Bioeng, 2008, 106: 8-15. |
| [52] |
Lu Y, Welsh JP, Chan W, et al. Escherichia coli-based cell free production of flagellin and ordered flagellin display on virus-like particles. Biotechnol Bioeng, 2013, 110: 2073-85. |
| [53] |
Lu Y, Chan W, Ko BY, et al. Assessing sequence plasticity of a virus-like nanoparticle by evolution toward a versatile scaffold for vaccines and drug delivery. Proc Natl Acad Sci U S A, 2015, 112: 12360-5. |
| [54] |
Plotkin S. History of vaccination. Proc Natl Acad Sci U S A, 2014, 111: 12283-7. |
| [55] |
Kobayashi T, Nakamura Y, Mikami S, et al. Synthesis of encephalomyocarditis virus in a cell-free system: from DNA to RNA virus in one tube. Biotechnol Lett, 2012, 34: 67-73. |
| [56] |
Colant N, Melinek B, Frank S, et al. Escherichia coli-based cell-free protein synthesis for iterative design of tandem-core virus-like particles. Vaccines, 2021, 9: 193. |
| [57] |
Lu Y, Welsh JP, Swartz JR. Production and stabilization of the trimeric influenza hemagglutinin stem domain for potentially broadly protective influenza vaccines. Proc Natl Acad Sci U S A, 2014, 111: 125-30. |
| [58] |
Hu VT, Kamat NP. Cell-free protein synthesis systems for vaccine design and production. Curr Opin Biotechnol, 2023, 79: 102888. |
| [59] |
Ranji Charna A, Des Soye BJ, Ntai I, et al. An efficient cell-free protein synthesis platform for producing proteins with pyrrolysine-based noncanonical amino acids. Biotechnol J, 2022, 17: e2200096. |
| [60] |
Kanter G, Yang J, Voloshin A, et al. Cell-free production of scFv fusion proteins: an efficient approach for personalized lymphoma vaccines. Blood, 2007, 109: 3393-9. |
| [61] |
Huang N, Shimomura E, Yin G, et al. Immunization with cell-free-generated vaccine protects from Porphyromonas gingivalis-induced alveolar bone loss. J Clin Periodontol, 2019, 46: 197-205. |
| [62] |
Ma D, Shen L, Wu K, et al. Low-cost detection of norovirus using paper-based cell-free systems and synbody-based viral enrichment. Synth Biol, 2018, 3: ysy018. |
| [63] |
Hadj Hassine I, Ben M' hadheb M, Almalki MA, et al. Virus-like particles as powerful vaccination strategy against human viruses. Rev Med Virol, 2024, 34: e2498. |
| [64] |
Herzog C, Hartmann K, Kunzi V, et al. Eleven years of inflexal V-a virosomal adjuvanted influenza vaccine. Vaccines, 2009, 27: 4381-7. |
| [65] |
Bovier PA. Recent advances with a virosomal hepatitis a vaccine. Expert Opin Biol Ther, 2008, 8: 1177-85. |
| [66] |
Pardee K, Green AA, Ferrante T, et al. Paper-based synthetic gene networks. Cell, 2014, 159: 940-54. |
| [67] |
Pardee K, Green AA, Takahashi MK, et al. Rapid, low-cost detection of Zika virus using programmable biomolecular components. Cell, 2016, 165: 1255-66. |
| [68] |
Vezeau GE, Gadila LR, Salis HM. Automated design of protein-binding riboswitches for sensing human biomarkers in a cell-free expression system. Nat Commun, 2023, 14: 2416. |
| [69] |
Wu K, Green AA. Detection of norovirus using paper-based cell-free systems. Methods Mol Biol, 2022, 2433: 375-90. |
| [70] |
Takahashi MK, Tan X, Dy AJ, et al. A low-cost paper-based synthetic biology platform for analyzing gut microbiota and host biomarkers. Nat Commun, 2018, 9: 3347. |
| [71] |
Takahashi MK, Tan X, Dy AJ. Cell-free paper-based analysis of gut microbiota and host biomarkers. Methods Mol Biol, 2022, 2433: 351-74. |
| [72] |
马武仁, 弓孟春, 戴辉, 等. 以ChatGPT为代表的大语言模型在临床医学中的应用综述. 医学信息学杂志, 2023, 44: 9-17. |
| [73] |
Fang RH, Hu CM, Luk BT, et al. Cancer cell membrane-coated nanoparticles for anticancer vaccination and drug delivery. Nano Lett, 2014, 14: 2181-8. |
| [74] |
Fang RH, Kroll AV, Zhang L. Nanoparticle-based manipulation of antigen-presenting cells for cancer immunotherapy. Small, 2015, 11: 5483-96. |
| [75] |
薛帅, 于袁欢, 叶海峰. 人工定制细胞与精准医疗的最新研究进展. 生命科学, 2019, 31: 402-12. |
| [76] |
Adamala KP, Martin-Alarcon DA, Guthrie-Honea KR, et al. Engineering genetic circuit interactions within and between synthetic minimal cells. Nat Chem, 2017, 9: 431-9. |
| [77] |
Aufinger L, Simmel FC. Artificial gel-based organelles for spatial organization of cell-free gene expression reactions. Angew Chem Int Ed, 2018, 57: 17245-8. |
| [78] |
Yang J, Kanter G, Voloshin A, et al. Rapid expression of vaccine proteins for B-cell lymphoma in a cell-free system. Biotechnol Bioeng, 2005, 89: 503-11. |
| [79] |
Newman DJ, Cragg GM. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod, 2020, 83: 770-803. |
| [80] |
Montalban-Lopez M, Scott TA, Ramesh S, et al. New developments in RiPP discovery, enzymology and engineering. Nat Prod Rep, 2021, 38: 130-239. |
| [81] |
Smanski MJ, Zhou H, Claesen J, et al. Synthetic biology to access and expand nature' s chemical diversity. Nat Rev Microbiol, 2016, 14: 135-49. |
| [82] |
Zhuang L, Huang S, Liu WQ, et al. Total in vitro biosynthesis of the nonribosomal macrolactone peptide valinomycin. Metab Eng, 2020, 60: 37-44. |
| [83] |
Liu WQ, Ji X, Ba F, et al. Cell-free biosynthesis and engineering of ribosomally synthesized lanthipeptides. Nat Commun, 2024, 15: 4336. |
| [84] |
Martin RW, Majewska NI, Chen CX, et al. Development of a CHO-based cell-free platform for synthesis of active monoclonal antibodies. ACS Synth Biol, 2017, 6: 1370-9. |
| [85] |
Murakami S, Matsumoto R, Kanamori T. Constructive approach for synthesis of a functional IgG using a reconstituted cell-free protein synthesis system. Sci Rep, 2019, 9: 671. |
| [86] |
Groff D, Armstrong S, Rivers PJ, et al. Engineering toward a bacterial " endoplasmic reticulum" for the rapid expression of immunoglobulin proteins. MAbs, 2014, 6: 671-8. |
| [87] |
Groff D, Carlos NA, Chen R, et al. Development of an E. coli strain for cell-free ADC manufacturing. Biotechnol Bioeng, 2022, 119: 162-75
|
| [88] |
Hershewe JM, Warfel KF, Iyer SM, et al. Improving cell-free glycoprotein synthesis by characterizing and enriching native membrane vesicles. Nat Commun, 2021, 12: 2363. |
| [89] |
Zichel R, Mimran A, Keren A, et al. Efficacy of a potential trivalent vaccine based on Hc fragments of botulinum toxins A, B, and E produced in a cell-free expression system. Clin Vaccine Immunol, 2010, 17: 784-92. |
| [90] |
Li J, Zhang L, Liu WQ. Cell-free synthetic biology for in vitro biosynthesis of pharmaceutical natural products. Synth Syst Biotechnol, 2018, 3: 83-9. |
| [91] |
Liu WQ, Wu C, Jewett MC, et al. Cell-free protein synthesis enables one-pot cascade biotransformation in an aqueous-organic biphasic system. Biotechnol Bioeng, 2020, 117: 4001-8. |
| [92] |
Feng J, Yang C, Zhao Z, et al. Application of cell-free protein synthesis system for the biosynthesis of L-theanine. ACS Synth Biol, 2021, 10: 620-31. |
| [93] |
Yang C, Liu Y, Liu WQ, et al. Designing modular cell-free systems for tunable biotransformation of L-phenylalanine to aromatic compounds. Front Bioeng Biotechnol, 2021, 9: 730663. |
| [94] |
Ji X, Liu WQ, Li J. Recent advances in applying cell-free systems for high-value and complex natural product biosynthesis. Curr Opin Microbiol, 2022, 67: 102142. |
| [95] |
Tian X, Liu WQ, Xu H, et al. Cell-free expression of NO synthase and P450 enzyme for the biosynthesis of an unnatural amino acid L-4-nitrotryptophan. Synth Syst Biotechnol, 2022, 7: 775-83. |
| [96] |
Moore SJ, Lai HE, Li J, et al. Streptomyces cell-free systems for natrual product discovery and engineering. Nat Prod Rep, 2023, 40: 228-36. |
| [97] |
刘晚秋, 季向阳, 许慧玲, 等. 限制性内切酶的无细胞快速制备研究. 合成生物学, 2023, 4: 840-51. DOI:10.12211/2096-8280.2022-048 |
| [98] |
Liu Y, Liu WQ, Huang S, et al. Cell-free metabolic engineering enables selective biotransformation of fatty acids to value-added chemicals. Metab Eng Commun, 2023, 16: e00217. |
| [99] |
Chen Y, Liu WQ, Zheng X, et al. Cell-free biosynthesis of lysine-derived unnatural amino acids with chloro, alkene, and alkyne groups. ACS Synth Biol, 2023, 12: 1349-57. |
| [100] |
Zhang R, Liu WQ, Ling S, et al. Combining cell-free expression and multifactor optimization for enhanced biosynthesis of cinnamyl alcohol. J Agric Food Chem, 2023, 71: 8551-7. |
| [101] |
Zhang Y, Liu WQ, Li J. Constructing an artificial short route for cell-free biosynthesis of the phenethylisoquinoline scaffold. Synth Syst Biotechnol, 2023, 8: 610-7. |
| [102] |
Lu H, Ouyang J, Liu WQ, et al. Enzyme-polymer-conjugate-based pickering emulsions for cell-free expression and cascade biotransformation. Angew Chem Int Ed, 2023, 62: e202312906. |
| [103] |
Liu Y, Huang S, Liu WQ, et al. An in vitro hybrid biocatalytic system enabled by a combination of surface-dispalyed, purified, and cell-free expressed enzymes. ACS Synth Biol, 2024, 13: 1434-41. |
| [104] |
Ma GL, Liu WQ, Huang F, et al. An enzymatic oxidation cascade converts δ-thiolactone anthracene to anthraquinone in the biosynthesis of anthraquinone-fused enediynes. JACS Au, 2024, 4: 2925-35. |
| [105] |
Lu Y, Zheng X, Liu WQ, et al. Chitin-functionalized cell-free system enables sustainable biocatalysis and gene expression. ACS Sustainable Chem Eng, 2024, 12: 18219-30. |
 2025, Vol. 37
2025, Vol. 37 

 李健,上海科技大学物质科学与技术学院研究员、博士生导师。2013年毕业于德国柏林工业大学,获得自然科学博士学位(Dr. rer. nat.);2013—2017年在美国西北大学从事博士后研究。目前担任中国生物工程学会青年工作委员会委员、上海市生物工程学会理事、上海市生物工程学会合成生物学专业委员会委员。主要研究方向是无细胞合成生物学、天然产物生物合成、生物催化与转化,以通讯作者(含共同)在Nature Communications、Cell Systems、Angewandte Chemie International Edition、Nucleic Acids Research、Metabolic Engineering、ACS Synthetic Biology等期刊发表论文80余篇,申请或授权国内外相关专利9项。主持科技部重点研发项目课题、国家自然科学基金面上项目、上海市浦江人才计划、上海市科委自然科学基金项目等多项科研项目。
李健,上海科技大学物质科学与技术学院研究员、博士生导师。2013年毕业于德国柏林工业大学,获得自然科学博士学位(Dr. rer. nat.);2013—2017年在美国西北大学从事博士后研究。目前担任中国生物工程学会青年工作委员会委员、上海市生物工程学会理事、上海市生物工程学会合成生物学专业委员会委员。主要研究方向是无细胞合成生物学、天然产物生物合成、生物催化与转化,以通讯作者(含共同)在Nature Communications、Cell Systems、Angewandte Chemie International Edition、Nucleic Acids Research、Metabolic Engineering、ACS Synthetic Biology等期刊发表论文80余篇,申请或授权国内外相关专利9项。主持科技部重点研发项目课题、国家自然科学基金面上项目、上海市浦江人才计划、上海市科委自然科学基金项目等多项科研项目。