合成生物学是一个快速发展的领域,结合了生物学、工程学和计算机科学的原理,旨在设计和构建具有新功能的生物系统。合成基因线路是指利用合成生物学的方法,对生物元件进行重新设计与组装,以创造出能够在活细胞中执行特定生物功能的系统。随着基础生物学研究的发展,更多的生物分子功能和基因表达调控机制被揭示,为合成基因线路的构建提供了潜在生物元件和设计指导。特别是哺乳动物细胞中基因表达调控机制的深入了解,以及CRISPR等基因编辑工具的快速发展,使合成基因线路在基础生物学研究、生命健康和医疗等领域表现出广泛的应用前景。合成基因线路的设计思路大致可分为两种:一种是设计出具有特定且相对独立于宿主细胞本身功能的基因线路,此时宿主细胞一般仅作为基因线路的承载者;另一种基因线路可以和宿主细胞进行良好互动,感知细胞内的生物分子信号并做出响应,从而对宿主细胞的生物功能进行调节。
RNA是细胞中一类非常重要的生物分子,其分为编码RNA和非编码RNA (ncRNA)两大类。编码RNA主要指信使RNA (mRNA),它们负责将遗传信息从DNA转录到蛋白质合成的过程。非编码RNA的种类较多,包括微小RNA (miRNA)、长非编码RNA (lncRNA)和环状RNA (circRNA)等。非编码RNA虽然不直接编码蛋白质,但其在多种生物过程中同样发挥着重要的调控作用[1-4]。例如,miRNA通过与靶mRNA结合,抑制其翻译或促进其降解,从而在基因表达调控中发挥重要作用[5, 6]。2024年诺贝尔生理学或医学奖颁给了两位科学家,以表彰他们发现miRNA及其在转录后基因调控中的作用。由于RNA本身具有重要生物学功能,并且很多RNA分子在不同细胞类型或疾病状态中的表达具有特异性,近年来RNA分子响应的基因线路设计越来越受到重视。这种类型基因线路的功能受宿主细胞自身表达的内源性RNA调节,根据目标RNA分子信号的表达情况,驱动信号输出从而实现特定的生物功能。
RNA响应的基因线路大致由三个模块组成(图 1)。(1)信号输入:通过整合基因元件感知细胞中特定RNA分子信号,例如人工插入目标miRNA的识别位点到设计区域,用于感知特定细胞中的内源靶miRNA信号;(2)信号处理:通过设计的基因线路对RNA信号的输入进行处理,如利用两个癌细胞特异性miRNA分别调控转录抑制蛋白的表达,两个miRNA同时存在时下游报告基因才能被激活,实现“与门”的功能;(3)信号输出:设计不同类型和功能的感兴趣基因作为输出信号从而实现特定的生物功能,如肿瘤细胞选择性杀伤。本文根据构建策略的不同分类,介绍了近年来发展的RNA分子响应基因线路并探讨了其生物医学应用,为以后理性设计RNA响应的基因线路提供新思路。
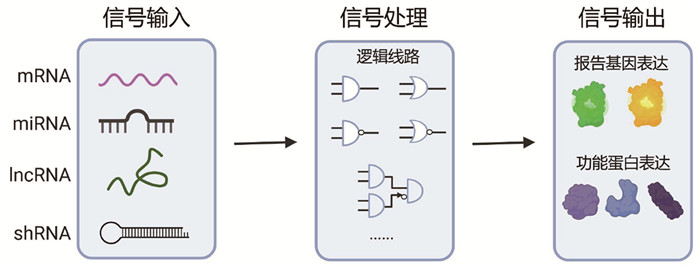
|
图 1 RNA分子响应的基因线路组成 |
CRISPR-Cas系统是一种强大的遗传操作工具,在基因调控、碱基编辑、表观遗传修饰和核酸成像等方面均展现出了极大应用潜力。CRISPR-Cas系统由引导RNA (gRNA)和Cas蛋白组成,其中gRNA可以是单个分子,也可以是两个独立的部分。在II型CRISPR系统如CRISPR-Cas9系统中,天然的gRNA由CRISPR RNA (crRNA)和反式激活CRISPR RNA (tracrRNA)两个部分组成,而在V型CRISPR系统如CRISPR/Cas12a系统中,其gRNA则仅由一条crRNA组成。
近年来,在CRISPR-Cas系统和合成生物学蓬勃发展的背景下,基于CRISPR-Cas系统构建的RNA响应基因线路不断被报道。根据所设计感应RNA分子的CRISPR组分不同,这些基因线路构建策略可分为三种:(1)构建RNA响应的功能性gRNA生成;(2)构建RNA响应的Cas蛋白开关;(3) CRISPR相关蛋白酶介导的蛋白剪切。这些基于CRISPR-Cas系统构建的基因线路,已被用于感应细胞内多种RNA (例如mRNA、miRNA和lncRNA),并可做出响应驱动信号输出,从而实现特定的生物功能。
1.1 设计RNA响应的功能性gRNA生成通过设计RNA激活的gRNA策略,很多不同RNA响应的CRISPR-Cas线路被设计出来用于特定RNA检测[7, 8]。这种构建策略工作模式如下:在没有目标RNA时,不产生功能性的gRNA,因此CRISPR系统不被激活;在有目标RNA时,生成功能性gRNA,CRISPR系统被激活,并根据设定驱动信号输出。这种基因线路的设计策略主要包括以下3种(图 2):(1) RNA响应的分裂型gRNA重组(图 2A);(2)支点开关调控gRNA生成(图 2B);(3) miRNA切割介导的gRNA生成(图 2C)。
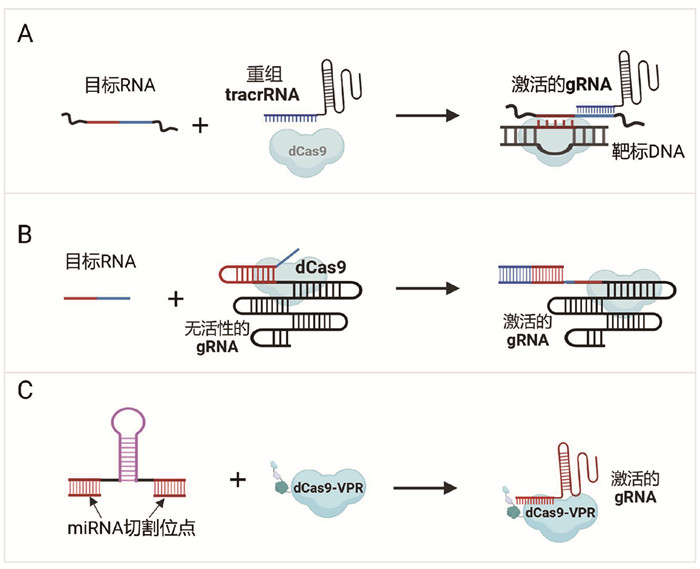
|
图 2 RNA响应的功能性gRNA生成的三种设计原理示意图 |
在CRISPR-Cas9系统中,天然的gRNA由crRNA和tracrRNA两部分组成,其中crRNA中包含识别目标位点的序列以及与tracrRNA配对的区域,当crRNA和tracrRNA配对时可以激活CRISPR-Cas9系统。近期研究表明,crRNA和tracrRNA的配对区序列可以被其他任意序列替代,并且人工设计的crRNA-tracrRNA对表现出了很好的正交性[9],这为构建RNA响应的功能性gRNA生成提供了理论基础。利用这种原理,Liu等[10]设计了一种tracrRNA,其能够响应大肠杆菌中的合成或天然RNA。在该CRISPR系统中,目标RNAs发挥类似crRNA的功能,促进功能性gRNA生成,通过进一步与核酸内切酶缺陷型Cas9 (dCas9)组装,触发GFP报告基因表达。此外,通过设计正反馈基因线路,这种基于tracrRNA的传感器对目标小rRNA或mRNA的响应表现出高达52.2倍的变化。在另外一项研究中,同样基于tracrRNA构建了一种RNA响应的基因线路[11]。不同的是,设计的tracrRNA通过与Cas9切口酶(nCas9)碱基编辑器进行组装,从而在感知内源性RNA后对合成的DNA靶标进行碱基编辑,使目标RNA的转录信息存储在编辑后的DNA中。这种通过基因编码的方式记录RNA转录本信息的方法被命名为TIGER (Transcribed RNAs Inferred by Genetically Encoded Records)。TIGER不仅能够精确测量某个基因的表达水平,还能够区分单核苷酸变异和同时记录多个RNA转录本信息。
然而,上述两种方法[10, 11]都只在原核细胞中显示出强大的RNA检测能力,尚未在真核细胞中被证明可行。Lin等[12]提出了一种自组装诱导的RNA线路,通过设计一种对目标RNA响应的重编程crRNA-tracrRNA双链来控制CRISPR功能。目标RNA和crRNA-tracrRNA形成三元复合体,促进crRNA-tracrRNA组装并激活dCas9,进而调控下游基因表达。该基因线路被证明在原核细胞和真核细胞中均能够响应目标RNA,但其在HEK293T细胞中表现出的检测灵敏度比在大肠杆菌中低。
1.1.2 支点开关调控gRNA生成上述几种构建方法均是利用分裂gRNA型重组的策略进行基因线路构建的。除此之外,基于链置换原理的支点开关也被广泛应用于构建条件型gRNA。较早的一项研究利用这种支点开关策略,通过结合CRISPR-Cas9系统,实现在体外以单碱基分辨率检测寨卡病毒[13]。之后,越来越多研究将链置换反应用于条件型gRNA构建,从而构建出多种RNA响应的基因线路。在这种设计中,gRNA的间隔序列(spacer sequence)[14-20]或支架区(scaffold region)[21-25]被设计为支点区域,只有目标RNA和支点区域发生链置换反应后才能形成功能性gRNA。在近期的一项研究中,基于链置换反应的功能性gRNA生成策略不仅被证明在原核细胞和真核细胞中均有效,而且可以响应多种RNA靶标,包括sRNA、miRNA和mRNA[25]。
然而,为了保证gRNA功能能够被目标RNA恢复,上述支点开关序列在设计上存在一些限制,因此理论上无法实现对任意感兴趣目标RNA的检测。有趣的是,通过设计一种自抑制的gRNA和靶RNA感应支点区,在理论上实现了RNA靶标与支点开关区域的相互作用没有序列限制[23]。在这项研究中,gRNA的5'端融合了两个不同且不重叠的结构域:一个结构域的碱基对与gRNA重复从而阻断Cas12a识别所需的发夹的形成,另一个结构域能够和靶RNA杂交,引起gRNA的重折叠和随后的功能性gRNA形成。这种设计极大扩大了可检测RNA序列的范围,但其表现出了较严重的信号泄漏问题,因此降低了检测的准确性和传感的动态范围。
1.1.3 miRNA切割介导的gRNA生成和以上构建策略不同,miRNA介导的切割也被用于功能性gRNA生成的调控[15, 16, 26]。在这种策略中,gRNA由聚合酶II转录,同时在gRNA上下游插入目标miRNA的识别位点。在没有目标miRNA的时候,转录本中的5'端帽子结构和3'端PolyA尾结构抑制gRNA功能;当存在目标miRNA时,目标miRNA会对靶标序列进行切割释放功能性gRNA,从而激活CRISPR功能并驱动下游报告基因表达,实现对目标miRNA分子的检测。例如,Wang等[26]利用这种miRNA介导的sgRNA释放策略和dCas9-VPR驱动红色荧光蛋白基因表达,实现了活细胞中miRNA表达变化的检测,并利用这套系统来监测干细胞的分化状态。有意思的是,另外一项研究通过利用miRNA介导的sgRNA释放策略和mRNA介导的链置换反应,设计出了一种响应目标miRNA和mRNA的“与门”基因线路,实现同时检测miRNA和mRNA[16]。这种新颖的“与门”设计被成功用于以RNA响应的方式诱导泛素体(ubiquibody)的表达,进而触发蛋白质降解[16]。
1.2 设计RNA响应的Cas蛋白开关在CRISPR-Cas系统中,除了gRNA之外,Cas蛋白是CRISPR-Cas系统发挥功能的另一必要组成部分。通过设计RNA响应的Cas蛋白开关,一些RNA响应的基因线路系统也被开发出来。这些基因线路的设计策略包括利用miRNA和分裂核酶来调控Cas蛋白这两种方式。
1.2.1 利用miRNA抑制Cas蛋白表达或活性通过利用miRNA介导的基因表达抑制机制,一些RNA响应的CRISPR-Cas系统也被构建出来用于RNA分子检测。在这种设计思路中,miRNA可以被设计为调控Cas9蛋白[27]或者抗Cas蛋白AcrllA4 [28, 29]表达(图 3A、B),通过RNA诱导沉默复合体(RISC)机制沉默Cas9蛋白或AcrllA4蛋白编码基因,从而调控CRISPR功能,进而实现对目标miRNA的表达检测。这种构建策略不仅能够用于活细胞水平miRNA检测[27-30],在小鼠体内也被证明同样有效[30]。此外,与直接利用miRNA调控Cas蛋白或抗Cas蛋白表达策略不同,He等[31]利用miRNA来抑制突变的tdMCP (突变的tdMCP竞争性抑制MS2-VPR与tdMCP结合)的表达,进而促进MS2-VPR与dCas9-tdMCP相互作用,从而通过激活下游报告基因来显示目标miRNA的表达(图 3C)。基于miR-21在HCT116细胞中高表达的特点,他们成功利用该系统分离出细胞混合物中的HCT116细胞[31]。
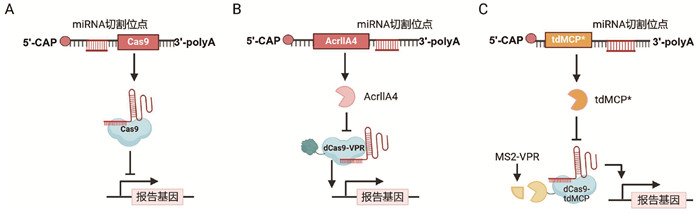
|
图 3 利用miRNA抑制Cas蛋白表达或活性的三种设计原理图 |
除了利用miRNA引发的RISC调控机制之外,最近的一项研究则采用了一种完全不同的方法。该研究创新性设计了一种分裂核酶系统,将RNA检测与dCas9蛋白输出连接起来,实现了对目标RNA分子的检测[32]。在这项设计中,完整的dCas9 mRNA被含有互补靶标RNA序列的内含子分开,当目标RNA与内含子序列配对后,形成模板组装分裂的核酶,引发反式剪接反应,产生功能完整的dCas9蛋白,进而对下游GFP报告基因进行调控。然而,该系统在没有目标RNA的情况下仍然存在泄漏活性,说明这种设计在阻止dCas9表达方面并不完全有效。此外,这种设计只在原核细胞中进行了验证,尚没有研究表明其在真核细胞中的有效性。
1.3 基于CRISPR相关蛋白酶构建的基因线路在CRISPR系统中,除了有Cas9或Cas12a等核酸酶之外,还存在一类CRISPR系统相关的蛋白酶[33, 34]。Cas7-11是一种III-E亚型CRISPR系统,由4个Cas7和1个Cas11结构域融合形成一个大的效应蛋白[35, 36]。Csx29是一种蛋白酶,可与Cas7-11形成功能复合物。这些CRISPR相关的蛋白酶也被成功用于构建RNA响应的基因线路。
在之前的两项研究中,研究者们基于Cas7-11-Csx29-Csx30蛋白酶系统,设计构建了一种RNA响应的蛋白酶活化和底物切割体系[37, 38]。在该系统中,Cas7-11-crRNA-Csx29核糖核蛋白复合物在与靶RNA结合后引起Csx29的构象变化,催化辅助蛋白Csx30的裂解。利用这一机制,将荧光蛋白[37]或重组酶[38]与底物Csx30蛋白融合,可以检测细菌和哺乳动物细胞中的靶标RNA (图 4A-B)。在这种设计中,预期是仅当与目标RNA结合时,Csx29切割Csx30激活下游报告基因表达。然而,由于系统灵敏度问题以及较为严重的泄漏问题,这两种RNA检测系统对靶标RNA的响应分别只有3倍和15倍的动态范围。此外,这种类型的RNA检测技术仅在合成RNA中被证明可行。
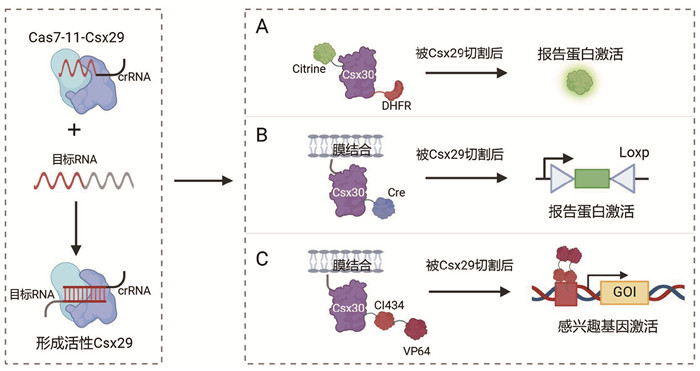
|
图 4 基于Cas7-11-Csx29-Csx30蛋白酶系统构建基因线路的原理图 |
近期,同样基于Cas7-11-Csx29-Csx30蛋白酶系统,人们构建出了一种不同的高分辨率RNA感应及响应基因线路[39]。在这项研究中,底物蛋白Csx30一端和细胞膜定位序列融合,另一端和效应蛋白dCas9-VP64融合;靶标RNA激活Cas7-11复合物中Csx29蛋白酶并切割Csx30,从而使效应蛋白dCas9-VR64从细胞膜释放,激活下游感兴趣基因(GOI)的表达(图 4C)。与之前的两项研究不同,这项研究证实了该基因线路不仅可以用于检测内源性靶标RNA,而且还可以用于检测RNA点突变,且检测的突变信号变化倍数高至94倍。这些研究说明了CRISPR相关蛋白酶系统在构建合成基因线路及RNA检测上的适用性。在未来研究中,基于crRNA的高度可编程性,Cas7-11-Csx29-Csx30蛋白酶系统在构建更复杂的基因线路上仍然具有潜在价值。
2 基于ADAR脱氨酶构建的RNA分子响应线路作用于RNA的腺苷脱氨酶(ADAR)以转录后腺苷(A)到肌苷(I)编辑的方式调节基因表达,在多种生理和病理过程中发挥重要作用[40, 41]。在哺乳动物细胞中,ADAR蛋白家族包含ADAR1、ADAR2和ADAR3三个成员,其中ADAR1和ADAR2具有脱氨酶活性,而ADAR3无催化活性。ADAR以结构依赖的方式作用于双链RNA (dsRNA),并催化A碱基发生脱氨反应生成I碱基(A-to-I编辑)。由于碱基I和鸟苷(G)之间的结构相似性,在mRNA翻译时碱基I会被细胞机制识别为碱基G,从而实现遗传信息的重写[42]。ADAR的这种RNA编辑功能被证明在治疗遗传性疾病,如杜氏肌营养不良症[43]、Hurler综合征[44]和Rett综合征[45]上具有应用潜力。
近年来,ADAR介导的A-to-I编辑被成功用于构建几种RNA响应型基因线路系统[46-50]。在这些基因线路中,目标RNA分子能够诱导生成可被ADAR编辑的双链RNA底物。双链RNA底物中的设计位点被ADAR编辑后能够驱动下游基因表达,从而实现对目标RNA分子的检测或表达变化响应。根据双链RNA底物中的两条RNA分子来源不同,这些基因线路的设计策略分为基于线性RNA的分子间双链底物和基于发夹RNA的分子内双链底物两种(图 5)。
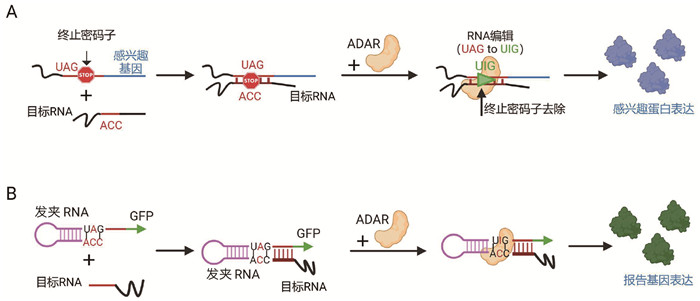
|
图 5 基于ADAR的RNA响应型基因线路的两种不同设计原理图 |
利用线性RNA分子间形成ADAR双链RNA底物的设计思路,一些RNA响应的基因线路系统被成功开发。在这些基因线路中,目标RNA分子能够和人工设计序列互补形成双链RNA。利用这种设计思路,一系列基于ADAR介导的A-to-I编辑的RNA分子感应及响应基因线路被报道,如CellREADR[46]、RADARS[47]、RADAR[48]和DART VADAR[49]等。在这些基因线路设计中,感兴趣基因上游通常会引入终止密码子UAG,使其翻译取决于内源性RNA转录物与感兴趣基因上游序列的结合及之后ADAR介导的终止密码子编辑(图 5A)。其中,RADARS感应器能够检测到表达水平低至13个TPM (transcripts per million)的内源性RNA转录本,并能够实现277倍的荧光素酶、荧光蛋白或重组酶等感兴趣基因的激活。
除了能够响应RNA转录本外,基于ADAR构建的RNA感应系统还能够区分疾病相关RNA转录本序列的改变,如点突变和融合[48]。此外,这类系统如RADAR可以被改造成“或门”和“与门”等逻辑线路,实现对两种RNA分子的检测。RADAR也被证明是一个独立发挥功能的系统,能够在哺乳动物及植物等不同生物类型中发挥作用[48]。而CellREADR被证实能够记录和控制小鼠特定类型的神经元,突出了该类系统在基于RNA的动物体内细胞监测和编辑的应用潜力。
2.2 基于发夹RNA的分子内ADAR底物设计在上述基于ADAR构建的RNA响应线路中,一种含UAG的线性感应区域与细胞内靶标RNA反向互补形成分子间dsRNA后即成为ADAR的编辑底物。然而,这种线性分子间底物设计存在一些固有的限制,如对RNA分子中的UAG-CCA错配中心序列的高度依赖性限制了目标序列的选择。
与之前的报道不同,最近的一项研究报道了一种发夹RNA传感器,它利用新机制产生分子内dsRNA底物,用于有效的ADAR招募和编辑,并将其应用于检测各种细胞内分子,包括mRNA、小分子和蛋白质[50]。在这项研究中,RNA感应器序列被设计为含UAG的RNA发夹结构,不能被ADAR编辑,只有细胞内靶标RNA分子存在时才能使其成为ADAR的底物(图 5B)。这种设计极大地增加了靶标RNA序列的可选择性,理论上能够实现对任意mRNA转录本的检测。此外,该RNA传感器还具有识别RNA单核苷酸突变的能力。更有意思的是,将相应RNA适配体整合到发夹RNA传感器中,通过简单的构象改变来编程发夹RNA的激活,实现了对活细胞中主要能量供应分子ATP和转录因子核因子κB (NF-κB)的检测。
3 其他类型RNA响应的基因线路除了上面基于CRISPR系统和ADAR脱氨酶构建的RNA分子响应线路外,合成生物学和生物改造技术的快速发展还提供了其他一些策略用于构建RNA响应基因线路。例如,基于RNA稳定的荧光蛋白策略、基于RNA响应的支点开关和基于miRNA的负反馈调控等几种设计策略,这些设计策略同样为以后理性设计RNA响应的基因线路及其应用提供了新方法和新思路。
3.1 基于RNA稳定的荧光蛋白策略荧光蛋白是合成基因线路中常用的信号输出报告分子,经常被用于检测细胞内RNA分子。有意思的是,通过构建一种RNA稳定的荧光蛋白系统,成功实现了外源性和内源性mRNA分子的表达检测[51, 52]。在这种系统中,一种包含蛋白降解子(degron)序列和Tat肽(一种RNA结合肽)的双功能肽和荧光蛋白融合,在反式激活反应元件(TAR)RNA发夹与Tat结合时才会阻止蛋白降解子募集蛋白水解所需的蛋白酶体。
例如,在Wu等[51]的设计中,将外源性β-actin和TAR进行融合表达,从而利用该系统实现了活细胞水平β-actin mRNA成像(图 6A)。而Zhou等[52]重新设计了一种RNA感应模块,目标RNA通过诱导RNA感应模块的构象变化,形成能与Tat肽作用的发夹结构,从而稳定降解子融合的荧光蛋白。这种设计被证明能够用于活细胞中内源性survivin mRNA丰度和动态的高对比度成像。此外,通过使用不同荧光蛋白可以将这种系统扩展成多色成像RNA,而通过采用分裂荧光蛋白策略可以设计成信号放大器。该RNA传感器还被进一步用于lncRNA MALAT-1及其在有丝分裂过程中的易位动力学成像[52]。
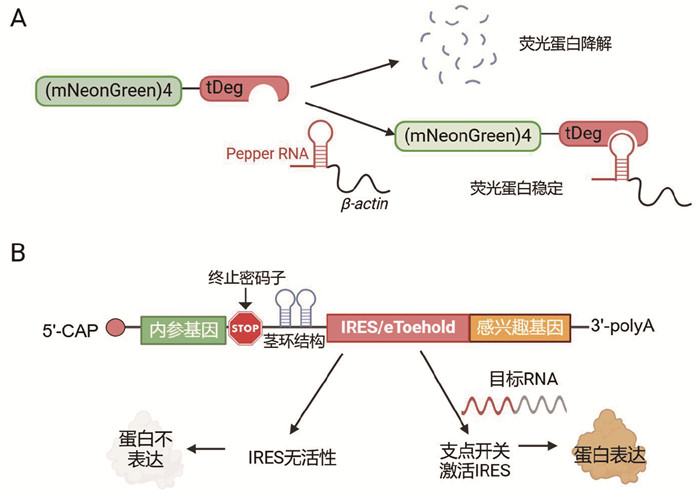
|
图 6 RNA稳定的荧光蛋白和RNA响应的支点开关设计原理图 |
支点开关是合成生物学系统中常用的一类基因开关,常常被用于不同类型的基因线路设计[53, 54]。早期的支点开关系统常常被应用于体外或原核生物基因表达调控[55, 56]。2021年,Zhao等[57]设计了一种基于内部核糖体进入位点(IRES)的真核生物支点开关(eToehold)系统,并将其用于哺乳动物细胞RNA转录本检测。在该系统中,缺乏目标RNA时,eToehold形成抑制环抑制IRES活性从而抑制下游基因表达;当目标RNA存在时,通过链置换反应打开支点开关,激活IRES介导的下游基因翻译(图 6B)。该系统被证实可以根据外源性或内源性RNA转录物的存在来识别病毒感染状态、基因表达信息和细胞类型。然而,eToehold响应外源性转录本时报告基因最高只显示出16倍的表达变化,对不同内源性RNA转录本的检测最高只显示9倍变化。因此,这种设计在响应灵敏度上还需要进一步提高。
3.3 基于miRNA的负反馈调控miRNA是一类长度约为19~25 nt的非编码RNA,具有抑制基因表达的属性。利用miRNA与其靶标RNA完全互补配对会导致靶标RNA的切割这一天然机制,一系列miRNA响应的基因线路被成功开发[58-63]。在这些基因线路中,可以将miRNA识别位点插入到一些基因表达抑制子的非编码区域,通过负反馈调控机制构建miRNA响应的基因线路系统。例如,通过将miRNA靶位点插入到转录抑制子LacI的非编码区域,并在报告基因上游插入LacI结合位点,便可以通过反馈调节机制实现对目标miRNA的检测与成像[59]。
除了调控转录抑制子之外,miRNA还可以被人工设计为调控一些在转录后水平发挥作用的抑制子,从而构建出不同基因线路用于miRNA检测或细胞类型特异性基因激活[60, 64-68]。例如,将miRNA的识别位点插入一种内切核糖核酸酶非编码区域,同时在感兴趣基因上游插入内切核糖核酸酶的特异性识别序列(图 7A),能够实现对目标miRNA的检测和响应[60]。此外,为了降低基因线路系统的泄漏表达,一种偶联转录和翻译调控的双重调控策略也被成功用于miRNA检测和功能基因调控(图 7B)[61]。然而,这些基因线路大多只能响应一个或两个miRNA分子,设计能够识别多个miRNA分子靶点的复杂基因线路在未来疾病诊断和治疗领域将更具意义。
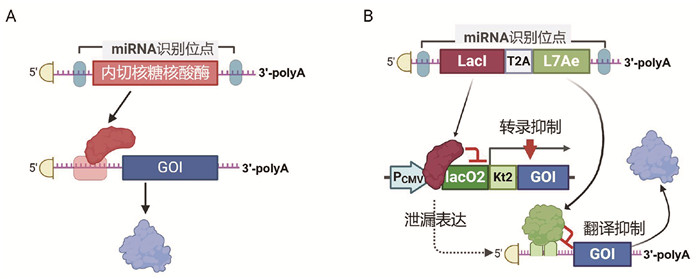
|
图 7 两种基于miRNA负反馈机制的RNA基因线路设计原理图 |
基于合成生物学方法构建的RNA响应型基因线路在生物医学领域展现出广阔的应用潜力,其核心在于通过动态感知细胞内RNA信号并触发特定基因表达,实现对细胞行为的精准调控。通过整合RNA感知与基因调控功能,这些RNA响应型基因线路不仅能够为基础生物学研究提供新工具,也为生物医学提供了从基础研究到临床转化的创新工具。
4.1 作为生物学基础研究的新工具作为最基本的用途,RNA响应的基因线路可以输出报告基因,从而实现对目标RNA分子的检测与成像。与传统RNA检测方法(如RNA探针)相比,基于合成基因线路构建的RNA分子检测策略在检测灵敏度、特异性和适用性等各方面都显著提高。因此,这些RNA分子响应的基因线路能够为生物学基础研究提供新工具。
例如,在一种mRNA响应的基因线路中,内源性RNA的转录信息被记录在编辑后的DNA中从而存储在活细胞中,这样能够建立当前细胞状态和过去转录状态之间的联系,揭示了单个细胞内复杂的细胞反应[11]。更有意义的是,基于miRNA细胞类型特异性表达的特征,一些miRNA响应的人工基因线路系统被用于细胞类型特异性调控CRISPR活性。这些基因线路能够用于细胞类型特异性基因组调控,从而更充分发挥CRISPR系统的应用潜力,为生物学研究提供新工具。利用miRNA响应的CRISPR系统进行筛选被认为是鉴定干细胞中miRNA活性异质性的一种有力工具[26]。
miRNA响应的基因线路系统还能被应用到细胞分类中。Xie等[69]最先将miRNA结合位点人工插入基因线路中的基因非编码区域,并基于逻辑线路设计从细胞混合物中识别HeLa细胞。目前这种以miRNA作为信号输入的人工基因线路已经被广泛应用到各种细胞分类问题上,如特定神经细胞的标记、分化细胞与多能干细胞的分选等[46, 66, 70, 71]。此外,通过监测特定miRNA或mRNA的表达变化,可实现动态监测干细胞分化过程[26, 39]。这些成果证明了miRNA在构建合成基因线路系统中的优势,也为开发更多人工可控的基因线路提供了基础。
4.2 在生物传感与疾病诊断中的应用RNA响应型基因线路在疾病诊断与生物传感中的应用核心在于将RNA信号转化为可检测的输出(如荧光信号)。例如在无细胞体系中,通过设计一种基于体外转录RNA的传感荧光报告基因,可用于在大流行性流感环境中快速灵敏地检测严重急性呼吸综合征冠状病毒2 (SARS-CoV2)。在原核生物中,通过设计用于检测大肠杆菌中各种环境反应基因的转录水平,可用于环境污染物(如砷、锌、铅、铜和镉)的现场评估[10]。在真核生物中,RNA响应的基因线路用于RNA分子检测在疾病诊断上同样具有重要意义。例如,很多RNA类型(如mRNA、miRNA、lncRNA)被认为是多种人类疾病的分子标志物,而RNA响应的基因线路可以很好地用于检测这些RNA分子的表达变化。此外,一些基因线路被证明不仅可以用于检测真核细胞RNA,并且能够区分其疾病相关突变体[39, 48, 50]。因此,基于合成基因线路的RNA分子检测技术在疾病诊断领和生物传感域具有很大应用潜力。
4.3 在精准医疗和细胞治疗领域的应用潜力RNA分子响应的基因线路可以输出具有生物功能的蛋白,从而实现设定生物功能,这为精准医疗和细胞治疗领域提供了新方法和新策略。例如,在一些响应细胞类型特异性表达的miRNA或者mRNA基因线路中,通过人工设计驱动毒性基因表达,可以实现细胞类型特异性肿瘤细胞杀伤[47, 61, 68, 70]。在最近报道的一种RNA-IN/RNA-OUT基因线路中,其能够高灵敏度地识别肿瘤相关基因的单碱基突变(如KRAS、TP53等),并触发效应模块激活凋亡信号通路,实现肿瘤细胞的特异性杀伤。该基因线路可将单点突变的检测灵敏度提升94倍,并成功应用于胰腺癌和肝癌细胞的靶向治疗[39]。该基因线路还被证明能够响应内源性RNA信号以激活内源性孕酮生物合成途径[39],这将赋予细胞新的生物功能,例如构建“智能细胞”用于激素替代治疗。此外,RNA响应的基因线路能够和CRISPR-Cas系统结合,从而在精准医疗领域发挥更大作用。
总的来说,RNA分子响应的基因线路理论上可以被设计成响应任意内源性mRNA或miRNA等编码或非编码RNA分子,并根据需求调控其他任意外源性或内源性基因表达,实现特定生物功能。这种灵活和可调节的基因线路在复杂的环境中具有巨大的应用潜力,例如在未来的临床治疗中,它可以根据疾病的变化情况控制效应分子的状态。因此,RNA响应的基因线路在未来精准医疗和细胞治疗领域中具有极大应用潜力。
5 总结和展望随着合成生物学的快速发展,近年来RNA分子响应的基因线路不断被报道(表 1),并在基础生物学研究和未来个性化医疗中表现出极大应用潜力。这些RNA响应的基因线路系统具有良好的模块化和可编程性,使系统能够精确地执行指定任务。特别是基于CRISPR系统构建的RNA分子响应线路为天然转录物和感兴趣基因之间建立人工连接提供了前所未有的机会。此外,RNA分子响应的基因线路也为构建其他更为复杂的基因线路提供了广泛的可能性。
| 表 1 RNA响应的基因线路设计策略 |
在已报道的RNA响应的基因线路中,大多只对一个或少数几个特定内源RNA分子进行了验证。然而,由于不同内源性RNA分子表达水平相差很大,RNA分子响应的基因线路的检测灵敏度和动态范围显得至关重要。目前大多数基于合成基因线路的RNA传感器均以线性的方式存在于细胞内,线性RNA固有的不稳定而快速降解性质可能是影响检测灵敏度和动态范围的因素之一。近期一项研究表明,可以仅用一个环状RNA分子就实现多个内源性miRNA的检测[70],这种设计方法为RNA响应基因线路设计提供了一种新思路。为了使RNA响应的基因线路更具应用性,未来仍有必要针对性开发灵敏度和检测范围更广的基因线路。此外,目前RNA响应的基因线路大多是以单一种类的一个或两个RNA分子作为信号输入。在临床诊断或治疗中,往往需要根据多个RNA分子靶点来提高检测结果的可靠性和提出个性化治疗方案,这就需要优化构建多个级联线路,并保持良好的灵敏度和可观察的信号输出。
| [1] |
Mattick JS, Amaral PP, Carninci P, et al. Long non-coding RNAs: definitions, functions, challenges and recom-mendations. Nat Rev Mol Cell Biol, 2023, 24: 430-47. |
| [2] |
Ali SA, Peffers MJ, Ormseth MJ, et al. The non-coding RNA interactome in joint health and disease. Nat Rev Rheumatol, 2021, 17: 692-705. |
| [3] |
Butz H, Patócs A, Igaz P. Circulating non-coding RNA biomarkers of endocrine tumours. Nat Rev Endocrinol, 2024, 20: 600-14. |
| [4] |
Nemeth K, Bayraktar R, Ferracin M, et al. Non-coding RNAs in disease: from mechanisms to therapeutics. Nat Rev Genet, 2024, 25: 211-32. |
| [5] |
Shang R, Lee S, Senavirathne G, et al. microRNAs in action: biogenesis, function and regulation. Nat Rev Genet, 2023, 24: 816-33. |
| [6] |
Wang X, He Y, Mackowiak B, et al. MicroRNAs as regulators, biomarkers and therapeutic targets in liver diseases. Gut, 2021, 70: 784-95. |
| [7] |
Liu Y, Liu W, Wang B. Engineering CRISPR guide RNAs for programmable RNA sensors. Biochem Soc Trans, 2023, 51: 2061-70. |
| [8] |
Galizi R, Duncan J N, Rostain W, et al. Engineered RNA-interacting CRISPR guide RNAs for genetic sensing and diagnostics. CRISPR J, 2020, 3: 398-408. |
| [9] |
Jiao C, Sharma S, Dugar G, et al. Noncanonical crRNAs derived from host transcripts enable multiplexable RNA detection by Cas9. Science, 2021, 372: 941-48. |
| [10] |
Liu Y, Pinto F, Wan X, et al. Reprogrammed tracrRNAs enable repurposing of RNAs as crRNAs and sequence-specific RNA biosensors. Nat Commun, 2022, 13: 1937. |
| [11] |
Jiao C, Reckstadt C, König F, et al. RNA recording in single bacterial cells using reprogrammed tracrRNAs. Nat Biotechnol, 2023, 41: 1107-16. |
| [12] |
Lin J, Wang WJ, Wang Y, et al. Building endogenous gene connections through RNA self-assembly controlled CRISPR/Cas9 function. J Am Chem Soc, 2021, 143: 19834-43. |
| [13] |
Pardee K, Green AA, Takahashi MK, et al. Rapid, low-cost detection of Zika virus using programmable biomolecular components. Cell, 2016, 165: 1255-66. |
| [14] |
Siu KH, Chen W. Riboregulated toehold-gated gRNA for programmable CRISPR-Cas9 function. Nat Chem Biol, 2019, 15: 217-20. |
| [15] |
Oesinghaus L, Simmel FC. Controlling gene expression in mammalian cells using multiplexed conditional guide RNAs for Cas12a*. Angew Chem Int Ed Engl, 2021, 60: 23894-902. |
| [16] |
Hunt VM, Chen W. A microRNA-gated thgRNA platform for multiplexed activation of gene expression in mammalian cells. Chem Commun (Camb), 2022, 58: 6215-18. |
| [17] |
Lin J, Liu Y, Lai P, et al. Conditional guide RNA through two intermediate hairpins for programmable CRISPR/Cas9 function: building regulatory connections between endogenous RNA expressions. Nucleic Acids Res, 2020, 48: 11773-84. |
| [18] |
Ying ZM, Wang F, Chu X, et al. Activatable CRISPR transcriptional circuits generate functional RNA for mRNA sensing and silencing. Angew Chem Int Ed Engl, 2020, 59: 18599-604. |
| [19] |
Hunt VM, Chen W. Deciphering the design rules of toehold-gated sgRNA for conditional activation of gene expression and protein degradation in mammalian cells. ACS Synth Biol, 2022, 11: 397-405. |
| [20] |
Pelea O, Mayes S, Ferry Q R V, et al. Specific modulation of CRISPR transcriptional activators through RNA-sensing guide RNAs in mammalian cells and zebrafish embryos. Elife, 2024. |
| [21] |
Li Y, Teng X, Zhang K, et al. RNA strand displacement responsive CRISPR/Cas9 system for mRNA sensing. Anal Chem, 2019, 91: 3989-96. |
| [22] |
Oesinghaus L, Simmel FC. Switching the activity of Cas12a using guide RNA strand displacement circuits. Nat Commun, 2019, 10: 2092. |
| [23] |
Collins SP, Rostain W, Liao C, et al. Sequence-independent RNA sensing and DNA targeting by a split domain CRISPR-Cas12a gRNA switch. Nucleic Acids Res, 2021, 49: 2985-99. |
| [24] |
Hochrein LM, Li H, Pierce NA. High-performance allosteric conditional guide RNAs for mammalian cell-selective regulation of CRISPR/Cas. ACS Synth Biol, 2021, 10: 964-71. |
| [25] |
Wang WJ, Lin J, Wu CQ, et al. Establishing artificial gene connections through RNA displacement-assembly-controlled CRISPR/Cas9 function. Nucleic Acids Res, 2023, 51: 7691-703. |
| [26] |
Wang XW, Hu LF, Hao J, et al. A microRNA-inducible CRISPR-Cas9 platform serves as a microRNA sensor and cell-type-specific genome regulation tool. Nat Cell Biol, 2019, 21: 522-30. |
| [27] |
Hirosawa M, Fujita Y, Parr CJC, et al. Cell-type-specific genome editing with a microRNA-responsive CRISPR-Cas9 switch. Nucleic Acids Res, 2017, 45: e118. |
| [28] |
Hirosawa M, Fujita Y, Saito H. Cell-type-specific CRISPR activation with microRNA-responsive AcrllA4 switch. ACS Synth Biol, 2019, 8: 1575-82. |
| [29] |
Hoffmann MD, Aschenbrenner S, Grosse S, et al. Cell-specific CRISPR-Cas9 activation by microRNA-dependent expression of anti-CRISPR proteins. Nucleic Acids Res, 2019, 47: e75. |
| [30] |
Lee J, Mou H, Ibraheim R, et al. Tissue-restricted genome editing in vivo specified by microRNA-repressible anti-CRISPR proteins. RNA, 2019, 25: 1421-31. |
| [31] |
He M, Zhou X, Li Z, et al. Programmable transcriptional modulation with a structured RNA-mediated CRISPR-dCas9 complex. J Am Chem Soc, 2022, 144: 12690-7. |
| [32] |
Gambill L, Staubus A, Mo KW, et al. A split ribozyme that links detection of a native RNA to orthogonal protein outputs. Nat Commun, 2023, 14: 543. |
| [33] |
Shmakov SA, Makarova KS, Wolf YI, et al. Systematic prediction of genes functionally linked to CRISPR-Cas systems by gene neighborhood analysis. Proc Natl Acad Sci U S A, 2018, 115: E5307-16. |
| [34] |
Makarova KS, Wolf YI, Iranzo J, et al. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol, 2020, 18: 67-83. |
| [35] |
Özcan A, Krajeski R, Ioannidi E, et al. Programmable RNA targeting with the single-protein CRISPR effector Cas7-11. Nature, 2021, 597: 720-25. |
| [36] |
van Beljouw SPB, Haagsma AC, Rodríguez-Molina A, et al. The gRAMP CRISPR-Cas effector is an RNA endonuclease complexed with a caspase-like peptidase. Science, 2021, 373: 1349-53. |
| [37] |
Kato K, Okazaki S, Schmitt-Ulms C, et al. RNA-triggered protein cleavage and cell growth arrest by the type III-E CRISPR nuclease-protease. Science, 2022, 378: 882-89. |
| [38] |
Strecker J, Demircioglu FE, Li D, et al. RNA-activated protein cleavage with a CRISPR-associated endopeptidase. Science, 2022, 378: 874-81. |
| [39] |
Zhang M, Zhang X, Xu Y, et al. High-resolution and programmable RNA-IN and RNA-OUT genetic circuit in living mammalian cells. Nat Commun, 2024, 15: 8768. |
| [40] |
Eisenberg E, Levanon EY. A-to-I RNA editing-immune protector and transcriptome diversifier. Nat Rev Genet, 2018, 19: 473-90. |
| [41] |
Booth BJ, Nourreddine S, Katrekar D, et al. RNA editing: expanding the potential of RNA therapeutics. Mol Ther, 2023, 31: 1533-49. |
| [42] |
Pfeiffer LS, Stafforst T. Precision RNA base editing with engineered and endogenous effectors. Nat Biotechnol, 2023, 41: 1526-42. |
| [43] |
Wang X, Zhang R, Yang D, et al. Develop a compact RNA base editor by fusing ADAR with engineered EcCas6e. Adv Sci (Weinh), 2023, 10: e2206813. |
| [44] |
Yi Z, Qu L, Tang H, et al. Engineered circular ADAR-recruiting RNAs increase the efficiency and fidelity of RNA editing in vitro and in vivo. Nat Biotechnol, 2022, 40: 946-55. |
| [45] |
Sinnamon JR, Kim SY, Fisk JR, et al. In vivo repair of a protein underlying a neurological disorder by programmable RNA editing. Cell Rep, 2020, 32: 107878. |
| [46] |
Qian Y, Li J, Zhao S, et al. Programmable RNA sensing for cell monitoring and manipulation. Nature, 2022, 610: 713-21. |
| [47] |
Jiang KY, Koob J, Chen XD, et al. Programmable eukaryotic protein synthesis with RNA sensors by harnessing ADAR. Nat Biotechnol, 2023, 41: 698-707. |
| [48] |
Kaseniit KE, Katz N, Kolber NS, et al. Modular, programmable RNA sensing using ADAR editing in living cells. Nat Biotechnol, 2022, 41: 482-87. |
| [49] |
Gayet RV, Ilia K, Razavi S, et al. Autocatalytic base editing for RNA-responsive translational control. Nat Commun, 2023, 14: 1339. |
| [50] |
Qin PP, Chen PR, Tan L, et al. Programming ADAR-recruiting hairpin RNA sensor to detect endogenous molecules. Nucleic Acids Res, 2025, 53: gkae1146. |
| [51] |
Wu J, Zaccara S, Khuperkar D, et al. Live imaging of mRNA using RNA-stabilized fluorogenic proteins. Nat Methods, 2019, 16: 862-65. |
| [52] |
Zhou WJ, Li H, Zhang KK, et al. Genetically encoded sensor enables endogenous RNA imaging with conformation-switching induced fluorogenic proteins. J Am Chem Soc, 2021, 143: 14394-401. |
| [53] |
Yarra SS, Ashok G, Mohan U. "Toehold switches; a foothold for synthetic biology". Biotechnol Bioeng, 2023, 120: 932-52. |
| [54] |
Li S, Zhu L, Lin S, et al. Toehold-mediated biosensors: Types, mechanisms and biosensing strategies. Biosens Bioelectron, 2023, 220: 114922. |
| [55] |
Green AA, Silver PA, Collins JJ, et al. Toehold switches: de-novo-designed regulators of gene expression. Cell, 2014, 159: 925-39. |
| [56] |
Takahashi MK, Tan X, Dy AJ, et al. A low-cost paper-based synthetic biology platform for analyzing gut microbiota and host biomarkers. Nat Commun, 2018, 9: 3347. |
| [57] |
Zhao EM, Mao AS, de Puig H, et al. RNA-responsive elements for eukaryotic translational control. Nat Biotechnol, 2021, 40: 539-45. |
| [58] |
Sano M, Morishita K, Oikawa S, et al. Live-cell imaging of microRNA expression with post-transcriptional feedback control. Mol Ther Nucleic Acids, 2021, 26: 547-56. |
| [59] |
Shu W J, Ma Z, Tian X J, et al. Near-infrared fluorescence imaging of miRNA using a transmembrane polypeptide-based genetic reporter. Small Methods, 2024, 8: e2300990. |
| [60] |
Wang L, Xu W, Zhang S, et al. Sensing and guiding cell-state transitions by using genetically encoded endori-bonuclease-mediated microRNA sensors. Nat Biomed Eng, 2024, 8: 1730-43. |
| [61] |
Shu WJ, Lee K, Ma Z, et al. A dual-regulation inducible switch system for microRNA detection and cell type-specific gene activation. Theranostics, 2023, 13: 2552-61. |
| [62] |
Zhang C, Zhang B, Tang C, et al. A ratiometric gene-switch system for miRNA sensing and gene regulation. Small Methods, 2024, 8: e2301266. |
| [63] |
Shi X, Hu C, Jiang Y, et al. Harnessing PUF-based reporters for noninvasive imaging of the microRNA dynamics in differentiation. Anal Chem, 2023, 95: 4786-94. |
| [64] |
Zhang C, Shi X, Tang C, et al. Engineering synthetic CopT/A-based genetic biosensors for miRNA imaging and functional gene regulation. Anal Chem, 2023, 95: 6748-56. |
| [65] |
Caddle LB, Guo M, Hay BA. A positive readout single transcript reporter for site-specific mRNA cleavage. PeerJ, 2017, 5: e3602. |
| [66] |
Fujita Y, Hirosawa M, Hayashi K, et al. A versatile and robust cell purification system with an RNA-only circuit composed of microRNA-responsive ON and OFF switches. Sci Adv, 2022, 8: eabj1793. |
| [67] |
Matsuura S, Ono H, Kawasaki S, et al. Synthetic RNA-based logic computation in mammalian cells. Nat Commun, 2018, 9: 4847. |
| [68] |
Lin MW, Tseng YW, Shen CC, et al. Synthetic switch-based baculovirus for transgene expression control and selective killing of hepatocellular carcinoma cells. Nucleic Acids Res, 2018, 46: e93. |
| [69] |
Xie Z, Wroblewska L, Prochazka L, et al. Multi-input RNAi-based logic circuit for identification of specific cancer cells. Science, 2011, 333: 1307-11. |
| [70] |
Ning H, Liu G, Li L, et al. Rational design of microRNA-responsive switch for programmable translational control in mammalian cells. Nat Commun, 2023, 14: 7193. |
| [71] |
Miki K, Endo K, Takahashi S, et al. Efficient detection and purification of cell populations using synthetic microRNA switches. Cell Stem Cell, 2015, 16: 699-711. |
 2025, Vol. 37
2025, Vol. 37 

 王福,西安交通大学基础医学院教授、博导,青年拔尖人才A类,陕西省杰出青年基金获得者,福建省“闽江学者”讲座教授,BMC Biology、BMC Cancer等杂志编委,中国光学学会生物医学光子学专委会委员。主要从事生物医学成像及分析传感领域的研究,先后主持包括国家自然科学基金项目(4项),科技部外国专家项目,国家“973计划”二级子课题,陕西省自然科学基金面上、重点、杰青项目,陕西省留学人员科技活动择优资助项目(优秀类,2项)等10余个国家、省部级基金项目。近年来以通讯作者在ACS Nano、Nano Letters、Biosensors and Bioelectronics、Analytical Chemistry等国际主流期刊发表SCI论文50余篇;申请/授权中国发明专利10余项;研究成果以第一完成人获得陕西省高等学校科学技术奖一等奖(2022、2016)等奖项
王福,西安交通大学基础医学院教授、博导,青年拔尖人才A类,陕西省杰出青年基金获得者,福建省“闽江学者”讲座教授,BMC Biology、BMC Cancer等杂志编委,中国光学学会生物医学光子学专委会委员。主要从事生物医学成像及分析传感领域的研究,先后主持包括国家自然科学基金项目(4项),科技部外国专家项目,国家“973计划”二级子课题,陕西省自然科学基金面上、重点、杰青项目,陕西省留学人员科技活动择优资助项目(优秀类,2项)等10余个国家、省部级基金项目。近年来以通讯作者在ACS Nano、Nano Letters、Biosensors and Bioelectronics、Analytical Chemistry等国际主流期刊发表SCI论文50余篇;申请/授权中国发明专利10余项;研究成果以第一完成人获得陕西省高等学校科学技术奖一等奖(2022、2016)等奖项