肠道微生物及肠道状态变化与多种疾病的发生和发展密切相关,包括炎症性肠病、结直肠癌和感染性疾病等[1-3]。早期精确检测这些肠道变化对于疾病的及时干预和症状控制十分重要。传统的内窥镜检查作为侵入性的诊断方法,易对患者造成生理和心理创伤,更重要的是难以实现肠道疾病的早期诊断和瞬态疾病标志物的变化分析[4-5]。尽管体外粪便分析发现某些生物标志物具有指示疾病存在的潜力,例如细胞因子、肠道菌群代谢物等,然而这些标志物通常是动态变化的,且难以可靠地预测疾病进展[6-7]。当前,多种基于粪便组学的研究揭示了肠道疾病与代谢组及微生物组变化之间的关联,但是这种方式具有非常高的技术要求且成本昂贵[8-10]。此外,基于粪便分析的检测方式难以准确反映肠道内的真实情况,也无法实现肠道疾病标志物的原位检测和分析测量。
微生物细胞在漫长进化过程中发展出大量感知胞内外信号的识别元件和基因传感线路,并以此调控自身行为。利用合成生物学技术在新的底盘中移植和重编程这些原始的传感机制,以创造能够感知和响应特定信号的基因工程细菌传感器,为多种肠道疾病的在体检测提供了一种低成本且非侵入性的策略[11-14]。当这些感知特定疾病信号的细菌传感器被部署在哺乳动物肠道内时,它们能够很轻易地探测肠道健康状态并通过人工定制的输出方式进行报告,包括荧光/生物发光信号、显色反应、色素蛋白以及由核酸编辑酶介导的DNA突变等。
近年来,结合生物信息学及多组学分析,新的疾病相关的分子标志物传感元件被不断挖掘。同时,快速扩展的遗传操作工具包,例如基于成簇规则间隔短回文重复序列及其相关蛋白(clustered regularly interspaced short palindromic repeats, CRISPR-Cas)技术,则不断拓展研究者对各种细菌底盘进行基因操作的能力[15-18]。在此背景下,越来越多的基因工程细菌传感器被开发并应用于肠道内特异性疾病生物标志物的检测。本文将介绍用于肠道疾病相关分子标志物检测的细菌传感器设计原理,并重点总结工程细菌传感器在炎症性肠病、结直肠癌、肠道出血、致病菌感染等肠道疾病检测中的研究进展,最后对其未来的研究和应用方向进行了展望。
1 工程细菌传感器识别和检测肠道分子标志物的原理微生物作为一种精巧的微型传感器,能够通过其识别元件和基因传感线路感知、处理并应对多种环境条件,从而调节自身的行为,如生长、代谢、运动和交流。基因工程细菌传感器是经过移植和重新组装信号识别和基因传感线路的完整微生物细胞,旨在感知特定环境信号并执行相应功能。
与传统传感器相比,基因工程细菌传感器具有多项独特优势:成本低廉,可再生,能够进行灵活设计及定制化信号输出,适合在哺乳动物肠道等复杂环境中部署和应用[19-21]。通常,应用于肠道疾病检测的工程细菌传感器主要由三个模块构成:检测胞外疾病信号输入的感应模块、将疾病信号输入整合并转换为自定义下游信号的处理模块、产生可检测信号或执行功能的输出模块(图 1)。
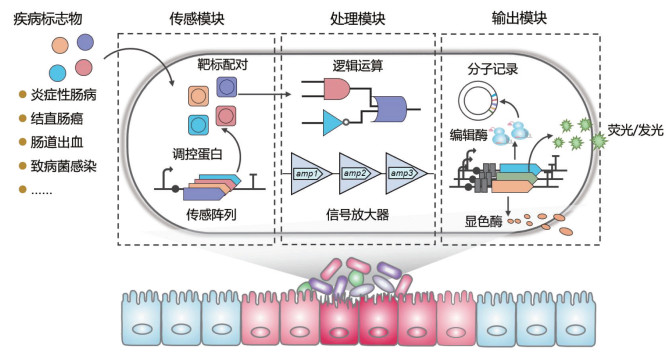
|
图 1 用于肠道疾病标志物检测的工程细菌传感器示意图 |
根据作用模式的不同,原核生物传感系统主要分为单组分传感系统(OCS)和双组分传感系统(TCS)[22]。OCS是最大的细菌信号转导通路家族和最常见的基因传感系统,典型的OCS由一个转录调控因子和相应的诱导型启动子组成。转录调控因子通常包括C端DNA结合结构域和N端传感结构域。转录调控因子的传感结构域与环境刺激分子结合,从而变构调节DNA结合结构域的活性,进而调节输出启动子的转录[23]。根据调控方式的不同,OCS可以进一步细分为阻遏型传感系统和激活型传感系统。
TCS是一类多步信号转导系统。典型的TCS传感器由组氨酸激酶(HK)、响应调节蛋白(RR)及输出启动子组成。HK结构包括N端的传感域、跨膜域和C端的激活域,输入信号的存在导致传感域的构象发生重排,从而增强激酶活性[24-25]。在激活状态下,激活域磷酸化RR中保守的天冬氨酸残基,进而激活靶基因转录[26]。相较于TCS,OCS更简单且元件库更加丰富,但其转录调节因子几乎全部位于细胞质中,因此仅能感知细胞内的输入信号且通常需要利用转运蛋白将胞外刺激分子转运到胞内。TCS的HK可定位于膜上或细胞质中,这使得TCS能够感知细胞外、膜内或细胞内的输入信号。
1.2 工程细菌传感器中的信号处理模块工程细菌传感器的信号处理器模块集成了更为复杂的遗传线路。肠道疾病的发生和发展是一个动态变化的过程,期间多种疾病标志物处于动态变化的过程中,这也导致多种肠道疾病的精确诊断十分困难[12]。通过在细菌底盘中设计布尔逻辑门(例如NAND、NOR)来整合和处理多种疾病信号的输入有望提升诊断的精确性[27-30]。当前正在细菌底盘中开发越来越多的布尔逻辑回路(例如OR、NAND、XOR及XNOR),以使工程细菌传感器具有更高阶的信号传感能力。一个里程碑式的例子是一种可用于自动设计复杂人工基因回路的计算机程序Cello,其利用布尔逻辑门设计电路,这些门技术由基于转录抑制子的NOT/NOR逻辑组成[31]。Cello系统内置丰富的基因元件库,包括启动子、转录调控蛋白等,便于快速构建所需的线路。用户输入所需的功能和特性,例如基因的表达水平、响应条件等,系统会根据需求自动生成可能的基因线路。在利用Cello自动设计的60个基因线路中,有45个能够按预期运行[31]。另一方面,肠道疾病分子标志物通常以较低的浓度存在,因此可以设计信号放大回路以提升可检测的信号强度。基于超灵敏激活因子的高增益转录放大器是一种具有代表性的模块化的级联信号放大策略,其可以提升传感器检测灵敏度及信号输出表达水平,将多个正交放大器串联,可以进一步提高传感性能[32]。
由于许多参与肠道疾病发生发展的分子标志物在排泄前具有局部集中分布、易降解、短暂存在的特征,使其很难在采样的粪便样品中被检测到。相比于传统的体外检测方法,工程细菌传感器具有原位感知和响应瞬时疾病线索的能力,通过在细菌传感器中构建双稳态开关、基因振荡器或基于核酸修饰酶的记录系统则能够长期记忆/记录感知到的信号。双稳态开关基于两个相互抑制的基因(A基因和B基因)进行调控。在初始状态下,A基因对B基因的表达施加抑制作用。当诱导剂1与A基因的表达产物结合时,A基因对B基因的抑制效果减弱;当标志物浓度超过特定阈值时,初始稳态将被完全逆转,形成新的稳态(图 2)[33]。基因振荡器则引入了第三个调控蛋白和信号刺激分子,以实现输出信号的长期规律振荡(图 2)[34]。在工程细菌传感器中引入DNA编辑酶(重组酶、Cas9蛋白、碱基编辑器等),则能够将细菌捕获的外界信息转换为自身DNA的永久突变,即使在信号消失后也能够通过测序技术方便地获取原始信息,这一过程称为分子记录[35-36]。
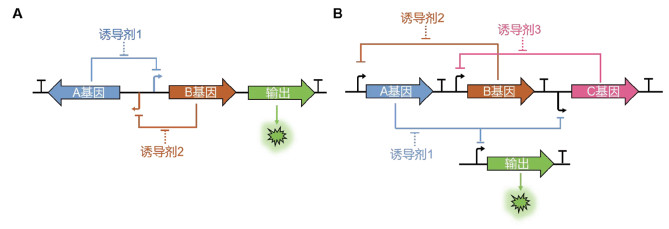
|
图 2 双稳态开关(A)和基因振荡器(B)设计示意图 |
工程细菌传感器设定报告系统表达通常是便于检测的,根据目标应用的不同,可以人为配置报告系统以评估是否存在输入信号(二进制,定性检测),存在输入信号的强度(模拟输出,定量检测)或在一定时间框架内存在的总信号强度(分子记录,量化检测)。常用的终端输出信号包括荧光/生物发光信号、显色反应、色素蛋白以及由DNA编辑酶介导的DNA突变等[37-38]。
2 基因工程细菌传感器检测肠道疾病的应用肠道疾病的及时干预和症状控制在很大程度上依赖于早期精确诊断。随着合成生物学的快速发展,已经针对炎症性肠病、结直肠癌和肠道出血等多种肠道疾病的早期精确诊断开发了一系列基因工程细菌传感器[39-41]。这些活体传感器能够被部署于肠道,通过在肠道内感知特定的疾病分子标志物,精准识别肠道内的病理变化,进而实现疾病的早期原位检测。
2.1 基因工程细菌传感器检测炎症性肠病炎症性肠病(inflammatory bowel disease, IBD)是涉及回肠、结肠和直肠的慢性肠道炎症,主要包括溃疡性结肠炎和克罗恩病[42-44]。IBD的发病率不断上升,如不进行早期干预,可能导致癌症或其他危及生命的并发症。在结肠炎期间,宿主酶将硫酸盐还原菌产生的大量H2S转化为硫代硫酸盐以减轻组织毒性[45-46]。炎症期间产生的活性氧(ROS)则将硫代硫酸盐转化为连四硫酸盐[47]。因此,硫代硫酸盐和连四硫酸盐的水平可能指示结肠炎的存在。鼠伤寒沙门氏菌(Salmonella typhimurium)利用TtrR/ TtrS TCS感知连四硫酸盐作为呼吸链电子受体,从而在发炎的肠道中获得生长优势[48]。Riglar等[49]在小鼠共生大肠杆菌菌株NGF-1中耦合TtrR/TtrS TCS与遗传拨动开关CI/Cro,创造了菌株PAS638。利用β-半乳糖苷酶基因作为报告基因可以方便地检测连四硫酸盐的存在(EC50=0.38~0.85 μmol/L)。该细菌传感器在长达六个月的时间内成功检测到小鼠肠炎模型中升高的连四硫酸盐水平。在发炎区域,硝酸盐含量增加,并且由于病灶部位通常靠近血液,O2水平较高[50],而S. typhimurium来源的TtrR/TtrS TCS受到O2和硝酸盐的抑制[51]。因此,TtrR/TtrS的交叉调控可能会影响其在肠道中的有效性。为此,Daeffler等[52]从Shewanella baltica OS195中鉴定出一种新型的TtrR/TtrS TCS,该系统不受硝酸盐影响且仅受O2的轻微抑制;此外,他们还在Shewanella halifaxensis HAW-EB4中发现了一种名为ThsS/ThsR的新型TCS,这是已知的第一个硫代硫酸盐传感器。作者将这一传感器移植到E. coli Nissle 1917 (EcN)菌株中,以荧光蛋白sfGFP作为报告分子成功构建了硫代硫酸盐响应的工程细菌传感器;同时,开发了一种口服灌胃工程细菌传感器和流式细胞术分析结肠和粪便样本的方法,结果显示小鼠结肠炎可以激活肠道中的硫代硫酸盐传感器。
肠道炎症可以上调宿主诱导型一氧化氮合酶的表达,从而导致一氧化氮(NO)产生增加[53-54],NO在肠道内被迅速转化为硝酸盐[55]。已有研究表明,结肠炎小鼠肠道内的硝酸盐/亚硝酸盐以及NO水平提高[56]。Archer等[57]利用对NO具有高度特异性的调控蛋白NorR开发了一种NO响应的细菌传感器。当NO与NorR结合时,其GAF结构域解除对AAA+结构域的抑制,使其能够结合σ54并启动norV启动子的转录。随后,作者利用NO传感器和重组酶构建了一个复合电路,将fimE重组酶基因置于PnorV的控制下,当传感器检测到NO时,细菌报告子从黄色荧光蛋白切换到青色荧光蛋白。此外,Woo等[58]利用NarX-NarL TCS在EcN中构建了一个对硝酸盐有响应的细菌传感器,并成功检测到葡聚糖硫酸钠(DSS)诱导的结肠炎小鼠体内升高的硝酸盐水平。组织炎症通常会引发宿主代谢变化,导致细胞外酸中毒。Cartwright等[59]构建了一种能够感知pH变化并在酸性条件下发光的工程细菌传感器,以检测由肠道炎症引起的酸中毒。
钙卫蛋白被认为是评估肠道炎症的临床金标准,通常在粪便样品中进行检测,以评估肠道炎症的存在和严重程度[60-62]。最近,Xia等[63]在EcN中发现了一系列对钙卫蛋白有响应的诱导启动子,这些启动子与细菌细胞内锌离子(Zn2+)调节相关,作者利用ykgMO启动子在EcN菌株中创建一种用于检测钙卫蛋白的工程细菌传感器,其对钙卫蛋白的检测下限达到25 µg/g,该浓度可以区分健康/炎症患者。在小鼠结肠炎模型中,研究者证明该细菌传感器在口服给药后可在体内激活;此外,该细菌传感器还可以被用来分析患者的粪便样本,以区分处于活性期或缓解期的IBD患者或无IBD患者。在另一项独立研究中,Zhu等[64]设计了一种利用ykgMO启动子检测钙卫蛋白的细菌传感器,并基于该传感器开发了一种自调节的结肠炎治疗方案。值得注意的是,ykgMO启动子并不能直接感知钙卫蛋白,而是感知由钙卫蛋白引起的锌限制条件[65-66]。大肠杆菌利用一种进化上保守的机制对锌缺乏产生反应。该机制依赖于转录因子Zur,Zur控制ykgM和ykgO基因的表达。当锌浓度足够时,Zur抑制下游基因转录,但当细胞内锌水平降到临界阈值以下时,它就会失去活性从而激活下游基因的表达[67-69]。钙卫蛋白对金属离子(如锌)具有很强的螯合能力,这可以被用来激活Zur下游基因的表达。
捕捉和记录肠道分子的动态变化是一个重大挑战,Zou等[70]在EcN底盘中利用硫代硫酸盐感应的ThsS/ThsR TCS开发了一种智能细菌传感器i-ROBOT,其可以报告并记录肠炎标志物硫代硫酸盐的存在。i-ROBOT利用单碱基编辑器将感知到的生物标志物刺激转化为基因组DNA中的永久性单核苷酸改变,从而导致功能蛋白(如LacZ)的翻译激活,通过简单的平板显色反应即可读取炎症信息,并且这些信息可以无损地传递给后代。此类耦合基因传感器与分子记录的创新策略为捕获和记录肠道内动态变化的疾病标志物提供了新的契机[71-73],这不仅对肠道疾病的精确诊断和监测具有重要意义,也有助于扩大我们对疾病发生发展机制的理解,为其治疗策略的开发提供参考。
2.2 基因工程细菌传感器检测结直肠癌结直肠癌(colorectal cancer, CRC)是指发生在结肠和直肠的恶性肿瘤。根据国际癌症研究机构(IARC)和世界卫生组织(WHO)的统计,CRC是全球第三大最常见的癌症,仅次于乳腺癌和肺癌,同时也是导致癌症相关死亡的第二大原因,并且年轻人群的发病率逐年上升[74]。CRC的早期检测面临诸多挑战,主要是其缺乏特异性症状,并且通常依赖入侵性强且费用昂贵的结肠镜检测方法,使得部分患者难以接受,并且易导致漏诊,尤其是在未进行定期筛查的人群中。因此,开发更为便捷高效的检测手段显得尤为重要。
最近的研究表明,工程细菌传感器可以作为无创检测CRC的新兴工具。多项研究指出,包括EcN和S. typhimurium在内的多种细菌,能够利用肿瘤微环境(TME)中的乏氧、坏死及免疫监视降低等特征,优先在肿瘤部位定植[75]。这一特性为将工程细菌改造为长期监测腺瘤或CRC的平台提供了契机。Gurbatri等[76]开发了一株能够有效定植于CRC腺瘤并持续产生水杨酸盐的工程化EcN,水杨酸盐可通过尿液方便地检测并且其在预防CRC方面具有潜在作用。为合成水杨酸盐的工程细菌,作者设计了一个菌株变体文库,以表达莽草酸盐途径的关键基因,利用该途径将细菌内源性分支酸转化为水杨酸盐。口服给药时,工程细菌传感器能够在CRC腺瘤内部成功定植并产生水杨酸盐。通过检测小鼠尿液中的水杨酸盐含量,可以方便地检测CRC腺瘤的存在。
在粪便样本中检测肿瘤脱落DNA是一种无创诊断CRC的方法,然而胞外DNA在肠道内会被很快分解,因此该方法的检测准确性较低。水平基因转移(HGT)是个体之间的遗传物质交换过程,这在微生物中很常见。贝氏不动杆菌(Acinetobacter baylyi)是一种能够从环境中摄取DNA的自然感受态细菌,其在健康人体内基本没有致病性并且可以定植小鼠肠道[77-78]。基于这种优势,Cooper等[79]在A. baylyi底盘中开发了一种称为靶向CRISPR识别水平基因转移的胞内检测方法(cellular assay for targeted, CRISPR-discriminated horizontal gene transfer, CATCH)。在该方法中,工程细菌传感器被设计用于识别和检测KRAS基因的突变序列KRASG12D,该基因在许多癌症中都会发生突变。当摄取到KRASG12D DNA序列时,细菌传感器能够利用其内源性CRISPR系统将序列进行基因组整合,整合后将激活报告基因,例如卡那霉素抗性基因,以方便检测。由于报告基因是模块化的,因此可以被替换成其他基因以产生感兴趣的输出信号。利用该生物传感器,作者在工程化的原位小鼠CRC模型以及在体外从天然CRC哺乳动物细胞中检测到靶标序列。总之,多项最新的临床前研究表明,工程细菌传感器有望为CRC的在体无创检测提供一种创新性的策略。
2.3 基因工程细菌传感器检测致病菌感染致病菌感染在全球范围内,尤其是在发展中国家,仍然是导致高死亡率的重要原因之一[80]。尽管医学技术不断进步,目前依然缺乏既经济又便捷的检测方法来快速识别致病菌感染,这一局面促使研究人员不断探索新兴技术。利用合成生物学的原理开发基因工程细菌传感器,为致病菌感染,尤其是肠道感染检测提供了一种新的策略。基因工程细菌传感器不仅成本较低,还能够根据特定的致病菌进行设计从而增强检测的特异性和准确性,其在肠道内能够实现对致病菌的快速识别和精确检测,从而为肠道致病菌感染的早期诊疗提供支持。
铜绿假单胞菌(Pseudomonas aeruginosa)能够在呼吸道和胃肠道中定植,尤其对免疫系统缺陷患者造成严重的感染风险[81]。Saeidi等[82]基于铜绿假单胞菌的I型群体感应分子AHL及其调控蛋白LasR开发了一种工程化大肠杆菌传感器,成功实现了对P. aeruginosa的快速检测。Li等[83]开发了一种工程化植物乳杆菌WCSF I以监测金黄色葡萄球菌(Staphylococcus aureus)感染。S. aureus是一种常见的革兰氏阳性致病菌,能够通过其AgrQS群体感应系统产生自诱导肽(AIP)以感知群体密度。基于S. aureus这种特有的群体感应机制,作者在植物乳杆菌(Lactobacillus plantarum) WCSF中构建了一个基于AIP的传感和AgrQS调节系统,从而开发出能够在纳摩尔浓度范围内检测外源AIP的工程化L. plantarum。
霍乱弧菌(Vibrio cholerae)是一种危及生命的胃肠道感染病原菌,若未能及时治疗,可能导致严重脱水,甚至在数小时内致死[84]。Holowko等[85]开发了一种基于V. cholerae群体感应诱导分子CAI-1及CRISPR干扰技术的工程细菌传感器。在缺乏CAI-1的情况下GFP的表达被抑制,而在CAI-1存在时CRISPRi的活性被解除,从而允许GFP的表达。进一步地,Mao等[86]对乳酸乳球菌进行了工程化改造,以特异性检测和报告肠道环境中的V. cholerae。V. cholerae能产生自诱导剂AI-2和CAI-1,并分别通过双组分组氨酸激酶受体LuxQ和CqsS进行特异性检测。作者利用CqsS对CAI-1的特异性结合以及组氨酸激酶受体的模块化特性,设计了一种乳酸乳球菌杂交受体(HR),该受体结合了CqsS的跨膜配体结合结构域与乳酸乳球菌中一种双组分受体NisK的信号转导结构域,从而成功开发出一种能够检测V. cholerae自诱导剂CAI-1的工程细菌传感器。该传感器可以特异性识别肠道中V. cholerae的群体感应信号,并触发一种在粪便样本中易于检测的显色酶基因的表达,从而实现V. cholerae感染的在体便捷检测。
2.4 基因工程细菌传感器检测消化道出血消化道出血是一种常见的临床症状,可能由多种因素导致,包括IBD、CRC及机械损伤等。粪便隐血测试是临床上用于检测消化道出血的常规方法,但其无法准确定位出血病灶[87]。虽然胃肠镜检查能够直接观察到出血部位,但该方法具有侵入性,且无法有效探测小肠区域。研究表明,大约5%的消化道出血病例被归类为不明原因消化道出血(OGIB),其中约75%的OGIB病灶位于小肠,这使得胃肠镜检查在此类病例中的应用受到限制[88-90]。
基因工程细菌传感器的独特功能为及时和精确诊断与定位胃肠道出血提供了一种非侵入式的新策略,尤其是在OGIB的情况下。出血部位的红细胞进入肠道后会很快裂解并释放出血红蛋白/血红素,因此血红素可以作为肠道出血检测的生物标志物。Zou等[91-92]在EcN底盘中开发了一种名为YES601的活菌传感器,用于胃肠道出血的检测和体内定位。YES601由乳酸乳球菌来源的血红素响应的阻遏蛋白HrtR及其诱导启动子、大肠杆菌O157:H7来源的血红素转运蛋白ChuA、一套遗传拨动开关以及精心设计的报告系统构成。YES601利用血红素转运蛋白ChuA摄取胞外血红素,进入胞内的血红素分子与调节蛋白HrtR结合使其发生构象变化从而降低其对下游基因的转录抑制,进而激活下游基因表达。此外,作者对产生发光信号的luxCDABE基因簇进行了彻底的拆分和重组,进一步提升了细菌传感器对血红素的检测能力,最终构建的传感器YES601能够响应低至0.12 ppm的血液,最大诱导比超过4 000倍[91]。利用YES601,作者成功实现了对机械损伤或结肠炎导致的小鼠消化道出血部位的精确诊断和无创定位[92]。此外,生物安全性评估结果表明YES601对小鼠的肝功能、免疫系统以及肠道菌群无显著影响,具有较高的应用安全性。
Tan等[93]基于血红素转运蛋白ChuA、血红素加氧酶-1 (HO1)以及远红外荧光蛋白BDFP 1.6开发了另一种血红素响应的细菌传感器。在该传感器中,进入胞内的血红素与HO1反应生成胆绿素(BV),BDFP1.6可以结合BV并发出远红外荧光信号。利用该传感器,作者成功在体检测了阿司匹林诱导的斑马鱼出血。目前临床上严重依赖胃肠镜来观察消化道出血病灶,难以定位和捕捉小肠的出血点。与这些侵入性技术相比,工程细菌传感器的开发为无创精确定位小肠出血点提供了一种有前景的策略。值得注意的是,由于当前报告分子的深层组织穿透能力有限,此类传感器在肠道疾病中的原位检测应用仍集中于小鼠、斑马鱼等小型动物。
3 基因工程细菌传感器封装及肠道疾病标志物检测应用工程细菌传感器在肠道疾病诊断中的应用前景备受关注,然而,将细菌传感器高效、安全地递送到肠道及病变部位仍然是一个紧迫的挑战。首先,胃酸、消化酶及肠道中的胆盐等不利环境因素可能显著降低细菌的存活率,从而限制活菌抵达胃肠道靶点的数量与活性。其次,细菌表面的抗原及其代谢产物可能引发宿主的免疫反应,导致过度炎症及组织损伤。此外,尽管细菌传感器能够在肠道内对疾病标志物作出原位响应,但其产生的信号往往较弱,无法有效传输至体外,这限制了其在实时监测和病灶定位中的应用效果。
顾臻团队在最近发表的一篇文章中定义了“宏观封装细菌”这一针对活菌疗法的新兴概念和策略[94]。该策略利用毫米至厘米级的材料或装置(水凝胶、微针以及电子胶囊等载体)封装工程活菌药物用于肠道或其他特定人体部位的递送。这种宏观封装策略能够通过物理隔离的方式保护细菌传感器,并防止其与宿主直接接触,提高了生物安全性。此外,经过精心设计的封装系统不会影响细菌传感器捕获与感知疾病分子标志物,甚至能够提高疾病标志物的局部浓度以增强响应。最后,将功能电子元件与活菌传感器结合,有望创造下一代集成细菌设备以实现肠道疾病的实时监测。
3.1 基于水凝胶的工程细菌传感器封装策略海藻酸盐水凝胶是一种具有高度生物相容性的网状结构聚合物材料,可以为细胞提供半透性保护屏障,使它们在与环境隔离的同时能够接收环境刺激[95]。Aghlara-Fotovat等[96]利用海藻酸盐水凝胶来封装感知结肠炎标志物硫代硫酸盐的细菌传感器。在大鼠结肠炎模型的检测应用中,作者证明了该水凝胶胶囊在大鼠肠道内保持形态完整且能够方便地从粪便样品中回收。利用流式细胞仪对回收的胶囊进行荧光检测,结果显示荧光强度与大鼠结肠炎疾病活动指数具有正相关性,表明该系统能够实现大鼠结肠炎的精确诊断。通常,摄入的细菌传感器会在约48 h内从胃肠道清除。为延长传感器的滞留时间并精确控制其在肠道中的位置,Liu等[97]开发了一种磁性水凝胶,该水凝胶通过将钕铁硼微粒掺入聚乙烯醇水凝胶中制备而成。利用该水凝胶封装血红素响应的细菌传感器,能够实现传感器在肠道的精确定位和长时间滞留。
3.2 基于微电子胶囊的工程细菌传感器封装策略将工程细菌传感器与超低功耗微电子技术相结合,有望实现肠道疾病相关分子标志物的原位实时检测和分析,为肠道中发生的动态分子变化提供了探测工具。Mimee等[98]描述了一种可摄入的微型生物电子设备,该设备将血红素响应细菌传感器与超低功耗微电子技术相结合,用于实时监测胃肠道出血。在该系统中,工程菌传感器响应血红素产生光信号,随后微电子设备将该光信号转换为电信号并传输至移动设备,以实现环境刺激的实时检测和读取。最近,Inda-Webb等[99]进一步升级了该电子胶囊,将其尺寸降低至1.4 cm3以符合人体安全摄入的大小。利用该集成系统,作者在猪小肠中监测了连四硫酸盐的存在和变化。尽管将工程细菌传感器与超低功耗微电子技术相结合具有很好的应用潜力和价值,但仍有必要评估其使用成本和患者对于这种新型检测方法的可接受度。
4 总结与展望随着合成生物学的发展,利用基因工程技术在新的底盘中重编程微生物的传感机制,已经开发了多种能够实时监测肠道健康状态的细菌传感器(表 1)。这些传感器可通过定制化的输出方式,报告特定的肠道分子信号,为非侵入性的肠道疾病检测提供了新方案。与内窥镜检查相比,工程细菌传感器提供了更早期且非侵入性的检测方法,从而显著减少了患者的不适感。此外,相较于体外粪便分析,细菌传感器能够在体实时监测肠道内的生物标志物,从而提供更为准确和及时的检测结果。然而,在临床应用之前,工程细菌传感器仍面临诸多重要挑战。未来的研究应集中于解决当前对细菌传感器在人体应用过程中的安全性、疾病诊断的准确性等方面的顾虑,同时需要进一步验证传感器在复杂生物环境中的稳定性和可靠性,以确保其在临床实践中的有效性。
| 表 1 用于检测肠道疾病的代表性基因工程细菌传感器 |
生物安全性是细菌传感器开发和应用中的关键性因素之一,应确保细菌传感器不会对宿主或环境产生负面影响,因此可以开发精准可控的基因传感线路,使其在完成任务后能够自然降解或失活[100]。例如,研究者已经开发了一株具有三个模块的工程菌传感器,它能够检测环境污染物水杨酸并将其降解为龙胆酸,最后在水杨酸耗尽时启动基于CcdB-CcdA的“毒素-抗毒素”自杀回路,使细菌自裂解,避免对环境造成影响[101]。此外,肠道疾病的发生发展是一个涉及多种疾病标志物的复杂过程,这使得依靠单一标志物进行准确诊断和治疗具有挑战性。为了解决这个问题,可以开发具有生物计算能力的“智能细菌传感器”以提高疾病诊断的精确性[102]。例如,AND门可以增强传感器对疾病信号响应的特异性,OR门可以帮助区分不同疾病阶段,NOT门则确保了额外的安全性。
当前适用于细菌传感器识别的疾病分子靶标及感应元件仍十分匮乏,未来的研究可以更多地整合多组学研究(如转录组学、代谢组学、基因组学和微生物组学)来挖掘可用于指示肠道疾病的分子标志物及其感知和响应元件,利用当前快速发展的CRISPR-Cas等基因编辑技术在不同底盘中进行组装和重构,以迅速开发一系列可用于肠道疾病检测的新型工程细菌传感器[16]。
在信号输出和检测方面,当前的传感器大多依赖于从粪便中分离细菌传感器进行分析,而无法准确定位肠道病灶部位。有少数利用依赖于荧光或生物发光的报告系统进行活体成像的报道,然而由于光在组织中的穿透性差、分辨率有限,其信号难以在大型动物或人类中进行原位解析。作为另一种输出,超声波很容易穿透组织,从而能够以百微米级的空间分辨率在几厘米的深度采集图像,该技术的发展使得肠道疾病病灶的原位无创定位成为可能[103-104]。
工程微生物与微电子设备的创新性结合为肠道健康实时监测提供了一种极具潜力的策略[98-99]。研究者已经在工程细菌和电子胶囊设备之间建立了一种双向的光通信语言[105]。精心设计的电子胶囊可以在实验猪的肠道内检测和传输工程细菌传感器产生的微弱生物发光信号,同时允许用户通过远程控制胶囊的LED灯开关以激活工程细菌的光传感元件从而释放治疗药物,进而形成诊疗一体化的闭环。这种“碳基”与“硅基”双向通信语言和工具为肠道疾病的实时监测和调节提供了一种全新的策略。在实际应用前,该系统仍有优化和改进的空间,例如:电子胶囊的器件应进一步集成化以降低尺寸并提升长期续航能力;可以拓展胶囊与细菌传感器间的多信号传输和交流能力;电子胶囊在胃肠道内的可控定位有望进一步提升系统对不同肠道疾病的适用性。
总之,细菌传感器在肠道疾病的检测中的发展潜力巨大。随着研究的深入及生物学、工程学、计算科学和医学等多学科的紧密合作,下一代的工程细菌传感器不仅有望实现肠道疾病的早期无创诊断,还有望为个体化医疗和公共卫生监测提供新的解决方案。
| [1] |
Zhang F, Aschenbrenner D, Yoo JY, et al. The gut mycobiome in health, disease, and clinical applications in association with the gut bacterial microbiome assembly. Lancet Microbe, 2022, 3: e969-83. |
| [2] |
Abdigazy A, Arfan M, Lazzi G, et al. End-to-end design of ingestible electronics. Nat Electron, 2024, 7: 102-18. |
| [3] |
Vos WM, Tilg H, Van Hul M, et al. Gut microbiome and health: mechanistic insights. Gut, 2022, 71: 1020-32. |
| [4] |
Clark-Snustad KD, Lee SD. Diagnosing inflammatory bowel disease and differentiating it from potential mimics. Tech Gastrointest Endosc, 2016, 18: 108-15. |
| [5] |
Kobayashi T, Siegmund B, Le Berre C, et al. Ulcerative colitis. Nat Rev Dis Primers, 2020, 6: 74. |
| [6] |
Tanna T, Ramachanderan R, Platt RJ. Engineered bacteria to report gut function: technologies and implementation. Curr Opin Microbiol, 2021, 59: 24-33. |
| [7] |
Park JH, Peyrin-Biroulet L, Eisenhut M, et al. IBD immunopathogenesis: a comprehensive review of inflammatory molecules. Autoimmun Rev, 2017, 16: 416-26. |
| [8] |
Lynch SV, Pedersen O. The human intestinal microbiome in health and disease. N Engl J Med, 2016, 375: 2369-79. |
| [9] |
Fan Y, Pedersen O. Gut microbiota in human metabolic health and disease. Nat Rev Microbiol, 2021, 19: 55-71. |
| [10] |
Lavelle A, Sokol H. Gut microbiota-derived metabolites as key actors in inflammatory bowel disease. Nat Rev Gastroenterol Hepatol, 2020, 17: 223-37. |
| [11] |
Hwang IY. Engineering live bacterial therapeutics to treat human diseases. Curr Opin Syst Biol, 2024, 37: 100492. |
| [12] |
Zou ZP, Zhang XP, Zhang Q, et al. Genetically engineered bacteria as inflammatory bowel disease therapeutics. Eng Microbiol, 2024, 4: 100167. |
| [13] |
Hicks M, Bachmann TT, Wang B. Synthetic biology enables programmable cell-based biosensors. Chemphyschem, 2020, 21: 132-44. |
| [14] |
Chiang AJ, Hasty J. Design of synthetic bacterial biosensors. Curr Opin Microbiol, 2023, 76: 102380. |
| [15] |
Pedrolli DB, Ribeiro NV, Squizato PN, et al. Engineering microbial living therapeutics: the synthetic biology toolbox. Trends Biotechnol, 2019, 37: 100-15. |
| [16] |
Chen Z, Jin W, Hoover A, et al. Decoding the microbiome: advances in genetic manipulation for gut bacteria. Trends Microbiol, 2023, 31: 1143-61. |
| [17] |
Zhang C, Liu H, Li X, et al. Modularized synthetic biology enabled intelligent biosensors. Trends Biotechnol, 2023, 41: 1055-65. |
| [18] |
Vigouroux A, Bikard D. CRISPR tools to control gene expression in bacteria. Microbiol Mol Biol Rev, 2020, 84: e00077-19. |
| [19] |
Tanniche I, Behkam B. Engineered live bacteria as disease detection and diagnosis tools. J Biol Eng, 2023, 17: 65. |
| [20] |
Liu Y, Zhu Z, Jiang L. Programming therapeutic probiotics by self-tunable sense-and-respond genetic circuits. Trends Microbiol, 2023, 31: 1099-101. |
| [21] |
Riglar DT, Silver PA. Engineering bacteria for diagnostic and therapeutic applications. Nat Rev Microbiol, 2018, 16: 214-25. |
| [22] |
Lazar JT, Tabor JJ. Bacterial two-component systems as sensors for synthetic biology applications. Curr Opin Syst Biol, 2021, 28: 100398. |
| [23] |
Ulrich LE, Koonin EV, Zhulin IB. One-component systems dominate signal transduction in prokaryotes. Trends Microbiol, 2005, 13: 52-6. |
| [24] |
Jacob-Dubuisson F, Mechaly A, Betton JM, et al. Structural insights into the signalling mechanisms of two-component systems. Nat Rev Microbiol, 2018, 16: 585-93. |
| [25] |
Kowallis KA, Silfani EM, Kasumu AP, et al. Synthetic control of signal flow within a bacterial multi-kinase network. ACS Synth Biol, 2020, 9: 1705-13. |
| [26] |
Gao R, Bouillet S, Stock AM. Structural basis of response regulator function. Annu Rev Microbiol, 2019, 73: 175-97. |
| [27] |
Rubens JR, Selvaggio G, Lu TK. Synthetic mixed-signal computation in living cells. Nat Commun, 2016, 7: 11658. |
| [28] |
Gao Y, Wang L, Wang B. Customizing cellular signal processing by synthetic multi-level regulatory circuits. Nat Commun, 2023, 14: 8415. |
| [29] |
Wang B, Kitney RI, Joly N, et al. Engineering modular and orthogonal genetic logic gates for robust digital-like synthetic biology. Nat Commun, 2011, 2: 508. |
| [30] |
Chien T, Harimoto T, Kepecs B, et al. Enhancing the tropism of bacteria via genetically programmed biosensors. Nat Biomed Eng, 2022, 6: 94-104. |
| [31] |
Nielsen AA, Der BS, Shin J, et al. Genetic circuit design automation. Science, 2016, 352: aac7341. |
| [32] |
Wan X, Volpetti F, Petrova E, et al. Cascaded amplifying circuits enable ultrasensitive cellular sensors for toxic metals. Nat Chem Biol, 2019, 15: 540-8. |
| [33] |
Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli. Nature, 2000, 403: 339-42. |
| [34] |
Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403: 335-8. |
| [35] |
Farzadfard F, Lu TK. Emerging applications for DNA writers and molecular recorders. Science, 2018, 361: 870-5. |
| [36] |
Sheth RU, Wang HH. DNA-based memory devices for recording cellular events. Nat Rev Genet, 2018, 19: 718-32. |
| [37] |
Lopreside A, Wan X, Michelini E, et al. Comprehensive profiling of diverse genetic reporters with application to whole-cell and cell-free biosensors. Anal Chem, 2019, 91: 15284-92. |
| [38] |
Ishiguro S, Mori H, Yachie N. DNA event recorders send past information of cells to the time of observation. Curr Opin Chem Biol, 2019, 52: 54-62. |
| [39] |
Rottinghaus AG, Amrofell MB, Moon TS. Biosensing in smart engineered probiotics. Biotechnol J, 2020, 15: e1900319. |
| [40] |
Selvakumar R, Kumar I, Onajobi GJ, et al. Engineering living therapeutics and diagnostics: a new frontier in human health. Curr Opin Syst Biol, 2024, 37: 100484. |
| [41] |
Landry BP, Tabor JJ. Engineering diagnostic and therapeutic gut bacteria. Microbiol Spectr, 2017, 5: 10.1128/microbiolspec.bad-0020-2017. |
| [42] |
Kaplan GG, Windsor JW. The four epidemiological stages in the global evolution of inflammatory bowel disease. Nat Rev Gastroenterol Hepatol, 2021, 18: 56-66. |
| [43] |
Bruner LP, White AM, Proksell S. Inflammatory bowel disease. Prim Care, 2023, 50: 411-27. |
| [44] |
Uhlig HH, Powrie F. Translating immunology into therapeutic concepts for inflammatory bowel disease. Annu Rev Immunol, 2018, 36: 755-81. |
| [45] |
Levitt MD, Furne J, Springfield J, et al. Detoxification of hydrogen sulfide and methanethiol in the cecal mucosa. J Clin Invest, 1999, 104: 1107-14. |
| [46] |
Furne J, Springfield J, Koenig T, et al. Oxidation of hydrogen sulfide and methanethiol to thiosulfate by rat tissues: a specialized function of the colonic mucosa. Biochem Pharmacol, 2001, 62: 255-9. |
| [47] |
Winter SE, Thiennimitr P, Winter MG, et al. Gut inflammation provides a respiratory electron acceptor for Salmonella. Nature, 2010, 467: 426-9. |
| [48] |
Hensel M, Hinsley AP, Nikolaus T, et al. The genetic basis of tetrathionate respiration in Salmonella typhimurium. Mol Microbiol, 1999, 32: 275-87. |
| [49] |
Riglar DT, Giessen TW, Baym M, et al. Engineered bacteria can function in the mammalian gut long-term as live diagnostics of inflammation. Nat Biotechnol, 2017, 35: 653-8. |
| [50] |
Winter SE, Winter MG, Xavier MN, et al. Host-derived nitrate boosts growth of E. coli in the inflamed gut. Science, 2013, 339: 708-11. |
| [51] |
Price-Carter M, Tingey J, Bobik TA, et al. The alternative electron acceptor tetrathionate supports B12-dependent anaerobic growth of Salmonella enterica serovar Typhimurium on ethanolamine or 1, 2-propanediol. J Bacteriol, 2001, 183: 2463-75. |
| [52] |
Daeffler KN, Galley JD, Sheth RU, et al. Engineering bacterial thiosulfate and tetrathionate sensors for detecting gut inflammation. Mol Syst Biol, 2017, 13: 923. |
| [53] |
Guslandi M. Nitric oxide and inflammatory bowel diseases. Eur J Clin Invest, 1998, 28: 904-7. |
| [54] |
McCafferty DM. Peroxynitrite and inflammatory bowel disease. Gut, 2000, 46: 436-9. |
| [55] |
Saijo F, Milsom AB, Bryan NS, et al. On the dynamics of nitrite, nitrate and other biomarkers of nitric oxide production in inflammatory bowel disease. Nitric Oxide, 2010, 22: 155-67. |
| [56] |
Kimura H, Miura S, Shigematsu T, et al. Increased nitric oxide production and inducible nitric oxide synthase activity in colonic mucosa of patients with active ulcerative colitis and Crohn's disease. Dig Dis Sci, 1997, 42: 1047-54. |
| [57] |
Archer EJ, Robinson AB, Süel GM. Engineered E. coli that detect and respond to gut inflammation through nitric oxide sensing. ACS Synth Biol, 2012, 1: 451-7. |
| [58] |
Woo SG, Moon SJ, Kim SK, et al. A designed whole-cell biosensor for live diagnosis of gut inflammation through nitrate sensing. Biosens Bioelectron, 2020, 168: 112523. |
| [59] |
Cartwright IM, Dowdell AS, Lanis JM, et al. Mucosal acidosis elicits a unique molecular signature in epithelia and intestinal tissue mediated by GPR31-induced CREB phosphorylation. Proc Natl Acad Sci U S A, 2021, 118: e2023871118. |
| [60] |
Jukic A, Bakiri L, Wagner EF, et al. Calprotectin: from biomarker to biological function. Gut, 70, 1978-88. |
| [61] |
Sands BE. Biomarkers of inflammation in inflammatory bowel disease. Gastroenterology, 2015, 149: 1275-85. |
| [62] |
Lichtenstein GR, Hanauer SB, Sandborn WJ, et al. Management of Crohn's disease in adults. Am J Gastroenterol, 2009, 104: 465-83. |
| [63] |
Xia JY, Hepler C, Tran P, et al. Engineered calprotectin-sensing probiotics for IBD surveillance in humans. Proc Natl Acad Sci U S A, 2023, 120: e2221121120. |
| [64] |
Zhu D, Galley J, Pizzini J, et al. Microbial biosensor for sensing and treatment of intestinal inflammation. bioRxiv, 2023. DOI:10.1101/2023.07.21.550106 |
| [65] |
Brophy MB, Hayden JA, Nolan EM. Calcium ion gradients modulate the zinc affinity and antibacterial activity of human calprotectin. J Am Chem Soc, 2012, 134: 18089-100. |
| [66] |
Nakashige TG, Zygiel EM, Drennan CL, et al. Nickel sequestration by the host-defense protein human calprotectin. J Am Chem Soc, 2017, 139: 8828-36. |
| [67] |
Gabriel SE, Helmann JD. Contributions of Zur-controlled ribosomal proteins to growth under zinc starvation conditions. J Bacteriol, 2009, 191: 6116-22. |
| [68] |
Liu JZ, Jellbauer S, Poe AJ, et al. Zinc sequestration by the neutrophil protein calprotectin enhances Salmonella growth in the inflamed gut. Cell Host Microbe, 2012, 11: 227-39. |
| [69] |
Gilston BA, Wang S, Marcus MD, et al. Structural and mechanistic basis of zinc regulation across the E. coli Zur regulon. PLoS Biol, 2014, 12: e1001987. |
| [70] |
Zou ZP, Du Y, Fang TT, et al. Biomarker-responsive engineered probiotic diagnoses, records, and ameliorates inflammatory bowel disease in mice. Cell Host Microbe, 2023, 31: 199-212. |
| [71] |
Schmidt F, Cherepkova MY, Platt RJ. Transcriptional recording by CRISPR spacer acquisition from RNA. Nature, 2018, 562: 380-85. |
| [72] |
Tanna T, Schmidt F, Cherepkova MY, et al. Recording transcriptional histories using Record-seq. Nat Protoc, 2020, 15: 513-39. |
| [73] |
Schmidt F, Zimmermann J, Tanna T, et al. Noninvasive assessment of gut function using transcriptional recording sentinel cells. Science, 2022, 376: eabm6038. |
| [74] |
Zhang J, Hasty J, Zarrinpar A. Live bacterial therapeutics for detection and treatment of colorectal cancer. Nat Rev Gastroenterol Hepatol, 2024, 21: 295-96. |
| [75] |
Fan JX, Niu MT, Qin YT, et al. Progress of engineered bacteria for tumor therapy. Adv Drug Deliv Rev, 2022, 185: 114296. |
| [76] |
Gurbatri CR, Radford GA, Vrbanac L, et al. Engineering tumor-colonizing E. coli Nissle 1917 for detection and treatment of colorectal neoplasia. Nat Commun, 2024, 15: 646. |
| [77] |
Chen TL, Siu LK, Lee YT, et al. Acinetobacter baylyi as a pathogen for opportunistic infection. J Clin Microbiol, 2008, 46: 2938-44. |
| [78] |
Nordgård L, Nguyen T, Midtvedt T, et al. Lack of detectable DNA uptake by bacterial gut isolates grown in vitro and by Acinetobacter baylyi colonizing rodents in vivo. Environ Biosafety Res, 2007, 6: 149-60. |
| [79] |
Cooper RM, Wright JA, Ng JQ, et al. Engineered bacteria detect tumor DNA. Science, 2023, 381: 682-6. |
| [80] |
Collaborators AR. Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. Lancet, 2022, 399: 629-55. |
| [81] |
Fujitani S, Sun HY, Yu VL, et al. Pneumonia due to Pseudomonas aeruginosa: part I: epidemiology, clinical diagnosis, and source. Chest, 2011, 139: 909-19. |
| [82] |
Saeidi N, Wong CK, Lo TM, et al. Engineering microbes to sense and eradicate Pseudomonas aeruginosa, a human pathogen. Mol Syst Biol, 2011, 7: 521. |
| [83] |
Li H, Jia M, Qi Q, et al. Engineered probiotic Lactobacillus plantarum WCSF I for monitoring and treatment of Staphylococcus aureus infection. Microbiol Spectr, 2023, 11: e0182923. |
| [84] |
Ali M, Nelson AR, Lopez AL, et al. Updated global burden of cholera in endemic countries. PLoS Negl Trop Dis, 2015, 9: e0003832. |
| [85] |
Holowko MB, Wang H, Jayaraman P, et al. Biosensing Vibrio cholerae with genetically engineered Escherichia coli. ACS Synth Biol, 2016, 5: 1275-83. |
| [86] |
Mao N, Cubillos-Ruiz A, Cameron DE, et al. Probiotic strains detect and suppress cholera in mice. Sci Transl Med, 2018, 10: eaao2586. |
| [87] |
Kim G, Soto JA, Morrison T. Radiologic assessment of gastrointestinal bleeding. Gastroenterol Clin North Am, 2018, 47: 501-14. |
| [88] |
Szold A, Katz LB, Lewis BS. Surgical approach to occult gastrointestinal bleeding. Am J Surg, 1992, 163: 90-2. |
| [89] |
Zuckerman GR, Prakash C, Askin MP, et al. AGA technical review on the evaluation and management of occult and obscure gastrointestinal bleeding. Gastroenterology, 2000, 118: 201-21. |
| [90] |
Pasha SF, Leighton JA, Das A, et al. Double-balloon enteroscopy and capsule endoscopy have comparable diagnostic yield in small-bowel disease: a meta-analysis. Clin Gastroenterol Hepatol, 2008, 6: 671-6. |
| [91] |
Zou ZP, Yang Y, Wang J, et al. Coupling split-lux cassette with a toggle switch in bacteria for ultrasensitive blood markers detection in feces and urine. Biosens Bioelectron, 2022, 214: 114520. |
| [92] |
Zou ZP, Wang J, Zhang Q, et al. Engineered bacteria for visualization and localization of gastrointestinal bleeding: a promising application. Anal Chem, 2024, 96: 13854-61. |
| [93] |
Tan ZZ, Li XD, Kong CD, et al. Engineering bacteria to monitor the bleeding of animals using far-red fluorescence. ACS Sens, 2021, 6: 1770-8. |
| [94] |
Lyu Y, Huang H, Su Y, et al. Macroencapsulated bacteria for in vivo sensing and therapeutics. Matter, 2024, 7: 1440-65. |
| [95] |
Nguyen DK, Son YM, Lee NE. Hydrogel encapsulation of cells in core-shell microcapsules for cell delivery. Adv Healthc Mater, 2015, 4: 1537-44. |
| [96] |
Aghlara-Fotovat S, Musteata E, Doerfert MD, et al. Hydrogel-encapsulation to enhance bacterial diagnosis of colon inflammation. Biomaterials, 2023, 301: 122246. |
| [97] |
Liu X, Yang Y, Inda ME, et al. Magnetic living hydrogels for intestinal localization, retention, and diagnosis. Adv Funct Mater, 2021, 31: 2010918. |
| [98] |
Mimee M, Nadeau P, Hayward A, et al. An ingestible bacterial-electronic system to monitor gastrointestinal health. Science, 2018, 360: 915-8. |
| [99] |
Inda-Webb ME, Jimenez M, Liu Q, et al. Sub-1.4 cm3 capsule for detecting labile inflammatory biomarkers in situ. Nature, 2023, 620: 386-92. |
| [100] |
Lee JW, Chan CTY, Slomovic S, et al. Next-generation biocontainment systems for engineered organisms. Nat Chem Biol, 2018, 14: 530-7. |
| [101] |
Liu H, Zhang L, Wang W, et al. An intelligent synthetic bacterium for chronological toxicant detection, biodegradation, and its subsequent suicide. Adv Sci, 2023, 10: e2304318. |
| [102] |
Armstrong A, Isalan M. Engineering bacterial theranostics: from logic gates to in vivo applications. Front Bioeng Biotechnol, 2024, 12: 1437301. |
| [103] |
Hurt RC, Buss MT, Duan M, et al. Genomically mined acoustic reporter genes for real-time in vivo monitoring of tumors and tumor-homing bacteria. Nat Biotechnol, 2023, 41: 919-31. |
| [104] |
Buss MT, Zhu L, Kwon JH, et al. Probiotic acoustic biosensors for noninvasive imaging of gut inflammation. bioRxiv, 2024. DOI:10.1101/2024.09.23.614598 |
| [105] |
Wang H, Zhang X, Feng Z, et al. A microbial-electronic optical communication language for intestinal monitoring and regulation. Research Square, 2024. DOI:10.21203/rs.3.rs-4904229/v1 |
 2025, Vol. 37
2025, Vol. 37 

 周英,华东理工大学教授,博士生导师。兼任上海市食品学会副秘书长,《微生物学通报》编委。曾赴荷兰瓦赫宁根大学微生物研究所访学。主要研究方向为可编程工程化微生物用于疾病的在体精准诊疗,即设计构建胞内传感、记录、调控等不同代谢功能模块,赋予微生物智能感应及原位诊疗功能,实现全细胞肠道疾病及代谢性疾病及时诊断和治疗的目标。代表性成果发表在Cell Host & Microbe、Proc Natl Acad Sci USA、Nucleic Acids Research、Analytical Chemistry、ACS Synthetic Biology、Applied & Environmental Microbiology等期刊。主持并参与多项国家自然科学基金项目、国家重点研发计划、上海市科委重点项目及企业横向课题等;
周英,华东理工大学教授,博士生导师。兼任上海市食品学会副秘书长,《微生物学通报》编委。曾赴荷兰瓦赫宁根大学微生物研究所访学。主要研究方向为可编程工程化微生物用于疾病的在体精准诊疗,即设计构建胞内传感、记录、调控等不同代谢功能模块,赋予微生物智能感应及原位诊疗功能,实现全细胞肠道疾病及代谢性疾病及时诊断和治疗的目标。代表性成果发表在Cell Host & Microbe、Proc Natl Acad Sci USA、Nucleic Acids Research、Analytical Chemistry、ACS Synthetic Biology、Applied & Environmental Microbiology等期刊。主持并参与多项国家自然科学基金项目、国家重点研发计划、上海市科委重点项目及企业横向课题等; 叶邦策,华东理工大学教授,博士生导师,现任华东理工大学生物工程学院院长。教育部“长江学者”特聘教授,教育部新世纪优秀人才支持计划学者,上海市曙光学者,上海市科委优秀学科带头人;Biotechnology & Applied Biochemistry副主编、Journal of Bacteriology编委、Biotechnology Journal编委、《合成生物学》期刊编委、《华东理工大学学报》编委。浙江省药学会常务理事,中国生物医药协会生物芯片委员会委员。曾获教育部自然科学奖二等奖、上海市技术发明二等奖、明治乳业生命科学奖等奖励。主要从事合成生物学与生物制造、智能生物系统设计与生物传感研究。近年来在Cell Host & Microbe、Proc Natl Acad Sci USA、Cell reports、Cell Chemical Biology、J Am Chem Soc、Angew Chem Int Ed等期刊发表SCI论文近400篇
叶邦策,华东理工大学教授,博士生导师,现任华东理工大学生物工程学院院长。教育部“长江学者”特聘教授,教育部新世纪优秀人才支持计划学者,上海市曙光学者,上海市科委优秀学科带头人;Biotechnology & Applied Biochemistry副主编、Journal of Bacteriology编委、Biotechnology Journal编委、《合成生物学》期刊编委、《华东理工大学学报》编委。浙江省药学会常务理事,中国生物医药协会生物芯片委员会委员。曾获教育部自然科学奖二等奖、上海市技术发明二等奖、明治乳业生命科学奖等奖励。主要从事合成生物学与生物制造、智能生物系统设计与生物传感研究。近年来在Cell Host & Microbe、Proc Natl Acad Sci USA、Cell reports、Cell Chemical Biology、J Am Chem Soc、Angew Chem Int Ed等期刊发表SCI论文近400篇