(2 上海吐露港生物科技有限公司,上海 200233)
(2 Tolo Biotechnology Co., Ltd., Shanghai 200233, China)
规律间隔成簇短回文重复序列及其关联蛋白(clustered regularly interspaced short palindromic repeats/CRISPR-associated proteins, CRISPR/Cas)系统是存在于大多数细菌和几乎所有古细菌中的重要获得性免疫系统,能够保护宿主免受外源遗传元件的入侵[1, 2]。2012年,Emmanuelle Charpentier和Jennifer Doudna团队合作解析了Cas9切割靶标双链DNA的分子机制[3],自此CRISPR技术在基因编辑领域获得了快速发展[4-8],并且两位科学家也于2020年成功获得了诺贝尔化学奖。除基因编辑外,CRISPR技术在体外诊断领域也展现出了巨大的应用潜力。相较之下,CRISPR在分子诊断上的应用虽然起步较晚,但是发展却非常迅速。
与CRISPR基因编辑应用时主要借助于Cas蛋白精准切割靶标核酸(nucleic acid, NA)的特性不同,CRISPR诊断应用主要依赖于Cas蛋白的反式切割活性[9-13]。当Cas12在向导RNA (guide RNA, gRNA)的引导下,特异结合靶标核酸并形成三元复合物之后,Cas蛋白除了能切割靶标核酸外,还会被激发出一种不依赖于序列的单链DNA切割活性,这种活性即Cas12的反式切割活性。类似地,Cas13在gRNA引导下,识别并结合靶标单链RNA后被激发出的针对非靶标单链RNA的随机切割活性,也被称为Cas13的反式切割活性(或旁路切割活性)。在开发一个基于Cas反式切割活性的CRISPR诊断(CRISPR diagnostics, CRISPR-Dx)体系时,通常在体系中加入针对靶标核酸的gRNA、Cas蛋白,以及经过精心设计的单链核酸报告探针;当靶标核酸存在时,Cas蛋白在gRNA的引导下,识别并结合靶标核酸,从而被激发出反式切割活性,切割报告探针,释放出可被检测的信号,最终完成检测过程。和传统的检测方法不同,CRISPR诊断技术的信号读取形式多样,包括荧光信号的变化、电化学信号的变化、体系颜色的变化,或结合侧向流层析技术,引起层析试纸条上的条带变化等。迄今为止,已有成百上千种基于Cas12和Cas13反式切割活性开发的CRISPR-Dx体系,其中,经典的体系包括基于Cas13的SHERLOCK[13]以及基于Cas12的HOLMES[11]和DETECTR[10, 14]。
目前,常用的分子诊断技术包括实时荧光定量PCR (quantitative real-time polymerase chain reaction, qPCR)、恒温扩增技术和下一代基因测序技术(next-generation sequencing, NGS)等。其中,qPCR技术具有灵敏准确的优势,但该技术依赖专业的设备和操作,对在资源匮乏地区的应用推广不太友好。恒温扩增技术种类众多,包括环介导恒温扩增(loop-mediated isothermal amplification, LAMP)、核酸依赖性扩增(nuclear acid sequence-based amplification, NASBA)、重组酶聚合酶扩增(recombinase polymerase amplification, RPA),以及与RPA类似的酶促重组恒温扩增(enzymatic recombinase amplification, ERA)等。恒温扩增方法虽然对设备的要求低,并可在恒温条件下指数扩增靶标核酸,但其受限于非特异性扩增和易产生假阳性结果等缺点。NGS擅长生成海量数据,但其成本较高、操作不便,且检测时间较长,因而会阻碍其在快速病原体诊断中的广泛应用。相比之下,CRISPR-Dx技术具有高特异性、快速、灵敏、易用和广泛适用性等优点,也被誉为下一代分子诊断技术,在传染病检测、癌症早期诊断和遗传病筛查等领域具有较大的应用前景。
通过联合核酸扩增技术,CRISPR-Dx技术可进一步提高检测的灵敏度;然而,这也会在一定程度上给检测操作带来挑战。例如,依赖扩增步骤的CRISPR-Dx方法早期主要为两步法反应,不仅增加了操作复杂性,也造成了气溶胶污染的风险,容易产生假阳性结果。为解决这些问题,研究人员将CRISPR-Dx核酸检测的研究重点集中于开发一步法检测体系和免扩增直接检测体系等方向。
除核酸(NA)靶标检测外,疾病特异性的非核酸(non-nucleic acid, NNA)生物标志物的检测在疾病的鉴定、早期诊断、预防和治疗过程监控等方面也有着非常重要的作用。针对NNA的检测,由于Cas蛋白不能直接识别靶标NNA,因此往往需要结合能够识别NNA的系统,并将识别信号转换为能够调控Cas反式切割活性的策略。因此,相较基于CRISPR-Dx技术的NA检测应用,NNA检测技术则仅利用了Cas的反式切割活性进行信号放大和输出。Cas蛋白的反式切割活性依赖Cas蛋白、gRNA和靶标激活序列(activator)共同形成的三元复合物,因此,可通过调控这三个元件来构建CRISPR NNA检测系统。近年来,研究人员拓展了CRISPR-Dx在诸多非核酸靶标检测中的应用,包括离子[15]、无机小分子[16, 17]、有机化合物[18, 19]、蛋白质[20, 21]和细菌[18]等。
本文总结了基于反式切割活性的CRISPR技术在医学诊断应用中的研究进展,聚焦基于反式切割活性的CRISPR-Dx核酸检测和非核酸检测应用。在NA检测策略方面,分别对CRISPR一锅法检测和免扩增检测等重点研究方向进行分析;而在NNA检测方面,则将目前CRISPR NNA检测策略进行归类分析,包括游离activator调控、gRNA生成调控及Cas蛋白活性重构等策略。本文最后还讨论了基于反式切割活性的CRISPR-Dx技术在临床诊断应用中存在的挑战及其应用展望。
1 基于反式切割活性的CRISPR核酸检测 1.1 经典CRISPR-Cas核酸检测系统基于反式切割活性的CRISPR-Dx核酸检测系统主要包括基于Cas12和Cas13的分子诊断体系(图 1)。
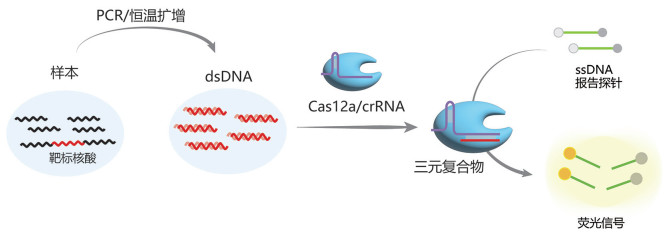
|
图 1 经典CRISPR-Cas12核酸检测系统示意图 |
在发现Cas12a具有单链DNA (single strand DNA, ssDNA)反式切割活性的同时[12],我们团队和美国Jennifer Doudna团队在同一时期分别研发出了基于Cas12反式切割活性的HOLMES[11] (An one-hour low- cost multipurpose highly efficient system)检测平台和DETECTR[10, 14](DNA endonuclease-targeted CRISPR trans reporter)检测平台(图 1),两个平台的底层技术原理完全一致。
HOLMES检测平台包括对核酸靶标的预扩增过程(PCR或恒温扩增),以及基于Cas12a的靶标识别和信号放大过程。首先针对靶标核酸序列进行PCR或恒温预扩增,然后在crRNA的引导下,Cas12a特异识别扩增产物中的靶标序列,形成Cas12a/crRNA和靶标序列的三元复合物,激活Cas12a的反式切割活性,切碎ssDNA报告探针。由于探针的两端分别被标记了荧光基团和荧光信号淬灭基团,当探针被切断后,淬灭基团便失去了淬灭效果,使得荧光基团可以发出可被检测的荧光信号,帮助完成检测。除了进行荧光检测外,ssDNA报告探针还可以进行不同的修饰,从而让HOLMES可以与诸如侧向流免疫层析试纸、电化学和颜色反应等信号输出方法结合,使得检测更加简单便捷。在靶标核酸预扩增过程中,若使用PCR技术,由于反应程序中存在高温步骤,因此扩增过程只能与Cas12a反应过程分成两步进行。相应地,如采用恒温扩增,则可与CRISPR反应进行整合,实现一步法CRISPR检测。HOLMES方法可实现阿摩尔(aM)级别的检测灵敏度,并具有极高的检测特异性,可区分靶标序列中的单碱基突变。
在HOLMES基础上,我们团队又进一步利用耐高温的Cas12b蛋白,结合LAMP恒温扩增技术,开发了首个Cas12b一步法CRISPR-Dx系统——HOLMESv2[22]。在HOLMESv2体系中,通过LAMP对靶标核酸进行扩增,产生的扩增产物可激活Cas12b的反式切割活性,释放检测信号。HOLMESv2兼具LAMP的灵敏性及CRISPR的特异性,构建了一步法核酸定量检测体系。而且,HOLMESv2无须在完成扩增反应后进行开盖移液等操作,从而避免了开盖导致的气溶胶污染风险。在HOLMESv2中,我们除了利用该技术对传染性病毒、miRNA和环状RNA进行常规检测外,还拓展了CRISPR诊断技术在包括核苷酸多态性鉴定和DNA甲基化定量测定等场景中的应用。
DETECTR[10]与HOLMES检测原理相同。DETECTR将RPA恒温扩增技术与Cas12a进行结合:首先使用核酸扩增技术对待测靶标进行初步放大,再通过Cas12a对待测靶标进行识别,并将信号转导为核酸酶活性,最终借助荧光报告探针对检测结果进行快速输出。DETECTR能够快速、特异地检测患者样本中的人类乳头瘤病毒(HPV),并对HPV-16和HPV-18亚型进行鉴定,从而为分子诊断提供了一个新的平台。
在Cas12家族中,Cas12a和Cas12b亚型在CRISPR-Dx中应用最为广泛。除了这两种蛋白外,Cas12f (旧称Cas14a)分子量较小,高度紧凑,可在crRNA-tracrRNA复合物或单链向导RNA (single-guide RNA, sgRNA)的引导下对靶标DNA进行识别和切割后,被激发出对ssDNA的反式切割活性[14, 23, 24]。除了上述效应蛋白外,近年来还发现了许多其他Cas12亚型[25] (如Cas12c、Cas12d、Cas12e、Cas12h、Cas12i、Cas12j、Cas12k、Cas12n、Cas12a2等),其中,除Cas12k[26]外,其余Cas12效应蛋白均有反式切割活性,理论上均可被用于构建基于反式切割活性的CRISPR-Dx系统。
1.1.2 CRISPR-Cas13核酸检测系统SHERLOCK[13]是首个基于Cas13的反式切割活性开发的靶标核酸快速检测方法。Cas13a在crRNA的引导下,特异性识别RNA靶标,并激活其对非靶标RNA的反式切割活性。SHERLOCK检测结合RPA恒温扩增和T7体外转录系统,使用的报告探针为单链RNA (single strand RNA, ssRNA)探针。与上面介绍的ssDNA报告探针类似,ssRNA报告探针的一端标记为荧光基团,另一端则标记淬灭基团。当存在靶标RNA时,Cas13a的反式切割活性被激活,切碎报告探针,发出荧光信号(图 2)。基于SHERLOCK方法,研究人员成功检测了寨卡病毒(ZIKV)、登革热病毒(DENV)、细菌分离株、抗性基因、人类DNA基因型和癌症突变等,实现了高特异、高灵敏的靶标核酸检测。
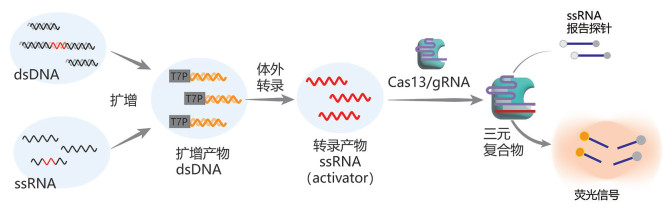
|
图 2 经典CRISPR-Cas13核酸检测系统示意图 |
除被广泛应用于CRISPR-Dx体系的Cas13a蛋白外,Cas13b、Cas13bt3、Cas13c和Cas13d等亚型均具有ssRNA反式切割活性,理论上亦可被应用于CRISPR-Dx系统的构建。
1.2 CRISPR一管法检测系统为避免因扩增产物移液操作而带来的气溶胶污染风险,目前多个研究团队已经成功开发出CRISPR一管法检测系统,可将扩增过程和CRISPR检测过程置于同一反应体系中,省去了扩增后的开盖移液等操作。一管法CRISPR诊断体系又可进一步分为不依赖Cas活性调节的物理隔离策略和直接混合策略(图 3)。
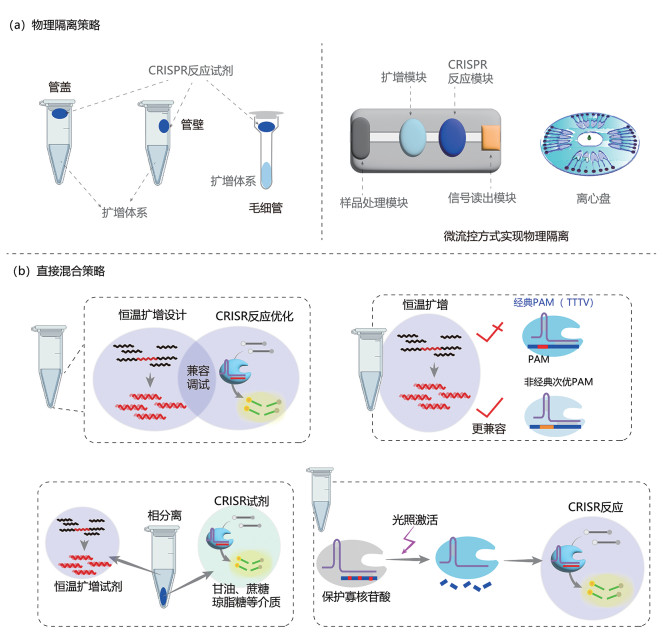
|
图 3 CRISPR一管法检测策略 |
基于物理隔离的一管法策略可通过空间分隔或微流控集成等方式实现。如将CRISPR试剂置于反应管盖或管壁,待扩增完成后进行微离心以混合CRISPR试剂和扩增产物来进行检测,其代表体系包括opvCRISPR[27]、Cas12aVDet[28]和Cas14VIDet[29]等。CRISPR一管法也可以微流控的方式[30-35]或毛细管的原理(CRISPCR)[36, 37]来实现扩增与检测的物理分隔。此外,利用赋型冷冻干燥技术可以分别将RPA扩增体系与CRISPR/Cas12a检测体系制备为独立的扩增微球与检测微球,并在检测微球表面包裹低熔点石蜡,制备为热启动CRISPR/Cas12a微球,以实现扩增和检测的物理分离[38]。这些物理分离的方法存在诸多优势,包括体系开发相对容易,而且灵敏度也较高;然而,物理分离的体系需要额外操作,检测时间也往往会比较长。
1.2.2 直接混合策略为开发更简便的诊断体系,研究人员后续便着手研究将CRISPR和扩增试剂整合到同一反应体系,从而实现一锅法CRISPR诊断。能够用于CRISPR一锅法体系的扩增技术主要集中于恒温扩增技术,包括RPA[39]、RCA[40]、NEAR[41]和LAMP[22]等。其中,LAMP或类似技术的扩增温度一般在60 ℃左右,与常用的Cas12a和Cas13蛋白的耐受温度不兼容,需适配能耐受较高温度的Cas蛋白,如Cas12b以及耐热型Cas12a和Cas13等[42-44]。通过对反应体系进行条件优化可以缓解CRISPR对扩增过程的抑制作用,进而提升检测的灵敏度和检测速度,其中优化的内容包括缓冲液组成成分、Cas酶的浓度和gRNA序列等[45-47]。例如,可以向反应体系中添加诸如L-脯氨酸和甘氨酸等成分来提高Cas蛋白的催化活性[48, 49]。
上述策略虽然可以极大程度上避免CRISPR切割对扩增过程的干扰,但Cas蛋白仍可能在扩增过程中切割扩增模板,影响检测灵敏度。为此,有研究人员便设计了利用Cas蛋白仅检测和切割扩增产物而不切割靶标模板的CRISPR诊断系统。例如,在基于RPA扩增和Cas12a的一锅法检测体系中,研究人员设计了只针对单链靶标的crRNA序列,其中单链产物可以由RPA扩增过程中释放的瞬时单链DNA[39],或者通过引入不对称扩增生成[50]。通过设计针对非经典前间隔序列邻近基序(protospacer adjacent motif, PAM)位点(如Cas12a的TTTV变体)的crRNA序列来降低切割效率,也可以减弱Cas蛋白的切割活性,减轻Cas切割对扩增的影响,保障扩增产物积累,提升检测灵敏度[51, 52]。此外,在利用Cas13a只识别和切割靶标RNA的特性开发的一锅法CRISPR检测体系[53, 54]中,也可以实现将CRISPR切割和靶标DNA扩增分离的目的;而利用该体系检测靶标RNA时,则也可能会发生Cas13a切割靶标并影响扩增和检测灵敏度的问题。近期研究发现,肝素钠可干扰Cas12a与crRNA的结合,因此可以通过调节其浓度来调控Cas12a的反式切割活性[55]。此外,通过在crRNA的5' 端引入RG4结构来精确调控Cas12a活性的新策略,同样解决了等温扩增与CRISPR反应不兼容的难题,显著提升了核酸检测的灵敏度和临床适用性[56]。
一锅法CRISPR检测还可通过相分离的方法来提升检测灵敏度。例如,通过将Cas12a与高黏稠度的甘油、蔗糖或琼脂糖等试剂预混合,实现CRISPR试剂和扩增试剂的有效隔离,延缓CRISPR切割对扩增过程的干扰。随着扩增反应的不断进行,包裹Cas反应体系的高黏稠度溶液便通过动态扩散实现均质化,进而启动CRISPR反式切割和信号输出[57-59]。虽然基于相分离的CRISPR一锅法体系相对简单,但其检测性能和重复性则严重依赖于对相分离体系的精细控制。
相比于相分离的策略,通过光控等物理信号输入的方式来控制Cas蛋白的切割活性,则可实现对CRISPR活性的更加精准的控制。例如,通过光裂解基团修饰crRNA序列[如掺入6-硝胡椒基氧甲基笼蔽胸腺嘧啶核苷(NPOM-dT)]或crRNA活性封闭序列(如在序列间加入PC-Linker基团)[60, 61],使得Cas蛋白的活性预先处于待激活状态。待完成扩增或需要激活Cas活性时,通过365 nm的紫外光短时间照射反应体系便可使修饰基团脱离或断裂,生成活性crRNA,激活Cas的切割活性和后续的CRISPR诊断信号输出。该方法不仅实现了封闭的一锅法CRISPR检测,而且其对CRISPR反应启动的调控更加精准,检测灵敏度也可以获得大幅度的提升。此外,crRNA的封闭策略还包括可被光照激活的酰化修饰,其原理和NPOM修饰类似,也通过光照来生成完整的crRNA,进而激活Cas12a的反式切割活性[62]。
1.3 CRISPR免扩增检测系统CRISPR技术因其可区分单碱基差异的高特异性、aM级别的检测灵敏度和快速便捷的优势,成为了分子诊断领域的革命性技术突破。传统CRISPR检测依赖靶标核酸的预扩增(如PCR或恒温扩增)来提升浓度,但扩增步骤不仅增加了检测时长和操作步骤,而且还可能会有气溶胶污染。相比之下,如能实现免扩增直接检测,则可有效解决气溶胶污染的风险,而且能够极大提高检测的速度和操作的简便性。因此,核酸免扩增直接检测也被广泛认为是分子诊断领域的终极目标。
基于CRISPR的免扩增直接检测技术因为省略了预扩增步骤,所以其核心难点在于如何提升低靶标浓度条件下的检测灵敏度。为解决这一难题,科学家们主要从以下几个方面来进行突破,包括优化gRNA[63, 64]与报告分子[65-67]序列、减小反应体积来提升靶标核酸的相对浓度[68-71]、采用更灵敏的信号检测技术(如电化学传感器[72-76]和表面增强拉曼光谱技术[77]等)、基于反式切割信号的级联放大[78-80]以及通过基因工程手段来改造并获得更灵敏的Cas蛋白[81]等方式(图 4)。
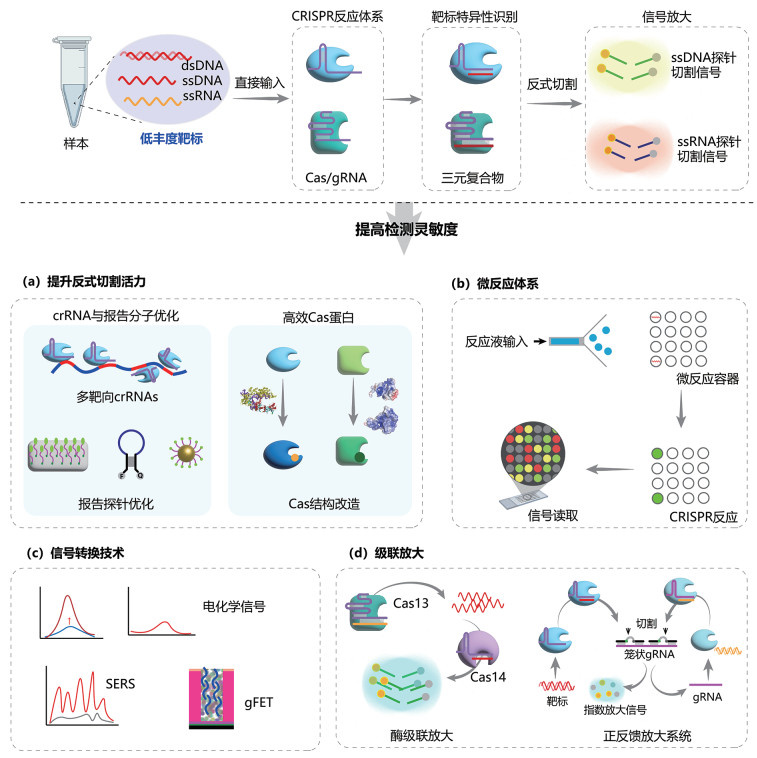
|
图 4 CRISPR免扩增检测策略 |
在CRISPR检测系统中,Cas蛋白的反式切割活性、gRNA和报告探针序列都是影响靶标识别与信号放大的关键组分,因此,提高免扩增CRISPR检测灵敏度的初始策略是优化gRNA和报告探针序列以及进化更高效的Cas蛋白。
靶向同一核酸不同区域的多条gRNA可协同激活Cas酶,提升信号强度。例如,相较于传统单crRNA的Cas12a检测系统,使用2~4条crRNA靶向同一段序列可以显著提高CRISPR检测系统的灵敏度[63]。类似地,在Cas13a检测系统中,选用两个最高效crRNA可将免扩增直接检测的灵敏度提升为270拷贝/毫升,并且检测系统具有极低的背景荧光信号[64]。
报告探针与CRISPR系统反式切割信号的输出直接相关。研究表明,Cas的催化效率受报告探针长度影响,如长度为22碱基的报告探针的检测灵敏度是5碱基的10倍[65]。除此之外,报告探针的二级结构也会影响Cas核酸酶的反式切割活性,其中,发夹型的DNA报告探针比线性探针具有更高的亲和力,其反式切割效率可提升5倍[66]。除优化报告探针的序列外,基于纳米材料或纳米颗粒辅助的报告探针也可以提高信号强度。通过将荧光染料标记的ssDNA(15 nt)均匀固定在纳米金(AuNP)核心上,形成球形核酸报告探针,可获得高信噪比,并实现低至飞摩尔(fM)级别的靶标检测灵敏度[82]。此外,对于球形核酸报告探针,通过电化学发光(ECL)[83]或金属增强荧光(MEF)[67]可进一步提高检测灵敏度,同时降低检测体系的背景信号。
通过对Cas蛋白进行改造,获得反式切割活性更高的Cas蛋白,也可以解决免扩增直接检测灵敏度不高的问题。例如,Yang等[81]利用蛋白质工程技术对LwaCas13a进行了结构导向的工程化改造,在LwaCas13a的结构域中插入RNA结合结构域,从而获得了两个高活性LwaCas13a突变体。相较于野生型LwaCas13a,突变体对靶标RNA免扩增直接检测的灵敏度提高了105倍,可直接检测aM级别浓度的靶标RNA。
1.3.2 基于微反应体系的免扩增CRISPR检测将高效的CRISPR与微反应体系相结合,可实现靶标DNA的免扩增定量直接检测。例如,Yue等[69]通过优化Cas12a反应条件参数,并将其与微液滴技术相结合,实现了基于CRISPR技术的无核酸扩增数字化DNA定量检测。利用该方法,作者成功地检测了非洲猪瘟病毒(血清样本)、人乙型肝炎病毒(血清样本)和EB病毒DNA等样本。
同样地,在基于Cas13的免扩增核酸检测研究中,Tian等[70]构建微液滴反应器来提高靶标和报告分子的局部浓度。相较常规的Cas13a检测体系,新方法的检测灵敏度提高了10 000倍,可实现单分子RNA的免扩增定量直接检测,且检测范围覆盖四个数量级。类似的方法还包括Shinoda等[68]开发的SATORI检测平台。该平台也是将Cas13与微液滴技术相结合,实现单分子水平ssRNA的准确检测。SATORI方法使用纳米孔聚碳酸酯膜将反应体系进行微液滴分割,利用荧光显微镜拍摄荧光图像,并进行分析,实现HIV-1 RNA的跨四个数量级的定量检测。基于Cas13的微反应体系还可以通过软蚀刻形成微孔,实现飞升级微孔反应来增强局部靶标浓度和信号强度,从而实现aM级别的靶标RNA检测灵敏度[71]。此外,还可将微流控芯片与智能手机进行集成,通过手机摄像头实现便携式荧光或比色信号读取[71]。
1.3.3 基于信号转换技术的免扩增CRISPR检测基于信号转换技术的免扩增CRISPR检测主要包括电化学传感器及表面增强拉曼光谱(surface-enhanced Raman spectroscopy, SERS)等技术,其中电化学传感器的原理是利用Cas酶切割探针引起电流变化,而SERS则通过结合纳米结构来提升检测信号的强度。
CRISPR系统与电化学法相结合,可实现靶标核酸的免扩增定量检测。例如,在Dai等[72]研发的Cas12a电化学生物传感器中,将ssDNA报告探针与亚甲基蓝金纳米离子(MB-AuNP)相连,并通过检测电化学信号的变化来检测靶标核酸。当反式切割活性被激活时,报告探针被剪切并产生电信号变化。后来,Zhang等[73]对该方法进行了优化,通过使用发夹DNA探针来提高Cas12a的剪切效率,并利用差分脉冲伏安法检测信号。除上述差分脉冲伏安法外,Suea-Ngam等[74]还构建了一种基于Cas12a系统的免扩增方波伏安法(square wave voltammetry, SWV)电化学CRISPR生物传感器(E-Si-CRISPR),并实现了耐甲氧西林葡萄球菌(MRSA) fM级别的免扩增检测灵敏度。
Bruch等[75]则通过集成Cas13a系统、电化学生物传感器和微流体技术,建立了免扩增的miRNA现场检测方案。该研究利用葡萄糖氧化酶(Gox)催化底物生成H2O2,产生可检电流信号,实现了对潜在肿瘤标志物miRNA-19b和miRNA-20a的pM级定量检测。类似地,Xu等[76]构建了一个基于CRISPR、三维四面体DNA框架探针和电化学生物传感器的miRNA检测方案(CRISPR-E)。
电化学发光法(ECL)是由电化学方法触发的化学放光,利用电子转移反应形成激发态,在电极表面发光,可主动控制,不需要外部光源。ECL与Cas12a相结合,可构建背景信号低的免扩增靶标核酸检测体系。如Liu等[84]利用金纳米团簇(Met-AuNC)构建了用于HPV-16检测的电化学发光生物传感器。在该方法中,二茂铁标记的巯基化ssDNA报告探针(SH-ssDNA-Fc)被固定在Met-AuNC电极表面,淬灭电化学发光信号。当靶标DNA存在时,Cas12a的反式切割活性被激活,切割SH-ssDNA-Fc成短片段,产生可检ECL信号。
石墨烯的高载流子迁移率使石墨烯场效应晶体管(graphene field-effect transistor, gFET)[85, 86]能够灵敏地探测表面发生的生物识别事件。gFET表现出V形传输特性,其中电荷中性点栅极电压的偏移用于确定转移特性的偏移,从而量化生物识别事件。据此,Weng等[87]报道了一种基于gFET和CRISPR-Cas12a的新型生物传感器CRISPR Cas12a-gFET。该方法主要利用Cas12a信号放大和gFET高性能传感能力的协同作用,使锚定在石墨烯表面的探针裂解,并能超灵敏地读取信号,以实现aM级灵敏度检测。
基于SERS技术也可实现靶标核酸的免扩增CRISPR检测。如Choi等[77]开发了一种SERS辅助的超灵敏Cas12a免扩增检测系统。该系统将被病毒DNA激活的Cas12a与由单链DNA固定的拉曼探针功能化金纳米粒子(RAuNPs)组成的拉曼敏感系统集成在氧化石墨烯(GO)/三角形金纳米花阵列上,提高了对多种病毒DNA (如HBV、HPV-16、HPV-18等)的检测灵敏度。
1.3.4 级联放大法免扩增CRISPR检测CRISPR免扩增直接检测还可以通过Cas反式切割信号的级联放大来实现,其既可以通过不同的Cas蛋白来实现,也可以使用同一种Cas蛋白。例如,Sha等[78]在构建的CRISPR体系中联合使用了Cas13a和Cas12f;其中,经Cas13a反式切割的产物可作为Cas12f的激活序列,激活Cas12f的反式切割活性,切割报告探针,释放出放大的荧光信号。
而在Shi等[79]构建的CRISPR自催化驱动的反馈放大系统(CONAN)中,则使用了单一的Cas12a蛋白来实现反式切割信号的级联放大。研究人员除了设计针对靶标序列的crRNA外,还设计了一种特殊的被DNA封闭的第二crRNA (可转化笼状向导RNA,scgRNA)。当靶标核酸存在时,靶向靶标序列的crRNA引导Cas12a对其识别并激发出Cas12a的反式切割活性,切断scgRNA中的封闭DNA,释放出有功能的第二crRNA,并在辅助DNA的帮助下,持续激活Cas12a的反式切割活性,在切割报告探针的同时,也持续释放更多的第二crRNA,达到一个正反馈的效果,快速放大信号,实现aM级别的靶标检测灵敏度。
近日,Lim等[80]提出一种类似的CRISPR-Cascade检测方法。该法通过整合正反馈回路实现无核酸扩增检测致病DNA,无须核酸预扩增,就能在10 min内从纳摩尔(nM)到aM的广泛靶标DNA浓度范围内实现高灵敏度和特异性检测,且检测靶标可覆盖多种血液感染病原体,如MRSA、大肠杆菌和乙型肝炎病毒(HBV)等。
2 基于Cas反式切割活性的NNA检测体系针对NNA的检测涉及临床诊断、生命科学、食品科学、环境监测等多个领域。传统NNA靶标的检测方法,如高效液相法(high-performance liquid chromatography, HPLC)、气相法(gas chromatography, GC)、质谱法(mass spectrum, MS)和酶联免疫吸附法(enzyme linked immunosorbent assay, ELISA)等,均需要依赖大型仪器及专业操作。目前,这些传统检测方法无法满足临床诊断、生命科学、食品科学和环境监测等领域中对NNA快速检测、现场检测、方便检测、及时检测的需求,而CRISPR诊断技术的问世也为这一难题带来了解决方案。基于CRISPR技术的NNA检测系统的关键是在NNA的特异识别信息与Cas蛋白反式切割活性的调控之间建立对应关系。由于Cas蛋白的反式切割活性依赖于Cas蛋白、gRNA和靶标序列之间的三元复合物的形成,因此,根据CRISPR NNA体系的构建原理,以反式切割活性调控的三个必要元件为分类依据,可将已报道的检测体系分为三大类,分别为:(1)通过游离activator调控的体系;(2)通过生成gRNA调控的体系;(3)通过重构Cas蛋白活性来调控的体系(图 5)。
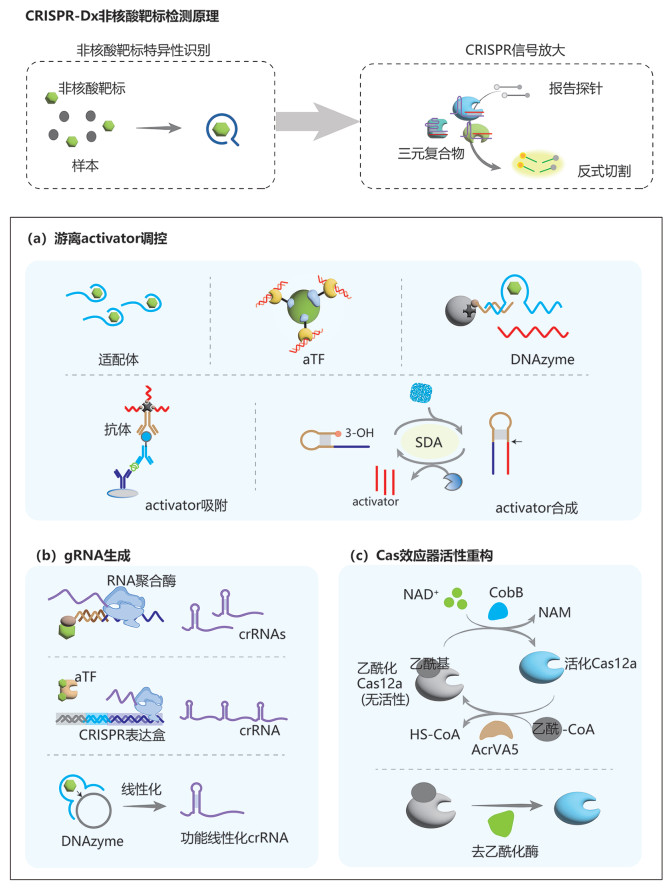
|
图 5 基于反式切割活性的CRISPR非核酸靶标检测 |
将NNA靶标信号识别转换为activator信号识别,是CRISPR-Dx非核酸靶标检测系统设计中应用最广的转换策略。游离activator的调控方法主要可细分为五大类:适配体介导的游离activator调控、变构转录因子(allosteric transcription factor, aTF)介导的游离activator调控、DNAzymes介导的游离activator调控、activator吸附和activator合成。
在适配体介导的游离activator调控中,适配体可以:(1)作为activator的“锁定链”[88]或连接activator与固定化载体的Linker[21],当适配体结合NNA靶标后会释放activator,激活Cas蛋白的反式切割活性,释放检测信号;(2)直接作为activator,当适配体与NNA靶标结合后,便不能激活Cas12a蛋白的反式切割活性,导致信号的降低[16]。
在aTF介导的游离activator调控系统中(如经典的CaT-Smelor检测系统[89]),固定化的aTF在靶标存在时可释放预捕获的activator,启动Cas蛋白的反式切割和信号释放。
DNAzymes介导的游离activator调控策略常用于离子靶标检测。该类检测系统通常包含特异的DNAzyme切割位点和阻断的activator序列。在靶离子存在的情况下,DNAzymes在切割位点切割,释放游离activator,激活Cas的反式切割活性,如基于Cas12a的Pb2+和Na+检测系统等[88, 90]。除了离子靶标,DNAzyme还可以被其他NNA激活。例如,在葡萄糖检测系统中,Gox首先氧化靶标葡萄糖产生H2O2,随后H2O2触发DNAzyme的自切割,导致游离activator的释放,产生输出信号[91]。
在activator吸附策略中,主要通过固定化的NNA响应捕获因子(如抗体、适配体等)调控游离activator的吸附。吸附的activator结合CRISPR的信号放大功能,提高靶标的检测灵敏度、检测范围等[92]。
合成activator的策略则主要依赖于工具酶,主要包括:(1)连接酶介导的activator修复(例如,NAD+/ATP的检测系统)[93];(2)末端转移酶/端粒酶/Klenow DNA聚合酶等介导的3' -OH延伸[94, 95];(3) aTF调控体外转录(in vitro transcription, IVT)生成Cas13 RNA activator[96]等。
2.2 gRNA生成调控策略除游离activator调控策略外,调控gRNA的合成也可实现对Cas蛋白反式切割活性的控制。在已报道的gRNA生成调控策略中,主要使用Cas12a效应蛋白,其gRNA (即crRNA)的合成调控体系通常可结合RNA聚合酶[97, 98]、外切核酸酶I (Exo I)[97]、aTF[98]和核酸酶(NAzyme)[99]等。例如,在链霉抗生物素蛋白和外泌体的检测中[97],研究人员设计了一种可被Exo I降解的IVT模板序列。当模板序列与NNA结合时,可避免被Exo I切割,随后通过IVT生成crRNA,并激活Cas蛋白的反式切割活性。通过在T7启动子和CRISPR阵列间设计aTF结合区,当NNA存在时,aTF解离,启动IVT合成crRNA[98]。靶标抗体与一对抗原偶联的DNA链结合,通过共定位诱导形成双分子复合物,该复合物可以与编码crRNA的不完整DNA模板链杂交,诱发无细胞转录,进而生成crRNA[100]。此外,NNA调控的NAzyme可切割无功能的环状crRNA,生成有活性的线性化crRNA;该方法可成功应用于ATP、大肠杆菌、肺炎克雷伯氏菌等靶标的检测[99]。
2.3 Cas蛋白的活性重构调控策略通过基于NNA靶标的乙酰化-去乙酰化修饰机制来调控Cas的蛋白反式切割活性也是一种可行的NNA靶标检测策略。Anti-CRISPR (Acr)系统是病毒进化出的CRISPR-Cas活性抑制系统。在迄今鉴定的十几种Acr系统中,乙酰转移酶AcrVA5通过乙酰化灭活Cas12a的顺式和反式切割活性[101]。有趣的是,细菌去乙酰化酶CobB能有效地将乙酰化修饰的Cas12a蛋白去乙酰,恢复其反式切割活性[102]。CobB去乙酰化需要NAD+辅因子,因此,该系统便可用于靶标NAD+的检测[103]。除了检测NAD+辅因子外,乙酰化的Cas12a也可以被用于直接检测待测体系中去乙酰化酶的活性,如组蛋白去乙酰化酶(Sirtuin-1)[104]。
3 总结与展望Cas12和Cas13对DNA或RNA的非特异性反式切割活性为CRISPR-Dx提供了基础。在核酸检测方面,通过联合核酸扩增技术,CRISPR-Dx技术可进一步提高检测的灵敏度;然而,依赖扩增步骤的CRISPR-Dx方法早期主要为两步法反应,不仅增加了操作复杂性,也造成了气溶胶污染的风险,容易产生假阳性检测结果。为解决这些问题,研究人员将CRISPR-Dx核酸检测的研究逐渐转向一锅法检测体系和免扩增直接检测体系的开发。
一锅法CRISPR检测策略的核心在于避免扩增反应和CRISPR反应之间的互相干扰,其中最直接的方式是将扩增和检测反应进行物理隔离,以保证扩增反应成功完成,之后再将CRISPR反应引入扩增后体系,进行信号放大与结果输出。此类策略虽然可以保证避免扩增反应和CRISPR反应之间的干扰,但在检测流程上往往需要额外操作,增加了检测步骤和时间。而对于直接混合策略,除相分离外,往往需要对CRISPR的活性进行精准调控,使得靶标扩增和CRISPR反应达到适当的平衡。虽然直接混合的CRISPR体系在方法上更加简便快速,但是其在系统稳定性、反应精细优化、检测灵敏度等方面仍存在诸多挑战。未来,通过开发抗基质和环境干扰的Cas蛋白突变体,有望促进粗提样本的直接检测应用。在信号输出方面,通过开发试纸条或比色法读取方式,可进一步降低设备依赖性和成本,提升CRISPR诊断应用的便捷性。未来,通过新材料、智能策略及多技术融合,有望推动其向低成本、便携化、高通量的方向发展,助力传染病监测、癌症早筛及环境监测等领域的革新。
实现核酸的免扩增直接检测被广泛认为是分子诊断领域的终极目标。CRISPR诊断技术是目前看来最有可能实现核酸免扩增直检的技术之一,其核心在于提升低靶标浓度条件下的检测灵敏度。CRISPR系统的各个模块,如靶标核酸识别、报告探针反式切割和信号输出等,均是影响检测灵敏度的关键步骤。首先,可以通过优化crRNA和报告探针序列的设计来提高检测的信噪比。此外,基于微流控技术的生物传感器、超灵敏数字检测平台以及信号级联放大系统等,也能够提升CRISPR检测技术的灵敏度。但是,目前基于CRISPR技术的免扩增直检技术仍存在一些不足之处,如靶标序列的识别受PAM位点的限制、检测体系对样本杂质的抗干扰性不强、反式切割反应受环境温度影响等。未来,除标准化、经济效益等方面考量外,集成样品预处理步骤与免扩增CRISPR/Cas检测是即时检验(point-of-care testing, POCT)应用中的关键。此外,多重检测一直是CRISPR诊断体系亟待解决的难题之一,引入基于物理分离的微流控技术或者构建具有反式切割序列偏好性的Cas蛋白突变体都有可能实现CRISPR一管多重的检测目的。
在非核酸检测方面,Cas12a蛋白的应用最为广泛。相较于其他Cas系统,Cas12a系统仅需合成短链crRNA和ssDNA报告探针,系统成分简单、稳定且成本低。在信号转化策略上,现有NNA检测系统往往受限于将NNA靶标转换为Cas蛋白可识别的核酸信号,限制了CRISPR检测系统的开发效率。基于乙酰化-去乙酰化机制的Cas蛋白活性调控策略,为基于“靶标-效应器”直接偶联机制的非核酸检测系统开发开拓了思路。未来通过优化Cas蛋白组合、开发新型信号模型(如逻辑门控设计)及融合数字检测技术,将开发出更高效、兼容多靶标且易于定量的CRISPR NNA检测技术,助力精准医疗与即时诊断。
| [1] |
Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea. Nature, 2012, 482: 331-8. |
| [2] |
Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 5819: 1709-12. |
| [3] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337: 816-21. |
| [4] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163: 759-71. DOI:10.11844/cjcb.2015.06.9001 |
| [5] |
Li S, Zhao G, Wang J. C-Brick: a new standard for assembly of biological parts using Cpf1. ACS Synth Biol, 2016, 5: 1383-8. |
| [6] |
Jiang Y, Qian F, Yang J, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun, 2017, 8: 15179. |
| [7] |
Li SY, Liu JK, Zhao GP, et al. CADS: CRISPR/Cas12a-assisted DNA steganography for securing the storage and transfer of DNA-encoded information. ACS Synth Biol, 2018, 7: 1174-8. |
| [8] |
Lei C, Li SY, Liu JK, et al. The CCTL (Cpf1-assisted cutting and Taq DNA ligase-assisted ligation) method for efficient editing of large DNA constructs in vitro. Nucleic Acids Res, 2017, 45: e74. |
| [9] |
Chertow DS. Next-generation diagnostics with CRISPR. Science, 2018, 360: 381-2. |
| [10] |
Chen JS, Ma E, Harrington LB, et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science, 2018, 360: 436-9. |
| [11] |
Li S, Cheng Q, Wang J, et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discov, 2018, 4: 20. |
| [12] |
Li S, Cheng Q, Liu J, et al. CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA. Cell Res, 2018, 28: 491-3. |
| [13] |
Gootenberg JS, Abudayyeh OO, Lee JW, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science, 2017, 356: 438-42. |
| [14] |
Harrington LB, Burstein D, Chen JS, et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science, 2018, 362: 839-42. |
| [15] |
Zhu F, Yu H, Zhao Q. CRISPR/Cas12a-amplified aptamer switch microplate assay for small molecules. Anal Chem, 2024, 96: 6853-9. |
| [16] |
Lin X, Li C, Meng X, et al. CRISPR-Cas12a-mediated luminescence resonance energy transfer aptasensing platform for deoxynivalenol using gold nanoparticle-decorated Ti3C2Tx MXene as the enhanced quencher. J Hazard Mater, 2022, 433: 128750. |
| [17] |
Hu J, Song H, Zhou J, et al. Metal-tagged CRISPR/Cas12a bioassay enables ultrasensitive and highly selective evaluation of kanamycin bioaccumulation in fish samples. Anal Chem, 2021, 93: 14214-22. |
| [18] |
Sheng A, Wang P, Yang J, et al. MXene coupled with CRISPR-Cas12a for analysis of endotoxin and bacteria. Anal Chem, 2021, 93: 4676-81. |
| [19] |
Wu P, Ye X, Wang D, et al. A novel CRISPR/Cas14a system integrated with 2D porphyrin metal-organic framework for microcystin-LR determination through a homogeneous competitive reaction. J Hazard Mater, 2022, 424: 127690. |
| [20] |
Liu ML, Li Y, Zhao ML, et al. Y-shaped DNA nanostructures assembled-spherical nucleic acids as target converters to activate CRISPR-Cas12a enabling sensitive ECL biosensing. Biosen Bioelectron, 2022, 214: 114512. |
| [21] |
Zhao X, Li S, Liu G, et al. A versatile biosensing platform coupling CRISPR-Cas12a and aptamers for detection of diverse analytes. Sci Bull (Beijing), 2021, 66: 69-77. |
| [22] |
Li L, Li S, Wu N, et al. HOLMESv2: a CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation. ACS Synth Biol, 2019, 8: 2228-37. |
| [23] |
Wu Q, Yi Z, Li H, et al. Harnessing noncanonical trans-cleavage characteristics of Cas12 and Cas13a to enhance CRISPR-based diagnostics. Commun Biol, 2024, 7: 1312. |
| [24] |
Aquino-Jarquin G. CRISPR-Cas14 is now part of the artillery for gene editing and molecular diagnostic. Nanomedicine, 2019, 18: 428-31. |
| [25] |
Tong B, Dong H, Cui Y, et al. The versatile type V CRISPR effectors and their application prospects. Front Cell Dev Biol, 2021, 8: 622103. |
| [26] |
Schmitz M, Querques I, Oberli S, et al. Structural basis for the assembly of the type V CRISPR-associated transposon complex. Cell, 2022, 185: 4999-5010.e17. |
| [27] |
Wang R, Qian C, Pang Y, et al. opvCRISPR: one-pot visual RT-LAMP-CRISPR platform for SARS-cov-2 detection. Biosens Bioelectron, 2021, 172: 112766. |
| [28] |
Wang B, Wang R, Wang D, et al. Cas12aVDet: a CRISPR/Cas12a-based platform for rapid and visual nucleic acid detection. Anal Chem, 2019, 91: 12156-61. |
| [29] |
Lai Y, Guo K, Zhu C, et al. Cas14VIDet: a visual instant method free from PAM restriction for antibiotic resistance bacteria detection. Biosens Bioelectron, 2025, 268: 116884. |
| [30] |
Ngamsom B, Iles A, Kamita M, et al. A sample-to-answer COVID-19 diagnostic device based on immiscible filtration and CRISPR-Cas12a-assisted detection. Talanta Open, 2022, 6: 100166. |
| [31] |
Wu H, Chen Y, Yang Q, et al. A reversible valve-assisted chip coupling with integrated sample treatment and CRISPR/Cas12a for visual detection of Vibrio parahaemolyticus. Biosens Bioelectron, 2021, 188: 113352. |
| [32] |
Zhou H, Xu Z, He L, et al. Coupling CRISPR/Cas12a and recombinase polymerase amplification on a stand-alone microfluidics platform for fast and parallel nucleic acid detection. Anal Chem, 2023, 95: 3379-89. |
| [33] |
Chen Y, Mei Y, Zhao X, et al. Reagents-loaded, automated assay that integrates recombinase-aided amplification and Cas12a nucleic acid detection for a point-of-care test. Anal Chem, 2020, 92: 14846-52. |
| [34] |
Chen Y, Zong N, Ye F, et al. Dual-CRISPR/Cas12a-assisted RT-RAA for ultrasensitive SARS-CoV-2 detection on automated centrifugal microfluidics. Anal Chem, 2022, 94: 9603-9. |
| [35] |
Zhang Y, Su R, Zhang Z, et al. An ultrasensitive one-pot Cas13a-based microfluidic assay for rapid multiplexed detection of microRNAs. Biosens Bioelectron, 2025, 274: 117212. |
| [36] |
Wang R, Chen R, Qian C, et al. Ultrafast visual nucleic acid detection with CRISPR/Cas12a and rapid PCR in single capillary. Sens Actuators B Chem, 2020, 326: 128618. |
| [37] |
Li H, Xiu L, Guo X, et al. Spatiotemporal dual-dimensional separation enables ultra-simple one-pot CRISPR-based molecular diagnostics. Chem Eng J, 2024, 482: 148872. |
| [38] |
Xie S, Shao S, Wang J, et al. Heat-triggered CRISPR/Cas12a microspheres for one-pot assays. Sci Bull (Beijing), 2025, 70: 1922-6. |
| [39] |
Ding X, Yin K, Li Z, et al. Ultrasensitive and visual detection of SARS-CoV-2 using all-in-one dual CRISPR-Cas12a assay. Nat Commun, 2020, 11: 4711. |
| [40] |
Yan H, Wen Y, Tian Z, et al. A one-pot isothermal Cas12-based assay for the sensitive detection of microRNAs. Nat Biomed Eng, 2023, 7: 1583-601. |
| [41] |
Bai L, Pang Y, Wang T, et al. SPEAR: CRISPR-mediated ultrasensitive, specific and rapid one-pot detection strategy for cancer-related SNPs. Theranostics, 2025, 15: 3275-88. |
| [42] |
Nguyen LT, Rananaware SR, Yang LG, et al. Engineering highly thermostable Cas12b via de novo structural analyses for one-pot detection of nucleic acids. Cell Rep Med, 2023, 4: 101037. |
| [43] |
Mahas A, Marsic T, Lopez-Portillo MM, et al. Characterization of a thermostable Cas13 enzyme for one-pot detection of SARS-CoV-2. Proc Natl Acad Sci U S A, 2022, 119: e2118260119. |
| [44] |
Nguyen LT, Macaluso NC, Pizzano B, et al. A thermostable Cas12b from Brevibacillus leverages one-pot discrimination of SARS-CoV-2 variants of concern. EBioMedicine, 2022, 77: 103926. |
| [45] |
Malcı K, Walls LE, Rios-Solis L. Rational design of CRISPR/Cas12a-RPA based one-pot COVID-19 detection with design of experiments. ACS Synth Biol, 2022, 11: 1555-67. |
| [46] |
Feng W, Peng H, Xu J, et al. Integrating reverse transcription recombinase polymerase amplification with CRISPR technology for the one-tube assay of RNA. Anal Chem, 2021, 93: 12808-16. |
| [47] |
Arizti-Sanz J, Freije CA, Stanton AC, et al. Streamlined inactivation, amplification, and Cas13-based detection of SARS-CoV-2. Nat Commun, 2020, 11: 5921. |
| [48] |
Li Z, Zhao W, Ma S, et al. A chemical-enhanced system for CRISPR-based nucleic acid detection. Biosens Bioelectron, 2021, 192: 113493. |
| [49] |
Wang Y, Chen H, Gao H, et al. CESSAT: a chemical additive-enhanced single-step accurate CRISPR/Cas13 testing system for field-deployable ultrasensitive detection and genotyping of SARS-CoV-2 variants of concern. Biosens Bioelectron, 2023, 229: 115238. |
| [50] |
Yang L, Chen G, Wu J, et al. A PAM-free one-step asymmetric RPA and CRISPR/Cas12b combined assay (OAR-CRISPR) for rapid and ultrasensitive DNA detection. Anal Chem, 2024, 96: 5471-7. |
| [51] |
Lu S, Tong X, Han Y, et al. Fast and sensitive detection of SARS-CoV-2 RNA using suboptimal protospacer adjacent motifs for Cas12a. Nat Biomed Eng, 2022, 6: 286-97. |
| [52] |
Bhardwaj P, Dhangur P, Kalichamy A, et al. RT-RPA assisted CRISPR/Cas12a based one-pot rapid and visual detection of the pan-dengue virus. J Med Virol, 2025, 97: e70219. |
| [53] |
Tong X, Zhang K, Han Y, et al. Fast and sensitive CRISPR detection by minimized interference of target amplification. Nat Chem Biol, 2024, 20: 885-93. |
| [54] |
Li H, Kielich D, Liu G, et al. Strategies to improve multi-enzyme compatibility and coordination in one-pot SHERLOCK. Anal Chem, 2023, 95: 10522-31. |
| [55] |
Cheng Z, Luo X, Yu S, et al. Tunable control of Cas12 activity promotes universal and fast one-pot nucleic acid detection. Nat Commun, 2025, 16: 1166. |
| [56] |
Huang W, Wang J, Wang C, et al. Expanding Cas12a activity control with an RNA G-quadruplex at the 5' end of CRISPR RNA. Adv Sci (Weinh), 2025, 12: e2411305. |
| [57] |
Lin M, Yue H, Tian T, et al. Glycerol additive boosts 100-fold sensitivity enhancement for one-pot RPA-CRISPR/Cas12a assay. Anal Chem, 2022, 94: 8277-84. |
| [58] |
Yin K, Ding X, Li Z, et al. Dynamic aqueous multiphase reaction system for one-pot CRISPR-Cas12a-based ultrasensitive and quantitative molecular diagnosis. Anal Chem, 2020, 92: 8561-8. |
| [59] |
Zhao L, Wang H, Chen X, et al. Agarose hydrogel-boosted one-tube RPA-CRISPR/Cas12a assay for robust point-of-care detection of zoonotic nematode Anisakis. J Agric Food Chem, 2024, 72: 8257-68. |
| [60] |
Hu M, Qiu Z, Bi Z, et al. Photocontrolled crRNA activation enables robust CRISPR-Cas12a diagnostics. Proc Natl Acad Sci U S A, 2022, 119: e2202034119. |
| [61] |
Chen Y, Xu X, Wang J, et al. Photoactivatable CRISPR/Cas12a strategy for one-pot DETECTR molecular diagnosis. Anal Chem, 2022, 94: 9724-31. |
| [62] |
Liu P, Lin Y, Zhuo X, et al. Universal crRNA acylation strategy for robust photo-initiated one-pot CRISPR-Cas12a nucleic acid diagnostics. Angew Chem Int Ed Engl, 2024, 63: e202401486. |
| [63] |
Zeng M, Ke Y, Zhuang Z, et al. Harnessing multiplex crRNA in the CRISPR/Cas12a system enables an amplification-free DNA diagnostic platform for ASFV detection. Anal Chem, 2022, 94: 10805-12. |
| [64] |
Fozouni P, Son S, Díaz de León Derby M, et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell, 2021, 184: 323-33.e9. |
| [65] |
He Q, Chen Q, Li F, et al. Amplification-free detection of viral RNA by super resolution imaging-based CRISPR/Cas13a system. bioRxiv, 2021. DOI:10.1101/2021.07.17.452803 |
| [66] |
Rossetti M, Merlo R, Bagheri N, et al. Enhancement of CRISPR/Cas12a trans-cleavage activity using hairpin DNA reporters. Nucleic Acids Res, 2022, 50: 8377-91. |
| [67] |
Choi JH, Lim J, Shin M, et al. CRISPR-Cas12a-based nucleic acid amplification-free DNA biosensor via Au nanoparticle-assisted metal-enhanced fluorescence and colorimetric analysis. Nano Lett, 2021, 21: 693-9. |
| [68] |
Shinoda H, Taguchi Y, Nakagawa R, et al. Amplification-free RNA detection with CRISPR-Cas13. Commun Biol, 2021, 4: 476. |
| [69] |
Yue H, Shu B, Tian T, et al. Droplet Cas12a assay enables DNA quantification from unamplified samples at the single-molecule level. Nano Lett, 2021, 21: 4643-53. |
| [70] |
Tian T, Shu B, Jiang Y, et al. An ultralocalized Cas13a assay enables universal and nucleic acid amplification-free single-molecule RNA diagnostics. ACS Nano, 2021, 15: 1167-78. |
| [71] |
Wang D, Wang X, Ye F, et al. An integrated amplification-free digital CRISPR/Cas-assisted assay for single molecule detection of RNA. ACS Nano, 2023, 17: 7250-6. |
| [72] |
Dai Y, Somoza RA, Wang L, et al. Exploring the trans-cleavage activity of CRISPR‐Cas12a (cpf1) for the development of a universal electrochemical biosensor. Angew Chem Int Ed Engl, 2019, 58: 17399-405. |
| [73] |
Zhang D, Yan Y, Que H, et al. CRISPR/Cas12a-mediated interfacial cleaving of hairpin DNA reporter for electrochemical nucleic acid sensing. ACS Sens, 2020, 5: 557-62. |
| [74] |
Suea-Ngam A, Howes PD, DeMello AJ. An amplification-free ultra-sensitive electrochemical CRISPR/Cas biosensor for drug-resistant bacteria detection. Chem Sci, 2021, 12: 12733-43. |
| [75] |
Bruch R, Baaske J, Chatelle C, et al. CRISPR/Cas13a-powered electrochemical microfluidic biosensor for nucleic acid amplification-free miRNA diagnostics. Adv Mater, 2019, 31: e1905311. |
| [76] |
Xu Y, Wang C, Liu G, et al. Tetrahedral DNA framework based CRISPR electrochemical biosensor for amplification- free miRNA detection. Biosens Bioelectron, 2022, 217: 114671. |
| [77] |
Choi JH, Shin M, Yang L, et al. Clustered regularly interspaced short palindromic repeats-mediated amplification-free detection of viral DNAs using surface-enhanced Raman spectroscopy-active nanoarray. ACS Nano, 2021, 15: 13475-85. |
| [78] |
Sha Y, Huang R, Huang M, et al. Cascade CRISPR/cas enables amplification-free microRNA sensing with fM-sensitivity and single-base-specificity. Chem Commun (Camb), 2021, 57: 247-50. |
| [79] |
Shi K, Xie S, Tian R, et al. A CRISPR-Cas autocatalysis-driven feedback amplification network for supersensitive DNA diagnostics. Sci Adv, 2021, 7: eabc7802. |
| [80] |
Lim J, Van AB, Koprowski K, et al. Amplification-free, OR-gated CRISPR-Cascade reaction for pathogen detection in blood samples. Proc Natl Acad Sci U S A, 2025, 122: e2420166122. |
| [81] |
Yang J, Song Y, Deng X, et al. Engineered LwaCas13a with enhanced collateral activity for nucleic acid detection. Nat Chem Biol, 2023, 19: 45-54. |
| [82] |
Fu X, Shi Y, Peng F, et al. Exploring the trans-cleavage activity of CRISPR/Cas12a on gold nanoparticles for stable and sensitive biosensing. Anal Chem, 2021, 93: 4967-74. |
| [83] |
Zhao K, Wang L, Liu P, et al. A signal-switchable electrochemiluminescence biosensor based on the integration of spherical nucleic acid and CRISPR/Cas12a for multiplex detection of HIV/HPV DNAs. Sens Actuators B Chem, 2021, 346: 130485. |
| [84] |
Liu PF, Zhao KR, Liu ZJ, et al. Cas12a-based electrochemiluminescence biosensor for target amplification- free DNA detection. Biosen Bioelectron, 2021, 176: 112954. |
| [85] |
Balderston S, Taulbee JJ, Celaya E, et al. Discrimination of single-point mutations in unamplified genomic DNA via Cas9 immobilized on a graphene field-effect transistor. Nat Biomed Eng, 2021, 5: 713-25. |
| [86] |
Hajian R, Balderston S, Tran T, et al. Detection of unamplified target genes via CRISPR-Cas9 immobilized on a graphene field-effect transistor. Nat Biomed Eng, 2019, 3: 427-37. |
| [87] |
Weng Z, You Z, Li H, et al. CRISPR-Cas12a biosensor array for ultrasensitive detection of unamplified DNA with single-nucleotide polymorphic discrimination. ACS Sens, 2023, 8: 1489-99. |
| [88] |
Li C, Zheng B, Li J, et al. Holographic optical tweezers and boosting upconversion luminescent resonance energy transfer combined clustered regularly interspaced short palindromic repeats (CRISPR)/Cas12a biosensors. ACS Nano, 2021, 15: 8142-54. |
| [89] |
Liang M, Li Z, Wang W, et al. A CRISPR-Cas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules. Nat Commun, 2019, 10: 3672. |
| [90] |
Li J, Yang S, Zuo C, et al. Applying CRISPR-Cas12a as a signal amplifier to construct biosensors for non-DNA targets in ultralow concentrations. ACS Sens, 2020, 5: 970-7. |
| [91] |
Zhang C, Yao H, Ma Q, et al. Ultrasensitive glucose detection from tears and saliva through integrating a glucose oxidase-coupled DNAzyme and CRISPR-Cas12a. Analyst, 2021, 146: 6576-81. |
| [92] |
Li Y, Deng F, Goldys EM. A simple and versatile CRISPR/Cas12a-based immunosensing platform: towards attomolar level sensitivity for small protein diagnostics. Talanta, 2022, 246: 123469. |
| [93] |
Zhao J, Tan Z, Wang L, et al. A ligation-driven CRISPR-Cas biosensing platform for non-nucleic acid target detections. Chem Commun, 2021, 57: 7051-4. |
| [94] |
Du Y, Wang S, Wang Y, et al. Terminal deoxynucleotidyl transferase combined CRISPR-Cas12a amplification strategy for ultrasensitive detection of uracil-DNA glycosylase with zero background. Biosens Bioelectron, 2021, 171: 112734. |
| [95] |
Chen X, Cao G, Zhang J, et al. An ultrasensitive and point-of-care strategy for enzymes activity detection based on enzyme extends activators to unlock the ssDNase activity of CRISPR/Cas12a (EdU-CRISPR/Cas12a). Sens Actuators B Chem, 2021, 333: 129553. |
| [96] |
Iwasaki RS, Batey RT. SPRINT: a Cas13a-based platform for detection of small molecules. Nucleic Acids Res, 2020, 48: e101. |
| [97] |
Kang X, Lei C, Shi J, et al. A versatile CRISPR/Cas12a-based biosensing platform coupled with a target-protected transcription strategy. Biosens Bioelectron, 2023, 219: 114801. |
| [98] |
Mahas A, Wang Q, Marsic T, et al. Development of Cas12a-based cell-free small-molecule biosensors via allosteric regulation of CRISPR array expression. Anal Chem, 2022, 94: 4617-26. |
| [99] |
Wu Y, Chang D, Chang Y, et al. Nucleic acid enzyme-activated CRISPR‐Cas12a with circular CRISPR RNA for biosensing. Small, 2023, 19: e2303007. |
| [100] |
Miceli F, Bracaglia S, Sorrentino D, et al. MAIGRET: a CRISPR-based immunoassay that employs antibody-induced cell-free transcription of CRISPR guide RNA strands. Nucleic Acids Res, 2025, 53: gkaf238. |
| [101] |
Dong L, Guan X, Li N, et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nat Struct Mol Biol, 2019, 26: 308-14. |
| [102] |
Kang X, Yin L, Zhuang S, et al. Reversible regulation of Cas12a activities by AcrVA5-mediated acetylation and CobB-mediated deacetylation. Cell Discov, 2022, 8: 45. |
| [103] |
Zhuang S, Hu T, Zhou H, et al. CRISPR-HOLMES-based NAD+ detection. Front Bioeng Biotechnol, 2024, 12: 1355640. |
| [104] |
Kang W, Liu L, Yu P, et al. A switchable Cas12a enabling CRISPR-based direct histone deacetylase activity detection. Biosens Bioelectron, 2022, 213: 114468. |
 2025, Vol. 37
2025, Vol. 37 

 王金,教授,上海吐露港生物科技有限公司总经理、首席科学家。2009年毕业于中国科学院上海生命科学研究院植物生理生态研究所,获得微生物学博士学位;2009—2012年在香港中文大学微生物学系从事博士后研究;2012—2022年先后任职于中国科学院上海生命科学研究院植物生理生态研究所、上海师范大学和深圳大学第一附属医院。其主要研究方向为基于CRISPR原理的“下一代分子诊断技术的开发及其应用”,主要成果包括:(1)首次鉴定CRISPR-Cas12蛋白的反式切割活性,并开发了名为“HOLMES/福尔摩斯”的新型CRISPR分子诊断系统;(2)开发了基于Cas12a的新型DNA体外无缝拼接/编辑技术,提高了大片段、复杂片段DNA的体外拼接/编辑效率和准确率;(3)开发了基于Cas12a的新型生物元器件拼接标准(C-Brick);(4)开发了基于CRISPR技术的微生物基因编辑/基因调控系统,建成了不依赖于PAM位点的基因组编辑方法和基于CRISPR阵列的多基因高效转录调控的新方法。近年来,以第一作者或通讯作者身份在Cell Research、Nucleic Acids Research、ACS Synthetic Biology、Cell Discovery、Trends in Biotechnology、EBioMedicine等杂志发表论文40多篇,并申请了多项发明专利。主持国家自然科学基金优秀青年科学基金项目、国家自然科学基金面上项目和中国科学院人才项目等
王金,教授,上海吐露港生物科技有限公司总经理、首席科学家。2009年毕业于中国科学院上海生命科学研究院植物生理生态研究所,获得微生物学博士学位;2009—2012年在香港中文大学微生物学系从事博士后研究;2012—2022年先后任职于中国科学院上海生命科学研究院植物生理生态研究所、上海师范大学和深圳大学第一附属医院。其主要研究方向为基于CRISPR原理的“下一代分子诊断技术的开发及其应用”,主要成果包括:(1)首次鉴定CRISPR-Cas12蛋白的反式切割活性,并开发了名为“HOLMES/福尔摩斯”的新型CRISPR分子诊断系统;(2)开发了基于Cas12a的新型DNA体外无缝拼接/编辑技术,提高了大片段、复杂片段DNA的体外拼接/编辑效率和准确率;(3)开发了基于Cas12a的新型生物元器件拼接标准(C-Brick);(4)开发了基于CRISPR技术的微生物基因编辑/基因调控系统,建成了不依赖于PAM位点的基因组编辑方法和基于CRISPR阵列的多基因高效转录调控的新方法。近年来,以第一作者或通讯作者身份在Cell Research、Nucleic Acids Research、ACS Synthetic Biology、Cell Discovery、Trends in Biotechnology、EBioMedicine等杂志发表论文40多篇,并申请了多项发明专利。主持国家自然科学基金优秀青年科学基金项目、国家自然科学基金面上项目和中国科学院人才项目等