蛋白质是生命体最重要的生物大分子,具有极其广泛的功能和作用。传统的生物技术通常是利用细胞等生命活体来合成需要的蛋白质。然而,由于细胞存在生长、维护、排斥、退化、变异,乃至污染等诸多限制因素,使用细胞生命形式作为蛋白质合成体系一直面临着很多限制。随着当今人工智能技术高速发展,大量设计新型蛋白质分子和大规模制造目标活性蛋白质的需求日盛,传统的活体细胞合成蛋白质技术更是捉襟见肘,成为现代生物制造和生物医药快速发展的瓶颈。
无细胞蛋白质合成技术就是突破了自然界的“细胞工厂”模式,将细胞内进行的蛋白质生产过程搬到了细胞外,从单个细胞各自为战的小作坊,变成了集团化生产的体外蛋白质合成大厂。由于该技术的简单性、开放性、便携性、通用性和易放大性,为蛋白质合成的工程化提供了极大的自由度,让“合成万物”成为可能。
无细胞合成(cell-free synthesis)的种类繁多,其中尤以无细胞蛋白质合成过程最为复杂,应用也最为广泛。无细胞蛋白质合成研究的起源可以追溯到70年前,自1954年起,经历了3个发展阶段:实验室发现、试剂盒研究、产业化制造(图 1)。
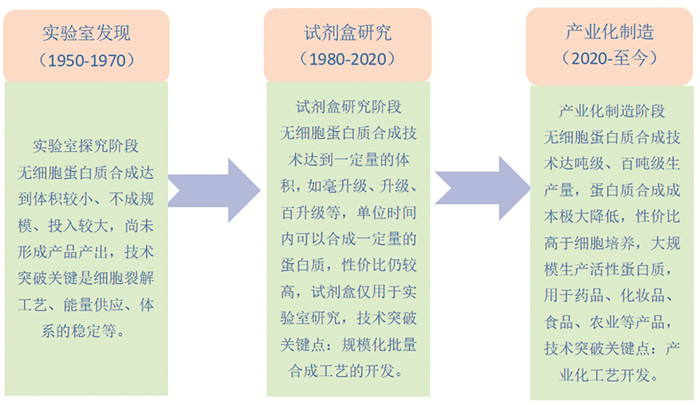
|
无细胞蛋白质合成技术出现70年来经历了3个发展阶段:实验室发现(1950‒1970)、试剂盒研究(1980‒2020)、产业化制造(2020‒至今)。 图 1 无细胞蛋白质合成技术的3个发展阶段 |
20世纪50年代初,第二次世界大战结束后,全球发达国家都致力于科技竞争,生命科学发展如火如荼。随着1953年Watson和Crick提出DNA双螺旋结构模型[1],DNA作为生命遗传信息的这一关键属性被揭示,生物学界开始聚焦于“遗传信息如何指导生命活动”这一核心问题。在这一背景下,哈佛大学的Zamecnik和Keller于1954年提出了突破性的实验构想——通过无细胞系统,在体外利用细胞中提取的活性物质,直接研究氨基酸聚合为蛋白质的反应过程,开创了体外蛋白质合成研究的先河[2]。
1961年,美国生物学家Nirenberg和Matthaei在使用大肠杆菌制备的S30提取物制作的无细胞体系中,加入人工合成的多聚尿嘧啶核苷酸(poly-U),成功翻译出一条仅含苯丙氨酸的多肽链。该实验验证了RNA而非DNA指导蛋白质合成的观点,并首次提及“无细胞蛋白质合成系统”(cell-free protein synthesis, CFPS)的概念[3]。CFPS作为一个通用平台则由Zubay等[4]在1973年的方法学综述中得到系统化阐述。1965年,来自美国威斯康星大学麦迪逊分校的Khorana团队合成了具有特定重复序列的RNA,如二聚体、三聚体、四聚体重复序列如(UCU)n、(CUC)n、(UAC)n、(ACU)n、(CUA)n、(…UAGUAG…)、(…UAAUAA…)、(…UGAUGA…),使用大肠杆菌S30提取物体系进行实验,成功破译了所有64个三联体密码子,并发现了UAA、UAG、UGA三个终止密码子[5]。同年,Holley团队使用酶促裂解加二维纸色谱的方法解析出第一个转运RNA (tRNA)的全序列,进一步证明tRNA在氨基酸插入过程中的关键作用[6]。1968年,美国生物学家Holley及其团队通过研究tRNA在蛋白质合成中的作用,使用氨基酸分析和多种分离技术(如凝胶电泳、色谱分析等)首次纯化并完整测定了单一tRNA——丙氨酸tRNA (tRNAAla)的序列,并证明tRNA的分子结构是由约70~80个核苷酸组成的(77个核苷酸),具有独特的“三叶草”结构和三维L型折叠。tRNA的发现及其在蛋白质合成中的作用,标志着分子生物学的一个重大突破,为氨基酸的准确插入提供了分子基础[7]。1968年,Nirenberg、Khorana和Holley因为在破译遗传密码方面的杰出贡献,共同获得诺贝尔生理学或医学奖[6]。无细胞蛋白质体外合成的实验技术,作为人们当时仅有的可控蛋白质合成手段,起到了重要的工具作用。至此,CFPS完成第一阶段的实验室进化。
2 无细胞蛋白质合成技术的商业化尝试:试剂盒研究从1958年开始到2000年的这40多年间,基于不同细胞来源的蛋白质体外合成实验陆续获得实现(表 1)。1958年,来自洛杉矶希望城(City of Hope)医院的Schweet团队首次利用兔网织红细胞提取物完成体外合成兔血红蛋白的实验,获得首个人工无细胞合成的活性蛋白质[8]。1964年,Pratt等[9]最早报道了小麦胚芽裂解物的蛋白质合成活性。2000年,Endo团队通过优化小麦胚芽提取物制备流程——去除抑制蛋白的ENDEXT技术,将脱脂小麦胚芽匀浆,经多次超速离心去除大颗粒及抑制因子,得到高活性提取液;补充能量再生体系(creatine phosphate/kinase),在双层或连续流动反应器中高效翻译,大幅提升了体系产量[10]。1972年,Sampson等[11]利用大鼠和小鼠肝细胞裂解物,实现了从小鼠和兔网织红细胞中提取的血红蛋白mRNA在体外的高效翻译,证明了哺乳动物组织提取物亦可支持完整蛋白质合成。1976年,Pelham等[12]进一步改良了兔网织红细胞体系,并发现HeLa、Krebs Ⅱ腹水瘤、CHO等多种来源的哺乳动物细胞均可通过制备无细胞系统,在体外高效翻译异源mRNA,为病毒RNA翻译及启动复合物研究提供了强有力工具。1978年,来自美国的Young团队报道了用酿酒酵母S-30裂解物体外翻译内源mRNA的研究,确立了酿酒酵母S-30裂解物具备翻译活性的重要事实,为后续酵母来源无细胞蛋白质合成系统的开发奠定了基础[13]。2009年,日本岛津公司的Suzuki等[14]使用基于Spodoptera frugiperda Sf21/Sf9细胞与含杆状病毒polyhedrin 5′-UTR增强子的表达载体,开发了昆虫Sf21/Sf9细胞来源的无细胞系统;2010年,岛津公司的Ezure等[15]进一步详述了Sf21/Sf9提取物体系的组分配置及应用示例,显示可以用于复杂蛋白质和多聚体的合成。
| 表 1 历史上典型的不同来源的蛋白质体外合成体系的实验室实现 |
随着无细胞蛋白质合成实验室技术的成熟,制造、销售实验室用无细胞蛋白质合成试剂盒的产品和企业逐步开始出现。最早提供商业化可购买的无细胞蛋白质合成试剂盒的公司,是来自美国威斯康星州麦迪逊市的Promega。Promega于1992年推出首款商业化无细胞蛋白合成试剂盒——TNT® Coupled Reticulocyte Lysate System。首发试剂盒产品中包括T7 Quick (Cat.# L1170)和SP6 Coupled (Cat.# L4600)两种体系,分别用T7 RNA多聚酶和SP6 RNA多聚酶转录DNA模板为mRNA。这款产品首次在商业化试剂盒中将DNA模板转录成mRNA和后续的蛋白质翻译反应集成于一个反应管中,从加入DNA模板开始,在30 ℃、60~90 min内即可快速产出模板编码的目标蛋白(常规产量可达约5 µg/mL),并可方便地添加辅因子、非天然氨基酸或其他如微粒体等以实现蛋白质翻译后修饰(post-translational modifications, PTM)。此款产品的主要优势在于操作简便、可定制化和支持信号肽剪切/糖基化等PTM。其“开放式”(open)体系还允许研究者对反应体系中的各种组分,如模板DNA/mRNA、氨基酸、能量系统、辅酶、缓冲液、修饰酶等进行灵活添加、调整或替换。虽然这套产品受限于当时的技术水平,具有体积小(50 μL)、产量有限(≤ 5 µg/mL)、对离子条件敏感、批次间活性可能波动以及单次成本较高等缺点,但作为无细胞蛋白质合成技术商业化的开创者,它无疑是一款成功的突破。自此,从20世纪90年代起,全球无细胞蛋白质体外合成领域迎来了小型研发试剂盒时代,无细胞蛋白质合成产业开始出现。
2001年,日本的Shimizu团队开发了一种名为PURE的系统(protein synthesis using recombinant elements)[16]。PURE系统的技术背景源于对传统细胞裂解提取液系统中存在的稳定性差和组分不确定性问题的解决,首次通过使用纯化的组分,在体外重构了一个原核来源的蛋白质合成系统。相较于传统路线,PURE系统实现了更高的可控性和稳定性,适用于基础生化研究、蛋白质工程、合成生物学等多个领域。PURE系统使用高度纯化的T7 RNA聚合酶和31种翻译因子(如起始因子、延伸因子、终止因子、氨酰tRNA合成酶等,均分别用大肠杆菌表达,通过亲和层析结合凝胶过滤方法纯化,获得纯度大于95%的原料),以及从大肠杆菌细胞中提取的70S核糖体(含54种蛋白质亚基和核糖核酸),再按照各个组分一定的浓度比例,进行体外重构而成。PURE系统还使用体外预先转录出的mRNA模板,既保证了相关因子、酶、tRNA、底物和能量分子的充足,又可以减少传统方法中的杂蛋白、核酸,以及核酸酶和蛋白酶等干扰因素,从而实现更加稳定、一致的体外蛋白质合成,成为优良的实验室研发体系。但PURE系统的分别制备高纯度原料的技术路线也造成了其量产难度大、反应体积小和制造成本高的缺点[17]。同时,其大肠杆菌来源的核糖体和其他酶因子,也使得其无法用于真核系统蛋白质合成研究。但无论如何,PURE系统开创了原料高质量制备,通过增加原料纯度和定制成分比例,以提高体外蛋白质合成系统稳定性和均一性的方法,为后来量产级高效体外合成系统的开发提供了重要线索。
基于Shimizu等2001年开发的PURE体系,日本的GeneFrontier (现由Cosmo Bio USA代理)于2011年进一步推出PUREfrex 1.0,可实现最高约160 µg/mL的蛋白产量,并在2015年升级为PUREfrex 2.0,将产量提升至约600 µg/mL[18];美国的Daicel Arbor Biosciences于2018年在SynBioBeta大会上发布首款myTXTL Linear DNA Expression System[19],并于2024年6月推出myTXTL Pro和Antibody/DS试剂盒,以“即加DNA,数小时内获得分析级产量”的简便流程助力抗体发现与蛋白质工程[20];Thermo Fisher Scientific则于约2011年推出1-Step Human Coupled IVT Kit,利用HeLa细胞裂解物在90 min内生成高达750 µg/mL的功能性蛋白,为真核蛋白研究提供了高产、高保真度的方案[21]。
无细胞蛋白质合成技术发展也经历了从简单到复杂的技术路线。早期的大部分无细胞蛋白质合成系统,其合成机器如核糖体、氨酰tRNA合成酶、翻译因子、折叠伴侣等主要来自原核细胞,如 E. coli等。目前常见的无细胞体外蛋白质合成体系来自原核细胞,如Bacillius megaterium[22]、Vibrio natriegens[23]、Pseudomonas putida[24]、Streptomyces venezuelae[25]。
与此相对,真核细胞来源的蛋白质翻译体系具有能够合成复杂蛋白质和复合物、更好的分子保真性、序列同源度更高、不含内毒素等多种优点。但是与原核细胞相比,真核细胞中的蛋白质合成体系构成更为复杂。首先,细胞中基本的、进化上保守的、负责完成蛋白质合成的酶、80S核糖体亚基、翻译起始因子、翻译延伸因子以及翻译终止因子等统称为翻译机器(translation machinery),而在真核细胞中,翻译机器的种类多达288种(以酵母细胞为例,哺乳动物的种类更多)[26],远远超过原核细胞中的翻译机器的种类数量(129种,以E. coli为例)(图 2)。其次,真核细胞中还存在大量的翻译调控因子(如GCN2、GCN4、mTOR等)和调控通路(如内质网应激引发的PERK通路、病毒感染引起的PKR通路、低铁离子引起的HRI通路、mRNA修饰通路等)[27, 28],以及更加复杂的分子伴侣和蛋白质质控系统(Hsp70、Hsp100、Zuo1、Ssb、Ssz1、Rqc1、Tae2、Ltn1、Cdc48、Npl4、Ufd1等)[29-31]。此外,真核细胞中天然的mRNA转录与蛋白质合成的物理空间和时间分割的特征,使得虽然真核细胞来源的体外蛋白质合成具有高质量、高纯度的优点,但同时也伴随着高难度、低效率、高成本的挑战,成为实现大规模体外蛋白质合成产业化的巨大壁垒。目前常见的无细胞体外蛋白质合成体系来自如酵母细胞[32]、小麦胚芽细胞[33]、兔网织红细胞[34]、中国仓鼠卵巢细胞[35]、昆虫细胞[36]、烟草细胞[37]等真核细胞。
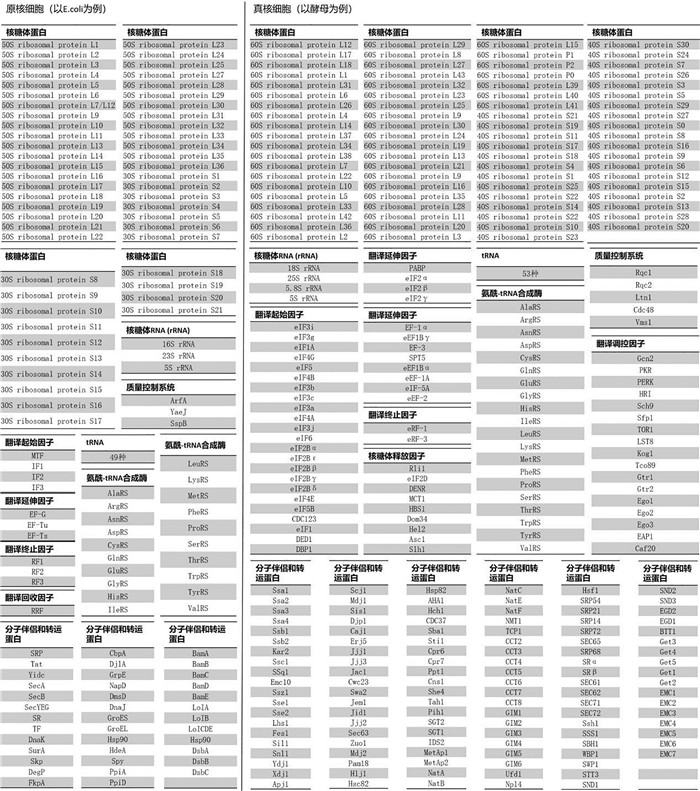
|
细胞中的蛋白质翻译过程可能是所有合成路径中最复杂的。从mRNA模板开始,蛋白质翻译过程中的分子机器主要包括核糖体、氨酰-tRNA合成酶、tRNA、翻译起始因子、翻译延伸因子、翻译终止因子、核糖体释放因子、质量控制系统、翻译调控因子,以及分子伴侣等。真核细胞还有更加复杂的调控,更多的tRNA种类。统一计算,原核细胞(如大肠杆菌)和真核细胞(酿酒酵母)中参与蛋白质合成和调控的这些关键翻译机器(蛋白质、酶、分子伴侣、rRNAs、tRNAs等)的总数分别达288种和129种。 图 2 真核与原核细胞中蛋白质翻译过程中的关键分子机器 |
基于原核和真核细胞中蛋白质合成机器的不同特性,利用不同的制备技术路线,过去20年中,全球生物公司陆续推出了数十种有代表性的商业化实验室级的CFPS试剂盒系统。目前上市的无细胞蛋白表达系统来源的系统包括大肠杆菌系统(E.coli extract, ECE)、酵母细胞(D2P)、兔网织红细胞(rabbit reticulocyte lysate, RRL)、麦胚(wheat germ extract, WGE)、昆虫(insect cell extract, ICE)和人源细胞系统(如HeLa cell)。参与制作和销售的代表公司包括Promega、Takara、Thermo、NEB以及康码-Healthcode等(表 2),他们将无细胞蛋白质合成反应带入了广大科研实验室,成为研究蛋白质合成机理、AI分子设计、高通量蛋白质合成、抗体药物开发等多方面的科研利器,大大促进了相关研究的进展。但这些产品,仍都具有反应体积小、单位价格高、性价比低、常温不稳定,以及缺乏专用合成设备和上下游配套的分离、纯化、定量系统等问题,导致无细胞蛋白质合成技术长期停留在实验室阶段。同时,这些试剂盒产品尚不足以支撑大规模、成体量的产业化下游产品制造和规模化市场应用。
| 表 2 商业化的实验室级无细胞蛋白质合成体系产品及代表企业 |
无细胞蛋白质合成系统(CFPS),具有多种应用场景,具有巨大的商业价值,如药物筛选、体外诊断、基础研究、药物制造及战场或现场应急等场景[38-40]。CFPS系统正在成为工业和高通量生产蛋白质疗法的强大平台,为解决生物制药工程中的问题提供了替代解决方案。此外,CFPS系统已成功应用于高通量药物筛选、非天然氨基酸导入、大规模蛋白质疗法生产、定制抗癌疫苗。这些成就突显了CFPS系统在未来的生物制药工程中具有巨大的应用潜力[41]。
目前,基于无细胞蛋白质体外合成技术,已经出现不少商业化应用公司,分布在全球多个国家,但鉴于大多数公司均处于产品研发、临床或提供对外服务阶段,尚未有成熟商业化的终端产品获批销售,这里选取了几家上市公司重点介绍。
3.1 Sutro Biopharma公司2003年,美国加州南旧金山的Sutro Biopharma是目前全球第一家上市的专注于无细胞蛋白生产商业化药物开发的公司(NASDAQ: STRO)。公司围绕专有的XpressCF/XpressCF+平台,使用大肠杆菌来源的无细胞蛋白质合成技术,专注于生物药物的研发。管线涵盖CD74和叶酸受体α靶向ADC (如STRO-001、STRO-002)、细胞因子、双特异性抗体等;生产工艺已从1 000 L (可产出≥500 L无细胞合成反应),计划放大至可实现5 000~10 000 L商业化批次[42],并与VGXI、Boehringer Ingelheim等CMO公司合作生产临床用质粒和蛋白质[43];Sutro通过“技术许可+里程碑/销售分成”与“大规模定制生产”并行的合作模式与包括Merck、Ipsen等,持续推动产能和管线扩张。Sutro目前已有2款产品获得临床审批,处于临床试验阶段[44]。自Sutro之后,陆续出现了多家以无细胞蛋白质合成技术合成蛋白质分子为目的进行商业化的上市公司(表 3)。
| 表 3 全球代表性商业化无细胞蛋白质合成系统 |
通过技术分拆,2015年,Sutro衍生出子公司Vaxcyte,专注于疫苗创新。通过使用现代合成技术,包括先进的化学技术和其无细胞蛋白质合成平台XpressCF (由Sutro Biopharma独家授权),该公司正在开发广谱结合疫苗和新型蛋白疫苗,以预防或治疗细菌性传染病。其主要候选疫苗为2款肺炎球菌结合疫苗(pneumococcal conjugate vaccine, PCV)。VAX-24是一种Ⅲ期就绪的24价广谱无载体PCV,正在开发用于预防侵袭性肺炎球菌病。VAX-31是该公司的下一代31价PCV,是目前临床上最广谱的PCV候选药物。Vaxcyte的其他在研管线还有新型A组链球菌疫苗、新型治疗性牙周炎疫苗以及新型志贺氏菌疫苗,但均处于临床前阶段[45]。
3.3 PeptideDream公司PeptiDream成立于2006年,总部位于日本神奈川县川崎市。公司利用其创始人Hiroaki Suga开发出的PDPS (peptide discovery platform system)技术,在体外实现含有天然和非天然氨基酸的大序列容量的多肽合成和筛选[46, 47]。PDPS技术通过构建包含数万亿种非标准肽的高多样性库,筛选出高亲和力和高选择性的大环肽候选物,开发用于治疗和诊断的肽类药物、小分子药物、肽-药物偶联物(PDC)和多功能肽偶联物(MPC)。此外,PeptiDream通过其子公司PDRadiopharma,在日本市场销售多种放射性药物和放射性诊断产品[48]。
4 中国无细胞蛋白质合成技术的研发近年来,全球无细胞领域研究进入快速发展期,2024年全球无细胞蛋白质合成相关论文发表数量是1970年的近20倍,几乎以每10年一倍的速度增加(图 3)。至2025年撰文时间为止,全球累计发表无细胞蛋白质合成相关论文21 5887篇。其中最早进行无细胞蛋白质合成研究的国家主要有美国、中国、英国、德国、日本、意大利、法国、加拿大等。
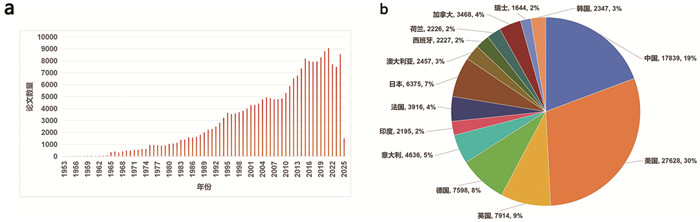
|
(a)过去70年中全球无细胞蛋白质合成科研文章数量曲线;(b)过去70年全球无细胞蛋白质合成科研文章数量的国家地区分布图 图 3 1953年来全球无细胞蛋白质科研文章 |
中国最早的无细胞蛋白质合成研究发表于1991年,此后快速发展,到目前为止已有相关学术论文17 839篇,占世界上无细胞蛋白质合成相关论文总数的8.26%,位居第2,论文占比最高的前六名国家分别为美国、中国、英国、德国、日本、意大利。
随着中国无细胞合成技术研究近20年的快速发展,涌现出了许多具有典型代表性的研发团队,例如中国科学院天津工业生物技术研究所、北京大学合成与功能生物分子中心、清华大学工业生物团队、厦门大学化学化工学院、上海交通大学、中国科学院深圳先进研究院等[49-52]。
不久前,清华大学工业生物催化教育部重点实验室卢元团队开发了一种通过动态阻遏器实现温度控制的无细胞表达系统并利用重组元素合成蛋白质[53, 54]。而华南理工大学生物科学与工程学院林影、郑穗平团队也搭建了基于无细胞蛋白合成技术的途径酶快速筛选体系[55]。中国科学院天津工业生物技术研究所张以恒研究员,首次提出体外生物转化(in vitro BioTransformation, ivBT)的概念,它是介于微生物发酵和酶催化之间的新质生物制造平台,实现了超限生物制造,可以从纤维素中体外无辅酶酶促合成淀粉,在保障粮食安全、推动新能源革命中具有战略价值[56]。ivBT是一个基于体外合成生物学的生物制造平台。多酶分子机器是ivBT的特有超高效生物催化剂。不会自我复制的多酶分子机器摆脱生物体繁殖的局限,超越细胞工厂合成极限,实现重要生物化学合成与超限生物能量转化,使得无细胞工厂成为未来生物制造的重要途径[57, 58]。作为体外生物转化合成中最复杂的技术分支,无细胞蛋白质合成技术通过70年的科技与产业积累,为未来体外生物转化平台的规模化发展提供了重要引领和参考。
中国无细胞蛋白质合成产业化的代表性企业是成立于2015年的康码(上海)生物科技有限公司(以下简称康码公司)。该公司是一家具备蛋白质合成底层核心技术,拥有自主独创的专利生物合成路线的中国生物技术企业[59]。康码公司自主开发出了DNA-to-Protein (D2P)体外蛋白质一步法合成技术,其蛋白质合成材料来源于真核细胞,且通用性强、产业化潜力大、技术工艺成熟。与传统的蛋白质合成技术相比,D2P无细胞蛋白质合成系统能够高效地制造复杂蛋白质、细胞因子,快速筛选抗体,同时有非天然氨基酸的特异性位点耦合技术,增加了药物的均质性和安全性,并能合成活体细胞难以合成的细胞毒素、癌症药物,以及AI设计的、自然界不存在的全新蛋白质分子。通过10年的技术研发和产业化积累,康码公司成功打破了传统细胞培养与细胞内表达蛋白质的体系,突破了全球无细胞蛋白质合成的产业化瓶颈,率先实现了百吨级批次产能和万吨级年产能,是目前世界上已知的拥有最大规模的无细胞蛋白质合成生产线的生物技术企业。
在抗击新冠疫情期间,康码公司联合湖北省疾控中心、中国科学院武汉病毒研究所、武汉大学、华中理工大学研发出了新冠病毒阻断剂康斯汀蛋白(Kansetin),该蛋白对新冠病毒SARS-CoV-2等呼吸道病毒具有极强亲和力。通过模拟人体ACE2结构,康斯汀蛋白高效结合新冠病毒,实现对病毒的物理黏连,达到高效阻断病毒侵染人体细胞的效果,且具有广谱抗病毒变种的能力[60]。康斯汀蛋白可有效阻断原始株、阿尔法、贝塔、德尔塔、奥密克戎等多种突变株[61]。与世界上其他已知的广谱抗新冠病毒变种的分子相比,康斯汀具有最强的体外抑制活性,对奥密克戎BA.1体外抑制常数IC50为121.9 pM,对奥密克戎BA.2为134.0 pM[62]。通过AI设计和D2P无细胞人工合成技术制造的康斯汀分子还具有极高的热稳定性,Tm > 82 ℃,常温可以保持活性2年以上,远远超过人体天然的ACE2蛋白分子[61]。2023年,“Lencoron®栏冠”鼻腔抗病毒喷雾医疗器械获得欧盟CE认证,出口土耳其、阿联酋、印尼、菲律宾和新加坡等国。2023年,康斯汀蛋白新冠阻断剂项目获上海市优秀科技成果转化项目奖。康斯汀蛋白新冠阻断剂是世界上第一款基于无细胞蛋白质合成技术大规模生产并销售的终端产品,是无细胞蛋白质合成产业化的先行者[63]。
2022年9月,康码公司基于5吨级无细胞蛋白质合成的产业化基础,启动第三代160吨级无细胞蛋白质合成产业化工厂的建设,并于2023年4月30日达产,成为世界上第一个百吨级无细胞蛋白质合成企业,真正实现了无细胞蛋白质合成技术的产业化突破。2023年10月,康码公司正式发布GMP级无细胞蛋白质合成专用的生物反应器,实现3小时高效蛋白质体外合成、满足GMP标准、单批次单体1 000 L反应、实时在线监控、定量合成、单位时间合成效率高达0.5 g/L/h,比传统细胞(如CHO细胞合成抗体)单位合成效率提高100倍以上。康码公司拥有目前世界上最大的吨级以上的无细胞蛋白质生产线:批次产能160吨级、年满载产能达3万吨。
利用康码公司合成的蛋白质产品和无细胞蛋白质合成生物反应设备,广大用户已成功开展各种药物开发、分子筛选、活性和结构实验等多种研究及应用[64-68]。康码公司基于量产级无细胞蛋白质合成技术和产能,陆续推出多款基于无细胞蛋白质合成的产品,获批上市的已达33款。其中包括19款国家和省级药监局备案审批的无细胞合成人源化蛋白原料和化妆品产品。这些产品都是全球市场上率先基于无细胞蛋白质合成技术批量化生产的终端产品(非研发试剂盒或开发服务),显示了无细胞蛋白质合成技术产业化后的强大产品能力和巨大市场潜力。
全球公开专利检索分析显示,从2015年康码公司进入无细胞蛋白质合成技术领域后,无细胞蛋白质合成专利申请迎来了第二次快速增长期,带来了产业的二次革新(图 4a、4b、4c)。康码公司目前拥有全球无细胞蛋白质合成技术路线最多的公开专利,其过去30年在该领域的专利贡献位居世界第一[69]。在全球有效专利申请排名前十的申请人中,康码公司的专利总数量位列第一(图 5a)。此外,康码公司维持有效专利(指在申请中的以及授权维持中的)的数量是第二名的2.3倍(图 5b)。按照技术的专利分类统计显示,康码公司在无细胞蛋白质合成领域的工程改造、制造工艺、生产设备、分子设计开发等四个技术维度的专利数量都远超其他申请人(图 5b、6a、6b)。
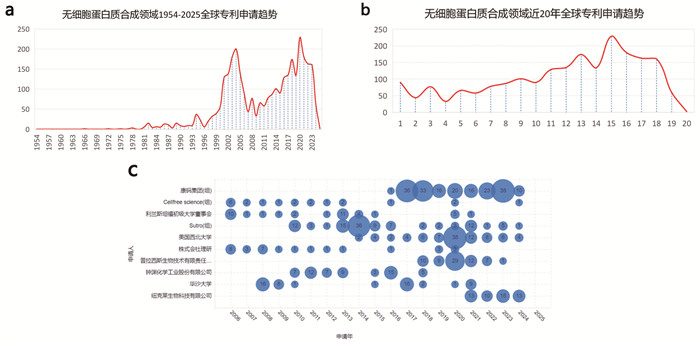
|
(a)近70年,全球无细胞蛋白质领域专利申请趋势图(1954‒2025);(b)近20年,全球无细胞蛋白质领域专利申请趋势图(2006‒2025);(c) 近20年,全球专利申请排名前十申请人的专利的申请趋势 图 4 无细胞蛋白质领域全球专利申请趋势和分布情况 |
|
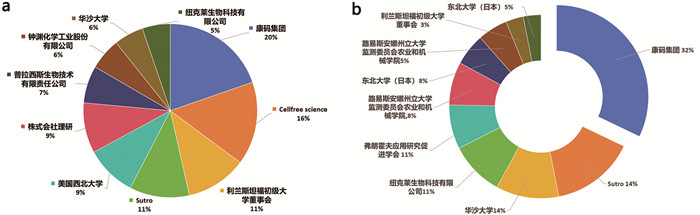
(a)截至2025年5月,全球所有专利排名前十的申请人分布图;(b)截至2025年5月,全球有效专利申请排名前十的申请人分布图 图 5 无细胞蛋白质领域全球专利申请人分布情况 |
|
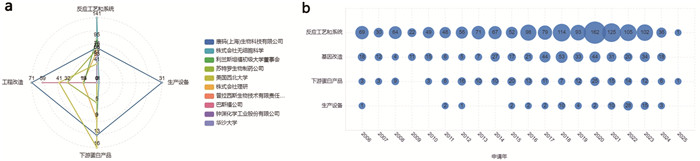
(a)截至2025年5月,无细胞蛋白质合成领域全球专利主要技术分类申请分布图;(b)过去20年,无细胞蛋白质合成领域全球专利主要技术分类申请数量年份分布图。图中数据仅统计截至统计日(2025年5月)的公开可检索专利数量,对于新提交的未公开专利申请(最长18个月)未予统计。 图 6 无细胞蛋白质合成领域全球专利主要技术分类申请分布情况 |
康码公司最近又与位于上海张江高科技园区的中国科学院上海高等研究院国家蛋白质科学研究(上海)设施开启合作,成立蛋白质智造联合实验室,计划在该实验室搭建康码公司最新开发的D2Pi-2.0 (DNA-to-Protein Intellgence 2.0)系统,形成全自动、高通量的蛋白质合成能力;其合成通量是同等体量细胞合成的100倍以上,预计每天能够合成数千种毫克级蛋白质分子。该系统建成后将对广大科研人员和产业界开放,为我国在生物医药研发、合成生物学等领域的基础研究和商业化开发的需求提供有力的技术支撑。
5 无细胞蛋白质合成技术面临的挑战与发展前景无细胞蛋白质合成技术仍然具有其特有的缺点和弱势。首先,无细胞系统的源头开发需要大量长期的人工构建,技术难度高;高效低成本合成体系成分复杂,开发周期长,技术壁垒高;对软硬件基础要求高,需要全产业链的整合。其次,基于无细胞蛋白质合成技术的单体蛋白质分子得率上限基本停留在1~5 g/L区间,其单位产能突破需要无细胞蛋白质生产系统持续优化,包括底盘细胞的进化更新、开发蛋白质合成的新型能源再生系统、稳定底物的供应、减少合成副产物、旁路循环控制和开发更加高效的体外正交翻译系统等[70]。再次,现阶段全球无细胞蛋白质合成的产业标准和质量标准不成熟,行业标准、国家标准和国际标准不完善,制约了行业发展。虽然无细胞蛋白质合成技术在速度、灵活性与可控性上的独特优势使其在生物医药、可持续制造及诊断技术中展现出不可替代的价值,正在重塑生物制造与合成生物学的研究范式,但是技术标准化、成本控制与复杂系统重构仍是未来突破的关键。随着人工智能、自动化与合成生物学的深度融合,无细胞蛋白质合成有望成为下一代生物经济的核心驱动力,推动从“实验室创新”到“产业化落地”的全面跨越[50, 71]。
随着AI技术的发展,研究者能够突破生物体基于基因编码蛋白质序列的限制,人类终于到了可以自主设计、创造全新非天然蛋白质的时代。人工无细胞体外合成蛋白质的产业化发展,对全球生物医药和生物制造产业具有重大意义[38, 72, 73]。大力发展新兴的无细胞蛋白质合成技术,建立无细胞工厂,一方面可以解决细胞合成蛋白质技术长期存在的菌株构建时间长、单位时间合成效率低、无法通用、合成成本高、制造设备复杂要求高、细胞排放环境污染多等诸多限制;另一方面可以利用其独有的开放性、通用性和先进性,进一步加大生物合成的效率和广度,像新能源汽车、光伏产业那样[73, 74],推进整体生物制造生产力发展。
| [1] |
Watson JD, Crick FHC. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid. Nature, 1953, 171: 737-8. |
| [2] |
Zamecnik PC, Keller EB. Relation between phosphate energy donors and incorporation of labeled amino acids into proteins. J Biol Chem, 1954, 209: 337-54. |
| [3] |
Nirenberg MW, Matthaei JH. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci U S A, 1961, 47: 1588-602. |
| [4] |
Zubay G. In‐vitro synthesis of protein in microbial systems. Annu Rev Genet, 1973, 7: 267-87. |
| [5] |
Khorana HG. Polynucleotide synthesis and the genetic code. Fed Proc, 1965, 24: 1473-87. |
| [6] |
Holley RW, Apgar J, Everett GA, et al. Structure of a ribonucleic acid. Science, 1965, 14: 1462-5. |
| [7] |
Holley RW, Apgar J, Everett GA, et al. Structure of a transfer RNA. Science, 1968, 160: 21-9. |
| [8] |
Schweet R, Lamfrom H, Allen E. The synthesis of hemoglobin in a cell-free system. Proc Natl Acad Sci U S A, 1958, 44: 1029-35. |
| [9] |
Pratt WB, Loomis CR. Protein synthesis by wheat germ ribosomes. Science, 1964, 146: 1003-4. |
| [10] |
Endo Y, Sawasaki T. ENDEXT: enhanced wheat germ cell-free protein expression system. J Biotechnol, 2000, 80: 1-7. |
| [11] |
Sampson J, Mathews MB, Osborn M, et al. Hemoglobin messenger ribonucleic acid translation in cell-free systems from rat and mouse liver and Landschutz ascites cells. Biochemistry, 1972, 11: 3636-40. |
| [12] |
Pelham HRB, Jackson RJ. An efficient rabbit reticulocyte system for mammalian mRNA translation. Eur J Biochem, 1976, 67: 247-56. |
| [13] |
Gallis BM, McDonnell JP, Hopper JE, et al. Endogenous messenger ribonucleic acid-directed polypeptide chain elongation in a cell-free system from the yeast Saccharomyces cerevisiae. J Bacteriol, 1975, 122: 719-26. |
| [14] |
Suzuki T, Ezure T, Ito M, et al. An insect cell-free system for recombinant protein expression using cDNA resources. Methods Mol Biol, 2009, 577: 97-108. |
| [15] |
Ezure T, Suzuki T, Shikata M, et al. A cell-free protein synthesis system from insect cells. Methods Mol Biol, 2010, 607: 31-42. |
| [16] |
Shimizu Y, Inoue A, Tomari Y, et al. Cell-free translation reconstituted with purified components. Sci Adv, 2001, 19: 751-5. |
| [17] |
GeneFrontier Corporation. PUREfrex®[EB/OL]. [2025-06-10]. https://purefrex.genefrontier.com/
|
| [18] |
GeneFrontier Corporation. PUREfrex®1.0[EB/OL]. [2025-06-10]. https://purefrex.genefrontier.com/products/cellfreeproteinsynthesiskits/purefrex1.0.html
|
| [19] |
Arbor Biosciences. Microsoft Word – 1801001_ArborBio_myTXTLLD_Press_Release. docx | Arbor Biosciences[EB/OL]. [2025-06-10]. https://arborbiosci.com/wp-content/uploads/2018/10/1801001_ArborBio_myTXTLLD_Press_Release.pdf
|
| [20] |
Arbor Biosciences. MyTXTL Pro Kit | Arbor Biosciences[EB/OL]. [2025-06-10]. Arbor Biosciences. https://arborbiosci.com/products/cell-free-protein-expression/mytxtl-pro-kit/
|
| [21] |
Thermo Scientific. Mammalian 1-Step IVT Systems | Thermo Scientific[EB/OL]. [2025-06-10]. https://www.thermofisher.cn/cn/zh/home/life-science/protein-biology/protein-expression/cell-free-protein-expression/mammalian-cell-free-protein-expression/overview-thermo-scientific-1-step-ivt-systems.html
|
| [22] |
Moore SJ, MacDonald JT, Wienecke S, et al. Rapid acquisition and model-guided engineering of product-tolerant microbial hosts. Proc Natl Acad Sci U S A, 2018, 115: E4340-9. |
| [23] |
Failmezger J, Scholz S, Blombach B, et al. Cell-free protein synthesis from fast-growing Vibrio natriegens. Front Microbiol, 2018, 9: 1146. |
| [24] |
Wang H, Li J, Jewett MC. Development of a Pseudomonas putida cell-free protein synthesis platform for rapid screening of gene regulatory elements. Synth Biol, 2018, 3: ysy003. |
| [25] |
Simon JM, Lai HN, Needham H, et al. Streptomyces venezuelae TX-TL - a next generation cell-free synthetic biology tool. Biotechnol J, 2017, 12: 1600678. |
| [26] |
Guo M, Schimmel P. Essential nontranslational functions of tRNA synthetases. Nat Chem Biol, 2013, 9: 145-53. |
| [27] |
Nahum S, Hinnebusch AG. Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell, 2009, 136: 731-45. |
| [28] |
Shut XE, Swanda RV, Qian S. Nutrient control of mRNA translation. Annu Rev Nutr, 2020, 40: 51-75. |
| [29] |
Brandman O, Stewart-Ornstein J, Wong DA, et al. A ribosome-bound quality control complex triggers degradation of nascent peptides and signals translation stress. Cell, 2012, 151: 1042-54. |
| [30] |
Kimpinga HH, Craig EA. The HSP70 chaperone machinery: J proteins as drivers of functional specificity. Nat Rev Mol Cell Biol, 2010, 11: 579-92. |
| [31] |
Kramer G, Shiber A, Bukau B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annu Rev Biochem, 2019, 88: 337-64. |
| [32] |
Hall CE, Johnson MC. Optimized extract preparation methods and reaction conditions for improved yeast cell-free protein synthesis. Biotechnol Bioeng, 2013, 110: 2643-54. |
| [33] |
Sawasaki T, Morishita R, Gouda MD, et al. Methods for high-throughput materialization of genetic information based on wheat germ cell-free expression system. Methods Mol Biol, 2007, 375: 95-106. |
| [34] |
Anastasina M, Terenin I, Butcher SA. A technique to increase protein yield in a rabbit reticulocyte lysate translation system. Biotechniques, 2014, 56: 36-9. |
| [35] |
Thoring L, Dondapati SK, Stech M, et al. High-yield production of "difficult-to-express" proteins in a continuous exchange cell-free system based on CHO cell lysates. Sci Rep, 2017, 7: 11710. |
| [36] |
Ezure T, Suzuki T, Higashide S, et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog, 2006, 22: 1570-7. |
| [37] |
Buntru M, Vogel S, Stoff K, et al. A versatile coupled cell-free transcription-translation system based on tobacco BY-2 cell lysates. Biotechnol Bioeng, 2015, 112: 867-78. |
| [38] |
Carlson ED, Gan R, Hodgman CE, et al. Cell-free protein synthesis: applications come of age. Biotechnol Adv, 2012, 30: 1185-94. |
| [39] |
Silverman AD, Karim AS, Jewett MC. Cell-free gene expression: an expanded repertoire of applications. Nat Rev Genet, 2020, 21: 151-70. |
| [40] |
Brookwell A, Oza JP, Caschera F. Biotechnology applications of cell-free expression systems. Life, 2021, 11: 1367. |
| [41] |
Jia X, Deng Z, Liu T. Progress of cell-free protein synthesis system and its applications in pharmaceutical engineering – a review. Wei Sheng Wu Xue Bao, 2016, 56: 530-42. |
| [42] |
Sutro Biopharma, Boehringer Ingelheim BioXcellence. Sutro Biopharma and Boehringer Ingelheim BioXcellence collaboration established first-in-class cell-free capabilities at commercial scale[EB/OL]. (2025-01-07)[2025-06-10]. https://www.sutrobio.com/sutro-biopharma-and-boehringer-ingelheim-bioxcellence-collaboration-established-first-in-class-cell-free-capabilities-at-commercial-scale
|
| [43] |
VGXI Inc. announces a strategic partnership with Sutro Biopharma Inc. to support growing clinical pipeline[EB/OL]. [2025-06-10]. https://vgxii.com/vgxi-inc-announces-a-strategic-partnership-with-sutro-biopharma-inc-to-support-growing-clinical-pipeline/
|
| [44] |
Sutro Biopharma, Inc. Annual Report 2024[EB/OL]. (2025-03-13)[2025-06-10]. https://ir.sutrobio.com/sec-filings/all-sec-filings/content/0000950170-25-038884/stro-20241231.htm
|
| [45] |
Vaxcyte. Vaxcyte Reports Second Quarter 2024 Financial Results and Provides Business Update[EB/OL]. (2024-08-06)[2025-06-10]. https://investors.vaxcyte.com/news-releases/news-release-details/vaxcyte-reports-second-quarter-2024-financial-results-and
|
| [46] |
Proprietary Drug Discovery Platform - System(PDPS)[EB/OL]. [2025-06-10]. https://www.peptidream.com/en/science/pdps/
|
| [47] |
PDR Radiopharma. PDR Radiopharma History[EB/OL]. (2022-03-28)[2025-06-10]. https://www.pdradiopharma.com/en/about/history
|
| [48] |
Curium Pharma, PeptiDream. Curium Announces Strategic Partnership with PeptiDream for Prostate Cancer Theranostics in Japan[EB/OL]. (2024-10-01)[2025-06-10]. https://www.curiumpharma.com/2024/10/01/s-strategic-partnership-for-prostate-cancer-theranostics
|
| [49] |
Li Z, Li Y, Lin X, et al. Supramolecular protein assembly in cell-free protein synthesis system. Bioresour Bioprocess, 2022, 9: 28. |
| [50] |
Chen Y, Xia W, Lu F, et al. Cell-free synthesis system: an accessible platform from biosensing to biomanufacturing. Microbiol Res, 2025, 293: 128079. |
| [51] |
Ji B, Qian Z, Xia X. Application of cell-free synthesis strategy in biomaterial research. Synth Biol J, 2021, 3: 658-7. |
| [52] |
Meyer C, Zhou C, Fang Z, et al. High-throughput experimentation using cell-free protein synthesis systems. Methods Mol Biol, 2022, 2433: 121-34. |
| [53] |
Cui Y, Chen X, Wang Z, et al. Cell-free PURE system: evolution and achievements. BioDesign Res, 2022, 9847014. |
| [54] |
Yang J, Wang C, Lu Y. A temperature-controlled cell-free expression system by dynamic repressor. ACS Synth Biol, 2022, 11: 1408-16. |
| [55] |
Wu L, Chang Y, Deng Z, et al. Efficient synthesis of gentamicin and its related products in industrial chassis cells. Synth Biol J, 2022, 3: 1277-91. |
| [56] |
Xu X, Zhang W, You Y, et al. Biosynthesis of artificial starch and microbial protein from agricultural residue. Sci Bull (Beijing), 2023, 68: 214-23. |
| [57] |
Shi T, Song Z, Song S, et al. In vitro BioTransformation (ivBT): a new frontier of industrial biomanufacturing. Synth Biol J, 2024, 5: 1437-60. |
| [58] |
Zhang YPJ, Zhu Z, You C, et al. In vitro BioTransformation (ivBT): definitions, opportunities, and challenges. Synth Biol Eng, 2023, 1: 10013. |
| [59] |
康码(上海)生物科技有限公司. 一种用于体外蛋白质合成的蛋白合成体系、试剂盒及其制备方法: 中国, CN108535489B[P]. 2019-04-19
|
| [60] |
Zhang H, Hu B, Lv P, et al. An ACE2-based decoy inhibitor effectively neutralizes SARS-CoV-2 Omicron BA.5 variant. Viruses, 2022, 14: 2387. |
| [61] |
Lv P, Hu B, Hua R, et al. A novelly designed protein antagonist confers potent neutralization against SARS-CoV-2 variants of concern. J Infect, 2022, 85: e72-6. |
| [62] |
Zhang H, Lv P, Jiang J, et al. Advances in developing ACE2 derivatives against SARS-CoV-2. Lancet Microbe, 2023, 4: e369-78. |
| [63] |
连线创始人. 康码生物创始人郭敏: 造出生物"光刻机" 实现蛋白质合成产业化[EB/OL]. (2023-02-03)[2025-06-10]. https://www.cls.cn/detail/1256283
|
| [64] |
Pulitzer JF, Colombo M, Ciaramella M, et al. New control elements of bacteriophage T4 pre-replicative transcription. J Mol Biol, 1985, 182: 249-63. |
| [65] |
Zhou Y, Wang M, Liu Y, et al. An HD-Zip Ⅲ transcription factor, BjPHVa, negatively regulates non-glandular trichome formation in Brassica juncea. Physiol Plant, 2024, 176: e14553. |
| [66] |
Zhang Y, Zhou J, Zhang Y, et al. The ABI3 transcription factor interaction and antagonism with ubiquitin E3 ligase ScPRT1 in Syntrichia caninervis. Genes, 2022, 13: 718. |
| [67] |
Tian Z, Shao D, Tang L, et al. Circular single-stranded DNA as a programmable vector for gene regulation in cell-free protein expression systems. Nat Commun, 2024, 15: 4635. |
| [68] |
Zhang K, Zhang K, Li X, et al. Systemic expression, purification, and initial structural characterization of bacteriophage T4 proteins without known structure homologs. Front Microbiol, 2021, 12: 674415. |
| [69] |
Meyer C, Nakamura Y, Raso BJ, et al. Analysis of the innovation trend in cell-free synthetic biology. Life, 2021, 11: 551. |
| [70] |
Liao C, Li H, Zheng H, et al. Optimization and application of cell-free system for protein expression. Chem Life, 2022, 42: 1493-501. |
| [71] |
Gao W, Cho E, Liu Y, et al. Advances and challenges in cell-free incorporation of unnatural amino acids into proteins. Front Pharmacol, 2019, 10: 611. |
| [72] |
Hodgman CE, Jewett MC. Cell-free synthetic biology: thinking outside the cell. Metab Eng, 2012, 14: 261-9. |
| [73] |
钱学森. 中国新能源汽车技术发展规划. 中国科学院院刊, 1986, 5: 23-31. |
| [74] |
Green M, Dunlop E, Hohl-Ebinger J, et al. Solar cell efficiency tables (Version 57). Prog Photovolt Res Appl, 2021, 29: 3-15. |
 2025, Vol. 37
2025, Vol. 37 

 郭敏,康码(上海)生物科技有限公司创始人、董事长。2005年中国科学技术大学博士毕业后赴美,在美国Scripps研究所任副教授、独立研究员、博士生导师,从事蛋白质翻译合成相关基础理论研究。先后获Sidney Kimmel学者奖、AAF学者奖及多国基金奖项;发表SCI论文70余篇,在Science、Nature、Nature Reviews Molecular Cell Biology等国际顶尖期刊发表论文30余篇,是蛋白质合成生物学基础理论及新功能应用研究领域的国际知名学者。获批国务院特殊津贴,全国归侨先进个人、上海市海外高层次人才、上海市领军人才、上海市优秀技术带头人、上海青年科技创业年度十大先锋。现任上海市侨联委员、浦东新区政协委员
郭敏,康码(上海)生物科技有限公司创始人、董事长。2005年中国科学技术大学博士毕业后赴美,在美国Scripps研究所任副教授、独立研究员、博士生导师,从事蛋白质翻译合成相关基础理论研究。先后获Sidney Kimmel学者奖、AAF学者奖及多国基金奖项;发表SCI论文70余篇,在Science、Nature、Nature Reviews Molecular Cell Biology等国际顶尖期刊发表论文30余篇,是蛋白质合成生物学基础理论及新功能应用研究领域的国际知名学者。获批国务院特殊津贴,全国归侨先进个人、上海市海外高层次人才、上海市领军人才、上海市优秀技术带头人、上海青年科技创业年度十大先锋。现任上海市侨联委员、浦东新区政协委员