在真核生物发育过程中,细胞类型的多样性依赖于特定基因在时间和空间上的特异性表达。这一复杂过程依赖于高度精细的基因表达调控机制,其中转录阶段的调控尤为关键。过去数十年的分子生物学与细胞生物学研究揭示了转录因子的组成、功能及其调控机制,为深入理解基因表达的分子基础奠定了坚实的理论基础和实验基础[1-4]。
转录过程的核心执行者是RNA聚合酶(RNA polymerase, RNAP),其以DNA为模板催化RNA的生物合成[5-7]。早在五十多年前,科学家从真核细胞中分离出三种RNA聚合酶,后续研究证实它们具有不同的基因转录分工:RNA聚合酶Ⅰ(Pol Ⅰ)主要负责合成核糖体RNA(rRNA)前体;RNA聚合酶Ⅱ(Pol Ⅱ)主要合成信使RNA(mRNA),是大多数蛋白质编码基因表达的关键;而RNA聚合酶Ⅲ(Pol Ⅲ)则转录转运RNA(tRNA)、5S rRNA和其他多种非编码RNA[8, 9] (图 1A)。
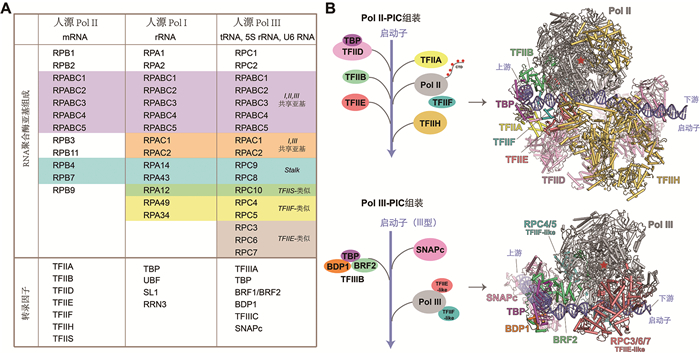
|
(A)人源Pol Ⅰ、Pol Ⅱ和Pol Ⅲ的组成成分(其中分别包含14、12和17个亚基)以及三类聚合酶转录过程涉及的转录因子。(B) Pol Ⅱ-PIC和Pol Ⅲ-PIC在启动子上顺序组装模式简图(左);Pol Ⅱ-PIC (PDB: 7EGB)和Pol Ⅲ-PIC (PDB: 8ITY)复合物结构(右)。 图 1 RNA聚合酶亚基组成和转录因子组成,以及转录前起始复合物结构 |
这三种RNA聚合酶不仅在亚基组成上各具特色,还各自依赖于一套特异的转录因子和调控机制[10-12]。在转录起始阶段,转录起始相关因子首先识别靶基因的启动子区域,随即招募并结合RNA聚合酶,形成转录前起始复合物(pre-initiation complex, PIC)。随后,相关转录因子协同作用解旋DNA双链,形成开放复合物(open complex, OC),并开始合成初生RNA。继而,聚合酶进入延伸阶段(elongation),稳定地延长RNA分子直至遇到转录终止信号,最终释放RNA转录产物及DNA模板,完成一个完整的转录周期[1, 13] (图 2)。
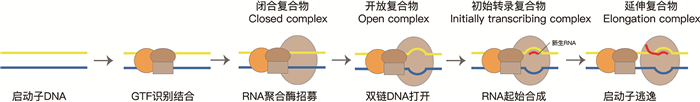
|
RNA聚合酶转录起始过程遵循以下基本步骤:(1)通用转录因子(GTFs)识别启动子,协助RNA聚合酶结合DNA并形成闭合复合物;(2) DNA局部解旋(“解链”),形成开放复合物;(3)随后开始合成短链RNA,形成初始转录复合物;(4) RNA聚合酶脱离启动子,形成延伸复合物,完成转录起始。 图 2 RNA聚合酶转录起始过程 |
转录过程是一个高度动态且多步骤协同的复杂生物学事件,从启动子识别、PIC组装、DNA双链打开、转录泡形成,到RNA合成起始及启动子逃逸,每一步都涉及大量因子的动态结合和解离,并伴随整体构象的重排[14, 15]。因此,系统解析其动态过程一直是转录研究领域的难点和核心热点。近期,冷冻电镜(Cryo-electron microscopy, Cryo-EM)技术革新带来的分辨率革命极大地推动了对RNA Pol Ⅱ/Pol Ⅲ转录起始过程中动态机制的探究。2023年,Chen等[16]通过改造天然的启动子序列,构建体外从头转录体系,在转录起始位点下游不同阶段精准地“卡住”Pol Ⅱ,结合冷冻电镜单颗粒结构解析,成功重构出16种连续构象状态的转录复合物(transcription complex, TC) TC2~TC17。该研究首次在单碱基分辨率下动态还原了Pol Ⅱ转录起始到早期延伸的全过程。近期,笔者团队采取类似的研究策略,展开对Pol Ⅲ转录起始的全过程研究,捕获了Pol Ⅲ转录从头起始到启动子逃逸进入转录延伸的多个关键状态[17]。这两项研究揭示了真核生物中两大核心转录系统在动态机制上的异同。本文将结合上述最近研究成果以及前期生物化学研究,系统阐述当前对真核基因转录起始过程的认知,尤其聚焦于Pol Ⅱ和Pol Ⅲ在转录起始与延伸阶段的分子机制。通过比较二者在招募起始因子、启动子识别、转录起始复合物组装及转录起始向延伸状态转变等方面的异同,旨在深化对真核转录调控网络的理解,并为解析细胞命运决定及其异常调控提供理论依据。
1 转录前起始复合物组装完整PIC的组装完成是转录起始的关键节点。PIC的形成是一个高度有序的过程,涉及RNAP与一系列通用转录因子(general transcription factors, GTFs)的动态协作,以确保转录能够在特定的位点准确起始。在真核生物中,被广为研究的是Pol Ⅱ介导的蛋白质编码基因转录,其PIC组装过程由TFⅡD对启动子区域的识别开始[18-20]。TFⅡD包含TBP (TATA-box binding protein)和多个TAFs (TBP-associated factors),在结合启动子后,进而招募TFⅡA以稳定TFⅡD-DNA复合物,TFⅡB则协助Pol Ⅱ和TFⅡF精准定位于转录起始位点(transcription start site, TSS)。随后,TFⅡE招募TFⅡH,后者具备易位酶/移位酶和激酶活性,负责打开DNA双链形成转录泡,并对Pol Ⅱ大亚基的C端(C-terminal domain, CTD)重复序列进行磷酸化,启动后续的转录过程[21] (图 1B)。值得注意的是,这一过程还受到中介复合物(Mediator)的动态调控,该复合物作为共激活因子在增强子与启动子之间建立联系,显著提高转录效率[22, 23]。
相比之下,Pol Ⅲ转录基因的启动子结构与其PIC组装机制皆与Pol Ⅱ体系不同。Pol Ⅲ主要负责细胞内大量短链非编码RNA的转录,其启动子可分为三种类型[24-27]:Ⅰ型(如5S rRNA基因)包含位于基因内部的序列元件A框、中间元件和C框;Ⅱ型(如tRNA基因)具有位于基因内部的特征性A框和B框序列元件;Ⅲ型(如U6 snRNA、7SK RNA和H1 RNA等基因)在高等脊椎动物中特有,不同于Ⅰ型和Ⅱ型,其特异性序列识别元件TATA框和近端序列元件(proximal sequence element, PSE)都位于基因外部。在Ⅱ型启动子的PIC组装中,TFⅢC首先识别这些基因内部的A框和B框,并招募TFⅢB (由TBP、TFⅡB-related factor 1 (BRF1)和B double prime 1 (BDP1)组成)至启动子上游区域,TFⅢB随即稳定结合DNA并进一步招募Pol Ⅲ。Ⅰ型启动子的转录起始则需要额外特异因子TFⅢA的参与以识别中间元件并进一步协助TFⅢC的结合。而Ⅲ型启动子主要依赖于SNAPc (small nuclear RNA activating protein complex)对上游PSE框的识别,随即招募TFⅢB至启动子上游区域[28, 29] (图 1B)。值得一提的是,BRF2代替BRF1,特异性地参与Ⅲ型启动子的转录起始。总之,与Pol Ⅱ不同,Pol Ⅲ-PIC的组装不依赖于TFⅡA、TFⅡD或TFⅡH等Pol Ⅱ所需的通用转录因子,也不涉及Mediator复合物,其PIC组装过程更为精简且高度特异化,这一精炼的PIC组装机制使其能快速响应细胞对大量短链非编码RNA的需求。
近年来,多个研究团队陆续报道了这两类聚合酶在PIC组装过程中关键节点的高分辨率结构。2021年,Chen等[30]报道了哺乳动物不同类型启动子上Pol Ⅱ-PIC复合物的冷冻电镜结构,系统地揭示了基于TFⅡD的PIC识别不同类型基因启动子并完成顺序装配的完整动态过程(图 1B)。同年,国际上三个研究团队几乎同期报道了Pol Ⅱ-PIC-Mediator超大复合物结构,首次在原子水平上解析了Mediator与Pol Ⅱ-PIC的相互作用界面,揭示了PIC-Mediator的动态组装过程以及Mediator调控Pol Ⅱ CTD磷酸化的分子机制[31-33]。2022年,基于+1核小体的Pol Ⅱ-PIC-Mediator复合物被报道,这一突破性研究首次在原子水平上揭示了转录前起始复合物与+1核小体的多个直接相互作用[34]。这一发现为理解染色质环境下转录起始的精确调控机制提供了关键结构基础,填补了从裸露DNA模板到染色质模板转录起始研究的空白。此外,对Pol Ⅲ-PIC的研究也取得了多项突破。2023年,两个研究团队分别报道了TFⅢA、TFⅢB和TFⅢC协同识别Ⅰ型启动子以及TFⅢB和TFⅢC协同识别Ⅱ型启动子进行转录起始的分子机制[35, 36]。两篇报道均系统揭示了TFⅢC亚基特异性识别基因内部启动子元件的结构基础,且探究了TFⅢC在招募下游因子和Pol Ⅲ中的重要作用。同时,SNAPc介导Ⅲ型启动子识别的完整PIC构象也被报道,揭示了SNAPc通过其SNAP3/4亚基识别启动子上游近端序列元件,并与TFⅢB协同招募Pol Ⅲ的分子机制[37-39] (图 1B)。这一系列突破性的结构研究深化了人们对真核生物转录起始调控核心机制的认知。
2 转录起始RNA聚合酶和GTFs在组装形成完整PIC之后,在转录起始阶段都遵循了DNA打开-转录泡形成-起始合成RNA的基本流程[1] (图 2)。转录起始的关键步骤之一是双链DNA局部打开,形成转录泡的结构区域,以暴露出模板单链供后续RNA的合成。在Pol Ⅱ体系中,DNA打开是一个高度依赖能量的过程,由TFⅡH介导。TFⅡH中的XPB亚基具有DNA易位酶/移位酶活性,其在ATP水解驱动下实现对启动子区双链DNA的局部解链,从而形成大约10~13个碱基对的转录泡[13, 30, 40]。与之不同,Pol Ⅲ体系中双链DNA的打开更为简化[41]。Pol Ⅲ转录基因的启动子区域常包含一段富含A-T碱基对的序列,该区域热稳定性较低,更容易发生局部解链[37]。这一特点使得Pol Ⅲ体系在缺乏TFⅡH等解旋因子的情况下,也能实现启动子区域的自发打开。此外,TFⅢB复合物与启动子DNA的紧密结合以及Pol Ⅲ本身的构象变化,也在DNA局部解链过程中起到了协助作用。这种“自主式”的DNA打开机制使Pol Ⅲ的转录起始更为高效。
转录泡一旦形成,模板链(template strand)DNA进入RNA聚合酶的活性中心空腔,被活性中心空腔的数个高度保守碱性氨基酸(如赖氨酸、精氨酸)通过静电作用稳定,以便于后续RNA的高效和高保真合成[16, 42, 43]。而非模板链(non-template strand)因未参与碱基配对,其结构柔性较高,因此在冷冻电镜结构解析中往往呈现为模糊、不连续,甚至完全缺失的密度(图 3)。Pol Ⅱ在转录起始阶段,TFⅡF的α亚基中一段碱性螺旋结构可能与非模板链相互作用,在转录起始阶段起到稳定转录泡的作用[16]。与此不同,Pol Ⅲ在转录起始阶段,其非模板链的稳定并不依赖GTFs,而是直接由Pol Ⅲ自身的lobe模块所形成的狭窄碱性空腔进行捕捉和稳定[17]。这一结构特征不仅在转录起始过程中发挥作用,也参与Pol Ⅲ转录终止过程,通过识别非模板链的poly-T序列,促进转录终止[44, 45]。
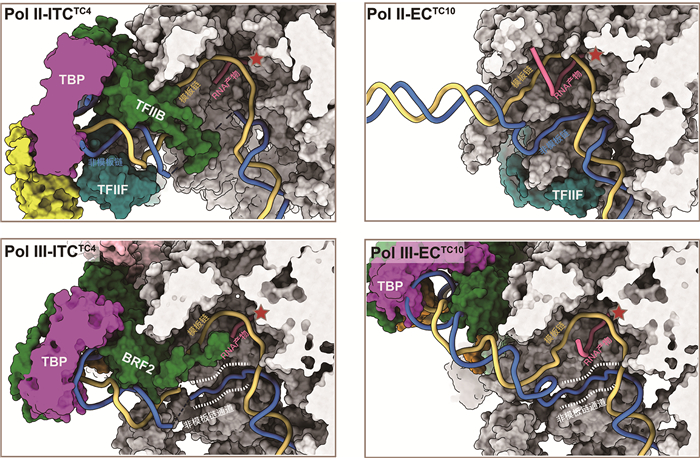
|
分别展示了转录4 nt和10 nt RNA产物时的整体构象。 图 3 Pol Ⅱ和Pol Ⅲ转录过程中ITC和EC构象比对 |
在转录起始早期阶段,RNA聚合酶开始催化合成短链RNA,但由于此时新生RNA与模板DNA链之间的结合不稳定,一部分新生RNA在延伸前就被释放,产生流产性RNA产物(abortive transcripts)[46, 47]。另外,在此阶段,下游DNA逐步被解链并进入RNA聚合酶中,伴随着RNA链的逐步延长,RNA-DNA配对逐渐稳定,转录泡持续扩张,但转录复合物的整体构象仍然和PIC的构象高度相似,此阶段被称为初始转录复合物(initially transcribing complex, ITC)。
3 转录起始向延伸转变转录起始向延伸的转变是转录循环的关键节点,标志着RNA聚合酶从启动子识别与RNA链初步合成状态,过渡到稳定、高效的RNA延伸状态[48]。这一过程伴随着转录泡局部坍塌、启动子的逃逸(promoter escape)和GTFs解离或重排等,从而完成从PIC到ITC,再到延伸复合物(elongation complex, EC)的动态构象转换。虽然早期的生化研究已揭示ITC到EC过渡期间转录复合物的组成和结构发生显著变化,但其精细的分子机制仍大多未明。Chen等[16]对Pol Ⅱ转录起始动态连续过程的研究为这一过程提供了新的见解:(1) Pol Ⅱ-TC2到TC9属于ITC状态,而从TC10之后则进入早期转录延伸状态,表明Pol Ⅱ转录ITC到EC的转变发生在10 nt RNA合成时;(2)该转变过程伴随转录泡的巨大变化。在RNA链合成至9 nt的过程中,转录泡由约13 nt逐渐扩大至21 nt,随着转录进入延伸阶段,转录泡迅速由21 nt“坍塌”至11 nt。同时,结合在pol Ⅱ上游的双链DNA发生明显位移,进入EC状态特有的稳定DNA通道,这一变化可能是实现启动子逃逸的关键;(3)该转变过程伴随GTFs的巨大构象变化。在ITC状态中,GTFs如TFⅡB、TFⅡF与Pol Ⅱ稳定结合,共同协助转录起始。随着转录进入延伸状态,部分GTFs与Pol Ⅱ的稳定互作被打破,便于进一步的延伸因子相关的调控(图 3、4)。
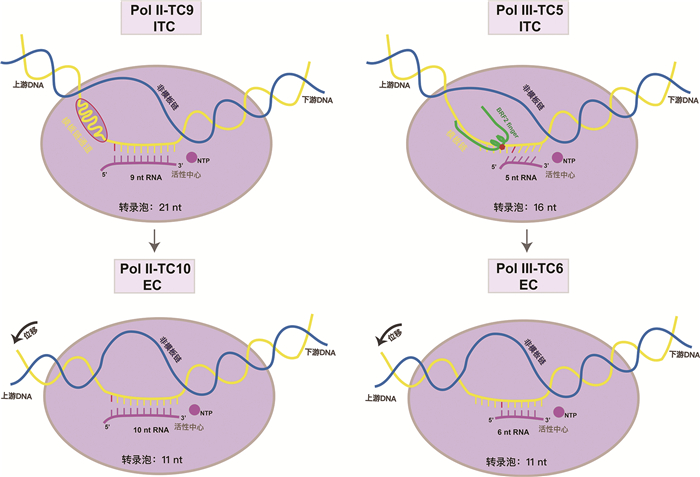
|
Pol Ⅱ转录ITC到EC转变发生在RNA合成9 nt之后,主要依赖模板链DNA在模板链通道中的“挤压”;而Pol Ⅲ转录ITC到EC转变更早,发生在RNA合成5 nt之后,且依赖于GTFs、Pol Ⅲ与启动子DNA的精确结合模式。模板链和非模板链分别显示为黄色和蓝色,转录起始位点标为红色。 图 4 Pol Ⅱ和Pol Ⅲ转录过程中ITC到EC转变驱动力对比模式图 |
笔者团队对人源Pol Ⅲ转录动态过程的研究表明,Pol Ⅲ转录起始向延伸转变这一过程与Pol Ⅱ呈现明显差异[17]。首先,Pol Ⅲ-TC4和TC5属于ITC状态,而从TC6之后则进入转录延伸状态,表明Pol Ⅲ转录ITC到EC的转变发生在6 nt RNA合成时,这一转折点早于Pol Ⅱ;其次,Pol Ⅲ在启动子逃逸之后,尽管其与GTFs之间的稳定相互作用被打破,但GTFs,如SNAPc、TFⅢB与启动子之间的相互作用依然维持稳定,从而通过这种方式确保转录的快速重新起始(图 3)。再次,Pol Ⅲ的ITC到EC转变过程中所依赖的分子驱动力机制与Pol Ⅱ也存在本质差异(详见下文)。
4 转录起始向延伸转变驱动力那么,推动ITC向EC过渡的“驱动力”究竟是什么?在Pol Ⅱ转录ITC阶段,下游DNA逐步解链并进入活性中心区域,RNA-DNA配对增加,同时模板链DNA逐渐进入Pol Ⅱ核心特定的模板链DNA通道T (channel T),该通道逐渐被占据并接近饱和。当RNA链延伸到10 nt时,通道T无法再容纳模板链DNA,迫使其扩展,从而触发构象变化并过渡到早期延伸[8]。此外,该转变过程显著依赖于RNA-DNA杂合体的稳定性以及核苷三磷酸(nucleotide triphosphate, NTP)水解反应的驱动。NTP的持续水解不仅推动RNA链延伸,同时也带动RNA-DNA杂合体在Pol Ⅱ活性空腔中沿5′→3′方向“前移”,这一推动力对模板链造成空间压力,是引发转录泡、GTFs重构和启动子逃逸的重要驱动力(图 4)。
值得注意的是,尽管Pol Ⅲ也具备高度保守的模板链通道结构,但其ITC向EC的转变机制却与Pol Ⅱ完全不同。笔者团队近期对Pol Ⅲ动态转录起始过程的研究表明,Pol Ⅲ向转录延伸的转变相较于Pol Ⅱ更早,且并不依赖于模板链DNA在模板链通道中的“挤压”,而是依赖于GTFs、Pol Ⅲ与启动子的精确结合模式[17]。具体来说,当Pol Ⅲ在Ⅲ型启动子上转录5 nt RNA时,其RNA-DNA杂合体呈现非经典的“倾斜”配对构象,此构象产生的原因是模板链DNA处于pre-translocate状态,而新生RNA处于post-translocate状态,由此在RNA- DNA杂合体内形成不对称形变(图 4)。这种构象被转录因子BRF2的finger结构域所稳定,该结构域嵌入RNA-DNA通道中,并与Pol Ⅲ核心直接相互作用,以这种方式阻碍模板链DNA的迁移。在NTP持续水解的驱动力下,第6个核苷酸被添加至RNA链3′端,在RNA-DNA杂合体“前移”推动下,BRF2的finger结构域从活性中心中撤离。这一结构域的撤离不仅解除了对模板链DNA的阻碍,也随即触发一系列转录因子与Pol Ⅲ之间的重构,即BRF2核心结构域及与其互作的TBP、BDP1和SNAPc的解离,从而完成Pol Ⅲ的启动子逃逸。
相较之下,细菌RNA聚合酶在ITC向EC转变过程中遵循“scrunching”机制,即DNA的压缩产生应力,从而促使不稳定的ITC中间体向EC转换并实现启动子逃逸[49]。尽管细菌RNA聚合酶和哺乳动物Pol Ⅱ遵循类似的原则,但分子机制存在差异:细菌RNA聚合酶依赖模板链和非模板链的推力,而哺乳动物Pol Ⅱ则主要依赖模板链;此外,细菌的σ因子与非模板链作用,而哺乳动物Pol Ⅱ的TFⅡB与模板链DNA接触,这些转录因子在各自系统中以不同的方式参与调控ITC向EC的转变。而Pol Ⅲ在此转换过程中采取的机制更加独特。在Ⅲ型启动子转录起始过程中,转录因子BRF2发挥至关重要的作用,而其同源蛋白BRF1参与Ⅰ型和Ⅱ型转录起始动态过程的机制仍不明确,有待进一步研究。
5 转录延伸阶段GFTs保留机制Chen等[16]研究表明,当Pol Ⅱ转录进入延伸阶段后,GTFs呈现分层式、动态性的解离模式。其中,TFⅡF、TFⅡE仍与Pol Ⅱ保持结合,不仅支持转录起始,在早期转录延伸阶段,还起到阻碍负调控因子(如DRB sensitivity-inducing factor, DSIF)和延伸因子(如super elongation complex, SEC)提前结合的作用,防止转录过早进入暂停状态,从而确保转录从早期延伸阶段平稳过渡至产RNA活跃的延伸状态。相较而言,TFⅡD是最早解离的因子。在早期延伸阶段,TFⅡD中的组成成分(如TAF1和TBP)已开始部分解离,而到晚期延伸阶段几乎完全解离。此外,TFⅡB、TFⅡE、TFⅡF、TFⅡH等在延伸过程中可能以较弱的方式保留,或与启动子区残留接触,但结合状态较不稳定。总体来看,GTFs在延伸复合物中的保留情况可能受到多种因素调控,包括启动子序列组成、Pol Ⅱ CTD的磷酸化状态、转录因子的协同作用以及染色质环境等。这些因素共同决定了不同GTFs在转录早期向延伸阶段过渡中的滞留程度及其功能意义。
Wang等[17]研究表明,当Pol Ⅲ转录进入延伸阶段后,其GTFs仍然稳定滞留在启动子上游区域,形成类似“占位”状态。长期以来,科学界一直推测Pol Ⅲ具备一种快速重复利用启动子的能力,以满足其在合成tRNA、5S rRNA等小RNA时的高转录频率需求[9, 50]。特别是在这一类短、无内含子的高表达基因上,Pol Ⅲ不依赖于复杂的染色质重构或新一轮装配,就能迅速重新起始转录,但这一机制始终缺乏直接结构证据支持。该研究首次以冷冻电镜结构形式呈现了转录起始因子在一次转录事件结束后仍保留在启动子附近的构象状态,从结构层面证实了Pol Ⅲ再起始模型的合理性。这类滞留状态下的起始因子与DNA仍保持稳定结合,而其空间位置适于重新招募Pol Ⅲ复合物,构成新一轮起始。这种类似“坐等”机制,使Pol Ⅲ得以在无须重新识别启动子或无需重组装因子的情况下,直接进入下一轮转录周期,极大提高了转录效率。因此,早期完成转录延伸转换和快速再起始机制,可能正是Pol Ⅲ在执行高频率转录任务中所采用的策略。
综上所述,转录起始向延伸的转变过程在Pol Ⅱ和Pol Ⅲ中均涉及转录泡动态重构、启动子逃逸及转录因子重排,但其发生时机、因子保留机制及驱动力存在显著差异,反映出不同RNA聚合酶系统对转录效率与调控精度的适应性机制。
6 总结与展望真核生物中Pol Ⅱ和Pol Ⅲ在转录起始过程中均体现出高度保守的基本原则,例如PIC组装顺序性、启动子特异性识别与启动子DNA的局部解旋,这些基本机制保证了转录起始的精准性和转录过程的有序进行。然而,尽管在这些基本原则上存在高度保守性,Pol Ⅱ和Pol Ⅲ在多个关键方面仍展现出显著差异,包括所依赖的转录因子类型、识别的启动子类型、复合物构象的动态变化以及调控策略等方面。具体来说,Pol Ⅱ主要介导mRNA的合成,其转录调控通常涉及多种启动子上游调控元件、增强子、染色质环境及染色质重塑因子,具有复杂性和可调性。这使得Pol Ⅱ系统在应对发育信号、环境刺激和细胞周期变化方面具有较强的响应能力。相对而言,Pol Ⅲ则主要负责合成如5S rRNA、tRNA和U6 snRNA等高丰度的非编码RNA,其转录调控更加高效和模块化,具备快速启动和高效转录的能力,以满足细胞对基础代谢RNA的稳定需求。这些机制方面的异同不仅体现出两种RNA聚合酶在功能定位和调控策略上的根本差异,也反映了真核转录系统在长期进化过程中演化的多样性和特异性。这种分工与协同的调控机制确保了细胞对不同类型RNA的合成效率与精确度。
未来,研究者需进一步引入更接近生理状态的染色质模板,以取代传统的裸DNA模板,从而解析染色质环境下转录因子组装、DNA构象变化及RNA聚合酶动态转录的真实过程。结合Cryo-EM、单分子成像(single-molecule imaging)、原位结构解析(in situ structural biology)和高分辨染色质免疫共沉淀等先进技术,将有助于构建一个更加动态、统一和结构功能关联明确的转录循环模型,进而深化对基因表达调控本质的理解。
| [1] |
Cramer P. Organization and regulation of gene transcription. Nature, 2019, 573: 45-54. |
| [2] |
Weidemuller P, Kholmatov M, Petsalaki E, et al. Transcription factors: bridge between cell signaling and gene regulation. Proteomics, 2021, 21: e2000034. |
| [3] |
Zhu H, Wang G, Qian J. Transcription factors as readers and effectors of DNA methylation. Nat Rev Genet, 2016, 17: 551-65. |
| [4] |
Roeder RG. The eukaryotic transcriptional machinery: complexities and mechanisms unforeseen. Nat Med, 2003, 9: 1239-44. |
| [5] |
Roeder RG, Rutter WJ. Multiple forms of DNA-dependent RNA polymerase in eukaryotic organisms. Nature, 1969, 224: 234-7. |
| [6] |
Roeder RG, Rutter WJ. Specific nucleolar and nucleoplasmic RNA polymerases. Proc Natl Acad Sci U S A, 1970, 65: 675-82. |
| [7] |
Roeder RG. 50+ years of eukaryotic transcription: an expanding universe of factors and mechanisms. Nat Struct Mol Biol, 2019, 26: 783-91. |
| [8] |
Cramer P, Armache KJ, Baumli S, et al. Structure of eukaryotic RNA polymerases. Annu Rev Biophys, 2008, 37: 337-52. |
| [9] |
Arimbasseri AG, Rijal K, Maraia RJ. Comparative overview of RNA polymerase Ⅱ and Ⅲ transcription cycles, with focus on RNA polymerase Ⅲ termination and reinitiation. Transcription, 2014, 5: e27639. |
| [10] |
Engel C, Neyer S, Cramer P. Distinct mechanisms of transcription initiation by RNA polymerases Ⅰ and Ⅱ. Annu Rev Biophys, 2018, 47: 425-46. |
| [11] |
Vannini A, Cramer P. Conservation between the RNA polymerase Ⅰ, Ⅱ, and Ⅲ transcription initiation machineries. Mol Cell, 2012, 45: 439-46. |
| [12] |
Turowski TW, Boguta M. Specific features of RNA polymerases Ⅰ and Ⅲ: structure and assembly. Front Mol Biosci, 2021, 8: 680090. |
| [13] |
Greber BJ, Nogales E. The structures of eukaryotic transcription pre-initiation complexes and their functional implications. Subcell Biochem, 2019, 93: 143-92. |
| [14] |
Shandilya J, Roberts SGE. The transcription cycle in eukaryotes: from productive initiation to RNA polymerase Ⅱ recycling. Biochim Biophys Acta, 2012, 1819: 391-400. |
| [15] |
Archuleta SR, Goodrich JA, Kugel JF. Mechanisms and functions of the RNA polymerase Ⅱ general transcription machinery during the transcription cycle. Biomolecules, 2024, 14: 176. |
| [16] |
Chen X, Liu W, Wang Q, et al. Structural visualization of transcription initiation in action. Science, 2023, 382: eadi5120. |
| [17] |
Wang Q, Ren Y, Jin Q, et al. Structural insights into human Pol Ⅲ transcription initiation in action. Nature, 2025. DOI:10.1038/s41586-025-09093-w |
| [18] |
Thomas MC, Chiang CM. The general transcription machinery and general cofactors. Crit Rev Biochem Mol Biol, 2006, 41: 105-78. |
| [19] |
Zawel L, Reinberg D. Initiation of transcription by RNA polymerase Ⅱ: a multi-step process. Prog Nucleic Acid Res Mol Biol, 1993, 44: 67-108. |
| [20] |
Buratowski S, Hahn S, Guarente L, et al. Five intermediate complexes in transcription initiation by RNA polymerase Ⅱ. Cell, 1989, 56: 549-61. |
| [21] |
Svejstrup JQ, Wang Z, Feaver WJ, et al. Different forms of TFⅡH for transcription and DNA repair: holo-TFⅡH and a nucleotide excision repairosome. Cell, 1995, 80: 21-8. |
| [22] |
Allen BL, Taatjes DJ. The Mediator complex: a central integrator of transcription. Nat Rev Mol Cell Biol, 2015, 16: 155-66. |
| [23] |
Soutourina J. Transcription regulation by the Mediator complex. Nat Rev Mol Cell Biol, 2018, 19: 262-74. |
| [24] |
White RJ. Transcription by RNA polymerase Ⅲ: more complex than we thought. Nat Rev Genet, 2011, 12: 459-63. |
| [25] |
Schramm L, Hernandez N. Recruitment of RNA polymerase Ⅲ to its target promoters. Genes Dev, 2002, 16: 2593-620. |
| [26] |
Geiduschek EP, Kassavetis GA. The RNA polymerase Ⅲ transcription apparatus. J Mol Biol, 2001, 310: 1-26. |
| [27] |
Lata E, Choquet K, Sagliocco F, et al. RNA polymerase Ⅲ subunit mutations in genetic diseases. Front Mol Biosci, 2021, 8: 696438. |
| [28] |
Mittal V, Ma B, Hernandez N. SNAPc: a core promoter factor with a built-in DNA-binding damper that is deactivated by the Oct-1 POU domain. Genes Dev, 1999, 13: 1807-21. |
| [29] |
Huang Y, Maraia RJ. Comparison of the RNA polymerase Ⅲ transcription machinery in Schizosaccharomyces pombe, Saccharomyces cerevisiae and human. Nucleic Acids Res, 2001, 29: 2675-90. |
| [30] |
Chen X, Qi Y, Wu Z, et al. Structural insights into preinitiation complex assembly on core promoters. Science, 2021, 372: eaba8490. |
| [31] |
Chen X, Yin X, Li J, et al. Structures of the human Mediator and Mediator-bound preinitiation complex. Science, 2021, 372: eabg0635. |
| [32] |
Abdella R, Talyzina A, Chen S, et al. Structure of the human Mediator-bound transcription preinitiation complex. Science, 2021, 372: 52-6. |
| [33] |
Rengachari S, Schilbach S, Aibara S, et al. Structure of the human Mediator-RNA polymerase Ⅱ pre-initiation complex. Nature, 2021, 594: 129-33. |
| [34] |
Chen X, Wang X, Liu W, et al. Structures of +1 nucleosome- bound PIC-Mediator complex. Science, 2022, 378: 62-8. |
| [35] |
Seifert-Davila W, Girbig M, Hauptmann L, et al. Structural insights into human TFⅢC promoter recognition. Sci Adv, 2023, 9: eadh2019. |
| [36] |
Talyzina A, Han Y, Banerjee C, et al. Structural basis of TFⅢC-dependent RNA polymerase Ⅲ transcription initiation. Mol Cell, 2023, 83: 2641-52.e7. |
| [37] |
Hou H, Jin Q, Ren Y, et al. Structure of the SNAPc-bound RNA polymerase Ⅲ preinitiation complex. Cell Res, 2023, 33: 565-8. |
| [38] |
Shah SZ, Perry TN, Graziadei A, et al. Structural insights into distinct mechanisms of RNA polymerase Ⅱ and Ⅲ recruitment to snRNA promoters. Nat Commun, 2025, 16: 141. |
| [39] |
Sun JF, Li X, Hou XB, et al. Structural basis of human SNAPc recognizing proximal sequence element of snRNA promoter. Nat Commun, 2022, 13: 6871. |
| [40] |
Schilbach S, Hantsche M, Tegunov D, et al. Structures of transcription pre-initiation complex with TFⅡH and Mediator. Nature, 2017, 551: 204-9. |
| [41] |
Kassavetis GA, Letts GA, Geiduschek EP. The RNA polymerase Ⅲ transcription initiation factor TFⅢB participates in two steps of promoter opening. EMBO J, 2001, 20: 2823-34. |
| [42] |
Bernecky C, Herzog F, Baumeister W, et al. Structure of transcribing mammalian RNA polymerase Ⅱ. Nature, 2016, 529: 551-4. |
| [43] |
Hoffmann NA, Jakobi AJ, Moreno-Morcillo M, et al. Molecular structures of unbound and transcribing RNA polymerase Ⅲ. Nature, 2015, 528: 231-6. |
| [44] |
Hou H, Li Y, Wang M, et al. Structural insights into RNA polymerase Ⅲ-mediated transcription termination through trapping poly-deoxythymidine. Nat Commun, 2021, 12: 6135. |
| [45] |
Girbig M, Xie J, Grotsch H, et al. Architecture of the yeast Pol Ⅲ pre-termination complex and pausing mechanism on poly(dT) termination signals. Cell Rep, 2022, 40: 111316. |
| [46] |
Bhargava P, Kassavetis GA. Abortive initiation by Saccharomyces cerevisiae RNA polymerase Ⅲ. J Biol Chem, 1999, 274: 26550-6. |
| [47] |
Luse DS, Jacob GA. Abortive initiation by RNA polymerase Ⅱ in vitro at the adenovirus 2 major late promoter. J Biol Chem, 1987, 262: 14990-7. |
| [48] |
Wade JT, Struhl K. The transition from transcriptional initiation to elongation. Curr Opin Genet Dev, 2008, 18: 130-6. |
| [49] |
Hsu LM. Promoter clearance and escape in prokaryotes. Biochim Biophys Acta, 2002, 1577: 191-207. |
| [50] |
Dieci G, Bosio MC, Fermi B, et al. Transcription reinitiation by RNA polymerase Ⅲ. Biochim Biophys Acta, 2013, 1829: 331-41. |
 2025, Vol. 37
2025, Vol. 37 

 徐彦辉,复旦大学附属肿瘤医院研究员,国家杰出青年科学基金获得者、教育部长江学者特聘教授、国家“万人计划”科技创新领军人才、首批“新基石研究员”,曾获“科学探索奖” “谈家桢生命科学奖(创新奖)”等奖项。团队建立了体外重构转录起始超大复合物的独特方法,解决了转录起始领域若干关键问题,被认为是分子生物学领域的重大突破性进展,成果入选“2021年度中国生命科学十大进展”和“中国2021年度重要医学进展”
徐彦辉,复旦大学附属肿瘤医院研究员,国家杰出青年科学基金获得者、教育部长江学者特聘教授、国家“万人计划”科技创新领军人才、首批“新基石研究员”,曾获“科学探索奖” “谈家桢生命科学奖(创新奖)”等奖项。团队建立了体外重构转录起始超大复合物的独特方法,解决了转录起始领域若干关键问题,被认为是分子生物学领域的重大突破性进展,成果入选“2021年度中国生命科学十大进展”和“中国2021年度重要医学进展”