植物在和病原微生物的长期互作中演化出了一套复杂且精细的免疫系统以抵御病原微生物的侵袭。植物免疫系统主要包括细胞表面识别受体控制的基础免疫(pattern-triggered immunity, PTI)和细胞内免疫蛋白控制的特异性免疫(effector-triggered immunity, ETI)。近年来,植物免疫领域的相关研究对植物免疫系统的工作机制的解析有了许多突破性的成果,其中钙通道和钙信号介导的免疫反应调控机制的解析就是其中之一。
作为细胞的重要第二信使,钙离子在基础免疫和特异性免疫中都发挥了关键作用,而介导钙离子运输的钙通道是植物免疫调控的重要节点。近年来,随着分子生物学和基因工程技术的发展,经典钙通道如环核苷酸门控离子通道(cyclic nucleotide-gated channel, CNGC)、高渗门控钙离子透过性通道(hyperosmolality-gate calcium-permeable channel, OSCA)、谷氨酸类受体通道(glutamate receptor-like channel, GLR)在免疫反应中的调控机制相继被解析。而一些重要的抗病相关蛋白,如跨膜锚蛋白(ankyrin repeat transmembrane protein, ANK-TM)、白粉病感病蛋白(mildew locus O, MLO)、核苷酸结合和富亮氨酸重复受体(nucleotide-binding domain and leucine-rich repeat-containing receptor, NLR)和卫青(WeiTsing, WTS)等,也被发现通过其钙通道活性调节植物免疫(图 1)。
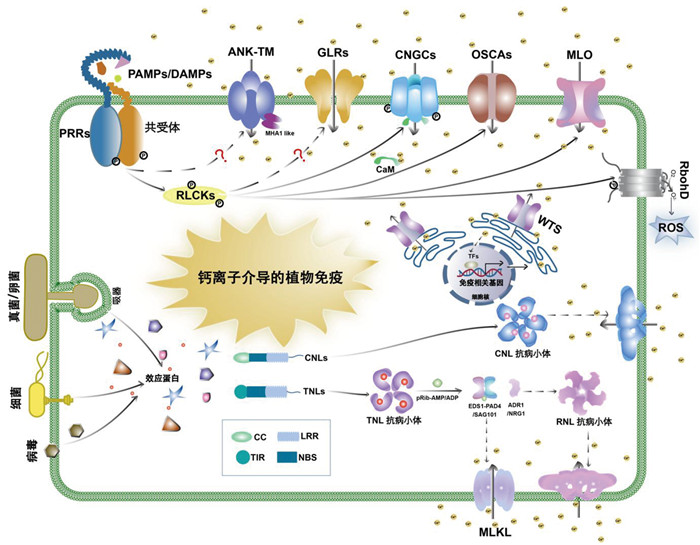
|
图 1 参与植物先天免疫的钙离子通道 |
本文综述了植物免疫调控中钙通道研究方向的最新进展,重点介绍了钙通道在植物免疫中的激活机制、病原菌效应蛋白通过攻击植物钙通道抑制植物免疫反应的机制,以及钙通道改造在植物抗病改良中的潜在应用,以期为作物抗病育种提供研究思路和理论借鉴。
1 植物基础免疫中的钙通道由细胞表面的模式识别受体(pattern recognition receptor, PRR)识别微生物相关分子模式(pathogen- associated molecular pattern, PAMP;例如细菌鞭毛蛋白和真菌细胞壁几丁质)和植物损伤相关分子模式(damage-associated molecular pattern, DAMP;例如胞外ATP和植物激发子小肽等)触发的免疫反应被称为植物的基础免疫。在基础免疫的激发过程中,模式受体识别胞外分子模式后,经共受体激酶磷酸化激活,进而磷酸化下游包括胞质类受体激酶(receptor-like cytoplasmic kinase, RLCK)和丝裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK)在内的信号传递组分,控制钙通道活性,调控植物免疫反应。现有研究表明,CNGC、OSCA和GLR等典型钙通道参与了植物基础免疫的调控。
1.1 环核苷酸门控离子通道环核苷酸门控离子通道(CNGC)是一类在动植物中广泛存在的非选择性阳离子通道。这类通道通常由四个具有六次跨膜结构的蛋白形成同源或异源复合体构成,其活性通过环核苷酸(如cAMP (cyclic adenosine monophosphate)和cGMP (cyclic guanosine monophosphate))的直接结合调控[1]。这些环核苷酸与通道上的特定结合位点结合,导致通道构象变化,从而调节其离子通透性[1]。另外,CNGC的活性也由钙调蛋白和激酶调控。例如,在拟南芥中,细胞膜上的AtCNGC2和AtCNGC4组装成一个异源钙通道。在静息状态时,钙调蛋白AtCaM7 (calmodulin 7)通过结合AtCNGC2/4的C末端抑制其活性;当病原菌入侵时,模式识别受体识别微生物相关分子模式并将信号传递给胞质类受体激酶AtBIK1 (botrytis-induced kinase 1),进而磷酸化激活AtCNGC2/4,胞外高浓度的钙离子内流引起胞内钙浓度增加,启动下游钙离子依赖的免疫反应[2]。类似地,在水稻中,模式识别受体通过激活OsRLCK185 (receptor-like cytoplasmic kinase 185) 进而磷酸化OsCNGC9激发其通道活性,从而调控对稻瘟病的抗性[3]。拟南芥AtCNGC19/20也是一种异源聚合体钙通道,但与AtCNGC2/4和OsCNGC9不同,AtCNGC19/20受到共受体AtBAK1 (brassinos- teroid insensitive 1 (BRI1)-associated receptor kinase)/AtSERK4 (somatic embryogenesis receptor kinase 4)和模式受体AtBTL2 (BAK-to-life 2) 的多重磷酸化调控[4, 5]。在静息态时,AtBAK1磷酸化AtBTL2和AtCNGC20以抑制AtBTL2的激酶活性并促进AtCNGC20的降解,进而抑制AtCNGC20介导的免疫反应;当AtBAK1/AtSERK4缺失时,AtCNGC20累积,并且被AtBTL2磷酸化激活,并通过AtEDS1 (enhanced disease susceptibility 1)-AtPAD4 (phytoalexin deficient 4)-AtADR1 (activated disease resistance 1)信号途径激发免疫信号,导致atbak1/atserk4出现生长受限、植株矮小、细胞死亡等自免疫表型[4, 5]。因此,CNGC家族成员通过相关蛋白复杂而精细的调控机制,在植物免疫反应中发挥重要作用。
1.2 高渗门控钙离子透过性通道高渗门控钙离子透过性通道(OSCA)是一类广泛分布于真核生物的非选择性钙渗透阳离子通道,最早在拟南芥中作为高渗胁迫感受器被发现[6, 7]。这类通道蛋白具有多次跨膜结构域,以单体或二聚体形式形成通道。在拟南芥保卫细胞中,细胞表面的模式受体AtFLS2 (flagellin sensitive 2)识别细菌模式分子flg22 (flagellin 22)后,通过AtBIK1磷酸化AtOSCA1.3的胞内Loop1结构域,激活其通道活性,释放胞外钙离子内流,导致胞内钙离子浓度升高,启动下游一系列反应,进而关闭气孔,以抵御病原菌从气孔入侵[8]。与其他钙通道不同,OSCA主要通过特异调节气孔开闭参与植物基础免疫。
1.3 谷氨酸类受体蛋白家族谷氨酸类受体蛋白家族(GLR)是动植物中广泛分布的一类钙通道。与其他钙通道家族不同,GLR具有胞外受体结构域,通过四聚体感受胞外谷氨酸信号[9],兼具受体和通道两种功能。与CNGC和OSCA相似,GLR在基础免疫中通过模式受体介导的信号控制钙离子内流,并受钙调蛋白的调控[10]。例如,拟南芥AtGLR2.7/2.8/2.9缺失导致多种PAMPs/DAMPs分子如flg22、elf18和pep1等触发的钙离子内流受损,但是具体调控机制未知[11]。类似地,棉花GhGLR4.8的自然变异或者敲除可以显著降低棉花对镰孢枯萎病的抗性[12]。与目前发现的其他钙通道的调控方式不同,AtGLR3.3和AtGLR3.6在系统性免疫信号,特别是在伤害信号的远距离传递中起着重要作用。AtGLR3.3和AtGLR3.6分别在韧皮部和木质部维管细胞中[13, 14],通过感知植物受伤细胞释放的谷氨酸信号,在阴离子通道MSL10 (MscS-like10)的协同作用下,以钙振荡的形式快速将伤害信号传递到其他未受伤的区域,并激活这些区域的免疫反应[15]。另外,AtGLR3.3和AtGLR3.6的通道活性也受胞外pH的调控[16]。
1.4 跨膜锚蛋白跨膜锚蛋白ANK-TM是一类在植物中广泛分布的抗病蛋白,以拟南芥AtACD6 (accelerated cell death 6)[17, 18]、玉米ZmACD6[19]、小麦Lr14a [20, 21]为主要代表,该蛋白家族包含多次跨膜结构域和一个介导蛋白互作的细胞质锚蛋白结构域。ANK-TM与动物TRPA (transient receptor potential ankyrin) 通道具有类似的三维蛋白质结构[18]。通过电生理和钙成像实验,拟南芥AtACD6和小麦Lr14a被证实在动物细胞中可以独立形成钙离子通道[18, 21]。与其他基础免疫中的钙通道不同,AtACD6通道并不直接通过模式受体介导的磷酸化激活,而是通过小肽AtMHA1L (modulator of hyperactive ACD6 1 like) 促发AtACD6复合体形成,进而激活其通道活性[18]。另外,与特异性免疫中NLR自免疫突变体的低温敏感表型类似,AtMHA1L过表达植物和AtACD6功能获得性突变体atacd6-1在低温下可以激发强烈的免疫反应[18, 22]。这表明AtMHAL-AtACD6模块可能与TRPA类似,在温度感应中发挥重要作用[23],但是AtMHAL-AtACD6模块中小肽信号如何接收来自模式受体的信号、AtACD6钙通道的结构基础以及AtACD6钙通道对低温敏感的机制有待进一步研究。
1.5 白粉病感病蛋白白粉病感病蛋白MLO是一类在植物中广泛存在的通道蛋白[24]。正如它的命名,MLO在不同植物中调控对白粉病的抗性,但是它是作为感病因子执行功能,MLO失活突变后表现为对白粉病的抗性。MLO蛋白具有典型的七次跨膜结构。与CNGC、OSCA和GLR类似,MLO通道的活性可以被模式受体激活,并被钙调蛋白抑制。例如,在花粉管发育中,模式受体复合体AtANX1/2 (Anxur1/2)- AtBUPS1/2 (Buddha’s paper seal 1/2)-AtLLG2/3 (Lorelei-like-GPI-anchored protein 2)识别小肽RALF4/19 (rapid alkalinization factor 4/19),通过磷酸化激活胞内激酶AtMARIS,进而激活AtMLO1/5/9/15,以控制花粉管的发育[25-27];而在助细胞中,模式受体复合体AtFER (Feronia)-AtLRE (Lorelei)在识别RALF4/19信号后激活AtMLO7,进而控制受精[28]。除了参与生殖发育的调控,MLO还与胞吐蛋白EXO70互作控制表皮毛细胞细胞壁的完整性,具体机制还不清楚[29]。总的来说,MLO在植物生长发育和植物免疫调控中发挥重要作用,但相比MLO在生殖发育调控中作用机制解析,MLO如何介导植物感白粉病的具体机制还是未知的。
2 植物特异性免疫中的钙通道NLR蛋白是植物特异性免疫中的核心受体,负责识别病原菌效应子引发细胞内钙离子流动,从而触发免疫信号级联反应,最终导致植物防御机制的激活。相比于基础免疫,特异性免疫中钙离子内流的持续时间更长。
NLRs在结构上通常包括三个部分:N端可变结构域、中央保守的核苷酸结合和寡聚化结构域(central conserved nucleotide binding and oligomerization domain, NOD) 以及C端的富亮氨酸重复结构域(leucine rich repeat, LRR)[30]。NOD区域的主要生化功能是作为核苷酸开关调控NLRs的聚合[31],LRR结构域参与蛋白质之间的相互作用和对效应蛋白的特异性识别,N端结构域则可以介导不同的免疫信号通路。根据N端结构域的不同,可以将NLRs划分为3种类型,即TNLs (Toll/interleukin-1 receptor (TIR) NLRs)、CNLs (coiled-coil (CC) NLRs)和RNLs (resistance to powdery mildew 8 (RPW8) NLRs) [32]。近年来基于结构生物学的研究证明了部分CNL和RNL通过聚合形成钙离子通道,进而通过介导钙离子内流调控植物免疫反应[33-35]。
2.1 CNL构成的钙通道与其激活方式CNL的CC结构域通常具有串联的4个α螺旋结构。以ZAR1 (HopZ-activated resistance 1)为例,在静息状态下,ZAR1与RKS1 (resistance related kinase 1) 组成性结合,ZAR1的LRR结构域与自身NOD区域结合,使ZAR1保持单体状态,此时CC结构域的4个α螺旋结构处于堆叠状态;在野油菜黄单胞菌侵染后,被效应蛋白AvrAC尿苷化的PBL2 (AvrPhB susceptible 1-like 2)通过与RKS1的相互作用结合到ZAR1-RKS1复合体中,通过NOD结构域内ADP到ATP的替换,促发ZAR1-RKS1-PBL2UMP复合体的聚合,形成轮状的五聚体结构,被称为抗病小体。在ZAR1抗病小体中,5个α1螺旋从折叠状态释放形成突出于轮状平面的漏斗状结构,并在中心形成孔道[36-38]。电生理实验表明,ZAR1是一种选择性的阳离子通道,激活后定位在细胞膜上以钙通道行使生物学功能[33]。与ZAR1相似,小麦中的CNL Sr35和对应的效应蛋白AvrSr35能够一起形成与ZAR1非常相似的五聚体抗病小体[34, 39]。虽然冷冻电镜成像结果并未获得Sr35抗病小体α1螺旋的结构,但是在AlphaFold3的预测结构中,Sr35抗病小体α1螺旋的结构形成了与ZAR1类似的漏斗结构[34, 39, 40]。与ZAR1抗病小体不同的是,Sr35抗病小体的阳离子通道不具有选择性,并且其阳离子通道活性不依赖于孔道内侧的酸性氨基酸残基。这可能是Sr35 N端结构域的结构和功能与ZAR1不同所导致的[34]。
2.2 RNL构成的钙通道与其激活方式目前鉴定的RNL主要包括ADR1、NRG1 (N requirement gene 1)和NRC (NLR-required for cell death)三个家族蛋白。RNL不直接识别病原菌效应蛋白,而作为辅助角色在TNL和部分CNL的下游传递信号,也被称为辅助性NLR (helper NLR, hNLR)[41]。与ZAR1相似,ADR1和NRG1家族蛋白的CCR结构域也具有4个α螺旋组成的堆叠结构[35]。在电生理实验中,拟南芥AtADR1和AtNRG1的自激活突变体可形成钙离子通透性的非选择性阳离子通道[35]。虽然目前没有获得ADR1和NRG1作为钙通道的全长蛋白质结构,但是在AlphaFold3的预测中,烟草NtADR1和NtNRG1也可以形成与ZAR1和Sr35相似的抗病小体结构,而NtNRG1的CCR结构在聚合体中形成了比ZAR1、Sr35和AtADR1更长的漏斗颈结构,这暗示NRG1可能具有与其他抗病小体不同的作用方式[40]。在植物中,ADR1和NRG1可以被TNL抗病小体产生的小分子信号激活。TNL的N端结构是具有NAD+水解酶活性的TIR结构,当TNL识别病原菌效应蛋白形成同源或者异源四聚体抗病小体时,其N端的TIR结构形成具有完整酶活的全酶,催化生成多种小分子化合物作为第二信使[42-48]。其中pRib-AMP/ADP与EDS1/PAD4结合并招募ADR1形成复合体,而ADPr-ATP/di-ADPR与EDS1/SAG101 (senescence-associated gene 101)结合并招募NRG1形成复合体,进而在未知机制的控制下激活ADR1和NRG1的通道活性[43, 44, 48, 49]。另外,虽然单子叶植物在演化过程中丢失了TNL,但是其基因组中还保留了一些TIR-only编码基因,并且这些TIR-only蛋白也是经由pRib-AMP/ADP信号分子通过EDS1-PAD4-ADR1复合体激活免疫反应[49]。有意思的是,最新研究结果表明,在烟草中,由flg22促发的基础免疫反应也可以通过TIR-only蛋白激活EDS1-PAD4-ADR1复合体[50]。
NRC是一类在茄科植物中特异存在的RNL,介导一系列CNL控制的免疫反应[51, 52]。在静息态时,NbNRC2可以形成二聚体、四聚体或者更高聚合状态的自抑制结构[53, 54];当上游CNL识别效应蛋白后,可能通过其NB结构域将信号传至NRC[55],促使其解除自抑制状态,并重新聚合生成同源六聚体轮状结构[40, 56],并促发下游的钙离子内流。与ZAR1、Sr35、ADR1和NRG1抗病小体不同,NRC并不能在动物细胞中独立控制钙离子的内流,暗示NRC可能需要植物中的其他组分参与钙信号的调控[56]。
2.3 卫青与其激活方式卫青WTS是在拟南芥中发现的油菜根肿病抗病蛋白。与其他具有复杂调控机制的钙通道不同,WTS的结构相对简单,仅包含四个跨膜结构域。因此,其调控机制与其他钙通道存在显著差异。具体而言,WTS的激活主要依赖于转录调控,在没有病原菌侵染的情况下WTS的表达受到严格控制,当油菜根肿菌(Plasmodiophora brassicae, Pb)侵染时,WTS在其特殊启动子的调控下在中柱鞘细胞中被特异激活转录、翻译成蛋白进而直接激活其钙通道活性介导免疫反应,有别于其他钙通道需要其他组分如激酶或者配体激发的方式;在亚细胞定位方面,WTS定位到内质网上,将内质网存储的钙离子释放到细胞质中,从而触发免疫反应;而目前发现的其他钙通道主要定位于细胞膜,通过释放细胞间隙的钙离子进入细胞质调控免疫反应;在蛋白质结构方面,WTS在内质网膜上聚合形成与ZAR1抗病小体类似的五聚体结构,但形成的孔道与跨膜蛋白形成的通道更类似[57]。另外,WTS是目前发现的最有效的根肿病抗病基因,由于其简单的激活机制,将在十字花科蔬菜的抗根肿病遗传改良中发挥重要作用,也能为其他作物的抗病遗传改良提供重要借鉴。
2.4 成孔蛋白MLKLMLKL (mixed lineage kinase domain-like)是一类广泛存在于动植物中的成孔蛋白。在植物中MLKL相关研究较少,目前已知其参与TNL介导的抗病反应[58]。而小麦重要的白粉病抗病基因Pm13 (powdery mildew resistance gene 13)也编码一个MLKL蛋白[59]。与ZAR1和ADR1等蛋白相似,MLKL的N端具有4个α螺旋组成的堆叠结构。与抗病小体的效应蛋白诱导钙通道形成机制不同,拟南芥MLKL在静息态可形成类似三角锥的同源四聚体结构[60]。TNL识别病原效应蛋白诱导产生的小分子激活EDS1/PAD4和EDS1/SAG101复合体,触发MLKL定位在细胞膜上,并转换为激发态行使钙通道功能,进而激活免疫反应[58]。虽然目前植物MLKL激发态的结构以及其具体的激活机制尚未解析,但是动物中的研究表明MLKL的激活可能需要胞质激酶介导的磷酸化参与[61]。
3 植物免疫钙通道的病原菌攻击机制及其改造应用病原微生物为了在植物中定殖,通过分泌效应蛋白攻击植物的免疫系统。相比病原物攻击其他免疫调节组分的研究,病原物攻击植物钙通道的研究较少。基于病原菌攻击钙通道机制解析,有助于进一步理解植物-病原物互作机制,并为设计规避病原菌攻击的钙通道进而提高作物抗病能力提供理论依据。
3.1 基础免疫钙通道的病原菌攻击与抗性改造卵菌RXLR (Arg-any amino acid-Leu-Arg)效应蛋白AVRblb2家族是广泛存在于卵菌的一类效应蛋白,可以通过靶标一系列植物免疫系统组件抑制植物免疫反应。其中钙信号通路是AVRblb2的重要靶标之一[62, 63],在烟草上AVRblb2可以与NbCaM、NbCML36 (calmodulin-like protein 36)、NbCNGC18互作,通过促进NbCaM和NbCNGC18的互作,抑制NbCNGC18的钙通道活性,进而抑制烟草的免疫反应[62]。在拟南芥上,黄单胞菌效应蛋白XopD通过转录调控诱导AtOSCA1.1的表达,进而诱导气孔关闭,增强细胞间隙湿度来创造一个有利于细菌繁殖的环境;而AtOSCA1.1缺失材料可以显著提高对黄单胞菌的抗性[64]。类似地,在番茄上SIOSCA4.1是凤果花叶病毒(Pepino mosaic virus, PepMV)关键的感病基因,敲除SIOSCA4.1后,番茄对凤果花叶病毒的抗性显著升高,但是目前PepMV通过SIOSCA4.1抑制植物免疫反应的机制还不清楚[65]。
虽然在植物-白粉病互作中MLO作为感病基因的具体工作机制还未被解析,但是MLO的沉默和敲除已成为提高作物抗病性的重要策略。在小麦[66-68]、葡萄[69]、黄瓜[70, 71]、大豆[72]和苹果[73]等作物中敲除或者沉默MLO可以显著提高作物对白粉病的抗性。由于MLO在植物生长发育中起着重要的作用,MLO的敲除可能会导致作物生长发育的缺陷,例如小麦tamlo1完全敲除体[66]和黄瓜mlo1/mlo8/mlo11均具有明显生长缺陷[70]。为了避免编辑MLO造成产量的损失,可能需要对MLO介导的感病机制进一步研究,通过更精准地编辑病原菌攻击位点或者协同编辑其他基因来创制理想的抗病材料。例如通过激活小麦TaTMT3B (tonoplast monosaccharide transporter 3)可以恢复小麦tamlo1完全敲除体的生长缺陷[66]。
3.2 NLR钙通道的效应蛋白抑制及其功能优化NLR在跨物种应用中往往不能实现预期抗性,提示NLR介导的特异抗性可能在不同病原-寄主中被抑制。根据目前的研究结果,病原菌通过分泌效应蛋白抑制NLR,特别是下游执行信号转导的hNLR,这可能是其中一个重要且普遍的机制。目前hNLR三大家族蛋白均有被病原菌效应蛋白攻击的案例报道。
在烟草中,通过筛选来自卵菌、细菌和线虫的效应蛋白,Derevnina等[74]发现来自大豆孢囊线虫的效应蛋白SS15 (SPRYSEC15)和来自卵菌的效应蛋白AVRcap1b可以抑制NbNRC2和NbNRC3介导的抗病反应。其中SS15可以结合NbNRC2和NbNRC3的NB结构域以抑制其聚合,从而抑制其介导的免疫反应[74, 75];然而SS15并不能攻击NbNRC2/3的同源蛋白NbNRC4。基于NbNRC2/3与NbNRC4序列的差异和SS15与SlNRC1NB-ARC复合体结构,研究人员发现NbNRC2的316位谷氨酸和317位天冬氨酸是与SS15互作的关键氨基酸残基;将NbNRC2的317位天冬氨酸突变为NRC4对应的赖氨酸即可在维持NbNRC2功能的基础上避免SS15对其寡聚化的抑制[76]。有意思的是,在对不同茄科植物NRC1/2/3进行分析时发现来自辣椒CaNRC3和番茄的SlNRC3不会被SS15攻击,而将NbNRC3的316位谷氨酸突变为赖氨酸时则可以在维持功能的基础上规避SS15的攻击[75, 76]。
在拟南芥中,AtADR1-L1/L2被来自丁香假单胞的效应蛋白AvrPtoB攻击,AvrPtoB以其E3泛素连接酶的功能对AtADR1-L1/L2的CCR结构域上的两个赖氨酸位点进行泛素标记,通过植物的26S蛋白酶体降解AtADR1-L1/L2,从而抑制AtADR1- L1/L2介导的免疫反应[77]。与之相似,来自青枯菌具有E3泛素连接酶活性的效应蛋白RipV2也可以泛素化并降解番茄SlNRG1,以促进病原菌的侵染[78]。有意思的是,AvrPtoB并不能攻击AtADR1- L1/L2的同源蛋白AtADR1[77]。通过对AtADR1- L1/L2和AtADR1序列差异的分析,发现CCR结构域上的两个赖氨酸残基变异是造成ADR1s三个同源蛋白功能差异的关键位点,进一步实验表明替换这两个赖氨酸位点则可以让AtADR1-L1/L2逃避AvrPtoB的攻击[77]。
3.3 钙通道的自然变异与定向改造策略从自然群体中寻找参与抗病调控的高活性的钙通道,或者基于自然变异对钙通道活性的影响机制的解析,对钙通道进行改造和利用可能是一种作物抗病性状改良的有效方法。例如,GhGLR4.8在棉花抗枯萎病中发挥重要作用,而在棉花感病品种中其第150位异亮氨酸到亮氨酸的突变导致其丧失功能[12];借助于基因编辑技术,有望编辑GhGLR4.8的150位氨基酸来恢复感病品种对棉花枯萎病的抗性。另外一个例子是ANK-TM,在拟南芥自然群体中存在高活性AtACD6Est-1,相比于AtACD6Col-0,跨膜区两个关键氨基酸的自然变异赋予了AtACD6Est-1更高的活性,能够激活更强的免疫反应[17],为改造和利用ANK-TM类似蛋白提供了重要借鉴。
对特异性免疫中枢RNL钙通道的攻击是病原菌致病的重要机制,因此帮助RNL规避病原菌的攻击将是提高作物抗病能力的有效手段。在研究效应蛋白AvrPtoB对AtADR1-L1/L2的攻击时,研究人员发现以上基于RNL自然变异的研究案例表明,在RNL与病原效应蛋白的共演化过程中,存在能规避效应蛋白攻击的自然变异,这使得我们可以在解析RNL与效应蛋白互作的基础上利用自然变异改造RNL以提高作物的抗病性。
以Sr35为代表的CNL通过其LRR结构域直接识别病原菌效应蛋白并通过其CC结构域形成的钙通道来调控免疫反应。由于CC结构域的高度保守性,使得交换不同CNL的LRR即可在不改变其功能的情况下改变其识别的效应蛋白。例如大麦抗白粉病抗病蛋白HvMLA10 (mildew resistance locus A10)和HvMLA13在更换Sr35的LRR结构域后,可以识别小麦杆锈菌效应蛋白AvrSr35[34]。而对于同源性更高的CNL,比如Sr35、TaSh1 (Sr35 homologue of wheat cultivar Chinese Spring)和HvSH1 (Sr35 homologue of barley),基于其蛋白质互作结构,交换TaSH1和HvSH1的关键氨基酸即可让TaSH1和HvSH1识别AvrSr35[34];类似地,将Sr50的部分关键识别氨基酸融合到Sr33中可以让Sr33识别效应蛋白AvrSr50[79];而基于Sr50-AvrSr50的互作结构预测,发现通过关键氨基酸改造可以让Sr50识别免疫逃逸的AvrSr50突变体[79, 80]。以上案例表明基于CNL钙通道的改造可以拓宽作物的效应蛋白识别谱,进而提高作物对病原菌的抗性。
4 总结与展望植物免疫调控领域内钙通道的不断发现及其功能与作用机制的深入解析,加深了我们对植物免疫机制的理解。虽然钙信号的下游免疫调控机制还有待解析,但是围绕钙通道的作用机制的研究结果已为作物抗病育种提供了重要的基因资源和潜在基因编辑位点。其中,病原菌对植物免疫调控中的RNL构成的核心钙通道的攻击可能是抗病育种中需要解决的重要问题。根据目前不同病原对钙通道蛋白特别是hNLR的抑制机制推测:攻击hNLR抑制寄主免疫可能是病原致病的普遍机制。解析其分子机制能够为创制逃避病原攻击的抗病作物提供重要理论基础;此外,通过分析不同物种来源的钙通道蛋白的功能和变异也能够为设计抗病蛋白提供重要借鉴。
| [1] |
Dietrich P, Moeder W, Yoshioka K. Plant cyclic nucleotide-gated channels: new insights on their functions and regulation. Plant Physiol, 2020, 184: 27-38. DOI:10.1104/pp.20.00425 |
| [2] |
Tian W, Hou C, Ren Z, et al. A calmodulin-gated calcium channel links pathogen patterns to plant immunity. Nature, 2019, 572: 131-5. DOI:10.1038/s41586-019-1413-y |
| [3] |
Wang J, Liu X, Zhang A, et al. A cyclic nucleotide-gated channel mediates cytoplasmic calcium elevation and disease resistance in rice. Cell Res, 2019, 29: 820-31. DOI:10.1038/s41422-019-0219-7 |
| [4] |
Yu X, Xie Y, Luo D, et al. A phospho-switch constrains BTL2-mediated phytocytokine signaling in plant immunity. Cell, 2023, 186: 2329-44. DOI:10.1016/j.cell.2023.04.027 |
| [5] |
Yu X, Xu G, Li B, et al. The receptor kinases BAK1/SERK4 regulate Ca2+ channel-mediated cellular homeostasis for cell death containment. Curr Biol, 2019, 29: 3778-90. DOI:10.1016/j.cub.2019.09.018 |
| [6] |
Yuan F, Yang H, Xue Y, et al. OSCA1 mediates osmotic-stress-evoked Ca2+ increases vital for osmosensing in Arabidopsis. Nature, 2014, 514: 367-71. DOI:10.1038/nature13593 |
| [7] |
Hou C, Tian W, Kleist T, et al. DUF221 proteins are a family of osmosensitive calcium-permeable cation channels conserved across eukaryotes. Cell Res, 2014, 24: 632-5. DOI:10.1038/cr.2014.14 |
| [8] |
Thor K, Jiang S, Michard E, et al. The calcium-permeable channel OSCA1.3 regulates plant stomatal immunity. Nature, 2020, 585: 569-73. DOI:10.1038/s41586-020-2702-1 |
| [9] |
Green MN, Gangwar SP, Michard E, et al. Structure of the Arabidopsis thaliana glutamate receptor-like channel GLR3.4. Mol Cell, 2021, 81: 3216-26. DOI:10.1016/j.molcel.2021.05.025 |
| [10] |
Yan C, Gao Q, Yang M, et al. Ca2+/calmodulin-mediated desensitization of glutamate receptors shapes plant systemic wound signalling and anti-herbivore defence. Nat Plants, 2024, 10: 145-60. DOI:10.1038/s41477-023-01578-8 |
| [11] |
Bjornson M, Pimprikar P, Nürnberger T, et al. The transcriptional landscape of Arabidopsis thaliana pattern-triggered immunity. Nat Plants, 2021, 7: 579-86. DOI:10.1038/s41477-021-00874-5 |
| [12] |
Liu S, Zhang X, Xiao S, et al. A single-nucleotide mutation in a GLUTAMATE RECEPTOR-LIKE gene confers resistance to Fusarium wilt in Gossypium hirsutum. Adv Sci, 2021, 8: 2002723. DOI:10.1002/advs.202002723 |
| [13] |
Nguyen CT, Kurenda A, Stolz S, et al. Identification of cell populations necessary for leaf-to-leaf electrical signaling in a wounded plant. Proc Natl Acad Sci USA, 2018, 115: 10178-83. DOI:10.1073/pnas.1807049115 |
| [14] |
Toyota M, Spencer D, Sawai-Toyota S, et al. Glutamate triggers long-distance, calcium-based plant defense signaling. Science, 2018, 361: 1112-5. DOI:10.1126/science.aat7744 |
| [15] |
Moe-Lange J, Gappel NM, Machado M, et al. Interdependence of a mechanosensitive anion channel and glutamate receptors in distal wound signaling. Sci Adv, 2021, 7: eabg4298. DOI:10.1126/sciadv.abg4298 |
| [16] |
Shao Q, Gao Q, Lhamo D, et al. Two glutamate- and pH-regulated Ca2+ channels are required for systemic wound signaling in Arabidopsis. Sci Signal, 2020, 13: eaba1453. DOI:10.1126/scisignal.aba1453 |
| [17] |
Todesco M, Balasubramanian S, Hu TT, et al. Natural allelic variation underlying a major fitness trade-off in Arabidopsis thaliana. Nature, 2010, 465: 632-6. DOI:10.1038/nature09083 |
| [18] |
Chen J, Li L, Kim JH, et al. Small proteins modulate ion-channel-like ACD6 to regulate immunity in Arabidopsis thaliana. Mol Cell, 2023, 83: 4386-97. DOI:10.1016/j.molcel.2023.10.030 |
| [19] |
Zhang Z, Guo J, Zhao Y, et al. Identification and characterization of maize ACD6-like gene reveal ZmACD6 as the maize orthologue conferring resistance to Ustilago maydis. Plant Signal Behav, 2019, 14: e1651604. DOI:10.1080/15592324.2019.1651604 |
| [20] |
Kolodziej MC, Singla J, Sánchez-Martín J, et al. A membrane-bound ankyrin repeat protein confers race-specific leaf rust disease resistance in wheat. Nat Commun, 2021, 12: 956. DOI:10.1038/s41467-020-20777-x |
| [21] |
Yue L, Wang L, Neuhäuser B, et al. Cytoplasmic calcium influx mediated by Lr14a regulates stomatal immunity against leaf rust in wheat. bioRxiv, 2024. DOI:10.1101/2024.12.10.627212 |
| [22] |
Todesco M, Kim ST, Chae E, et al. Activation of the Arabidopsis thaliana immune system by combinations of common ACD6 alleles. PLoS Genet, 2014, 10: e1004459. DOI:10.1371/journal.pgen.1004459 |
| [23] |
Zhang H, Wang C, Zhang K, et al. The role of TRPA1 channels in thermosensation. Cell Insight, 2022, 1: 100059. DOI:10.1016/j.cellin.2022.100059 |
| [24] |
Kusch S, Panstruga R. mlo-based resistance: an apparently universal "Weapon" to defeat powdery mildew disease. Mol Plant Microbe Interact, 2017, 30: 179-89. DOI:10.1094/MPMI-12-16-0255-CR |
| [25] |
Meng JG, Liang L, Jia PF, et al. Integration of ovular signals and exocytosis of a Ca2+ channel by MLOs in pollen tube guidance. Nat Plants, 2020, 6: 143-53. DOI:10.1038/s41477-020-0599-1 |
| [26] |
Gao Q, Wang C, Xi Y, et al. RALF signaling pathway activates MLO calcium channels to maintain pollen tube integrity. Cell Res, 2023, 33: 71-9. DOI:10.1038/s41422-022-00754-3 |
| [27] |
Ge Z, Bergonci T, Zhao Y, et al. Arabidopsis pollen tube integrity and sperm release are regulated by RALF-mediated signaling. Science, 2017, 358: 1596-600. DOI:10.1126/science.aao3642 |
| [28] |
Gao Q, Wang C, Xi Y, et al. A receptor-channel trio conducts Ca2+ signalling for pollen tube reception. Nature, 2022, 607: 534-9. DOI:10.1038/s41586-022-04923-7 |
| [29] |
Huebbers JW, Caldarescu GA, Kubátová Z, et al. Interplay of EXO70 and MLO proteins modulates trichome cell wall composition and susceptibility to powdery mildew. Plant Cell, 2024, 36: 1007-35. DOI:10.1093/plcell/koad319 |
| [30] |
Förderer A, Yu D, Li E, et al. Resistosomes at the interface of pathogens and plants. Curr Opin Plant Biol, 2022, 67: 102212. DOI:10.1016/j.pbi.2022.102212 |
| [31] |
Duxbury Z, Wu CH, Ding P. A comparative overview of the intracellular guardians of plants and animals: NLRs in innate immunity and beyond. Annu Rev Plant Biol, 2021, 72: 155-84. DOI:10.1146/annurev-arplant-080620-104948 |
| [32] |
Shao ZQ, Xue JY, Wu P, et al. Large-scale analyses of angiosperm nucleotide-binding site-leucine-rich repeat genes reveal three anciently diverged classes with distinct evolutionary patterns. Plant Physiol, 2016, 170: 2095-109. DOI:10.1104/pp.15.01487 |
| [33] |
Bi G, Su M, Li N, et al. The ZAR1 resistosome is a calcium-permeable channel triggering plant immune signaling. Cell, 2021, 184: 3528-41. DOI:10.1016/j.cell.2021.05.003 |
| [34] |
Förderer A, Li E, Lawson AW, et al. A wheat resistosome defines common principles of immune receptor channels. Nature, 2022, 610: 532-9. DOI:10.1038/s41586-022-05231-w |
| [35] |
Jacob P, Kim NH, Wu F, et al. Plant "helper" immune receptors are Ca2+-permeable nonselective cation channels. Science, 2021, 373: 420-5. DOI:10.1126/science.abg7917 |
| [36] |
Wang J, Hu M, Wang J, et al. Reconstitution and structure of a plant NLR resistosome conferring immunity. Science, 2019, 364: aav5870. DOI:10.1126/science.aav5870 |
| [37] |
Wang J, Wang J, Hu M, et al. Ligand-triggered allosteric ADP release primes a plant NLR complex. Science, 2019, 364: aav5868. DOI:10.1126/science.aav5868 |
| [38] |
Hu M, Qi J, Bi G, et al. Bacterial effectors induce oligomerization of immune receptor ZAR1 in vivo. Mol Plant, 2020, 13: 793-801. DOI:10.1016/j.molp.2020.03.004 |
| [39] |
Zhao YB, Liu MX, Chen TT, et al. Pathogen effector AvrSr35 triggers Sr35 resistosome assembly via a direct recognition mechanism. Sci Adv, 2022, 8: eabq5108. DOI:10.1126/sciadv.abq5108 |
| [40] |
Madhuprakash J, Toghani A, Contreras MP, et al. A disease resistance protein triggers oligomerization of its NLR helper into a hexameric resistosome to mediate innate immunity. Sci Adv, 2024, 10: eadr2594. DOI:10.1126/sciadv.adr2594 |
| [41] |
Castel B, Ngou PM, Cevik V, et al. Diverse NLR immune receptors activate defence via the RPW8-NLR NRG1. New Phytol, 2019, 222: 966-80. DOI:10.1111/nph.15659 |
| [42] |
Ma S, Lapin D, Liu L, et al. Direct pathogen-induced assembly of an NLR immune receptor complex to form a holoenzyme. Science, 2020, 370: abe3069. DOI:10.1126/science.abe3069 |
| [43] |
Jia A, Huang S, Song W, et al. TIR-catalyzed ADP-ribosylation reactions produce signaling molecules for plant immunity. Science, 2022, 377: eabq8180. DOI:10.1126/science.abq8180 |
| [44] |
Huang S, Jia A, Song W, et al. Identification and receptor mechanism of TIR-catalyzed small molecules in plant immunity. Science, 2022, 377: eabq3297. DOI:10.1126/science.abq3297 |
| [45] |
Yu D, Song W, Tan EYJ, et al. TIR domains of plant immune receptors are 2', 3'-cAMP/cGMP synthetases mediating cell death. Cell, 2022, 185: 2370-86. DOI:10.1016/j.cell.2022.04.032 |
| [46] |
Martin R, Qi T, Zhang H, et al. Structure of the activated ROQ1 resistosome directly recognizing the pathogen effector XopQ. Science, 2020, 370: abd9993. DOI:10.1126/science.abd9993 |
| [47] |
Yang Y, Furzer OJ, Fensterle EP, et al. Paired plant immune CHS3-CSA1 receptor alleles form distinct hetero-oligomeric complexes. Science, 2024, 383: eadk3468. DOI:10.1126/science.adk3468 |
| [48] |
Yu H, Xu W, Chen S, et al. Activation of a helper NLR by plant and bacterial TIR immune signaling. Science, 2024, 386: 1413-20. DOI:10.1126/science.adr3150 |
| [49] |
Wu Y, Xu W, Zhao G, et al. A canonical protein complex controls immune homeostasis and multipathogen resistance. Science, 2024, 386: 1405-12. DOI:10.1126/science.adr2138 |
| [50] |
Wang H, Tan J, Cui X, et al. Switch of TIR signaling by a Ca2+ sensor activates ADR1 recognition of pRib-AMP-EDS1-PAD4 for stomatal immunity. bioRxiv, 2024. DOI:10.1101/2024.10.29.620780 |
| [51] |
Wu CH, Abd-El-Haliem A, Bozkurt TO, et al. NLR network mediates immunity to diverse plant pathogens. Proc Natl Acad Sci USA, 2017, 114: 8113-8. DOI:10.1073/pnas.1702041114 |
| [52] |
Wu CH, Derevnina L, Kamoun S. Receptor networks underpin plant immunity. Science, 2018, 360: 1300-1. DOI:10.1126/science.aat2623 |
| [53] |
Ma S, An C, Lawson AW, et al. Oligomerization-mediated autoinhibition and cofactor binding of a plant NLR. Nature, 2024, 632: 869-76. DOI:10.1038/s41586-024-07668-7 |
| [54] |
Selvaraj M, Toghani A, Pai H, et al. Activation of plant immunity through conversion of a helper NLR homodimer into a resistosome. PLoS Biol, 2024, 22: e3002868. DOI:10.1371/journal.pbio.3002868 |
| [55] |
Contreras MP, Pai H, Thompson R, et al. The nucleotide-binding domain of NRC-dependent disease resistance proteins is sufficient to activate downstream helper NLR oligomerization and immune signaling. New Phytol, 2024, 243: 345-61. DOI:10.1111/nph.19818 |
| [56] |
Liu F, Yang Z, Wang C, et al. Activation of the helper NRC4 immune receptor forms a hexameric resistosome. Cell, 2024, 187: 4877-89. DOI:10.1016/j.cell.2024.07.013 |
| [57] |
Wang W, Qin L, Zhang W, et al. WeiTsing, a pericycle-expressed ion channel, safeguards the stele to confer clubroot resistance. Cell, 2023, 186: 2656-71. DOI:10.1016/j.cell.2023.05.023 |
| [58] |
Shen Q, Hasegawa K, Oelerich N, et al. Cytoplasmic calcium influx mediated by plant MLKLs confers TNL-triggered immunity. Cell Host Microbe, 2024, 32: 453-65. DOI:10.1016/j.chom.2024.02.016 |
| [59] |
Li H, Men W, Ma C, et al. Wheat powdery mildew resistance gene Pm13 encodes a mixed lineage kinase domain-like protein. Nat Commun, 2024, 15: 2449. DOI:10.1038/s41467-024-46814-7 |
| [60] |
Mahdi LK, Huang M, Zhang X, et al. Discovery of a family of mixed lineage kinase domain-like proteins in plants and their role in innate immune signaling. Cell Host Microbe, 2020, 28: 813-24. DOI:10.1016/j.chom.2020.08.012 |
| [61] |
Meng Y, Garnish SE, Davies KA, et al. Phosphorylation-dependent pseudokinase domain dimerization drives full-length MLKL oligomerization. Nat Commun, 2023, 14: 6804. DOI:10.1038/s41467-023-42255-w |
| [62] |
Lee S, Lee HY, Kang HJ, et al. Oomycete effector AVRblb2 targets cyclic nucleotide-gated channels through calcium sensors to suppress pattern-triggered immunity. New Phytol, 2024, 241: 1277-91. DOI:10.1111/nph.19430 |
| [63] |
Naveed ZA, Bibi S, Ali GS. The Phytophthora RXLR effector Avrblb2 modulates plant immunity by interfering with Ca2+ signaling pathway. Front Plant Sci, 2019, 10: 374. DOI:10.3389/fpls.2019.00374 |
| [64] |
You Y, Koczyk G, Nuc M, et al. The eINTACT system dissects bacterial exploitation of plant osmosignalling to enhance virulence. Nat Plants, 2023, 9: 128-41. |
| [65] |
Ruiz-Ramón F, Rodríguez-Sepúlveda P, Bretó P, et al. The tomato calcium-permeable channel 4.1 (SlOSCA4.1) is a susceptibility factor for pepino mosaic virus. Plant Biotechnol J, 2023, 21: 2140-54. DOI:10.1111/pbi.14119 |
| [66] |
Li S, Lin D, Zhang Y, et al. Genome-edited powdery mildew resistance in wheat without growth penalties. Nature, 2022, 602: 455-60. DOI:10.1038/s41586-022-04395-9 |
| [67] |
Acevedo-Garcia J, Spencer D, Thieron H, et al. mlo-based powdery mildew resistance in hexaploid bread wheat generated by a non-transgenic TILLING approach. Plant Biotechnol J, 2017, 15: 367-78. DOI:10.1111/pbi.12631 |
| [68] |
Wang Y, Cheng X, Shan Q, et al. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nat Biotechnol, 2014, 32: 947-51. DOI:10.1038/nbt.2969 |
| [69] |
Pessina S, Lenzi L, Perazzolli M, et al. Knockdown of MLO genes reduces susceptibility to powdery mildew in grapevine. Hortic Res, 2016, 3: 16016. DOI:10.1038/hortres.2016.16 |
| [70] |
Ma M, Yang L, Hu Z, et al. Multiplex gene editing reveals cucumber MILDEW RESISTANCE LOCUS O family roles in powdery mildew resistance. Plant Physiol, 2024, 195: 1069-88. DOI:10.1093/plphys/kiae047 |
| [71] |
Shnaider Y, Elad Y, Rav-David D, et al. Development of powdery mildew resistance in cucumber using CRISPR/Cas9-mediated mutagenesis of CsaMLO8. Phytopathology, 2023, 113: 786-90. DOI:10.1094/PHYTO-06-22-0193-FI |
| [72] |
Bui TP, Le H, Ta DT, et al. Enhancing powdery mildew resistance in soybean by targeted mutation of MLO genes using the CRISPR/Cas9 system. BMC Plant Biol, 2023, 23: 533. DOI:10.1186/s12870-023-04549-5 |
| [73] |
Pessina S, Angeli D, Martens S, et al. The knock-down of the expression of MdMLO19 reduces susceptibility to powdery mildew (Podosphaera leucotricha) in apple (Malus domestica). Plant Biotechnol J, 2016, 14: 2033-44. DOI:10.1111/pbi.12562 |
| [74] |
Derevnina L, Contreras MP, Adachi H, et al. Plant pathogens convergently evolved to counteract redundant nodes of an NLR immune receptor network. PLoS Biol, 2021, 19: e3001136. DOI:10.1371/journal.pbio.3001136 |
| [75] |
Contreras MP, Pai H, Selvaraj M, et al. Resurrection of plant disease resistance proteins via helper NLR bioengineering. Sci Adv, 2023, 9: eadg3861. DOI:10.1126/sciadv.adg3861 |
| [76] |
Sugihara Y, Kourelis J, Contreras MP, et al. Helper NLR immune protein NRC3 evolved to evade inhibition by a cyst nematode virulence effector. bioRxiv, 2024. DOI:10.1101/2024.06.16.598756 |
| [77] |
Wang MY, Chen JB, Wu R, et al. The plant immune receptor SNC1 monitors helper NLRs targeted by a bacterial effector. Cell Host Microbe, 2023, 31: 1792-803. DOI:10.1016/j.chom.2023.10.006 |
| [78] |
Qi P, Zhang D, Zhang Y, et al. Ubiquitination and degradation of plant helper NLR by the Ralstonia solanacearum effector RipV2 overcome tomato bacterial wilt resistance. Cell Rep, 2024, 43: 114596. DOI:10.1016/j.celrep.2024.114596 |
| [79] |
Tamborski J, Seong K, Liu F, et al. Altering specificity and autoactivity of plant immune receptors Sr33 and Sr50 via a rational engineering approach. Mol Plant Microbe Interact, 2023, 36: 434-46. DOI:10.1094/MPMI-07-22-0154-R |
| [80] |
Seong K, Wei W, Vega B, et al. Engineering the plant intracellular immune receptor Sr50 to restore recognition of the AvrSr50 escape mutant. bioRxiv, 2024. DOI:10.1101/2024.08.07.607039 |
 2025, Vol. 37
2025, Vol. 37 

 朱旺升,中国农业大学植物保护学院教授、博士生导师,玉米生物育种全国重点实验室PI。2005年本科毕业于华中农业大学,2008年硕士毕业于东华大学,2014年在澳大利亚蒙纳士大学获博士学位,2014—2019年在德国马普学会生物学研究所从事博士后研究,2019年10月入职中国农业大学。现任植物生物安全系主任、院学术委员会和学位委员会委员,兼任第十二届中国植物病理学会理事/副秘书长、《植物病理学报》青年编委会主任和Journal of Integrative Plant Biology编委。主要研究方向为植物抗病遗传,发现了一类调控植物先天免疫的小肽门控钙通道(2023, Molecular Cell);揭示了病原菌靶标植物关键免疫节点的分子机制和植物演化的攻防策略(2023, Cell Host & Microbe)。近年来,致力于玉米真菌病害抗性基因挖掘和利用,并开展禾谷镰孢-玉米分子互作研究,鉴定了玉米抗病抗虫双重抗性基因ZmBGLU17(2024, Plant Biotechnology Journal)和广谱抗病基因ZmLecRK1 (2024, Molecular Plant)
朱旺升,中国农业大学植物保护学院教授、博士生导师,玉米生物育种全国重点实验室PI。2005年本科毕业于华中农业大学,2008年硕士毕业于东华大学,2014年在澳大利亚蒙纳士大学获博士学位,2014—2019年在德国马普学会生物学研究所从事博士后研究,2019年10月入职中国农业大学。现任植物生物安全系主任、院学术委员会和学位委员会委员,兼任第十二届中国植物病理学会理事/副秘书长、《植物病理学报》青年编委会主任和Journal of Integrative Plant Biology编委。主要研究方向为植物抗病遗传,发现了一类调控植物先天免疫的小肽门控钙通道(2023, Molecular Cell);揭示了病原菌靶标植物关键免疫节点的分子机制和植物演化的攻防策略(2023, Cell Host & Microbe)。近年来,致力于玉米真菌病害抗性基因挖掘和利用,并开展禾谷镰孢-玉米分子互作研究,鉴定了玉米抗病抗虫双重抗性基因ZmBGLU17(2024, Plant Biotechnology Journal)和广谱抗病基因ZmLecRK1 (2024, Molecular Plant)