(2 中国科学院上海药物研究所,上海 201203)
(2 Shanghai Institute of Materia Medica, Chinese Academy of Sciences, Shanghai 201203, China)
生命现象展现出了独特的复杂性和多样性,涵盖了从分子、细胞到个体乃至生态系统等多个层级之间的相互关联。每一个层次不仅构成了庞大而复杂的网络结构,而且遵循着自身特有的运行规律。面对这种复杂性和多样性,人工智能技术在处理生命科学数据方面取得了显著进展。2024年,人工智能在生命科学领域的应用更加广泛和深入。这一年,诺贝尔物理学奖和化学奖被授予在人工智能领域,特别是在生物计算方面取得突破性成就的科学家,这一举措标志着人工智能正在重塑生命科学研究的范式[1]。本文将从研究范式的革新、新理论的发现以及新场景应用的视角,通过分析近年来尤其是2024年以来的研究新进展,深入探讨人工智能驱动下生命科学研究的新态势,以期为生命科学领域的研究提供新的思路和方向。
1 人工智能驱动生命科学研究范式的革新研究范式是研究者在科学活动中遵循的基本框架,包括研究的目标、方法、工具、组织形式和评估标准等要素。传统生命科学研究范式历经了从早期基于观察和描述的经验科学,到利用数学模型构建理论的理论科学阶段,再到借助计算机模拟的计算科学时代,以及当前以数据分析为核心的数据科学时期。面对生命系统的高度复杂性,如多层次结构、动态变化和非线性关系等,传统范式在处理海量生物数据和揭示复杂生命规律方面面临诸多挑战,然而人工智能的出现为生命科学研究带来了新的范式。
1.1 “假说驱动”与“数据驱动”的融合人工智能正在快速改变生命科学领域的数据获取、分析挖掘与深度应用的方式,为“假说驱动”与“数据驱动”研究范式的融合提供了手段[2]。一方面,数据驱动的方法可以为假说驱动的研究提供新的线索和方向。通过对大量生命科学文献的文本挖掘和数据分析,发现潜在的疾病机制和治疗靶点,为科学家提出假设提供依据。另一方面,假说驱动的实验结果又可以为数据驱动的模型提供训练数据,不断优化模型的性能。这种双向互动使得生命科学研究更加高效和精准,加速了科学发现的进程[3]。
2024年以来,人工智能驱动生命科学新的研究方法与工具不断涌现。新的研究方法,如基于Transformer架构的深度学习模型LucaProt,能够快速准确地挖掘环境样本中的RNA病毒基因序列,预测出超过16万种可能的“暗物质病毒”,这不仅加快了病毒发现的速度和准确性,也预示着病毒学研究范式的潜在转变[4]。同时,自动化实验平台的发展,包括全自主实验操作机器人的应用,实现了实验操作的加速及结果获取的即时性,并通过人工智能实时分析处理数据,提高了实验效率和可靠性,尤其是在药物研发过程中对候选药物的快速筛选[5]。
1.2 解析低维与高维生物信息的复杂关联人工智能可以从海量数据中识别出低维数据与高维信息之间的关联,解析复杂的非线性关系,揭示生物大分子结构的生成规律、基因表达的调控机制,以及个体发育和衰老等多因素交互作用的底层规律[6]。例如,美国团队开发的“Evo”多模态机器学习模型,通过分析数百万微生物基因组,能够高精度地解码和设计DNA、RNA和蛋白质序列,同时预测核苷酸序列微小变化对生物体适应性的影响,生成符合实际的基因组长度序列,并据此设计全新的生物系统[7]。随着组学测序技术向多尺度、跨模态方向发展,人工智能技术被用来整合多种类型的组学数据,如蛋白质组学、基因组学、代谢组学[8]和空间组学[9]等。基于人工智能的多组学分析,已成为肿瘤精准医学领域的一个关键研究方向,特别是在诊断、分类、早期筛查、反应评估和预后预测等方面的应用[10]。此外,多模态数据融合的能力得到了增强,生命科学领域内不同来源的数据(如基因表达、蛋白质相互作用、临床症状等)得以更好地整合,促进了对生命现象的全面理解。例如,HEALNet是一种灵活的多模态融合架构,能够处理图像、表格和图形数据,通过保留特定模态的结构信息并捕获跨模态交互,成功地在癌症基因组图谱数据集的生存分析任务中实现了平均7%的性能提升[11]。
1.3 AI跨界融合赋能生命科学多维拓展人工智能与多种技术的交叉融合,将促进研究与应用水平的进一步提升。首先,人工智能与合成生物学的结合,进一步推动了研究和生产范式的变革。在大模型支持下,合成生物学从传统的“设计-构建-测试(DBT)”循环升级为“设计-构建-测试-学习(DBTL)”循环后,进一步迭代为“设计-构建-测试-学习-预测(DBTLP)”循环,这让微生物细胞工厂等领域的复杂手动设计和繁琐测试分析不再成为瓶颈[12],使得RNA药物等生物药的开发效率得以大幅提升[13]。其次,在追求高精度和准确性的研究与诊断工具过程中,微流控设备因能够显著减少样本量和研究成本而被视为具有巨大潜力的解决方案。然而,这些设备所生成的数据复杂且处理过程耗时较长,因此研究人员逐渐转向集成了人工智能技术的微流控平台,即所谓的“AI集成微流控”,由此促进了研究效率和效能的提升[14]。再次,在应用领域中,“纳米治疗诊断学”(nanotheranostics)作为一种新兴范式,其广泛采纳仍面临纳米颗粒合成耗时、对纳米-生物相互作用理解不足等障碍,而人工智能的应用则提供了一套能够执行耗时和结果感知任务的工具,为这一领域带来了新的发展机遇[15]。此外,在无标记多光子显微镜等领域中,人工智能的应用也为打造新型诊断框架提供了可能[16]。
2 人工智能驱动生命科学理论的发展生命科学理论作为对生命现象和过程的系统性阐释,其目的在于深入探寻生命的本质、起源、进化路径及生命活动的内在运作机制。它是在长期的大量实验观察与数据分析积累的基础上逐步构建而成的,在生命科学研究进程中发挥着至关重要的指导框架与理论基石的作用。如今,人工智能凭借其强大的数据处理与预测分析能力强势介入生命科学领域,为生命科学理论的进一步发展注入了新的活力。
人工智能与合成生物学的深度融合,催生了生成式生物学(Generative Biology)。生成式生物学的核心在于利用生成式人工智能(Generative AI)从大量数据中提取模式,并基于这些模式进行创新性设计[17]。以AI在生物序列设计中的应用为例,DNA序列、RNA序列、蛋白质序列的设计因为AI的赋能,使得科学家能够在计算机上模拟出大量生物分子的组合。通过“数字实验”极大地减少了实际实验室工作中的试错成本,加快了从理论到实践的转化速度,快速筛选出最有潜力的序列加以实际验证[18]。由此,主动学习与闭环反馈得以融合,用于帮助科学家解析基因组的复杂调控网络,预测基因表达模式,理解蛋白质相互作用,并洞察生理和病理的规律。
通过虚拟实验与物理实验的迭代反馈循环,这种模式显著提高了生物系统设计的效率和准确性。2024年4月,美国Profluent推出了首个开源的人工智能生成基因编辑器OpenCRISPR-1,该基因编辑器完全由人工智能从头设计,并首次成功地开展了人类基因组精确编辑[19]。此外,生成对抗网络和大语言模型(如GPT-4)在蛋白质结构预测[20]和基因调控元件设计[21-22]中也发挥了重要作用。在具体应用方面,生成式生物学已经展现出广泛的应用潜力。例如,华盛顿大学蛋白质设计研究所所长David Baker团队首次使用生成式人工智能从头设计出全新的抗体,有望推动AI在抗体药物市场的应用,为新型疫苗、非天然氨基酸蛋白质、新型药物输送载体等领域提供支持[23]。北京理工大学团队开发出生成式DNA大语言模型,为从头设计以噬菌体为代表的完整生物体基因组奠定了新的理论基础[24]。
人工智能技术在整合生物学中发挥重要作用,进一步加深了从分子水平到细胞、组织乃至整个有机体的理解。在生命科学的各研究层级上,人工智能的赋能带来了诸多的突破。例如,利用深度学习算法整合了病理影像、基因和临床数据,以此来预测乳腺癌患者的无病生存期[25]。在细胞结构和功能的整合研究中,结合人工智能的结构生物学方法可以帮助解析细胞内复杂的分子结构和相互作用[8]。通过结合冷冻电镜技术和人工智能预测算法,可以更准确地确定蛋白质和核酸的三维结构,从而深入了解细胞内的分子机制[26]。同时,AI还可用于整合生物系统的不同时间尺度和空间尺度的信息,通过建立多尺度模型,将细胞内的分子过程与组织和器官水平的生理功能联系起来,实现对生命系统的全面理解[27]。
更为关键的是,人工智能的应用开启了跨层级研究的新纪元,为揭示跨层级的统一性规律提供了可能,推动了整合生物学(Integrative Biology)的进一步发展。人工智能不仅能够帮助科学家识别生物系统中的复杂模式和非线性关系,还使得跨层级的生物过程预测和模拟成为可能,揭示数据背后可能隐藏的模式,使研究人员能够从更深层次理解基因表达调控、细胞分化、疾病发生发展等生命过程,为生命科学的研究拓展了新的视野。在代谢研究中,人体代谢涉及从分子到个体的跨层级的调控,依托传统方法处理这些高通量、高维度、高度复杂的数据捉襟见肘,但AI的应用使得分析检测、数据预处理、生物标志物发现、预测建模和多组学数据集成得以高效展开[28]。在胰腺癌[29]、肝细胞癌[30]、膀胱癌[31]等肿瘤治疗领域,人工智能展现出其在识别临床预测生物标志物方面的强大能力,推动了全球精准肿瘤学的普及。再如,在帕金森病[32]、阿尔茨海默病[33]等尚无良好治愈方法的神经退行性疾病研究与诊疗中,AI的应用可以促成更为精准的生物标志物评估体系的构建,从而实现更有效的诊断、治疗和预后建模。在帕金森病的诊断与治疗过程中,人工智能技术的应用能够精确解析大脑与肠道之间的相互作用,即“脑-肠轴”关系,从而为探索源自肠道的非侵入性生物标志物,以实现对帕金森病的早期诊断提供了新的途径[34]。在对混合性痴呆与阿尔茨海默病进行鉴别诊断的过程中,结合人工智能模型与神经科医师的评估,相较于仅依赖神经科医师的独立评估,展现出更优的诊断效能[35]。在精神疾病领域,AI通过分析大量的医疗数据和患者的行为模式,可以辅助医生在早期阶段就发现潜在的精神健康问题,从而提供及时的干预和治疗[36]。
3 人工智能驱动生命科学新场景的应用人工智能推动了“干湿”实验的深度整合,在生命科学领域催生了一系列创新应用,涵盖了从分子尺度到个体尺度的多个层面(图 1)。“干实验”指的是利用计算机模拟和数据分析来进行的研究;而“湿实验”则是指传统的实验室操作。两者的有机结合带来了诸如数字细胞、数字器官,甚至整个数字生命的概念。这类虚拟模型可以在体外重现真实的生理过程,极大地促进了个性化医疗的发展,并为药物筛选与临床试验[37]、活性肽等功能食品[38]、智能医疗器械开发等各类健康场景,生物制造、作物育种等农业场景,以及污水处理[39]等生态环境场景的应用提供了高效的平台。
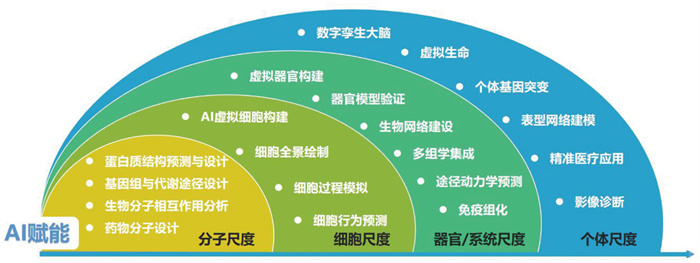
|
图 1 人工智能在生命科学不同尺度上的应用 |
生物大分子如蛋白质、核酸等在生命活动中起着至关重要的作用。这些生物大分子的结构与功能会随着细胞环境的变化而动态调整[40],深入理解它们的结构与功能对于探索生命的奥秘、开发新药及治疗方案至关重要。
近年来,人工智能在优化基因编辑技术、蛋白质结构预测等方面取得了显著成果。借助深度学习算法,AI不仅能够辨识基因组内的细微模式,还能够构建完整的基因序列,为基因编辑技术及新药研发领域提供技术支撑。例如,博德研究所等利用机器学习合成了数千个全新的顺式调节元件,可实现对基因表达更精确的控制[41]。在蛋白质结构预测领域,AI的发展尤为突出。DeepMind于2024年发布的AlphaFold 3,已经能够准确预测蛋白质、DNA、RNA等分子的结构及其相互作用[42]。另外,人工智能方法还可以从大型序列和结构数据集中训练,从头开始“编写”具有新形状和分子功能的新型蛋白质,而不依赖于自然界中发现的蛋白质[43]。尽管如此,捕捉蛋白质结构变化的信息仍然是一个挑战。研究人员正在探索结合物理原理的新方法来改进这一问题,以更准确地预测蛋白质的动力学行为[44]。
受此启发,科学家们正致力于开发可以预测更广泛生物分子间相互作用的AI模型,涵盖从RNA结构预测到蛋白质-小分子对接等多个方面。新兴的全原子模型,如RoseTTAFold All-Atom和AlphaFold3展示了处理复杂生物分子相互作用的潜力,但仍需进一步整合物理定律和扩大数据集以提高预测准确性[45]。此外,人工智能在生物分子动力学模拟领域也取得了重要进展。微软研究院推出的AI²BMD系统,结合了机器学习与传统物理学原理,实现了生物分子动力学模拟的高精度和高效性[46]。
人工智能技术的应用范围已超越了单一的生物大分子研究领域,进一步促进了生物分子复合体[47]及亚细胞层次的研究进展。以外泌体研究为例,其在肿瘤诊断等领域的应用潜力备受瞩目。然而,在过往研究中,外泌体数据集的复杂性处理一直是一个挑战。可解释的人工智能通过揭示决策过程,增强了诊断的透明度和可靠性;而不可解释的人工智能则通过其“黑匣子”式的工作机制,提供了强大的数据处理能力及高诊断准确性[48]。
3.2 虚拟细胞:人工智能模拟生命的新前沿从DNA序列到蛋白质结构,再到整个细胞的功能,人工智能正在重塑科学家们理解和操控生命基本单元的方式[49]。为了深入理解细胞的功能,科学家正构建虚拟细胞模型,以模拟、预测并指导细胞行为。基于这一理念,有研究采用“虚拟细胞” (virtual cell)这一术语来定义模拟细胞的生物功能和相互作用的计算模型[50]。
传统上,这些模型基于规则系统,结合生物学机制的假设与实验数据,依赖于数学或计算方法如微分方程和随机模拟。随着人工智能和组学技术的进步,现在可以利用数据直接构建人工智能驱动的虚拟细胞(AI virtual cell, AIVC),使得空间组学、时空组学的更大应用潜力得以释放[51]。AIVC是一种多尺度、多模态的大型神经网络模型,能够表示并模拟分子、细胞和组织在不同状态下的行为。这种模型整合了多种数据类型,提供对生命系统复杂性的深刻理解,并推动生物医学研究的重大进展。例如,清华大学团队开发的scFoundation细胞大模型,基于海量细胞基因表达数据训练而成,能够在“虚拟药物试验”等多种生物医学任务中展现卓越性能,为单细胞研究领域提供了新的范式[52]。此外,陈-扎克伯格倡议(Chan Zuckerberg Initiative, CZI)于2024年3月启动了AI咨询小组和驻留计划,旨在加速虚拟细胞模型的开发[53]。
人工智能的应用,不仅提升了数字细胞的构建能力,还为未来开发生成式的数字细胞提供了可能。由此,细胞生物学的“干实验”与“湿实验”的双向链接通路或得以贯通,从而激活更高层级的研究[54]。
3.3 数字器官:人工智能迈向器官级精准模拟的新阶段细胞层面的功能构成了组织和器官生理学的基础。人类细胞图谱(HCA)项目利用人工智能技术,已经初步描绘出人体已知细胞类型的轮廓,在骨骼[55]、脊椎[56]、肢体[57]、肺[58-59]、皮肤[60]、视网膜[61-62]等领域的研究成果显著。在这一过程中,人工智能在数据处理[63]、注释分类[64-65]、模型训练评估[66-68]、数据整合[69-70]及可视化[71-73]等方面发挥关键作用,极大地促进了对器官生理过程的理解。此外,人工智能在胸腺[74]、胃肠道[75]、肺部[76]、牙齿[77]、大脑[78-79]、子宫[80]、皮肤[81]等器官特异性研究中取得重要进展,为数字器官构建提供了坚实支撑。
在精确多尺度计算模型的驱动下,对人体器官的组成、形态及功能进行数字化建模,进而开发出能够模拟其生理和病理状态的数字器官。这一过程实现了生命科学、生物技术、数字技术、仿生技术等领域的科技融合,为揭示生命本质规律、推动新药研发、预防重大疾病提供了赋能。从数字器官发展所需的生物技术看,分子层面的基因编辑等技术、细胞层面的干细胞等技术日渐成熟,为数字器官的系统构建提供了数据基础。在此基础上,数字孪生整合多尺度数据与计算模型,可在细胞、组织/器官及生物体水平精准预测模拟,提高人类对生物系统的认知和控制能力,实现对生命活动的精确调控。在这种场景中,多模态数据得以有机融合和集成应用[82]。例如,人工智能在心血管领域的数字孪生应用,能够将人口统计学、症状特征、心电图结果、实验室值、生物标志物水平和影像学等数据融入其中[83],从而扩展心血管筛查和监测的范围,使心血管护理更加个性化、精确和有效[84]。北京智源人工智能研究院官网显示,智源研究院实现了数字孪生心脏电功能的超实时仿真,为医学基础研究及临床应用,如射频消融方案规划、心脏起搏器植入方案提供了重要支持[85]。数坤科技官网报道,其推出的“数字肝”算法具备超过40亿个神经元,能够处理超过4 000张图像,并整合了“数字心”“数字脑”等部位的数字化产品,形成了基于数字人体技术平台的智慧解决方案[86]。人工智能在类器官研究中也发挥了重要作用,提升了研究的客观性、准确性和速度,并通过图像分析、组学数据处理等技术,实现了对类器官的精确识别和分类[87]。未来,在克服高质量生物医学数据采集处理、准确构建器官系统计算表示等挑战后,可精准模拟、可量化预测、可动态评估的数字器官或将得以应用[88]。
值得关注的是,随着生物技术与人工智能的融合发展,数字器官的模拟将不仅仅局限于传统的“低维”层面,而是可以体现复杂的肿瘤微环境等高维的信息。在这些研究中,基因调控机制、细胞动力学、组织环境的特性等多维信息得以全面呈现,为攻克肿瘤等复杂疾病提供了更为坚实的支撑[89]。
3.4 虚拟生命:数字生命与人工智能的融合虚拟生命代表了数字生命与人工智能深度融合的新阶段,通过模拟生命的核心特征,在数字世界中创建、演化与互动,形成一种高度智能化、自适应的生命形态。以新加坡南洋理工大学、商汤科技和上海AI实验室共同发起的“Digital Life Project”为例,该项目利用AI技术和动作合成技术创造出具有自主性的3D虚拟角色。这些角色不仅能够进行交流对话,还具备独立的人格特质,能够感知社交环境并作出相应的情感表达和反应。项目的两大核心组成部分是SocioMind数字大脑和MoMat-MoGen数字身体,前者模拟人类的思维和决策过程,后者则负责控制角色的3D身体动作,确保角色能够依据不同场景做出恰当的反应[90]。
数字孪生大脑概念的出现进一步推动了这一融合进程。通过整合神经影像学和神经生物学数据,并结合人工智能技术,数字孪生大脑能够模拟生物脑的功能,从而构建出类似脑的人工智能系统。复旦大学的研究团队已成功模拟再现人脑860亿神经元,与原脑相似度高达90%[91]。此项技术可用于模拟老年性痴呆等脑相关疾病的病理过程,进而预测疾病的发展态势。在构建出老年性痴呆患者的数字脑模型后,研究者可以通过施加不同强度和频率的模拟刺激,获取数字脑的反馈,为生物脑的病理治疗提供指导[92]。
4 展望展望未来,人工智能与生命科学的深度融合,将开启多模态[93]、多维度、多尺度、多学科研究的新范式[94],驱动理论发现和实践应用的革新。在研究范式方面,数据驱动与假说驱动的深度融合将持续推动生命科学研究的高效化和精准化。新的研究方法和工具将不断涌现,进一步提升实验效率和可靠性,为科学发现提供更多的线索和方向。在理论发展方面,人工智能与合成生物学、整合生物学等新学科的结合,将继续推动生命科学理论的深入发展。在新场景应用方面,数字细胞、数字器官和虚拟生命等概念将不断拓展和深化。人工智能在生物大分子解析与设计中的应用将为新药研发和疾病治疗提供更多的可能性。虚拟细胞模型将更加准确地模拟细胞行为,为生物医学研究提供新的范式。数字器官的精准模拟将为个性化医疗和精准医学提供更强大的工具[95],同时也需要克服数据采集和模型构建等方面的挑战。虚拟生命的发展将为教育、娱乐、医疗康复等领域带来新的机遇和挑战。
在推动生命科学研究发展的历程中,人工智能与量子计算等新兴技术的融合进一步推动生命科学理论的发展[96]。量子计算作为一项颠覆性技术,其与人工智能的结合正在解决传统计算难以克服的问题。量子计算通过量子位的独特特性,能够同时处理多个状态,从而实现比传统计算机更快、更强大的计算能力[97]。2024年量子计算纠错技术的进步,说明了这种融合应用的广阔前景[98]。此外,生物活动和生命现象的研究本身,反过来也会启发人工智能的发展,由此促成了多方联动的正向循环反馈的升级[99]。
随着人工智能技术的不断进步,其在生命科学领域的应用前景变得日益广阔和深入。这不仅要求科学家们在技术上不断创新,还必须重视智能化工作流程和知识体系的构建[100],以推动“可共享的人工智能”应用的发展[101]。在伦理和法律层面,深入地探讨和规范变得至关重要。一方面,人工智能领域广泛采用的机器学习与深度学习算法本质上依赖于训练数据集的质量,构建负责任的机器学习数据集显得尤为重要[102];另一方面,确保个人隐私,防止数据滥用,以及处理人工智能产生的生物信息可能引发的知识产权问题,已成为亟待解决的重要议题。同时,随着虚拟生命和数字器官等概念的实现,如何界定这些虚拟实体的法律地位和权利,亦成为亟待解决的问题。跨学科合作的重要性日益凸显,生命科学、计算机科学、物理学等多学科的深度融合将为解决复杂问题提供新的思路和方法。例如,在临床研究中,要将人工智能安全地融入临床环境,通常需要进行随机临床试验,这涉及伦理学、转化医学、生物统计学以及人工智能开发等不同领域的专业知识。在这一过程中,必须考虑如何衡量社会价值、建立科学有效性、确保公平选择参与者、评估不同患者亚组的风险收益比,以及解决知情同意和数据使用条款固有的复杂性等问题[103]。因此,在这些应用过程中,选择性预测方面的理想解决方案显得尤为重要[104]。
总之,人工智能在生命科学领域的应用前景广阔,将不断推动生命科学的进步,为人类健康和生活带来更多福祉。然而,仍需关注其在技术、伦理和法律等方面面临的挑战,通过跨学科合作和规范管理,确保其健康、可持续地发展。
| [1] |
Abriata LA. The Nobel Prize in Chemistry: past, present, and future of AI in biology. Commun Biol, 2024, 7: 1409. DOI:10.1038/s42003-024-07113-5 |
| [2] |
Embedding AI in biology. Nat Methods, 2024, 21: 1365-6
|
| [3] |
李丹丹, 马征远, 张博文, 等. 人工智能驱动生命科学研究的发展态势. 生命科学, 2024, 36: 122-8. DOI:10.13376/j.cbls/2024015 |
| [4] |
Hou X, He Y, Fang P, et al. Using artificial intelligence to document the hidden RNA virosphere. Cell, 2024, 187: 6929-42. DOI:10.1016/j.cell.2024.09.027 |
| [5] |
Angelopoulos A, Cahoon JF, Alterovitz R. Transforming science labs into automated factories of discovery. Sci Robot, 2024, 9: eadm6991. DOI:10.1126/scirobotics.adm6991 |
| [6] |
李鑫, 于汉超. 人工智能驱动的生命科学研究新范式. 中国科学院院刊, 2024, 39: 50-8. |
| [7] |
Nguyen E, Poli M, Durrant MG, et al. Sequence modeling and design from molecular to genome scale with Evo. Science, 2024, 386: 729-30. DOI:10.1126/science.adt3007 |
| [8] |
Bifarin OO, Fernández FM. Automated machine learning and explainable AI (AutoML-XAI) for metabolomics: improving cancer diagnostics. J Am Soc Mass Spectrom, 2024, 35: 1089-100. DOI:10.1021/jasms.3c00403 |
| [9] |
Ang KS, Chen J. A machine learning tool for spatial multi-omics. Nat Methods, 2024, 21: 1593-4. DOI:10.1038/s41592-024-02358-8 |
| [10] |
Zadeh Shirazi A, Tofighi M, Gharavi A, et al. The application of artificial intelligence to cancer research: a comprehensive guide. Technol Cancer Res Treat, 2024, 23: 15330338241250324. DOI:10.1177/15330338241250324 |
| [11] |
Hemker K, Simidjievski N, Jamnik M. HEALNet: multimodal fusion for heterogeneous biomedical data. arXiv, 2024. DOI:10.48550/arXiv.2311.09115 |
| [12] |
Gong X, Zhang J, Gan Q, et al. Advancing microbial production through artificial intelligence-aided biology. Biotechnol Adv, 2024, 74: 108399. DOI:10.1016/j.biotechadv.2024.108399 |
| [13] |
Hill A, True JM, Jones CH. Transforming drug development with synthetic biology and AI. Trends Biotechnol, 2024, 42: 1072-5. DOI:10.1016/j.tibtech.2024.01.008 |
| [14] |
Asadian E, Bahramian F, Siavashy S, et al. A review on recent advances of AI-integrated microfluidics for analytical and bioanalytical applications. Trends Analyt Chem, 2024, 181: 118004. DOI:10.1016/j.trac.2024.118004 |
| [15] |
Rao L, Yuan Y, Shen X, et al. Designing nanotheranostics with machine learning. Nat Nanotechnol, 2024, 19: 1769-81. DOI:10.1038/s41565-024-01753-8 |
| [16] |
Wang S, Pan J, Zhang X, et al. Towards next-generation diagnostic pathology: AI-empowered label-free multiphoton microscopy. Light Sci Appl, 2024, 13: 254. DOI:10.1038/s41377-024-01597-w |
| [17] |
Mock M, Langmead CJ, Grandsard P, et al. Recent advances in generative biology for biotherapeutic discovery. Trends Pharmacol Scie, 2024, 45: 255-67. DOI:10.1016/j.tips.2024.01.003 |
| [18] |
Zhang P, Wei L, Li J, et al. Artificial intelligence-guided strategies for next-generation biological sequence design. Natl Sci Rev, 2024, 11: nwae343. DOI:10.1093/nsr/nwae343 |
| [19] |
Ruffolo JA, Nayfach S, Gallagher J, et al. Design of highly functional genome editors by modeling the universe of CRISPR-Cas sequences. bioRxiv, 2024. DOI:10.1101/2024.04.22.590591 |
| [20] |
Ille AM, Markosian C, Burley SK, et al. Generative artificial intelligence performs rudimentary structural biology modeling. Sci Rep, 2024, 14: 19372. DOI:10.1038/s41598-024-69021-2 |
| [21] |
Zhang P, Wang H, Xu H, et al. Deep flanking sequence engineering for efficient promoter design using DeepSEED. Nat Commun, 2023, 14: 6309. DOI:10.1038/s41467-023-41899-y |
| [22] |
Hu M, Alkhairy S, Lee I, et al. Evaluation of large language models for discovery of gene set function. Nat Methods, 2024, 22: 82-91. |
| [23] |
Bennett NR, Watson JL, Ragotte RJ, et al. Atomically accurate de novo design of single-domain antibodies. bioRxiv, 2024. DOI:10.1101/2024.03.14.585103 |
| [24] |
Shao B, Yan J. A long-context language model for deciphering and generating bacteriophage genomes. Nat Commun, 2024, 15: 9392. DOI:10.1038/s41467-024-53759-4 |
| [25] |
Wang Z, Lin R, Li Y, et al. Deep learning-based multi-modal data integration enhancing breast cancer disease-free survival prediction. Precis Clin Med, 2024, 7: pbae012. DOI:10.1093/pcmedi/pbae012 |
| [26] |
李宽莹, 王闻雪, 朱赟, 等. 综述与专论: 原位冷冻电镜技术和可视蛋白质组学前沿. 生物化学与生物物理进展, 2024, 51: 2456-77. |
| [27] |
Karagöz Z. Dynamic computational models of cell-extracellular matrix and cell-cell interactions[D]. Maastricht: Maastricht University, 2024
|
| [28] |
Chi J, Shu J, Li M, et al. Artificial intelligence in metabolomics: a current review. Trends Analyt Chem, 2024, 178: 117852. DOI:10.1016/j.trac.2024.117852 |
| [29] |
Osipov A, Theodorescu D. Artificial intelligence uses multi-omic data to predict pancreatic cancer outcomes. Nat Cancer, 2024, 5: 226-7. DOI:10.1038/s43018-023-00698-6 |
| [30] |
Jiang M, Wu P, Zhang Y, et al. Artificial intelligence-driven platform: unveiling critical hepatic molecular alterations in hepatocellular carcinoma development. Adv Healthc Mater, 2024, 13: e2400291. DOI:10.1002/adhm.202400291 |
| [31] |
Shkolyar E, Zhou SR, Carlson CJ, et al. Optimizing cystoscopy and TURBT: enhanced imaging and artificial intelligence. Nat Rev Urol, 2024, 22: 46-54. |
| [32] |
Reddy A, Reddy RP, Roghani AK, et al. Artificial intelligence in parkinson's disease: early detection and diagnostic advancements. Ageing Res Rev, 2024, 99: 102410. DOI:10.1016/j.arr.2024.102410 |
| [33] |
Kale MB, Wankhede NL, Pawar RS, et al. AI-driven innovations in Alzheimer's disease: integrating early diagnosis, personalized treatment, and prognostic modelling. Ageing Res Rev, 2024, 101: 102497. DOI:10.1016/j.arr.2024.102497 |
| [34] |
Li Y, Ren HX, Chi CY, et al. Artificial intelligence-guided gut-microenvironment-triggered imaging sensor reveals potential indicators of Parkinson's disease. Adv Science, 2024, 11: e2307819. DOI:10.1002/advs.202307819 |
| [35] |
Artificial intelligence for differential diagnosis of dementia. Nat Med, 2024, 30: 2737-8
|
| [36] |
Tononi G, Raison C. Artificial intelligence, consciousness and psychiatry. World Psychiatry, 2024, 23: 309-10. DOI:10.1002/wps.21222 |
| [37] |
Zhang Y, Mastouri M, Zhang Y. Accelerating drug discovery, development, and clinical trials by artificial intelligence. Med, 2024, 5: 1050-70. DOI:10.1016/j.medj.2024.07.026 |
| [38] |
Chang J, Wang H, Su W, et al. Artificial intelligence in food bioactive peptides screening: recent advances and future prospects. Trends Food Sci Technol, 2024, 156: 104845. |
| [39] |
Oh S, Byeon H, Wijaya J. Machine learning surveillance of foodborne infectious diseases using wastewater microbiome, crowdsourced, and environmental data. Water Res, 2024, 265: 122282. DOI:10.1016/j.watres.2024.122282 |
| [40] |
Liu G, Niu T, Qiu M, et al. DeepETPicker: fast and accurate 3D particle picking for cryo-electron tomography using weakly supervised deep learning. Nat Commun, 2024, 15: 2090. DOI:10.1038/s41467-024-46041-0 |
| [41] |
Gosai SJ, Castro RI, Fuentes N, et al. Machine-guided design of cell-type-targeting cis-regulatory elements. Nature, 2024, 634: 1211-20. DOI:10.1038/s41586-024-08070-z |
| [42] |
Abramson J, Adler J, Dunger J, et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature, 2024, 630: 493-500. DOI:10.1038/s41586-024-07487-w |
| [43] |
Kortemme T. De novo protein design — From new structures to programmable functions. Cell, 2024, 187: 526-44. DOI:10.1016/j.cell.2023.12.028 |
| [44] |
Guan X, Tang QY, Ren W, et al. Predicting protein conformational motions using energetic frustration analysis and AlphaFold2. Proc Natl Acad Sci U S A, 2024, 121: e2410662121. DOI:10.1073/pnas.2410662121 |
| [45] |
Baek M. Towards the prediction of general biomolecular interactions with AI. Nat Methods, 2024, 21: 1382-3. DOI:10.1038/s41592-024-02350-2 |
| [46] |
Wang T, He X, Li M, et al. Ab initio characterization of protein molecular dynamics with AI2BMD. Nature, 2024, 635: 1019-27. DOI:10.1038/s41586-024-08127-z |
| [47] |
Siebenmorgen T, Menezes F, Benassou S, et al. MISATO: machine learning dataset of protein-ligand complexes for structure-based drug discovery. Nat Comput Sci, 2024, 4: 367-78. DOI:10.1038/s43588-024-00627-2 |
| [48] |
Lin X, Zhu J, Shen J, et al. Advances in exosome plasmonic sensing: device integration strategies and AI-aided diagnosis. Biosens Bioelectron, 2024, 266: 116718. DOI:10.1016/j.bios.2024.116718 |
| [49] |
Abudayyeh OO, Gootenberg JS. Programmable biology through artificial intelligence: from nucleic acids to proteins to cells. Nat Methods, 2024, 21: 1384-6. DOI:10.1038/s41592-024-02338-y |
| [50] |
Bunne C, Roohani Y, Rosen Y, et al. How to build the virtual cell with artificial intelligence: priorities and opportunities. Cell, 2024, 187: 7045-63. DOI:10.1016/j.cell.2024.11.015 |
| [51] |
Coleman K, Schroeder A, Li M. Unlocking the power of spatial omics with AI. Nat Methods, 2024, 21: 1378-81. DOI:10.1038/s41592-024-02363-x |
| [52] |
Hao M, Gong J, Zeng X, et al. Large-scale foundation model on single-cell transcriptomics. Nat Methods, 2024, 21: 1481-91. DOI:10.1038/s41592-024-02305-7 |
| [53] |
Chan Zuckerberg Initiative. AI Advisory Group to Accelerate Virtual Cell Development[EB/OL]. (2024-03-21)[2024-12-17]. https://chanzuckerberg.com/newsroom/ai-advisory-group-accelerate-virtual-cell-development/
|
| [54] |
Uhler C. Building a two-way street between cell biology and machine learning. Nat Cell Biol, 2024, 26: 13-4. DOI:10.1038/s41556-023-01279-6 |
| [55] |
To K, Fei L, Pett JP, et al. A multi-omic atlas of human embryonic skeletal development. Nature, 2024, 635: 657-67. DOI:10.1038/s41586-024-08189-z |
| [56] |
Lawrence JEG, Roberts K, Tuck E, et al. HOX gene expression in the developing human spine. Nat Commun, 2024, 15: 10023. DOI:10.1038/s41467-024-54187-0 |
| [57] |
Zhang B, He P, Lawrence JEG, et al. A human embryonic limb cell atlas resolved in space and time. Nature, 2023, 635: 668-78. |
| [58] |
Sariyar S, Sountoulidis A, Hansen JN, et al. High-parametric protein maps reveal the spatial organization in early-developing human lung. Nat Commun, 2024, 15: 9381. DOI:10.1038/s41467-024-53752-x |
| [59] |
Quach H, Farrell S, Wu MJM, et al. Early human fetal lung atlas reveals the temporal dynamics of epithelial cell plasticity. Nat Commun, 2024, 15: 5898. DOI:10.1038/s41467-024-50281-5 |
| [60] |
Gopee NH, Winheim E, Olabi B, et al. A prenatal skin atlas reveals immune regulation of human skin morphogenesis. Nature, 2024, 635: 679-89. DOI:10.1038/s41586-024-08002-x |
| [61] |
Zuo Z, Cheng X, Ferdous S, et al. Single cell dual-omic atlas of the human developing retina. Nat Commun, 2024, 15: 6792. DOI:10.1038/s41467-024-50853-5 |
| [62] |
Dorgau B, Collin J, Rozanska A, et al. Single-cell analyses reveal transient retinal progenitor cells in the ciliary margin of developing human retina. Nat Commun, 2024, 15: 3567. DOI:10.1038/s41467-024-47933-x |
| [63] |
Schuster V, Dann E, Krogh A, et al. multiDGD: a versatile deep generative model for multi-omics data. Nat Commun, 2024, 15: 10031. DOI:10.1038/s41467-024-53340-z |
| [64] |
Ergen C, Xing G, Xu C, et al. Consensus prediction of cell type labels in single-cell data with popV. Nat Genet, 2024, 56: 2731-8. DOI:10.1038/s41588-024-01993-3 |
| [65] |
Sumanaweera D, Suo C, Cujba AM, et al. Gene-level alignment of single-cell trajectories. Nat Methods, 2024, 22: 68-81. |
| [66] |
Wang J, Fonseca GJ, Ding J. scSemiProfiler: advancing large-scale single-cell studies through semi-profiling with deep generative models and active learning. Nat Commun, 2024, 15: 5989. DOI:10.1038/s41467-024-50150-1 |
| [67] |
Zinati Y, Takiddeen A, Emad A. GRouNdGAN: GRN-guided simulation of single-cell RNA-seq data using causal generative adversarial networks. Nat Commun, 2024, 15: 4055. DOI:10.1038/s41467-024-48516-6 |
| [68] |
Megas S, Lorenzi V, Marioni J C. EmptyDropsMultiome discriminates real cells from background in single-cell multiomics assays. Genome Biol, 2024, 25: 121. DOI:10.1186/s13059-024-03259-x |
| [69] |
Li T, Horsfall D, Basurto-Lozada D, et al. WebAtlas pipeline for integrated single-cell and spatial transcriptomic data. Nat Methods, 2024, 22: 3-5. |
| [70] |
Hao M, Luo E, Chen Y, et al. STEM enables mapping of single-cell and spatial transcriptomics data with transfer learning. Commun Biol, 2024, 7: 56. DOI:10.1038/s42003-023-05640-1 |
| [71] |
Heimberg G, Kuo T, DePianto DJ, et al. A cell atlas foundation model for scalable search of similar human cells. Nature, 2024. DOI:10.1038/s41586-024-08411-y |
| [72] |
Gao H, Hua K, Wu X, et al. Building a learnable universal coordinate system for single-cell atlas with a joint-VAE model. Commun Biol, 2024, 7: 977. DOI:10.1038/s42003-024-06564-0 |
| [73] |
Putri G H, Howitt G, Marsh-Wakefield F, et al. SuperCellCyto: enabling efficient analysis of large scale cytometry datasets. Genome Biol, 2024, 25: 89. DOI:10.1186/s13059-024-03229-3 |
| [74] |
Yayon N, Kedlian V R, Boehme L, et al. A spatial human thymus cell atlas mapped to a continuous tissue axis. Nature, 2024, 635: 708-18. DOI:10.1038/s41586-024-07944-6 |
| [75] |
Oliver A J, Huang N, Bartolome-Casado R, et al. Single-cell integration reveals metaplasia in inflammatory gut diseases. Nature, 2024, 635: 699-707. DOI:10.1038/s41586-024-07571-1 |
| [76] |
Nyirenda J, Hardy OM, Silva Filho JD, et al. Spatially resolved single-cell atlas unveils a distinct cellular signature of fatal lung COVID-19 in a Malawian population. Nat Med, 2024, 30: 3765-77. |
| [77] |
Easter QT, Fernandes Matuck B, Beldorati Stark G, et al. Single-cell and spatially resolved interactomics of tooth-associated keratinocytes in periodontitis. Nat Commun, 2024, 15: 5016. |
| [78] |
Steyn C, Mishi R, Fillmore S, et al. A temporal cortex cell atlas highlights gene expression dynamics during human brain maturation. Nat Genet, 2024, 56: 2718-30. |
| [79] |
Chen X, Huang Y, Huang L, et al. A brain cell atlas integrating single-cell transcriptomes across human brain regions. Nat Med, 2024, 30: 2679-91. |
| [80] |
Marečková M, Garcia-Alonso L, Moullet M, et al. An integrated single-cell reference atlas of the human endometrium. Nat Genet, 2024, 56: 1925-37. |
| [81] |
Li R, Strobl J, Poyner EFM, et al. Cutaneous T cell lymphoma atlas reveals malignant TH2 cells supported by a B cell-rich tumor microenvironment. Nat Immunol, 2024, 25: 2320-30. |
| [82] |
Warner E, Lee J, Hsu W, et al. Multimodal machine learning in image-based and clinical biomedicine: survey and prospects. Int J Comput Vis, 2024, 132: 3753-69. |
| [83] |
Lüscher TF, Wenzl FA, D'Ascenzo F, et al. Artificial intelligence in cardiovascular medicine: clinical applications. Eur Heart J, 2024, 45: 4291-304. |
| [84] |
Khera R, Oikonomou E K, Nadkarni G N, et al. Transforming cardiovascular care with artificial intelligence: from discovery to practice: JACC state-of-the-art review. J Am Coll Cardiol, 2024, 84: 97-114. |
| [85] |
北京智源人工智能研究院. 世界首次!智源研究院实现数字孪生心脏电功能超实时仿真[EB/OL]. (2024-11-28)[2024-12-13]. https://mp.weixin.qq.com/s/H2Rgb0pVgo-BLRIWo6qAEsA
|
| [86] |
数坤科技. 数坤科技携"数字人体3.0"亮相CMEF2024, 实力诠释医疗新质生产力[EB/OL]. (2024-04-15)[2024-12-13]. https://mp.weixin.qq.com/s?__biz=Mzk0MTUyMTg3Ng==&mid=2247484619&idx=1&sn=8ce66e78c6e8d220ca1bc5e48a20d8a6&chksm=c2d06a70f5a7e3662907201bb615ee70b7026755891af76a869628ca96c194a7caa1a0c240c7&token=1140625305&lang=zh_CN#rd
.
|
| [87] |
Bai L, Wu Y, Li G, et al. AI-enabled organoids: construction, analysis, and application. Bioact Mater, 2024, 31: 525-48. |
| [88] |
Hansen J, Jain AR, Nenov P, et al. From transcriptomics to digital twins of organ function. Front Cell Dev Biol, 2024, 12: 1240384. |
| [89] |
Fan JL, Nazaret A, Azizi E. A thousand and one tumors: the promise of AI for cancer biology. Nat Methods, 2024, 21: 1403-6. |
| [90] |
Cai Z, Jiang J, Qing Z, et al. Digital Life Project: autonomous 3D characters with social intelligence. arXiv, 2023. DOI:10.48550/arXiv.2312.04547 |
| [91] |
Lu W, Zeng L, Wang J, et al. Imitating and exploring the human brain's resting and task-performing states via brain computing: scaling and architecture. Natl Sci Rev, 2024, 11: nwae080. |
| [92] |
冯凡凡, 王晶. 生物脑与人工智能融合——数字生命的崭新时代. 科学, 2024, 76: 17-20. |
| [93] |
Lei W, Fuster-Barceló C, Reder G, et al. BioImage. IO Chatbot: a community-driven AI assistant for integrative computational bioimaging. Nat Methods, 2024, 21: 1368-70. |
| [94] |
A multiscale approach for biomedical machine learning. Nat Mach Intell, 2024, 6: 989
|
| [95] |
Ghebrehiwet I, Zaki N, Damseh R, et al. Revolutionizing personalized medicine with generative AI: a systematic review. Artif Intell Rev, 2024, 57: 128. |
| [96] |
Pyrkov A, Aliper A, Bezrukov D, et al. Complexity of life sciences in quantum and AI era. Wiley Interdiscip Rev Comput Mol Sci, 2024, 14: e1701. |
| [97] |
Klusch M, Lässig J, Müssig D, et al. Quantum artificial intelligence: a brief survey. KI-Künstliche Intelligenz, 2024. DOI:10.1007/s13218-024-00871-8 |
| [98] |
Google Quantum AI and Collaborators. Quantum error correction below the surface code threshold. Nature, 2024. DOI:10.1038/s41586-024-08449-y |
| [99] |
Farisco M, Baldassarre G, Cartoni E, et al. A method for the ethical analysis of brain-inspired AI. Artif Intell Rev, 2024, 57: 133. |
| [100] |
Perez-Lopez R, Ghaffari Laleh N, Mahmood F, et al. A guide to artificial intelligence for cancer researchers. Nat Rev Cancer, 2024, 24: 427-41. |
| [101] |
Kehl KL, Jee J, Pichotta K, et al. Shareable artificial intelligence to extract cancer outcomes from electronic health records for precision oncology research. Nat Commun, 2024, 15: 9787. |
| [102] |
Mittal S, Thakral K, Singh R, et al. On responsible machine learning datasets emphasizing fairness, privacy and regulatory norms with examples in biometrics and healthcare. Nat Mach Intell, 2024, 6: 936-49. |
| [103] |
Youssef A, Nichol AA, Martinez-Martin N, et al. Ethical considerations in the design and conduct of clinical trials of artificial intelligence. JAMA Netw Open, 2024, 7: e2432482. |
| [104] |
Goetz L, Seedat N, Vandersluis R, et al. Generalization — a key challenge for responsible AI in patient-facing clinical applications. NPJ Dig Med, 2024, 7: 126. |
 2025, Vol. 37
2025, Vol. 37 

 李丹丹,中国科学院上海营养与健康研究所生命科学信息中心馆员,中国科学院知识产权专员,上海科技翻译学会会员,专注于生物领域的产业与技术情报研究工作,服务了多家政府机构、高校院所与企业。主持了多项上海市科委和卫健委等机构的生物医药领域项目,其中部分成果获得上海市科技情报成果奖和华东地区科技情报成果奖,个人获“上海地区优秀科技情报工作者”称号;
李丹丹,中国科学院上海营养与健康研究所生命科学信息中心馆员,中国科学院知识产权专员,上海科技翻译学会会员,专注于生物领域的产业与技术情报研究工作,服务了多家政府机构、高校院所与企业。主持了多项上海市科委和卫健委等机构的生物医药领域项目,其中部分成果获得上海市科技情报成果奖和华东地区科技情报成果奖,个人获“上海地区优秀科技情报工作者”称号; 郑明月,中国科学院上海药物研究所研究员,课题组长。担任中国计算机学会数字医学分会执行委员,中国化学会计算机化学专委会委员,中国生物信息学学会药物发现专委会委员。担任Molecular Diversity、Artificial Intelligence in the Life Sciences期刊副主编,2023年机械工业出版社《人工智能与药物设计》主编。研究方向是基于大数据和人工智能的药物设计技术开发、发展机器学习算法和模型用于活性化合物的作用机制和靶点发现,新靶点活性化合物的发现和成药性优化。近年来,在Nat Method、Immunity等杂志发表SCI论文200余篇,申请发明专利和软件著作权20余项。获得国家杰出青年科学基金,获得药明康德生命化学研究奖等奖励和荣誉;
郑明月,中国科学院上海药物研究所研究员,课题组长。担任中国计算机学会数字医学分会执行委员,中国化学会计算机化学专委会委员,中国生物信息学学会药物发现专委会委员。担任Molecular Diversity、Artificial Intelligence in the Life Sciences期刊副主编,2023年机械工业出版社《人工智能与药物设计》主编。研究方向是基于大数据和人工智能的药物设计技术开发、发展机器学习算法和模型用于活性化合物的作用机制和靶点发现,新靶点活性化合物的发现和成药性优化。近年来,在Nat Method、Immunity等杂志发表SCI论文200余篇,申请发明专利和软件著作权20余项。获得国家杰出青年科学基金,获得药明康德生命化学研究奖等奖励和荣誉; 陈大明,中国科学院上海营养与健康研究所生命科学信息中心研究馆员,从事生命科学及其交叉学科的科技情报研究。近年来,带领团队完成了20多项产业规划研究、知识产权分析、软科学研究项目,研究成果获华东地区科学技术情报成果奖一等奖等多个奖项
陈大明,中国科学院上海营养与健康研究所生命科学信息中心研究馆员,从事生命科学及其交叉学科的科技情报研究。近年来,带领团队完成了20多项产业规划研究、知识产权分析、软科学研究项目,研究成果获华东地区科学技术情报成果奖一等奖等多个奖项