(2 教育部系统生物工程重点实验室,天津 300072)
(3 教育部合成生物学前沿科学中心,天津 300072)
(4 天津大学生物安全战略研究中心,天津 300072)
(2 Key Laboratory of Systems Bioengineering, Ministry of Education of China, Tianjin 300072, China)
(3 Frontier Science Center for Synthetic Biology, Tianjin 300072, China)
(4 Center for Biosafety Research and Strategy, Tianjin University, Tianjin 300072, China)
蓝细菌是地球上唯一可以进行放氧光合作用的原核生物,其出现的历史可追溯至35亿年前[1]。自蓝细菌诞生以来,其极大地影响了地球气候、生态系统,不仅促进了地球含氧环境的形成,还使大气中CO2含量相对稳定,其每年固定CO2的总量约占全球的四分之一[2]。此外,叶绿体起源于蓝细菌的“内共生”假说目前被科学界广泛接受[3],蓝细菌和植物叶绿体的相似性表明在蓝细菌中开展光合固碳等相关机制研究对植物的遗传改造同样具有借鉴意义。近些年,生物技术在蓝细菌中的应用使其展现出作为光合自养人工细胞工厂的潜力。蓝细菌以光能为能源、以CO2为碳源的特点赋予其环境友好、成本低廉的优点。即便如此,蓝细菌的问题是其化学品产量往往显著低于酿酒酵母(Saccharomyces cerevisiae)和大肠杆菌(Escherichia coli)等异养模式菌株;而制约蓝细菌产量的主要因素是其较低的生长速度及干重积累。例如,相比于大肠杆菌20 min的倍增时间[4],集胞藻6803 (Synechocystis sp. PCC 6803)倍增时间最快为4.3 h。在细胞干重方面,聚球藻7942 (Synechococcus elongatus PCC 7942)培养16 h仅实现0.2 mg/mL左右干重增加[5]。究其根本原因,蓝细菌相对低效的光能利用以及CO2固定效率使其不能像补充葡萄糖等糖源的异养微生物一样快速进行生长繁殖和细胞代谢。
合成生物学是一门依据工程学思想,结合分子生物学、计算机科学、物理学等技术,重新设计和标准化生物元件,进而“改造生命”,甚至“创造生命”的颠覆性学科。2013年,研究人员利用合成生物学技术在酿酒酵母中实现了25 g/L的青蒿酸(青蒿素前体)合成,为治疗疟疾特效药的大规模生产做出重大贡献[6];2014年,Lin等[7]通过将烟草中编码核酮糖1, 5-二磷酸羧化酶/加氧酶基因替换成聚球藻7942来源的相关基因,使烟草的光合速率和生物量得到了大幅提升,为植物改良提供了新思路[8];此外,基于合成生物学的第三代生物炼制,正努力实现从CO2到绿色新能源的转化,以逐步取代日益枯竭的石油等不可再生能源[9-10]。利用合成生物学手段对蓝细菌开展光合固碳相关的机制研究,并对其光合固碳系统优化改造,是加快蓝细菌生长速率和提升蓝细菌人工细胞工厂效能的关键(图 1)。
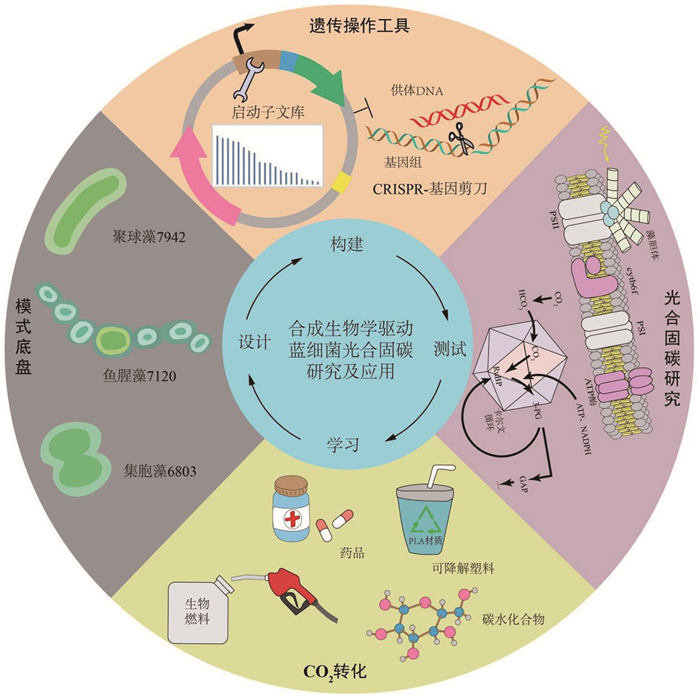
|
图 1 合成生物学驱动蓝细菌光合固碳研究及应用进展汇总图 |
为促进对合成生物学在蓝细菌中应用的理解,本文首先介绍蓝细菌常用的模式底盘(model chassis)和近些年的新兴底盘、底盘多样性;接着介绍蓝细菌的遗传操作工具开发,总结蓝细菌光合固碳系统优化重构的具体方向;最后总结通过合成生物学改造蓝细菌将光能和CO2转化为高价值化学品的案例,展望未来的研究方向,以期促进蓝细菌光合固碳机制研究,从而培育出可以适应于相关工业化生产要求的蓝细菌细胞。
1 模式蓝细菌蓝细菌中常用的模式底盘主要为集胞藻6803和聚球藻7942,目前对它们已有相对完善的基因操作方法,它们是开展蓝细菌光合固碳机制探索的主要底盘。集胞藻6803早在1968年由美国科学家从淡水环境中分离得到,并于1996年完成其全基因组测序工作[11],聚球藻7942也于2007年完成全基因组测序。这两种蓝细菌生长速率过慢、容易受户外环境因素影响,其倍增时间分别为4.3 h和4.9 h[12];当出现40 ℃以上高温时,集胞藻6803生长被抑制[13],在44 ℃以上温度、400 μmol•m-2•s-1以上光照强度下,聚球藻7942生长停滞[14],因此户外高温、高光照条件不利于这两种蓝细菌生长。
除了集胞藻6803和聚球藻7942这两种模式蓝细菌外,近年涌现一些具有优良特性的新蓝细菌。例如,与聚球藻7942基因组相似度达99.8%的聚球藻2973 (Synechococcus elongatus UTEX 2973)倍增时间最短为1.5 h,且能耐受聚球藻7942所无法承受的42 ℃的高温和光子通量密度为1 500 μmol•m-2•s-1的光照强度,通过半连续培养,聚球藻2973最高可以达到23.4 g/L DCW (dry cell weight,细胞干重)生物量[15]。Włodarczyk等[16]在2020年分离并表征了海洋聚球藻11901 (Synechococcus sp. PCC 11901)相关特性,发现其具有耐受43 ℃高温和600 μmol•m-2•s-1高光照强度的能力,最短倍增时间仅需2.1 h,其干重累计可以突破蓝细菌最高纪录,到达32.6 g/L DCW。该菌株较大的缺陷是培养过程中需要外源添加维生素B12,因此增加了培养的成本。Jaiswal等[17]在2018年在印度湖泊中分离出聚球藻11801 (Synechococcus elongatus PCC 11801),其在41 ℃、1 000 μmol•m-2•s-1光照强度的最适培养条件下的倍增时间为2.3 h。新发现的聚球藻11802 (Synechococcus elongatus PCC 11802)与聚球藻11801基因组相似度达97%,在1% CO2、1 000 μmol•m-2•s-1光照强度的培养条件下倍增时间为2.8 h,该菌株在高温、高CO2的环境中生长速度优于聚球藻11801[18]。
2 蓝细菌遗传改造工具近些年,蓝细菌中质粒载体、启动子、核糖体结合位点、CRISPR (clustered regularly interspaced short palindromic repeats)、碱基编辑等遗传改造工具的不断研发,为实现更高效光合固碳奠定了方法学基础。在蓝细菌中发掘不同强度的启动子、不同强度的核糖体结合位点以及不同拷贝数的稳定质粒载体是稳定过表达异源基因的关键。对此,Sun等[19]在2018年较全面地总结了蓝细菌中常用的传统组成型启动子和诱导型启动子、核糖开关等。Berla等[20]在2013年介绍了对蓝细菌中穿梭质粒载体的研究工作。在内源启动子基础上,Sengupta等[21]在2020年通过易错PCR技术对中等强度启动子PrbcL和强启动子PcpcB进行随机突变从而建立48个突变启动子文库,PrbcL启动子的部分突变体甚至表现出高于PcpcB的转录活性,该突变启动子文库拓宽了蓝细菌内启动子高、中、低转录活性范围,并在应用于其他蓝细菌如聚球藻11801和聚球藻11802后仍然具有相应的转录活性。目前该研究的部分启动子,例如PrbcLm17、PrbcLm15等已被应用于外源基因的表达[22]。Seo等[23]在2023年利用集胞藻6803天然启动子序列和dRNA-seq (differential RNA sequencing)数据构建机器学习预测模型,生成了10 000个可预测强度的人工启动子,并通过体外细胞转录实验评测人工启动子转录活性,发掘出转录强度高于蓝细菌内源强启动子PpsbA2S的4个人工启动子,随后在蓝细菌中利用这些人工启动子表达黄色荧光蛋白(yellow fluorescent protein)验证了其在蓝细菌中的适配性,这些基于机器学习生成的人工启动子或将有助于拓宽蓝细菌低、中、高强度启动子遗传库。
目前,蓝细菌基因组的遗传改造主要依靠同源重组方法,但其存在转化效率偏低、依靠质粒进行同源重组可能出现单交换等问题;此外,因为蓝细菌基因组普遍存在多拷贝[24],该方法很难在短时间得到纯合的编辑菌株,在蓝细菌中开发CRISPR工具或许能实现更加有效、时间节约型的基因编辑。Li等[25]在2016年尝试在集胞藻6803中使用CRISPR-Cas9敲除glgc的同时引入gltA、ppc来构建琥珀酸生产菌株,但由于Cas9蛋白对蓝细菌存在严重毒性,相关工作并未得到广泛应用。相较于Cas9蛋白,Cpf1对于蓝细菌具有更低的细胞毒性,Niu等[26] 于2019年在鱼腥藻PCC 7120 (Anabaena sp. PCC 7120)中构建了CRISPR-Cpf1工具,实现最多118 kb片段的敲除。此外,Cas3是一种同时具有解旋酶和核酸外切酶活性的Cas蛋白,Sengupta等[27]于2024年在聚球藻2973中利用CRISPR-Cas3删除了基因组上55 kb的大片段,使其生长和生物量分别提高了23%和22.7%,该工具未来可应用于蓝细菌基因组的大规模精简优化。上述工作表明开发低毒性Cas蛋白或许是未来CRISPR可以完美适配蓝细菌的关键。
此外,无核酸内切酶活性的CRISPR-dCas12a已在蓝细菌中被应用于靶向性抑制目的基因,由于dCas12a较低的细胞毒性,该工具目前在集胞藻6803、聚球藻7942等取得了相较于CRISPR-Cas9更广泛的应用[28-29]。目前在蓝细菌中常用的目的基因靶向性抑制方法还有sRNA (small regulatory RNA)干扰,Sun等[30]于2018年在集胞藻6803中利用Hfq-Micc类型的sRNA工具靶向抑制了丙二酰辅酶A的竞争途径,将细胞内丙二酰辅酶A含量提高了41%;Li等[31]于2018年在聚球藻2973中利用Hfq依赖的sRNA靶向抑制编码参与营养饥饿条件下藻胆体降解的nblA基因,达到了70%左右的抑制效果;Li等[32]于2021年对蓝细菌中各类sRNA工具进行了总结。上述两种抑制目的基因的方法,对蓝细菌中无法进行基因敲除的必需基因研究具有极大的帮助。
除了CRISPR系统,碱基编辑也是近些年取得重大突破的基因编辑方法。相较于CRISPR,碱基编辑无需DNA双链断裂和供体DNA,它是一种由CRISPR/Cas引导脱氨酶对目标靶点精准的C→T或A→G的碱基替换工具[33-34]。目前碱基编辑已在大肠杆菌、哺乳动物细胞等中广泛应用[35-36],但在蓝细菌中的报告比较少。2023年,Wang等[37]在聚球藻7942中首次报告碱基编辑在蓝细菌中的应用,通过对glgp、glgx两个基因同时进行提前引入终止密码子的编辑而使其失活,有效地提高了菌株的糖原积累,菌株的编辑效率高达80.77%。在大肠杆菌等其他物种的碱基编辑工具中,通常将尿嘧啶DNA糖基化酶抑制剂(UGI)与胞嘧啶脱氨酶等融合表达,以抑制尿嘧啶DNA糖基化酶(UDG)对目标靶点编辑的修复[38]。Lee等[39]于2023年通过结构比对和进化树分析发现聚球藻7942的尿嘧啶DNA糖基化酶属于IV型UDG,其受枯草芽孢杆菌噬菌体PBS2来源的UGI抑制的效果较差,进而该团队通过CRISPRi-dCas12a抑制UDG活性,实现了蓝细菌中更高效的碱基编辑效果。
3 蓝细菌光合固碳系统研究蓝细菌光合系统通常由多亚基蛋白复合物PSI、PSII和细胞色素b6f (Cyt b6f)组成,当有400~700 nm的可见光照射时,PSI、PSII反应中心的叶绿素a分子受光子激发,发生电荷分离释放电子,Cyt b6f主要负责将PSII产生的电子传递至PSI,以便于后续NADPH的生成,而此电子传递的过程同时会推动ATP的合成,上述同化力形成过程生成的NADPH和ATP将为碳固定提供能量。此外,蓝细菌进化出巨大的蛋白复合物藻胆体,其上附有大量具有捕光能力的藻胆蛋白[40],提高了其在低光环境中的生存能力。蓝细菌固碳系统的核心是核酮糖1, 5-二磷酸羧化酶/加氧酶(Rubisco),该酶被羧酶体包裹以便隔绝氧气,避免Rubisco进行加氧反应。羧酶体上有多类负责运输CO2和HCO3-的转运蛋白,使羧酶体成为一个CO2丰富,有利于碳固定的微环境。
目前蓝细菌光合机构的主要问题是当室外光照过强时,过量的光能会打破蓝细菌能量供应与消耗之间的平衡,富余的电子将导致活性氧(ROS)的大量生成,而后者对蓝细菌具有较大的破坏作用。近些年利用合成生物学方法改造菌株应对高光引起的细胞损害,主要集中在截短藻胆体捕光天线、过表达光保护(photoprotection)机制的策略。Bersanini等[41]通过过表达黄素二铁蛋白(flavodiiron proteins)相关基因flv4-2,该蛋白在PSII的替代电子传递途径中发挥作用,从而减轻PSII的激发压力。
蓝细菌固碳系统最大的问题是碳固定效率太低,主要表现为Rubisco酶相对低效[42],羧酶体上运输CO2、HCO3-相关蛋白的转运效率仍有提高的空间[43]。针对以上问题,研究人员主要通过超量表达以Rubisco酶为核心的卡尔文-本森-巴萨姆(Calvin-Benson-Bassham, CBB)循环相关酶减少碳损失、提高CO2转运力等方式提高碳固定效率。例如,Liang等[44]于2016年通过在集胞藻6803中过表达CBB循环中几个酶[Rubisco、景天庚酮糖1, 7-二磷酸酶(SBPase)、果糖二磷酸醛缩酶(FBA)和转酮醇酶(TK)],显著提高光合放氧速率、生物质积累和生长速率。Tharasirivat等[45]在PCC 6803中过表达Rubisco及其伴侣蛋白,显著提高聚羟基丁酸酯(PHB)产量。在这个循环中,当三碳产物向乙酰辅酶A转化时,有一个固定的碳以CO2形式丧失,Yu等[46]于2018年通过引入苹果酰辅酶A-甘油酸(MCG)途径,有效弥补CBB循环的碳损失,同时能有效提高乙酰CoA含量,将聚球藻7942的碳酸氢盐同化效率提高大约2倍。Gupta等[47]通过在聚球藻7002 (Synechococcus sp. PCC 7002)中过表达无机碳转运蛋白SbtA、BicA将生物量提高50%,胞内糖原含量提高50%。此类工作通过在蓝细菌中增强碳固定相关酶、碳转运蛋白表达提高菌株生物量和产品产量,表明碳固定相关酶活性仍有很大提升空间,针对相关酶的定向进化未来或有助于进一步提高碳固定效率。
近年来,研究人员多采用适应性驯化来获取具有耐受高光照等逆境能力的菌株[48-50],并通过耐受菌株的机制研究揭示对逆境抗性增强有帮助的基因及相关机制。适应性驯化是一种将微生物培养于逐渐提高的选择压力下,通过长期培养获得具有耐受相关胁迫条件的菌株的方法[51]。但由于蓝细菌生长周期长且适应性驯化依赖于细胞自身较低频率的基因随机突变,蓝细菌适应性驯化过程往往十分漫长,一般需要长达数年时间。为了加快菌株驯化速度,常采用物理或化学诱变[52]、常温常压等离子诱变[53]等方法,但这些方法通常会对细胞生长造成较大的影响。近期,Sun等[54]在2023年通过敲除聚球藻7942复制保真相关基因muts以及过表达引起DNA错误复制的recA基因将细胞随机突变频率提高1 000倍以上,实现较短时间内获得耐受高温、高光照的驯化菌株,大大缩短了适应性驯化的时间。对耐受高温、高光照菌株分析表明,synpcc7942_0894基因突变增强了莽草酸激酶的表达,该酶参与催化多种氨基酸合成。在蓝藻中,莽草酸途径可涉及光合作用的限速步骤,因此聚球藻7942中莽草酸激酶的过表达可提高光合作用的效率和稳定性。相较于传统诱变在细胞内引入突变后再进行耐受性能测试,该方法还具有可以让随机突变和胁迫条件筛选两个过程同时进行的优点。
除了上述挖掘光合固碳相关基因的方法,CRISPRi抑制文库的建立或对蓝细菌光合固碳研究具有指导作用。Yao等[55]2020年在集胞藻6803中建立了包含10 498个sgRNA的CRISPRi抑制文库,通过对生长表现良好的菌株进行下一代测序分析,鉴定出与乳糖耐受等胁迫响应相关的多个关键基因。该研究工具目前在解析蓝细菌光合固碳机制方面得到了广泛应用,例如Miao等[56]于2023年对集胞藻6803每个基因都设计了5对sgRNA以尽可能避免出现针对某些基因设计的sgRNA靶向性差的问题,从而构建21 705个sgRNA组成的更庞大的CRISPRi抑制文库并应用于氮缺乏、不同CO2浓度、不同光照强度、添加葡萄糖等相关应激条件的机制解析,揭示出蛋白磷酸酶、3-磷酸甘油醛脱氢酶、小蛋白CP12在混合营养、光异养培养中发挥重要作用。
4 用合成生物学改变CO2转化蓝细菌作为以CO2为碳源的光能自养微生物,具有将CO2转化为生物燃料、可降解塑料、药品、碳水化合物等富有经济价值化学品的能力,在我国大力发展“碳达峰、碳中和”的背景下具有广阔的应用前景。目前利用蓝细菌合成的化学品已多达上百种,本文因篇幅限制,仅列举代表性工作,感兴趣的读者可参见相关综述文献[57-58]。
在生物燃料方面,目前研究人员已实现在蓝细菌中合成2, 3-丁二醇、乙醇、异丁醇等化学品[59-62]。其中,Atsumi等[59]于2009年通过在聚球藻7942中引入外源基因实现了异丁醇和异丁醛的生产,并过表达Rubisco提高产量,分别实现在8 d内生产1.1 g/L异丁醛和在6 d内产生450 mg/L异丁醇的产量。Gao等[60]于2012年通过在集胞藻6803中引入运动发酵单胞菌(Zymomonas mobilis)来源的丙酮酸脱羧酶,以及过表达内源乙醇脱氢酶、阻断聚-β-羟基丁酸酯生物合成途径,实现5.5 g/L的乙醇产量。Oliver等[61]于2013年在聚球藻7942中构建2, 3丁二醇的生物合成途径,在21 d内获得2.38 g/L的2, 3丁二醇产量;该团队随后采用添加葡萄糖的混碳培养模式,并通过增强OPP途径(oxidative pentose phosphate pathway)增加来自葡萄糖的碳通量,然后将其重定向到由Rubisco催化的碳固定步骤,12 d内将2, 3丁二醇的产量提高至12.6 g/L[62]。
在可降解塑料方面,目前在蓝细菌中已实现3-羟基丙酸(3-HP)、乳酸、聚乳酸(PLA)、聚羟基丁酸酯(PHB)等物质的合成[63-66]。Wang等[63]于2016年在集胞藻6803中构建了3-HP合成途径,实现了6 d内生产837.18 mg/L的3-HP产量。Hidese等[64] 于2020年通过敲除集胞藻6803中的醋酸激酶并过表达乳酸脱氢酶、内源苹果酸脱氢酶,实现3 d内达到26.6 g/L乳酸的产量。Khetkorn等[65]通过在集胞藻6803中过表达PHB合成相关的phaAB基因,并在缺氮、乙酸盐补充的条件下培养,实现35% (DCW)的PHB产量。此外,Tan等[66]于2022年在聚球藻7942中构建PLA的人工细胞工厂,通过启动子优化、碳流重定向等代谢工程策略和高密度培养,将10 d内PLA的产量提升至108.0 mg/L。该法生产出的PLA的分子量是目前文献报告中最高的,具有很好的应用前景。
在药物合成方面,研究人员已在蓝细菌中实现将CO2转化合成虾青素、肌醇等药品。虾青素是一类具有抗肿瘤、消炎等作用的类胡萝卜素[67]。Diao等[68]于2020年在集胞藻6803中引入β-类胡萝卜素酮醇酶和羟化酶并调控这两种酶的表达水平,将不同来源的上述酶结合起来,确定并改进β-胡萝卜素转化为虾青素途径中的限速步骤,以及增加内源甲基赤藓糖醇4-磷酸(MEP)途径的碳通量,实现了10 d内29.6 mg/g细胞干重的虾青素产量。肌醇是一种具有抗癌、辅助治疗抑郁症的六碳环结构的多元醇[69]。Sun等[70]于2023年在快速生长的聚球藻2973中通过sRNA下调肌醇合成以外的竞争通路,以及开发基于肌醇传感器的自调节系统平衡肌醇生产和菌株生长之间的关系,实现了7 d内262.6 mg/L的肌醇产量。
在碳水化合物等食品和添加剂方面,目前在蓝细菌中已经实现蔗糖、果糖、贝壳杉烯酸等食品及食品添加剂前体的有效合成[71-74]。其中吕雪峰团队专注于糖类化合物合成,目前该团队已实现蓝细菌中的蔗糖、果糖、葡萄糖的高产量合成。例如,Zhang等[71]于2023年通过敲除聚球藻7942葡萄糖激酶消除葡萄糖下游消耗以及加强蔗糖合成、优化碳流等策略,使其菌株葡萄糖产量在18 d内达到2 g/L,并依靠半连续培养在36 d内达到5 g/L的葡萄糖产量。Sun等[72] 2023年通过灭活聚球藻7942的果糖激酶、改造蓝细菌蔗糖代谢途径和优化细胞生理功能,在12 d内实现了约3.9 g/L的果糖产量;该团队还将该策略应用于聚球藻7002,可在单批次培养16 d左右达到6.3 g/L的果糖产量。
5 总结与展望通过合成生物学技术在蓝细菌中的应用,前人已经开展了众多蓝细菌光合固碳机制的研究工作,通过工程改造实现其光合、固碳效率的提升,并且已构建多个蓝细菌光合固碳生成高价值化学品的自养型人工细胞工厂。然而,蓝细菌相较于其他工业化生产的模式生物仍然具有生长速率缓慢等缺陷,因此蓝细菌的合成生物学研究仍面临不少挑战。未来在原材料细胞发掘、工业化生产以及基因编辑工具和机制研究方法等方面都有深入研究、提升的空间。
5.1 发掘新型蓝细菌现有蓝细菌普遍存在生长速度慢、细胞稳定性差等缺陷,作为有数十亿年演化史的光合微生物,自然界中必有可待挖掘的具有优良应用价值的原材料细胞。例如Schubert等[75] 在2023年从具有高CO2浓度的火山口附近海域分离出一株具有生长快、耐高温等特性的菌株UTEX 3222,其倍增时间为2.35 h,在分批补料培养下可形成超过31 g/L生物质干重;该菌株可以耐受45 ℃的高温,且具有快速沉降的特点;有助于工业产品的分离。该菌株的快速生长、耐高温能力表明其具有成为高效的蓝细菌人工细胞工厂的潜力,对其相关特性的机制解析或将有助于提升蓝细菌生长速度和耐高温能力。该团队的工作表明从极端环境中筛选菌株往往可以得到一些具有优良特性的蓝细菌原材料。
5.2 构建混菌发酵体系构建蓝细菌与其他模式微生物的混菌发酵体系,将光合微生物和异养微生物两者优势相互结合,是未来蓝细菌工业化应用的研究方向。例如Li等[76]于2023年构建了蓝细菌聚球藻7942与具有极快生长速度的一种海洋嗜盐菌——需钠弧菌(Vibrio natriegens)的混菌体系,蓝细菌光合作用生产和分泌蔗糖,需钠弧菌以此为碳源代谢生产乳酸、2, 3丁二醇等化学品。然而,混菌培养可能会出现竞争关系,Sun等[77]于2023年合成一种可以一定程度隔绝微生物但是可以进行物质交换的水凝胶,使两种微生物在各自区室中生长。此外,Cournoyer等[78]利用细胞融合方法,将聚球藻7942引入呼吸作用阻断的酿酒酵母,实现了蓝细菌为酿酒酵母提供ATP,酿酒酵母为蓝细菌提供代谢产物的共生体。该共生体系统在光照条件下可繁殖15~20代。后者通过创造蓝细菌、酵母耦合的新细胞为混菌培养提供了新思路。
5.3 开发高效的使能工具目前蓝细菌的高效基因编辑工具仍然匮乏,未来利用深度学习、定向进化等方法研发低毒性的CRISPR系统或开展蓝细菌先导编辑研究,将有助于蓝细菌快速基因编辑工具的开发。蓝细菌光合固碳机制研究工具库仍需要进一步扩展,例如Gallagher等[79] 在大肠杆菌、铜绿假单胞菌中将作用于双链DNA的胞嘧啶脱氨酶DddA与转录因子融合表达,通过转录因子引导DddA到DNA结合区域产生特征突变,从而依靠全基因组高通量测序追踪转录因子在DNA上的相互作用位点,该方法比传统的转录因子研究手段ChIP-seq操作更为简便,缺点是DddA有较大的细胞毒性。未来通过改造、筛选毒性较低的DddA蛋白或采用渗漏性低的诱导系统有望将该转录因子研究工具应用于蓝细菌。
5.4 重构光合固碳系统目前,在大肠杆菌、酵母中进行基因组大规模的修饰、改造技术已较为成熟。例如天津大学、清华大学和华大基因研究院于2017年实现酿酒酵母中4条染色体的从头优化和人工合成,为蓝细菌等微生物基因组理性设计改造提供了方向[80-83];Zimmermann等[84] 在酿酒酵母中通过将具有解旋酶活性的Cas3与胞苷脱氨酶融合表达,实现对55 kb基因区域靶向性的随机突变,有效地提高了其番茄红素产量,该技术有应用于蓝细菌光合、固碳相关基因区域靶向诱变修饰的潜力;Chen等[85]于2018年将叶绿素合成相关的12个外源基因转入大肠杆菌,首次实现卟啉IX到叶绿素a的合成;Schwander等[86]于2016年将来自9个物种的17种碳固定相关酶在体外组装,其新的固碳途径的效率是天然碳固定途径的5倍。此类工作表明,未来在蓝细菌的光合固碳系统中引入内源或外源基因的大规模改造,或将有助于大幅度提升其光合固碳效率。
| [1] |
Schopf JW. Microfossils of the early archean apex chert: new evidence of the antiquity of life. Science, 1993, 260: 640-6. DOI:10.1126/science.260.5108.640 |
| [2] |
Zwirglmaier K, Jardillier L, Ostrowski M, et al. Global phylogeography of marine Synechococcus and Prochlorococcus reveals a distinct partitioning of lineages among oceanic biomes. Environ Microbiol, 2008, 10: 147-61. DOI:10.1111/j.1462-2920.2007.01440.x |
| [3] |
Archibald JM. Endosymbiosis and eukaryotic cell evolution. Curr Biol, 2015, 25: R911-21. DOI:10.1016/j.cub.2015.07.055 |
| [4] |
Gibson B, Wilson DJ, Feil E, et al. The distribution of bacterial doubling times in the wild. Proc Biol Sci, 2018, 285: 20180789. |
| [5] |
Yu J, Liberton M, Cliften PF, et al. Synechococcus elongatus UTEX 2973, a fast growing cyanobacterial chassis for biosynthesis using light and CO2. Sci Rep, 2015, 5: 8132. DOI:10.1038/srep08132 |
| [6] |
Paddon CJ, Westfall PJ, Pitera DJ, et al. High-level semi-synthetic production of the potent antimalarial artemisinin. Nature, 2013, 496: 528-32. DOI:10.1038/nature12051 |
| [7] |
Lin MT, Occhialini A, Andralojc PJ, et al. A faster Rubisco with potential to increase photosynthesis in crops. Nature, 2014, 513: 547-50. DOI:10.1038/nature13776 |
| [8] |
Bailey-Serres J, Parker JE, Ainsworth EA, et al. Genetic strategies for improving crop yields. Nature, 2019, 575: 109-18. DOI:10.1038/s41586-019-1679-0 |
| [9] |
Gleizer S, Ben-Nissan R, Bar-On YM, et al. Conversion of Escherichia coli to generate all biomass carbon from CO2. Cell, 2019, 179: 1255-63. DOI:10.1016/j.cell.2019.11.009 |
| [10] |
Chen FYH, Jung HW, Tsuei CY, et al. Converting Escherichia coli to a synthetic methylotroph growing solely on methanol. Cell, 2020, 182: 933-46. DOI:10.1016/j.cell.2020.07.010 |
| [11] |
Kaneko T, Sato S, Kotani H, et al. Sequence analysis of the genome of the unicellular cyanobacterium Synechocystis sp. strain PCC6803. Ⅱ. Sequence determination of the entire genome and assignment of potential protein-coding regions. DNA Res, 1996, 3: 109. DOI:10.1093/dnares/3.3.109 |
| [12] |
van Alphen P, Najafabadi HA, dos Santos FB, et al. Increasing the photoautotrophic growth rate of Synechocystis sp. PCC 6803 by identifying the limitations of Its cultivation. Biotechnol J, 2018, 13: e1700764. DOI:10.1002/biot.201700764 |
| [13] |
Sheng J, Kim HW, Badalamenti JP, et al. Effects of temperature shifts on growth rate and lipid characteristics of Synechocystis sp PCC6803 in a bench-top photobioreactor. Bioresour Technol, 2011, 102: 11218-25. DOI:10.1016/j.biortech.2011.09.083 |
| [14] |
Zhang SS, Zheng SN, Sun JH, et al. Rapidly improving high light and high temperature tolerances of cyanobacterial cell factories through the convenient introduction of an AtpA-C252F mutation. Front Microbiol, 2021, 12: 647164. DOI:10.3389/fmicb.2021.647164 |
| [15] |
Ungerer J, Lin PC, Chen HY, et al. Adjustments to photosystem stoichiometry and electron transfer proteins are key to the remarkably fast growth of the cyanobacterium Synechococcus elongatus UTEX 2973. Mbio, 2018, 9: e02327-17. |
| [16] |
Wlodarczyk A, Selao TT, Norling B, et al. Newly discovered Synechococcus sp. PCC 11901 is a robust cyanobacterial strain for high biomass production. Commun Biol, 2020, 3: 215. DOI:10.1038/s42003-020-0910-8 |
| [17] |
Jaiswal D, Sengupta A, Sohoni S, et al. Genome features and biochemical characteristics of a robust, fast growing and naturally transformable cyanobacterium Synechococcus elongatus PCC 11801 isolated from India. Sci Rep, 2018, 8: 16632. DOI:10.1038/s41598-018-34872-z |
| [18] |
Jaiswal D, Sengupta A, Sengupta S, et al. A novel cyanobacterium Synechococcus elongatus PCC 11802 has distinct genomic and metabolomic characteristics compared to its neighbor PCC 11801. Sci Rep, 2020, 10: 191. DOI:10.1038/s41598-019-57051-0 |
| [19] |
Sun T, Li S, Song X, et al. Toolboxes for cyanobacteria: recent advances and future direction. Biotechnol Adv, 2018, 36: 1293-307. DOI:10.1016/j.biotechadv.2018.04.007 |
| [20] |
Berla BM, Saha R, Immethun CM, et al. Synthetic biology of cyanobacteria: unique challenges and opportunities. Front Microbiol, 2013, 4: 246. |
| [21] |
Sengupta A, Madhu S, Wangikar PP. A library of tunable, portable, and inducer-free promoters derived from cyanobacteria. ACS Synth Biol, 2020, 9: 1790-801. DOI:10.1021/acssynbio.0c00152 |
| [22] |
Pritam P, Sarnaik AP, Wangikar PP. Metabolic engineering of Synechococcus elongatus for photoautotrophic production of mannitol. Biotechnol Bioeng, 2023, 120: 2363-70. DOI:10.1002/bit.28479 |
| [23] |
Seo E, Choi YN, Shin YR, et al. Design of synthetic promoters for cyanobacteria with generative deep-learning model. Nucleic Acids Res, 2023, 51: 7071-82. DOI:10.1093/nar/gkad451 |
| [24] |
Griese M, Lange C, Soppa J. Ploidy in cyanobacteria. FEMS Microbiol Lett, 2011, 323: 124-31. DOI:10.1111/j.1574-6968.2011.02368.x |
| [25] |
Li H, Shen CR, Huang CH, et al. CRISPR-Cas9 for the genome engineering of cyanobacteria and succinate production. Metab Eng, 2016, 38: 293-302. DOI:10.1016/j.ymben.2016.09.006 |
| [26] |
Niu TC, Lin GM, Xie LR, et al. Expanding the potential of CRISPR-Cpf1-based genome editing technology in the cyanobacterium Anabaena PCC 7120. ACS Synth Biol, 2019, 8: 170-80. DOI:10.1021/acssynbio.8b00437 |
| [27] |
Sengupta A, Bandyopadhyay A, Sarkar D, et al. Genome streamlining to improve performance of a fast-growing cyanobacterium Synechococcus elongatus UTEX 2973. mBio, 2024, 15: e0353023. DOI:10.1128/mbio.03530-23 |
| [28] |
Choi SY, Woo HM. CRISPRi-dCas12a: a dCas12a-mediated CRISPR interference for repression of multiple genes and metabolic engineering in cyanobacteria. ACS Synth Biol, 2020, 9: 2351-61. DOI:10.1021/acssynbio.0c00091 |
| [29] |
Shabestary K, Anfelt J, Ljungqvist E, et al. Targeted repression of essential genes to arrest growth and increase carbon partitioning and biofuel titers in cyanobacteria. ACS Synth Biol, 2018, 7: 1669-75. DOI:10.1021/acssynbio.8b00056 |
| [30] |
Sun T, Li SB, Song XY, et al. Re-direction of carbon flux to key precursor malonyl-CoA via artificial small RNAs in photosynthetic Synechocystis sp PCC 6803. Biotechnol Biofuels, 2018, 11: 26. DOI:10.1186/s13068-018-1032-0 |
| [31] |
Li SB, Sun T, Xu CX, et al. Development and optimization of genetic toolboxes for a fast-growing cyanobacterium Synechococcus elongatus UTEX 2973. Metab Eng, 2018, 48: 163-74. DOI:10.1016/j.ymben.2018.06.002 |
| [32] |
Li S, Sun T, Chen L, et al. Designing and constructing artificial small RNAs for gene regulation and carbon flux redirection in photosynthetic cyanobacteria. Methods Mol Biol, 2021, 2290: 229-52. |
| [33] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533: 420-4. DOI:10.1038/nature17946 |
| [34] |
Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A.T to G.C in genomic DNA without DNA cleavage. Nature, 2017, 551: 464-71. DOI:10.1038/nature24644 |
| [35] |
Zeng J, Wu Y, Ren C, et al. Therapeutic base editing of human hematopoietic stem cells. Nat Med, 2020, 26: 535-41. DOI:10.1038/s41591-020-0790-y |
| [36] |
Zhao DD, Li J, Li SW, et al. Glycosylase base editors enable C-to-A and C-to-G base changes. Nat Biotechnol, 2021, 39: 35-40. DOI:10.1038/s41587-020-0592-2 |
| [37] |
Wang SY, Li X, Wang SG, et al. Base editing for reprogramming cyanobacterium Synechococcus elongatus. Metab Eng, 2023, 75: 91-9. DOI:10.1016/j.ymben.2022.11.005 |
| [38] |
Wang Y, Liu Y, Zheng P, et al. Microbial base editing: a powerful emerging technology for microbial genome engineering. Trends Biotechnol, 2021, 39: 165-80. DOI:10.1016/j.tibtech.2020.06.010 |
| [39] |
Lee M, Heo YB, Woo HM. Cytosine base editing in cyanobacteria by repressing archaic type IV uracil-DNA glycosylase. Plant J, 2023, 113: 610-25. DOI:10.1111/tpj.16074 |
| [40] |
Sengupta A, Bandyopadhyay A, Schubert MG, et al. Antenna modification in a fast-growing cyanobacterium Synechococcus elongatus UTEX 2973 leads to improved efficiency and carbon-neutral productivity. Microbiol Spectrum, 2023, 11: e0050023. DOI:10.1128/spectrum.00500-23 |
| [41] |
Bersanini L, Battchikova N, Jokel M, et al. Flavodiiron protein Flv2/Flv4-related photoprotective mechanism dissipates excitation pressure of PSⅡ in cooperation with phycobilisomes in cyanobacteria. Plant Physiol, 2014, 164: 805-18. DOI:10.1104/pp.113.231969 |
| [42] |
Durao P, Aigner H, Nagy P, et al. Opposing effects of folding and assembly chaperones on evolvability of Rubisco. Nat Chem Biol, 2015, 11: 148-55. DOI:10.1038/nchembio.1715 |
| [43] |
Kamennaya NA, Ahn S, Park H, et al. Installing extra bicarbonate transporters in the cyanobacterium Synechocystis sp PCC6803 enhances biomass production. Metab Eng, 2015, 29: 76-85. DOI:10.1016/j.ymben.2015.03.002 |
| [44] |
Liang F, Lindblad P. Effects of overexpressing photosynthetic carbon flux control enzymes in the cyanobacterium Synechocystis PCC 6803. Metab Eng, 2016, 38: 56-64. DOI:10.1016/j.ymben.2016.06.005 |
| [45] |
Tharasirivat V, Jantaro S. Increased biomass and polyhydroxybutyrate production by Synechocystis sp. PCC 6803 overexpressing RuBisCO genes. Int J Mol Sci, 2023, 24: 6415. DOI:10.3390/ijms24076415 |
| [46] |
Yu H, Li X, Duchoud F, et al. Augmenting the Calvin-Benson-Bassham cycle by a synthetic malyl-CoA-glycerate carbon fixation pathway. Nat Commun, 2018, 9: 2008. DOI:10.1038/s41467-018-04417-z |
| [47] |
Gupta JK, Rai P, Jain KK, et al. Overexpression of bicarbonate transporters in the marine cyanobacterium Synechococcus sp. PCC 7002 increases growth rate and glycogen accumulation. Biotechnol Biofuels, 2020, 13: 17. DOI:10.1186/s13068-020-1656-8 |
| [48] |
Yoshikawa K, Ogawa K, Toya Y, et al. Mutations in hik26 and slr1916 lead to high-light stress tolerance in Synechocystis sp. PCC6803. Commun Biol, 2021, 4: 343. DOI:10.1038/s42003-021-01875-y |
| [49] |
Hu L, He J, Dong M, et al. Divergent metabolic and transcriptomic responses of Synechocystis sp. PCC 6803 to salt stress after adaptive laboratory evolution. Algal Res, 2020, 47: 101856. DOI:10.1016/j.algal.2020.101856 |
| [50] |
Wang Y, Shi M, Niu X, et al. Metabolomic basis of laboratory evolution of butanol tolerance in photosynthetic Synechocystis sp PCC 6803. Microb Cell Fact, 2014, 13: 151. DOI:10.1186/s12934-014-0151-y |
| [51] |
Zheng Y, Hong K, Wang B, et al. Genetic diversity for accelerating microbial adaptive laboratory evolution. ACS Synth Biol, 2021, 10: 1574-86. DOI:10.1021/acssynbio.0c00589 |
| [52] |
Dann M, Ortiz EM, Thomas M, et al. Enhancing photosynthesis at high light levels by adaptive laboratory evolution. Nat Plants, 2021, 7: 681-95. DOI:10.1038/s41477-021-00904-2 |
| [53] |
Fang M, Jin L, Zhang C, et al. Rapid mutation of Spirulina platensis by a new mutagenesis system of atmospheric and room temperature plasmas (ARTP) and generation of a mutant library with diverse phenotypes. PLoS One, 2013, 8: e77046. DOI:10.1371/journal.pone.0077046 |
| [54] |
Sun H, Luan G, Ma Y, et al. Engineered hypermutation adapts cyanobacterial photosynthesis to combined high light and high temperature stress. Nat Commun, 2023, 14: 1238. DOI:10.1038/s41467-023-36964-5 |
| [55] |
Yao L, Shabestary K, Bjork SM, et al. Pooled CRISPRi screening of the cyanobacterium Synechocystis sp PCC 6803 for enhanced industrial phenotypes. Nat Commun, 2020, 11: 1666. DOI:10.1038/s41467-020-15491-7 |
| [56] |
Miao R, Jahn M, Shabestary K, et al. CRISPR interference screens reveal growth-robustness tradeoffs in Synechocystis sp. PCC 6803 across growth conditions. Plant Cell, 2023, 35: 3937-56. DOI:10.1093/plcell/koad208 |
| [57] |
Velmurugan R, Incharoensakdi A. Metabolic transformation of cyanobacteria for biofuel production. Chemosphere, 2022, 299: 134342. DOI:10.1016/j.chemosphere.2022.134342 |
| [58] |
Choi YN, Lee JW, Kim JW, et al. Acetyl-CoA-derived biofuel and biochemical production in cyanobacteria: a mini review. J Appl Phycol, 2020, 32: 1643-53. DOI:10.1007/s10811-020-02128-x |
| [59] |
Atsumi S, Higashide W, Liao JC. Direct photosynthetic recycling of carbon dioxide to isobutyraldehyde. Nat Biotechnol, 2009, 27: 1177-80. DOI:10.1038/nbt.1586 |
| [60] |
Gao Z, Zhao H, Li Z, et al. Photosynthetic production of ethanol from carbon dioxide in genetically engineered cyanobacteria. Energy Environ Sci, 2012, 5: 9857-65. DOI:10.1039/C2EE22675H |
| [61] |
Oliver JW, Machado IM, Yoneda H, et al. Cyanobacterial conversion of carbon dioxide to 2, 3-butanediol. Proc Natl Acad Sci U S A, 2013, 110: 1249-54. DOI:10.1073/pnas.1213024110 |
| [62] |
Kanno M, Carroll AL, Atsumi S. Global metabolic rewiring for improved CO2 fixation and chemical production in cyanobacteria. Nat Commun, 2017, 8: 14724. DOI:10.1038/ncomms14724 |
| [63] |
Wang Y, Sun T, Gao X, et al. Biosynthesis of platform chemical 3-hydroxypropionic acid (3-HP) directly from CO2 in cyanobacterium Synechocystis sp PCC 6803. Metab Eng, 2016, 34: 60-70. DOI:10.1016/j.ymben.2015.10.008 |
| [64] |
Hidese R, Matsuda M, Osanai T, et al. Malic enzyme facilitates D-lactate production through increased pyruvate supply during anoxic dark fermentation in Synechocystis sp. PCC 6803. ACS Synth Biol, 2020, 9: 260-8. DOI:10.1021/acssynbio.9b00281 |
| [65] |
Khetkorn W, Incharoensakdi A, Lindblad P, et al. Enhancement of poly-3-hydroxybutyrate production in Synechocystis sp. PCC 6803 by overexpression of its native biosynthetic genes. Bioresour Technol, 2016, 214: 761-8. DOI:10.1016/j.biortech.2016.05.014 |
| [66] |
Tan C, Tao F, Xu P. Direct carbon capture for the production of high-performance biodegradable plastics by cyanobacterial cell factories. Green Chem, 2022, 24: 3850. DOI:10.1039/D2GC90034C |
| [67] |
Higuera-Ciapara I, Félix-Valenzuela L, Goycoolea FM. Astaxanthin: a review of its chemistry and applications. Crit Rev Food Sci Nutr, 2006, 46: 185-96. DOI:10.1080/10408690590957188 |
| [68] |
Diao J, Song X, Zhang L, et al. Tailoring cyanobacteria as a new platform for highly efficient synthesis of astaxanthin. Metab Eng, 2020, 61: 275-87. DOI:10.1016/j.ymben.2020.07.003 |
| [69] |
Chhetri DR. Myo-inositol and its derivatives: their emerging role in the treatment of human diseases. Front Pharmacol, 2019, 10: 1172. DOI:10.3389/fphar.2019.01172 |
| [70] |
Sun T, Li Z, Li S, et al. Exploring and validating key factors limiting cyanobacteria-based CO2 bioconversion: case study to maximize myo-inositol biosynthesis. Chem Eng J, 2023, 452: 139158. DOI:10.1016/j.cej.2022.139158 |
| [71] |
Zhang S, Sun J, Feng D, et al. Unlocking the potentials of cyanobacterial photosynthesis for directly converting carbon dioxide into glucose. Nat Commun, 2023, 14: 3425. DOI:10.1038/s41467-023-39222-w |
| [72] |
Sun J, Zhang Z, Zhang S, et al. Engineering cyanobacterial cell factories for photosynthetic production of fructose. ACS Synth Biol, 2023, 12: 3008-19. DOI:10.1021/acssynbio.3c00338 |
| [73] |
Ko SC, Woo HM. Biosynthesis of the calorie-free sweetener precursor ent-kaurenoic acid from CO2 using engineered cyanobacteria. ACS Synth Biol, 2020, 9: 2979-85. DOI:10.1021/acssynbio.0c00523 |
| [74] |
Lai MJ, Tsai JC, Lan EI. CRISPRi-enhanced direct photosynthetic conversion of carbon dioxide to succinic acid by metabolically engineered cyanobacteria. Bioresour Technol, 2022, 366: 128131. DOI:10.1016/j.biortech.2022.128131 |
| [75] |
Schubert MG, Tang TC, Goodchild-Michelman IM, et al. Cyanobacteria newly isolated from marine volcanic seeps display rapid sinking and robust, high density growth. bioRxiv, 2023, 10. |
| [76] |
Li C, Wang R, Wang J, et al. A highly compatible phototrophic community for carbon-negative biosynthesis. Angew Chem Int Ed, 2023, 62: e202215013. DOI:10.1002/anie.202215013 |
| [77] |
Sun Z, Wen H, Di Z, et al. Photosynthetic living fiber fabrication from algal-bacterial consortia with controlled spatial distribution. ACS Biomater Sci Eng, 2023, 9: 6481-9. DOI:10.1021/acsbiomaterials.3c00884 |
| [78] |
Cournoyer JE, Altman SD, Gao Yl, et al. Engineering artificial photosynthetic life-forms through endosymbiosis. Nat Commun, 2022, 13: 2254. DOI:10.1038/s41467-022-29961-7 |
| [79] |
Gallagher LA, Velazquez E, Peterson SB, et al. Genome-wide protein-DNA interaction site mapping in bacteria using a double-stranded DNA-specific cytosine deaminase. Nat Microbiol, 2022, 7: 844-55. DOI:10.1038/s41564-022-01133-9 |
| [80] |
Wu Y, Li BZ, Zhao M, et al. Bug mapping and fitness testing of chemically synthesized chromosome X. Science, 2017, 355: eaaf4706. DOI:10.1126/science.aaf4706 |
| [81] |
Zhang W, Zhao G, Luo Z, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome. Science, 2017, 355: eaaf3981. DOI:10.1126/science.aaf3981 |
| [82] |
Xie ZX, Li BZ, Mitchell LA, et al. "Perfect" designer chromosome V and behavior of a ring derivative. Science, 2017, 355: eaaf4704. DOI:10.1126/science.aaf4704 |
| [83] |
Shen Y, Wang Y, Chen T, et al. Deep functional analysis of synⅡ, a 770-kilobase synthetic yeast chromosome. Science, 2017, 355: eaaf4791. DOI:10.1126/science.aaf4791 |
| [84] |
Zimmermann A, Prieto-Vivas JE, Cautereels C, et al. A Cas3-base editing tool for targetable in vivo mutagenesis. Nat Commun, 2023, 14: 3389. DOI:10.1038/s41467-023-39087-z |
| [85] |
Chen GE, Canniffe DP, Barnett SFH, et al. Complete enzyme set for chlorophyll biosynthesis in Escherichia coli. Sci Adv, 2018, 4: eaaq1407. DOI:10.1126/sciadv.aaq1407 |
| [86] |
Schwander T, von Borzyskowski LS, Burgener S, et al. A synthetic pathway for the fixation of carbon dioxide in vitro. Science, 2016, 354: 900-4. DOI:10.1126/science.aah5237 |
 2024, Vol. 36
2024, Vol. 36 

 孙韬,博士,天津大学生物安全战略研究中心副教授,从事以光合微生物为代表的合成生物学及生物安全相关研究,在Science Advances、Metabolic Engineering、Chemical Engineering Journal、ACS Nano等专业杂志发表论文70余篇,主持国家自然科学基金项目2项、国家重点研发计划子课题2项,担任Blue Biotechnology、Discover Applied Sciences等杂志编委及中国医药生物技术协会生物安全专委会常务委员;
孙韬,博士,天津大学生物安全战略研究中心副教授,从事以光合微生物为代表的合成生物学及生物安全相关研究,在Science Advances、Metabolic Engineering、Chemical Engineering Journal、ACS Nano等专业杂志发表论文70余篇,主持国家自然科学基金项目2项、国家重点研发计划子课题2项,担任Blue Biotechnology、Discover Applied Sciences等杂志编委及中国医药生物技术协会生物安全专委会常务委员; 张卫文,博士,天津大学化工学院教授,博士生导师,生物安全战略研究中心主任,教育部第八届“科学技术委员会”委员,国家重点研发计划“合成生物学”重点专项首席科学家。长期从事光合微生物的合成生物学,以及生物安全相关研究,牵头中美合成生物学生物安全“二轨”战略对话。已在Science Advances、Metabolic Engineering、ACS Synthetic Biology、Biotechnology Advances等专业杂志发表论文200余篇,现担任8个SCI杂志副主编和编委
张卫文,博士,天津大学化工学院教授,博士生导师,生物安全战略研究中心主任,教育部第八届“科学技术委员会”委员,国家重点研发计划“合成生物学”重点专项首席科学家。长期从事光合微生物的合成生物学,以及生物安全相关研究,牵头中美合成生物学生物安全“二轨”战略对话。已在Science Advances、Metabolic Engineering、ACS Synthetic Biology、Biotechnology Advances等专业杂志发表论文200余篇,现担任8个SCI杂志副主编和编委