(2 工业互联网创新中心(上海)有限公司, 上海 200131)
(2 Industrial Internet Innovation Center (Shanghai) Co., Ltd., Shanghai 200131, China)
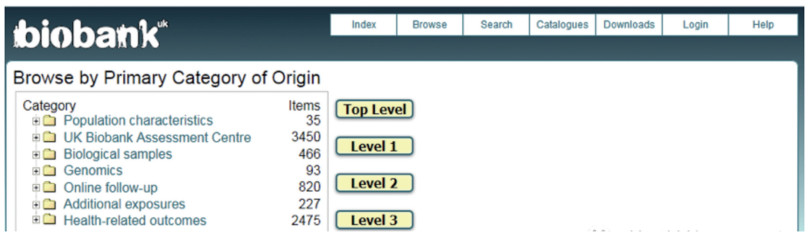
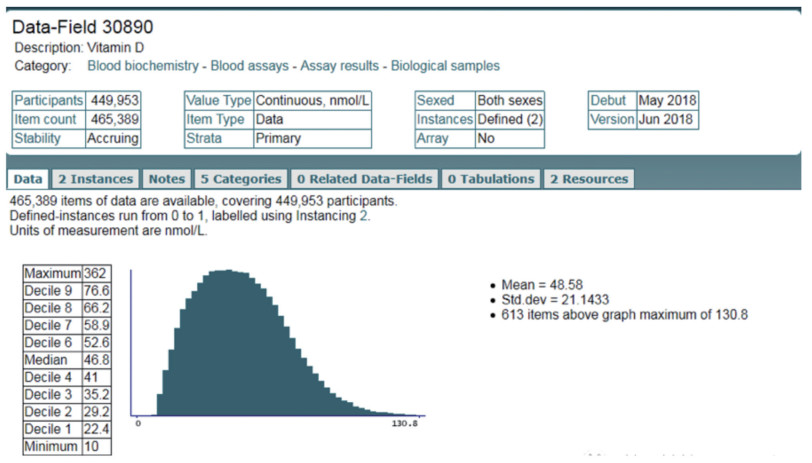
队列研究是流行病学及临床研究的金标准[1, 2],能够前瞻性地、纵向观察某一种疾病从发生发展到预后的整个过程,且可以追踪暴露因素的影响;相比于横断面研究,队列研究通过对人群随时间的变化情况进行观察,明确了暴露-结果的发生顺序,更好地证明了因果关系[3],应用此类方法更有助于客观评价相关性。多组学队列的大样本量(N > 10 000)、多时间点采样以及表型刻画等三大特点[4],使得多维数据集可以很好地发现疾病的分子基础,也可以很好地反映环境因素和遗传背景之间的相互作用。作为经典的多组学队列之一,UK Biobank至今已经收集了超过50万位参与者的完整表型和多层次组学信息,为慢性病机制研究提供了更好的平台[5](图 1、2)。
|
图 1 UK Biobank的数据目录 |
|
图 2 UK Biobank的数据域及队列信息 |
大模型的快速发展给队列研究带来了范式变革的机会[6, 7],主要包括以下三个方面:首先,队列研究产生的大量非结构化数据需要综合应用大模型多模态处理能力,大模型通过预训练学到了丰富的语义表征和跨模态理解能力,可以把临床指标数据、医学影像数据、电子病历(electronic medical record, EMR)数据等多源数据映射到同一语义空间中[8, 9],有利于从多种来源整合信息开展队列研究。其次,队列数据的时间动态属性与基于自注意力机制的Transformer架构相适应[10],可以通过自注意力机制和时间位置编码实现不规则采样间隔下的长距离上下文交互,兼顾各种尺度的时间关联关系[11]。最后,人群亚组异质性分析对模型表征学习能力和聚类能力有着较高的要求,能够发现数据内在的亚组结构,识别出具有相似分子特征但临床表现不同的疾病亚型[12]。
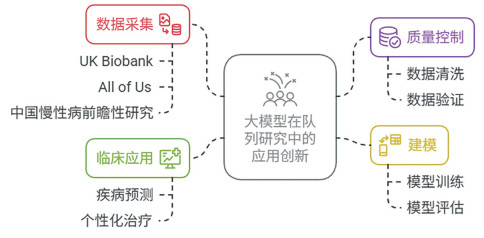
1.3 综述框架创新关于队列研究完整流程的大模型应用创新,在从数据采集到数据质量控制再到建模及临床应用等方面给出了大模型的整体技术路线图,如图 3所示。在此基础上着重分析了三个具有代表性的队列:UK Biobank[5]因含有大量的影像学、基因组数据等而被广泛使用;All of Us[13]特别强调多民族、多场景;中国慢性病前瞻性研究(CKB)[14]则针对亚洲人群特有的疾病危险因素。通过对以上三个队列的剖析(表 1),不仅能体现大模型应用于不同环境下的优势,同时也可以看出其在不同文化中的普适性。
|
图 3 整体性技术路线图 |
| 表 1 代表性大型队列研究的特点比较c |
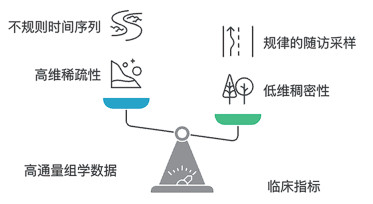
队列多组学数据具有自身的维度特征和时序特征(图 4)。从维度上看:高通量组学数据具有高维稀疏性(pn)的特点,传统统计方法难以直接处理;临床指标则具有相对低维稠密性的特点。从时间上看:队列很少能够做到规律的随访采样,形成不规则的时间序列;不同组学的采集频率与时序分辨率差异较大。不同组学之间代表的是生物系统从基因到表型的不同层面,在不同水平上反映了生物体不同层次的调控网络和调控机制,相互之间可以互相补充和印证。这种异质性带来数据整合挑战,大模型的表征学习能力为克服这一难题提供了新可能[15, 16]。
|
图 4 队列多组学数据的特点 |
批次效应及缺失数据处理是队列研究质量控制的两个难点。批次效应来源于样本采集、处理平台、操作人员的不同,有可能掩盖真实生物信号。大模型利用自监督学习[17]可以发现数据中存在的潜在规律,从而能区分技术变异和生物变异。对于缺失数据来说,队列研究随访脱落带有偏倚性,是非随机缺失。生成对抗网络(GANs)[18]和变分自编码器(VAEs)等生成模型可以学习数据分布特性,用生成模型自动生成与真实数据一致的样本来补齐缺失值,这样能更好地保留各变量之间的复杂关系。
2.3 伦理与隐私考量随着队列研究的增多,队列研究中涉及伦理和隐私的问题会更加突出,特别是动态知情同意和跨中心的数据共享的问题。动态知情同意允许参与者通过数字平台实时查看数据使用情况并调整授权范围,All of Us研究计划[13]率先采用了这一模式。对于跨中心的数据共享,联邦学习[19, 20]使得原始数据可保存在各个机构,仅向各个机构传递模型参数,与差分隐私[21]和同态加密等技术结合,在保障隐私安全的同时也可以实现协同分析的目的。
基于Transformer的联邦框架将模型性能损失控制在3%以内,同时满足各中心的隐私约束。值得注意的是,差分隐私[21]的引入需要权衡隐私保护强度与研究结果的统计效力,在慢性病预测任务中,ε=5时的ROC-AUC下降3.2%,而ε=10时可控制在1.5%以内,提示需要针对不同研究问题开展参数优化研究。
大模型应用给多中心队列研究带来了更加明显的伦理与隐私缺陷,比如:虽然All of Us项目[13]通过开发的电子同意平台并借助区块链技术完成了同意的状态实时更新、全程可溯源的工作,相比以往传统流程降低了21%的撤回率,但是在面临多模态数据的场景下,出现了一些新问题。其中最重要的两个问题:一是在二次使用生物样本的过程中可能会存在一些组学的衍生数据的所有权问题;二是因为影像数据存在匿名化不够完全(如体态特征的重识别风险)的风险,故都迫切需要提出新的方法来解决此类问题。另一个突出的难点是多个中心间数据共享过程中隐私保护以及算法在各个平台上的同步运行的问题,即面临着如何做到跨平台数据同构的问题。对此,现有联邦学习框架在协同训练过程中实现了分层架构上的优化[19]。在UK Biobank和BioMe两平台间的联合研究任务中,通过设计动态参数聚合策略区分组学特征的全局共享与临床特征的本地保留,基于Transformer的联邦框架能够实现整体模型准确率下降在3%内的效果,同时满足各中心的隐私约束。值得注意的是,差分隐私[21]的引入需要权衡隐私保护强度与研究结果的统计效力。在慢性病预测任务中,ε=5时的ROC-AUC下降3.2%,而ε=10时可控制在1.5%以内,提示需要开展针对不同研究问题的参数优化研究。
对于深度隐私保护而言,同态加密等新技术为基因组-表型相关分析提供可能。CKB项目利用加法和乘法密文操作,在保证χ²检验误差控制在1e-6水平[22],成功实现了全基因组关联研究(GWAS)的安全外包计算。但这种技术带来的计算开销(运行时间增加50~100倍)并不适合大范围推广。目前,生物医学领域数据尤其是生物医学文本数据隐私问题还未得到足够重视,NLP模型对EHR数据的细粒度重建实验表明,仅用住院时长、实验室检验项目等结构化字段,即可推断患者身份信息(准确度达72%),这要求开发文本解缠表示技术以分离临床语义特征和潜在身份标识符。
3 大模型在队列研究中的方法学创新 3.1 时间感知的建模架构Transformer架构[10]利用自注意力机制来克服RNN处理长序列梯度消失的问题,非常适合处理队列研究中的长周期随访数据。时间位置编码[11]是改造Transformer处理队列数据的关键创新,直接将绝对时间信息编码到模型中,使模型能感知事件的确切时间点和间隔。记忆增强网络通过显式外部记忆模块补充了注意力机制,能够存储历史观测中的关键信息。例如,在UK Biobank的心血管疾病预测研究中,时间感知Transformer模型通过精细的时间编码,整合了不同时间点的多源数据,显著提高了预测性能[11]。此外,基于Transformer的因果推断框架能更准确地估计动态处理效应,区分直接效应和中间生物标志物介导的间接效应[23]。
Transformer架构在多时间点队列数据分析中的优势已得到充分证实,但其对不规则间隔时间序列的处理仍存挑战。最新研究提出将离散时间编码扩展为两种互补机制[11]:基于Weibull分布的时间间隔嵌入方法可以捕获随访间期的变化模式,联合周期基函数用于提取周期性/生理周期(如昼夜及节气)信息。在UK Biobank心血管疾病预测中的混合编码可以使得预测能力提高约6个百分点。时间动态建模的另一个进展是多尺度注意力机制:采用分层次构建一系列分层的Transformer以不同比例的周、月、年建立本地化注意力窗口,以观测内部的信息,而跨越尺度链接能够捕捉长程依赖关系。这种方法显著提高了糖尿病视网膜病变进展预测中微血管变化的早期识别能力。
在处理多组学时序数据时,记忆增强网络具有独特优势。例如,当存储模块经过训练掌握组学指标间的动力学规律后,它可以利用学到的知识推测未知时间点的生物标记物插补值,且结果更符合真实情况。本文对CKB代谢组数据的测试表明,该方法使1年间隔外的外推预测误差降低31%。在因果推断方面,基于结构因果模型的变体Transformer (SCM-Transformer)通过显式建模组学时序变量间的因果关系约束,在识别疾病进展驱动因素方面的特异性比传统方法提升18%。这种架构通过可微D分离算法自动识别混杂路径[23],为高维组学数据的因果发现提供了新工具。
3.2 异质性人群分析技术基于注意力的亚群发现方法运用了Transformer的自注意力机制进行无监督分层[12],能够自适应性地为不同的特征分配权重,发掘亚群间极其细微的差异。在糖尿病队列的研究中,该方法发现具有不同并发症风险、对不同药物有不同反应的亚群[12](图 5)。大模型驱动的反事实推理框架通过学习高维协变量空间中的潜在表征,建立更为精准的反事实预测模型[23, 24],尤其适用于评估长期干预的效果;异质性治疗效应分析借助元学习框架,能够在未指定亚组的情况下发现异质性模式,并在欧洲多中心糖尿病队列研究中发现了对药物反应存在明显差异的亚群[10]。
|
图 5 糖尿病亚群发现方法 |
异质性分析深度学习方法改变了以往划分亚组的传统定义方式。基于动态原型网络的方法,每个样本能依据注意力权重聚合多种原型表征,以更好地刻画连续谱系式疾病亚型。例如在NASH患者的分层分析中,我们利用该方法发现有3个具有差异纤维化进展风险的亚群,其临床终点预测C-index达0.73,远超传统聚类方法(0.61)[12]。针对治疗效果异质性,最新型的反事实平衡表征框架利用对抗训练来消除协变量和治疗之间的虚假相关,在All of Us降压药疗效分析中,将处理效应估计偏差从0.12降至0.04标准差单位。
元学习方法在自动发现异质性模式方面取得突破。基于MAML-HTE框架的元学习方法可以在不需要分布转移的情况下,利用HTE任务中个体间治疗响应差异的模式来学习,当转移到新队列时只需要少量样本就可以根据分布差异调整对应的参数。
3.3 小样本迁移学习策略预训练-微调范式是迁移学习的成功实践,在队列研究中被广泛应用[8]。模型首先可以在大规模公开数据库如UK Biobank上预训练,然后用小规模的地方队列或者是针对一些特殊的疾病亚型微调,达到改善模型在小样本场景下的效果。通过领域适应技术可以学习到源域和目标域中均不发生变化的特征表示,从而实现队列间知识迁移,使得模型能够适用于其他队列数据。自监督学习[17]利用设计好的辅助任务,从海量无标记的组学数据中学习有用的表示,因此能够有效利用队列研究中大量的无标记组学数据。对比学习,通过最大化同一样本不同视图表征之间最大化正相关性来学习低维表示,能在小样本情况下获得优异表现。表 2归纳了大模型在队列研究中的方法学创新。
| 表 2 大模型在队列研究中的方法学创新及其应用 |
大模型驱动的动态风险评估模型能够追踪个体的风险变化规律,为临床上找到风险较小时的最恰当的干预时机[11, 12]。例如,在糖尿病前期人群研究中,基于Transformer的模型可以同时完成总体风险预测和风险模式及高危节点的定位问题,相较于传统的Cox模型有大约15%的提升。多组学早诊标志物挖掘通过整合多维数据,识别出疾病的前临床信号。在阿尔茨海默病研究中,发现了早于疾病出现10年内可以检测到的病发组合早期标志物[8],使得疾病的早诊准确性得到了显著的提升。大模型还能将总体风险分解为不同组分的贡献,通过注意力机制和特征归因分析(如SHAP值[27]),为靶向干预提供精准指导。
4.2 治疗响应预测在药物基因组学队列研究中,大模型整合了基因组变异、表达谱、代谢组特征和临床参数等信息,形成了高精度的个体化剂量预测模型。例如,利用深度学习模型将华法林剂量预测准确度提高30%以上,显著降低了并发症风险[28]。在肿瘤免疫治疗预测领域,大模型整合肿瘤微环境的多维信息,显著提高了预测准确性。在肺癌免疫治疗队列研究中,多模态大模型将AUC提高至0.85,显著优于传统生物标志物[8]。在药物不良反应监测方面,基于Transformer的模型结合纵向健康记录与基因组学数据,能够准确识别基因型特异性的不良反应模式,并适应时间延迟效应和累积剂量效应,挖掘一些传统的不良反应监测方法不易监测到的长期的安全性信号[29]。
4.3 公共卫生决策大模型支持的人群分层干预策略是根据队列数据的风险分布以及干预的响应模式,确定最受益于干预的亚群,据此提供更加科学的干预。采用精准分层策略可在糖尿病预防研究中提高可预防病例的比例约40%。同时,集成队列研究数据、实时监测信号及环境因素等建立了流行病学预警系统,能提前2~3周预测传播水平的变化及相应医疗资源的需求情况;健康不平等分析通过揭示社会决定因素与健康结果之间错综复杂的关系,指导制定减少健康差距的相关政策。例如在All of Us研究[13]中,利用因果推断增强的大模型可量化解构影响健康差距的因素当中能被改变的社会因素部分,为今后精准的政策干预措施提供参考价值。表 3清晰展示出大模型在疾病风险预测、治疗响应预测、公共卫生决策3种临床应用场景下的临床意义。
| 表 3 大模型驱动的临床应用场景对比 |
随访丢失可能导致严重的生存分析偏倚。大模型可以采用基于生成模型的合成数据填充方法来保留各个变量之间的复杂关联;自适应设计队列实时预测某参与者的随访丢失概率,并动态调整决策的顺序,在All of Us研究计划[13]中应用了这样一套系统的做法,可以提高长期随访的留存率约15%;多源数据融合是利用现有数据和外部数据来补充失访参与者的相关结局。组学技术发展的不同步会造成数据异质化,以领域对抗神经网络或周期一致性生成对抗网络[26]为基础可以解决该类问题,使得长期队列研究不仅可以在数据本身应用最新的技术,同时也能保证数据纵向的可比性。
5.2 模型可解释性需求临床环境中对模型的可解释性需求高于一般的场景[30, 31]。针对临床医生这一人群需求,SHAP值[27]、LIME[32]等局部可解释性方法可以量化出特征对于预测的贡献大小,但是由于面对的对象是患者,因此不能使用大量晦涩难懂的专业名词去阐述。同时需要突出其中的具体的易操作的要点,层层递进地展开。而为了满足监管部门的要求,反事实解释[33]能够直接告诉我们当输入变量有变动的时候,会得到什么结果,这种方式能够更加直接且真实地展现模型的行为,而算法审计工具则是通过一系列的检测来推断算法是否公平、鲁棒。
5.3 转化医学瓶颈从实验室转化到临床尚存在很大距离,如何将大模型驱动的大队列研究变为临床实用已成为亟待解决的问题[34, 35]。学术上对于评价指标的评价很高,例如AUC(曲线下面积)、灵敏度(sensitivity)与特异度(specificity),但这些指标并不能作为临床有效的证据,因为它们未能完全反映实际临床中面临的情况与资源约束问题[34]。为此,一些临床影响评估框架被陆续开发出来。①临床决策修改率:直接量化模型预测对临床决策的影响,即通过事先决定后听取模型结论再修改最初决定的概率来衡量。此项指标是基于前瞻性的临床决策研究所获取。②需治疗/检测人数(number needed to treat/test, NNT)与净重分类指数(net reclassification index, NRI),这两个指标是从资源分配的角度去估计其有用价值,以及确定要发现1个真正阳性需要多少人筛查或检测(图 6)。比如,心血管风险预测模型评估数据显示,在纳入多组学标志物后NNT可由25降至10,即为预防1例心血管事件需要干预的患者人数减少60%以上,降低了筛查所需的实际样本数。
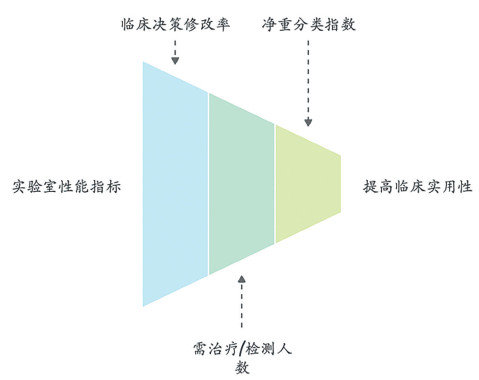
|
图 6 从实验室到临床的转化 |
实施科学(Implementation Science) 研究为大模型从研发到临床应用提供了一个核心框架,在此过程中会遇到诸多阻碍因素[35],也能识别出一些有利的促进因素。这样可以让潜在问题提前显现,并提出相应对策来规避不利因素可能造成的影响,包括评估大模型接入临床工作的难度、用户接受度、成本效益以及所需的基础设施支撑等。实施科学方法(如RE-AIM框架)能够全面评估临床转化过程中的干预措施,并不仅仅只是单纯评价其技术效果,例如整合基因组风险评分的糖尿病预防模型的技术在有效性的维度表现得较好,但尚需考虑模型是否具有足够可及性(Reach),模型的应用能否维持较长时长(Maintenance),针对这种情况可以采取简化检测流程或建立自动化维护体系的办法去改善转化成效,全面地对大模型临床试验的全过程加以检视,才能挖掘出产生问题的关键瓶颈,从而更好地提升模型临床试验的接受度以及持续性。
真实世界验证(RWV)是转化过程的最后一个阶段,也是难度最大的一步,在此阶段需要保证模型即便是在现实情况而非最理想的运行环境下也可以维持良好的表现水平[36, 37]。科研队列和临床实践环境数据相差较大,科研队列样本同质性强,数据完备,随访完整;临床实践环境中数据噪声大、随访不规律、人群异质性大。为了解决上述问题,研究者分步开展验证研究,从科研队列至多中心EHR数据库[36, 37],再到前瞻性的临床实施应用,逐步检验模型在接近实际情况的环境中能否实现相应的价值。为了使模型适用于真实世界场景,还需要进行特定的模型适应策略,如增量学习(incremental learning),即在上线后可以不断地从新数据中学习与更新;再有就是正则化(regularization),保证模型更新时性能不会有太大变化,从而使患者体验相对平稳。例如,一项前列腺癌风险预测模型的真实世界验证结果表明,在通过增量学习适应后,其稳定性保持较好,未做适应的学习性能在各亚群之间波动很大,因此,在真实世界的使用中可能存在疗效失常的风险。严格的RWV过程既是获得监管批准的关键,也是建立医生信任、用于大规模推广的重要基础[38]。根据以上内容表 4总结了大模型在队列研究中面临的挑战与对策。
| 表 4 大模型在队列研究中面临的挑战与对策 |
嵌入式AI队列是把人工智能由被动分析的工具转化为队列设计、运行的主要组成部分,利用算法自适应地进行队列设计与运行,并根据已有数据持续学习并迭代优化数据采集的方法和方式。同时,在实时采集数据的过程中也不断地更新模型,实现模型的动态学习;自适应采样,针对不同的亚组设定最佳的数据采集方案;自学习假设生成可以自动从数据中挖掘出一些新问题的研究假设。数字孪生队列[39]通过创建参与者的数字化复制体,实现了前所未有的自由度实验方式。虚拟对照生成给已干预的患者加上一个假的对照组,模拟该患者是否进行干预后的不同结局情况;虚拟临床试验模拟可以在虚拟世界中做实验来预测试验的结果,然后再用于真实的临床试验设计上,以此方式显著减少试错成本[39, 40]。
6.2 方法学突破点因果表征学习[23]通过将数据映射到解缠表征空间,实现了混杂因素的自动识别和调整,在糖尿病前期进展研究中成功将基因型、代谢状态等因素分离到不同维度,实现对不同干预路径的精确评估。反事实因果推断[23, 24, 33]根据建模后的个性化潜在结果模型,可以对每一个体在未来采用不同的干预后的不同可能效果做出个性化推断,这也是精准医学的关键所在。多模态大模型统一框架[8, 9]不仅可以有效整合各种模态多源异构的数据,而且还可以在共享表征空间和不同模态的数据中使用不同的编码器来协同学习、转移知识;随着计算能力及算法的发展[15, 41],未来能够接受更多的模态,并且可运用于更加庞大的队列数据。
6.3 伦理框架演进动态风险告知的伦理规范需要将知情同意从一次性的事后告知变为持续过程,并允许参与人员根据自己的实际情况获取不同风险等级的信息;同时采用可理解的方式清楚准确地传达置信区间、预测区间和其他可能的解释,避免“假精确性”。为保证算法公平性,提出了基于多维公平性的算法公平性定义;在模型训练阶段加入了公平感知的预训练方法,结合两个不同群体的样本点来调节模型的权重大小,使不同群体的人能被公平对待,从而显著提升了公平均衡效果。公众参与是伦理规范演变的主导力量,以实现伦理规范能够契合各利益方的价值与期许为原则,确保伦理标准不断完善与变革。根据以上内容,表 5总结了大模型驱动队列研究的未来发展方向。
| 表 5 大模型驱动队列研究的未来发展方向 |
本综述系统梳理了大模型驱动的多组学队列数据整合与疾病预测的前沿进展。时间感知的建模架构、异质性人群分析技术和小样本迁移学习策略等方法学创新使队列研究从描述性分析向精准预测和因果推断转变。在临床应用方面,大模型驱动的队列分析已在多个关键领域展现价值:动态风险评估模型和早诊标志物挖掘提高了疾病早期识别的准确性;治疗响应预测和药物基因组学分析促进了个性化治疗决策;人群分层干预策略和流行病学预警系统优化了公共卫生资源分配。这些应用不仅提高了医疗决策的精准度,还为医疗系统的成本效益带来了实质性改善。尽管面临随访丢失、技术异质性、模型可解释性和转化医学瓶颈等挑战,嵌入式AI队列、数字孪生队列、因果表征学习和多模态统一框架等创新方向正开辟新的可能性。
大模型驱动的多组学队列研究是技术手段的革新,也是医学科学方法论的革命。通过整合生物学知识、计算方法和临床实践,有望重构从生物技术到医疗范式的创新链条,朝着以预防为主的精准治疗迈进。今后随着技术的发展以及应用经验的积累,我们期待这一领域在未来产生更多突破性成果,最终实现从数据到知识、从知识到行动的转化,为人类健康做出实质性贡献。
| [1] |
Caruana E, Roman M, Hernandez-Sanchez J, et al. Longitudinal studies. J Thorac Dis, 2015, 7: E567-70. |
| [2] |
Coggon D, Rose G, Barker DJP. Chapter 7. Longitudinal studies[M]//Epidemiology for the Uninitiated. 4th ed. London, UK: BMJ Books, 1997
|
| [3] |
Capilli B, Anastasi JK. Overview: cohort study designs. Am J Nurs, 2021, 121: 45-8. |
| [4] |
Manolio TA, Weis BK, Cowie CC, et al. New models for large prospective studies: is there a better way?. Am J Epidemiol, 2012, 175: 859-66. DOI:10.1093/aje/kwr453 |
| [5] |
Sudlow C, Gallacher J, Allen N, et al. UK biobank: an open access resource for identifying the causes of a wide range of complex diseases of middle and old age. PLoS Med, 2015, 12: e1001779. DOI:10.1371/journal.pmed.1001779 |
| [6] |
Topol EJ. High-performance medicine: the convergence of human and artificial intelligence. Nat Med, 2019, 25: 44-56. DOI:10.1038/s41591-018-0300-7 |
| [7] |
Rajkomar A, Dean J, Kohane I. Machine learning in medicine. N Engl J Med, 2019, 380: 1347-58. DOI:10.1056/NEJMra1814259 |
| [8] |
Zitnik M, Nguyen F, Wang B, et al. Machine learning for integrating data in biology and medicine: principles, practice, and opportunities. Inf Fusion, 2019, 50: 71-91. DOI:10.1016/j.inffus.2018.09.012 |
| [9] |
Banerjee I, Ling Y, Chen MC, et al. Comparative effectiveness of convolutional neural network (CNN) and recurrent neural network (RNN) architectures for radiology text report classification. Artif Intell Med, 2019, 97: 79-88. DOI:10.1016/j.artmed.2018.11.004 |
| [10] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need[C]//Advances in Neural Information Processing Systems 30 (NIPS 2017). Curran Associates, Inc., 2017: 5998-6008
|
| [11] |
Cheng L, Varghese N, Imran A, et al. Temporal representation learning for health trajectories: applications to electronic health records and biobank data. Nat Commun, 2022, 13: 7612. DOI:10.1038/s41467-022-35289-z |
| [12] |
Alaa AM, van der Schaar M. Attentive state-space modeling of disease progression[C]//Advances in Neural Information Processing Systems 32 (NeurIPS 2019). Curran Associates, Inc., 2019: 11338-48
|
| [13] |
Denny JC, Rutter JL, Goldstein DB, et al. The "All of Us" research program. N Engl J Med, 2019, 381: 668-76. DOI:10.1056/NEJMsr1809937 |
| [14] |
Chen Z, Lee L, Chen J, et al. Cohort profile: the Kadoorie Study of Chronic Disease in China (KSCDC). Int J Epidemiol, 2005, 34: 1243-9. DOI:10.1093/ije/dyi174 |
| [15] |
Aggarwal CC. Neural Networks and Deep Learning: Cham: Springer, 2018.
|
| [16] |
Ching T, Himmelstein DS, Beaulieu-Jones BK, et al. Opportunities and obstacles for deep learning in biology and medicine. J R Soc Interface, 2018, 15: 20170387. DOI:10.1098/rsif.2017.0387 |
| [17] |
Kipf TN, Welling M. Semi-supervised classification with graph convolutional networks[C]//International Conference on Learning Representations, 2017
|
| [18] |
Yoon J, Jordon J, van der Schaar M. GANITE: Estimation of individualized treatment effects using generative adversarial nets[C]//International Conference on Learning Representations, 2018
|
| [19] |
McMahan HB, Moore E, Ramage D, et al. Communication-efficient learning of deep networks from decentralized data[C]//Artificial Intelligence and Statistics. PMLR, 2017: 1273-82
|
| [20] |
Kairouz P, McMahan HB, Avent B, et al. Advances and open problems in federated learning. Found Trends Mach Le, 2021, 14: 1-210. DOI:10.1561/2200000083 |
| [21] |
Dwork C, Roth A. The algorithmic foundations of differential privacy. Found Trends Theor Comput Sci, 2014, 9: 211-407. DOI:10.1561/0400000042 |
| [22] |
Raisaro JL, Troncoso-Pastoriza JR, Misbach M, et al. MedCo: enabling secure and privacy-preserving exploration of distributed clinical and genomic data. IEEE/ACM Trans Comput Biol Bioinform, 2019, 16: 1328-41. DOI:10.1109/TCBB.2018.2854776 |
| [23] |
Prosperi M, Guo Y, Sperrin M, et al. Causal inference and counterfactual prediction in machine learning for actionable healthcare. Nat Mach Intell, 2020, 2: 369-75. DOI:10.1038/s42256-020-0197-y |
| [24] |
Pearl J. Causality: Models, Reasoning, and Inference: 2nd ed. Cambridge: Cambridge University Press, 2009.
|
| [25] |
Shalit U, Johansson FD, Sontag D. Estimating individual treatment effect: generalization bounds and algorithms[C]//Proceedings of the 34th International Conference on Machine Learning. PMLR, 2017: 3076-85
|
| [26] |
Fortin JP, Parker D, Tunç B, et al. Harmonization of multi-site diffusion tensor imaging data. Neuroimage, 2017, 161: 149-70. DOI:10.1016/j.neuroimage.2017.08.047 |
| [27] |
Lundberg SM, Lee SI. A unified approach to interpreting model predictions[C]//Advances in Neural Information Processing Systems 30 (NIPS 2017). Curran Associates, Inc., 2017: 4765-74
|
| [28] |
Zou J, Huss M, Abid A, et al. A primer on deep learning in genomics. Nat Genet, 2019, 51: 12-8. DOI:10.1038/s41588-018-0295-5 |
| [29] |
Wang Y, Wang L, Rastegar-Mojarad M, et al. Clinical information extraction applications: a literature review. J Biomed Inform, 2018, 77: 34-49. DOI:10.1016/j.jbi.2017.11.011 |
| [30] |
Rudin C. Stop explaining black box machine learning models for high stakes decisions and use interpretable models instead. Nat Mach Intell, 2019, 1: 206-15. DOI:10.1038/s42256-019-0048-x |
| [31] |
Ahmad MA, Eckert C, Teredesai A. Interpretable machine learning in healthcare[C]//Proceedings of the 2018 ACM International Conference on Bioinformatics, Computational Biology, and Health Informatics. New York: Association for Computing Machinery, 2018: 559-60
|
| [32] |
Ribeiro MT, Singh S, Guestrin C. "Why should I trust you?": Explaining the predictions of any classifier[C]//Proceedings of the 22nd ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. New York: Association for Computing Machinery, 2016: 1135-44
|
| [33] |
Wachter S, Mittelstadt B, Russell C. Counterfactual explanations without opening the black box: automated decisions and the GDPR. Harv JL Tech, 2017, 31: 841-922. |
| [34] |
Yu MK, Ma J, Fisher J, et al. Visible machine learning for biomedicine. Cell, 2018, 173: 1562-5. DOI:10.1016/j.cell.2018.05.056 |
| [35] |
Ghassemi M, Naumann T, Schulam P, et al. A review of challenges and opportunities in machine learning for health. AMIA Summits Transl Sci Proc, 2020, 2020: 191-200. |
| [36] |
Johnson AEW, Pollard TJ, Shen L, et al. MIMIC-III, a freely accessible critical care database. Sci Data, 2016, 3: 160035. DOI:10.1038/sdata.2016.35 |
| [37] |
Jha AK, DesRoches CM, Campbell EG, et al. Use of electronic health records in U.S. hospitals. N Engl J Med, 2009, 360: 1628-38. DOI:10.1056/NEJMsa0900592 |
| [38] |
Beam AL, Kohane IS. Big data and machine learning in health care. JAMA, 2018, 319: 1317-8. DOI:10.1001/jama.2017.18391 |
| [39] |
Björnsson B, Borrell A, Bru G, et al. Digital twins to personalize medicine. Genome Med, 2020, 12: 4. DOI:10.1186/s13073-019-0701-3 |
| [40] |
Corral-Acero J, Margara F, Marciniak M, et al. The 'digital twin' to enable the vision of precision cardiology. Eur Heart J, 2020, 41: 4556-64. DOI:10.1093/eurheartj/ehaa159 |
| [41] |
Miotto R, Wang F, Wang S, et al. Deep learning for healthcare: review, opportunities and challenges. Brief Bioinform, 2018, 19: 1236-46. DOI:10.1093/bib/bbx044 |
 2025, Vol. 37
2025, Vol. 37 

 方金武,中国信通院上海工创中心副总经理,数字健康事业部总经理,兼任中国人工智能产业发展联盟医学人工智能委员会副秘书长、中国互联网协会互联网医疗健康工作委员会副秘书长、国家卫生健康委能力建设和继续教育中心放射影像标准数据库专家、上海市产医融合战略咨询委员会人工智能医疗器械专业委员会委员、上海市生物医药行业协会生物医药数字化专委会秘书长、复旦大学智能医学研究院智能医学产业合作中心主任。作为高级工程师,重点关注人工智能医疗、医疗大模型、类脑智能、医疗数据、医疗元宇宙、医疗具身智能、智慧医院、AI医药等领域,参与相关的标准制订、政策预研、产业规划、测试评价等相关工作,先后多次承担省部级课题,参与制订标准十余项;参与主导智慧医疗白皮书等研究报告十余项;发表英文SCI、SSCI期刊论文及中文权威期刊9篇
方金武,中国信通院上海工创中心副总经理,数字健康事业部总经理,兼任中国人工智能产业发展联盟医学人工智能委员会副秘书长、中国互联网协会互联网医疗健康工作委员会副秘书长、国家卫生健康委能力建设和继续教育中心放射影像标准数据库专家、上海市产医融合战略咨询委员会人工智能医疗器械专业委员会委员、上海市生物医药行业协会生物医药数字化专委会秘书长、复旦大学智能医学研究院智能医学产业合作中心主任。作为高级工程师,重点关注人工智能医疗、医疗大模型、类脑智能、医疗数据、医疗元宇宙、医疗具身智能、智慧医院、AI医药等领域,参与相关的标准制订、政策预研、产业规划、测试评价等相关工作,先后多次承担省部级课题,参与制订标准十余项;参与主导智慧医疗白皮书等研究报告十余项;发表英文SCI、SSCI期刊论文及中文权威期刊9篇