肿瘤的发生和发展是一个涉及复杂分子机制的过程,其显著的特征在于肿瘤细胞之间以及不同肿瘤个体间的异质性[1, 2]。这种异质性体现在形态、转录谱、代谢途径以及转移潜能等多个方面,是导致肿瘤对治疗产生抵抗、更易发生转移以及疾病复发的重要原因[1-3]。肿瘤的异质性不仅存在于基因层面,也受到表观遗传修饰和肿瘤微环境的动态影响及其交互作用[4]。深入理解肿瘤异质性的驱动因素,包括基因组的不稳定性、表观遗传的改变以及微环境的相互作用,对于设计能够应对耐药性的治疗策略至关重要[2, 4]。近年来,单细胞和空间基因组学等新兴技术的发展,正在不断深化我们对肿瘤异质性的认识,并为实现更加精准的个性化治疗提供了新的研究和干预路径[1]。
1.2 基因组学与人工智能在肿瘤研究中的作用在理解肿瘤的发生和进展过程中,肿瘤基因数据发挥关键作用[5]。基因组学方法不仅能够识别个体化的靶向治疗靶点,还可以评估肿瘤的突变负荷,这对于预测患者对免疫检查点抑制剂的治疗反应具有重要意义[6]。此外,基因组学分析还能发现肿瘤特异性的抗原,为设计个性化的抗癌疫苗提供信息。随着研究的深入,肿瘤基因组学在临床实践中的应用也日益广泛,包括监测患者对治疗的反应以及深入理解肿瘤耐药机制。基因组学方法能够提供的信息远超传统的单一靶点分析,例如突变负荷等指标,对于预测更广泛的治疗效果至关重要。临床实践中基因组学应用的普及,也凸显了推广基因检测可及性的必要性,以使更多患者能够从中获益[6]。
人工智能技术(AI)已经成为解决包括医学成像在内的复杂数据分析挑战的强大工具[7]。在肿瘤基因组数据分析领域,人工智能展现出巨大的潜力,能够实现疾病的早期诊断、高效筛查,并根据患者的基因特征制定个性化的治疗方案[8-10]。尤其值得关注的是,大语言模型(LLMs)在生物医学领域已经显示出非凡的能力,它们能够有效地从大规模、多样化的数据集中学习,并执行各种复杂的下游任务[11]。人工智能在处理高维度、复杂的生物医学数据方面表现出卓越的能力。大语言模型不仅在自然语言处理领域取得了突破性进展,其在生物医学数据分析中的潜力也正被积极探索和挖掘[12]。人工智能驱动的个性化医疗有望显著提高肿瘤治疗的有效性,并最终改善患者的预后[13, 14]。
1.3 本研究的目标与方法本文旨在系统综述人工智能技术在肿瘤基因数据分析和个性化治疗中的应用现状、关键技术路径与面临的挑战,聚焦其在识别变异、风险评估、个性化推荐等任务中的实际表现与潜力。文章以“人工智能如何赋能肿瘤个性化治疗”为主线,重点探讨从基因层面出发的智能分析技术,而不涉及传统的治疗手段如手术、化疗和放疗等。通过梳理近年来相关技术的演进脉络与代表性研究,本文力图展现AI在推动个性化肿瘤治疗中的独特价值,并为未来研究与临床落地提供思路参考。
为提高本综述的客观性与规范性,我们设定了文献筛选策略与纳入标准。文献主要检索自PubMed、EMBASE、Web of Science等主流生物医学数据库,检索时间范围主要限定为近十年,关键词包括“人工智能”“肿瘤基因”“个性化治疗”等。纳入标准包括:(1)研究内容聚焦AI在肿瘤基因数据分析或个体化治疗中的应用;(2)研究设计合理,优先纳入随机对照试验、队列研究等高质量研究;(3)报告完整,提供可复核的方法与统计信息;(4)符合伦理规范。
2 肿瘤基因数据的特征与挑战 2.1 基因组学技术与多组学整合高通量测序技术的飞速发展,尤其是下一代测序(NGS)技术的广泛应用,彻底改变了肿瘤研究的格局,使得研究人员能以前所未有的规模和精度探索基因变异,包括单核苷酸多态性(SNPs)、拷贝数变异(CNVs)、基因表达谱、microRNA表达、蛋白质水平表达等遗传变异信息[13]。近年来,多组学方法逐渐成为主流[15-17],它整合了来自基因组学、蛋白质组学、转录组学和代谢组学等不同层面的数据[18-20],旨在为肿瘤的复杂性提供更全面的理解[15]。不同类型的组学数据,例如基因组、转录组和蛋白质组,提供了关于肿瘤不同层面的信息,而整合这些信息对于深入理解疾病的发生机制至关重要[17, 21]。多组学数据的出现,也为开发新的肿瘤预后、诊断和治疗方法开辟了广阔的前景[15, 16, 22, 23]。
2.2 肿瘤基因数据库与资源为了存储和共享这些海量的肿瘤基因数据,全球范围内建立了多个重要的数据库。癌症基因组图谱(TCGA)是由美国国家癌症研究所(NCI)和美国国家人类基因组研究所(NHGRI)共同发起的一项里程碑式的癌症基因组学计划。TCGA旨在全面编目和发现超过30种人类主要癌症类型的关键基因组改变,通过对超过20 000个原发性肿瘤和匹配的正常样本进行大规模基因组测序和整合的多维度分析,为研究人员提供了前所未有的数据资源[24, 25]。AACR Project GENIE (Genomics Evidence Neoplasia Information Exchange)是一个国际性的数据共享联盟,其目标是通过整合全球多家顶级癌症中心治疗的数万名癌症患者的临床级别基因组数据和临床结局数据,为精准癌症医学的研究奠定坚实的证据基础[26, 27]。cBioPortal for Cancer Genomics则是一个开放存取、交互式的资源,提供了来自TCGA、ICGC等多个大型癌症基因组学项目的数据,方便研究人员进行泛癌研究和特定癌症的深入分析[28, 29]。此外,基因组数据共享中心(GDC)也作为NCI的一个重要组成部分,存储、标准化并共享大量的癌症基因组、转录组和表观基因组数据,供全球研究人员使用[30]。这些大型公共数据库的建立,为癌症研究者提供了宝贵的资源,涵盖了不同类型的癌症和不同组学的数据,为泛癌研究和特定癌症的深入探索提供了坚实的基础。数据共享联盟如GENIE的出现,通过促进跨机构的数据整合和分析,显著加速了精准医疗研究的进展。表 1对这些主流的肿瘤基因数据库进行了系统性的总结,详细列出了各个数据库的名称(如癌症基因组图谱TCGA、国际癌症基因组联盟ICGC等)、其主要功能描述、所包含的关键数据类型(如基因组学、转录组学数据)、大致的样本或研究数量以及公开的访问途径,为研究人员快速了解和利用这些资源提供了便利。
| 表 1 主要肿瘤基因数据数据库 |
然而,肿瘤基因数据本身也具有一些显著的特点,给数据分析带来了诸多挑战。首先,这些数据通常是高维度的,例如,一个基因组可能包含数百万个基因变异,而基因表达谱则涉及数万个基因的表达水平[31]。其次,如前所述,肿瘤内部以及不同患者肿瘤之间的基因组变异存在显著的差异,导致数据的高度异质性[1, 3]。第三,由于获取全面且带标签的临床数据往往成本较高且耗时,肿瘤基因数据通常存在标签稀缺的问题,这限制了监督学习方法的应用。最后,患者的基因信息属于高度敏感的隐私数据,一旦泄露可能会对个人及其家庭造成不良影响,因此需要采取严格的措施进行保护[32]。肿瘤基因数据的复杂性给传统的数据分析方法带来了巨大的挑战。数据异质性意味着需要开发能够适应不同患者和肿瘤特征的分析方法[3],而标签稀缺和隐私问题则限制了可用于模型训练的数据量和类型。
2.4 人工智能解决方案的潜力与局限面对这些挑战,人工智能技术的介入显得尤为必要。人工智能,特别是机器学习和深度学习算法以及近几年快速发展的大语言模型,能够处理和学习这些高维度、异构的数据集,从而为肿瘤基因数据的分析提供强有力的工具[18, 33]。然而,在将人工智能应用于生物医学数据集时,仍然存在一些显著的挑战,例如数据隐私的保护、不同来源数据的融合以及模型结果的解释性等[20, 32, 34]。将人工智能全面融入医疗保健领域,还需要克服技术、伦理和社会等多重障碍,包括如何保障患者的隐私和数据的安全、如何确保人工智能系统的安全性和有效性[35, 36]、如何获得患者的知情同意、如何控制应用的成本、如何保证技术的广泛可及性以及如何评估其真正的临床有效性等问题[37, 38]。因此,尽管人工智能在肿瘤基因数据分析领域潜力巨大,但在实际应用中仍面临诸多挑战,需要研究人员仔细权衡并积极寻求解决方案。解决这些挑战需要跨学科的合作和创新,结合生物学、医学、计算机科学和伦理学等多方面的专业知识。
3 人工智能在肿瘤基因数据处理中的典型任务与案例 3.1 人工智能技术在肿瘤基因分析中的应用概述人工智能技术包括机器学习和深度学习方法,已被广泛应用于肿瘤基因数据的分析,其中患者的诊断是最常见且最重要的应用之一[10, 39]。图 1直观地展示了人工智能在肿瘤研究中的典型工作流程。该流程概括了从基础研究(例如a部分所示的蛋白质结构预测、高通量分析、基因组结构识别)到转化研究以及临床研究的各个阶段(例如a部分同样提及的病理标志物识别、响应评估和多模态数据结合)。图 1的b部分具体描绘了人工智能(AI)图像处理的三种主要方法。方法一是标准的图像分析流程,涵盖了从3D切片到亚细胞级别的多尺度分析。方法二则阐述了构建机器学习管道的关键步骤,包括样本准备、模型训练、参数调整、测试验证及最终部署。值得注意的是,方法三强调了基础模型(foundation models)的应用潜力,它们能够利用海量数据和预训练能力,通过少样本学习、零样本学习或直接应用来解决特定问题。除了传统的机器学习方法,图神经网络(GNNs)作为一种在非欧几里得空间进行深度学习的分支,在处理具有复杂关系结构的图数据方面表现尤为出色,并已迅速成为生物信息学领域中不可或缺的重要工具[40]。知识图谱则通过将生物医学领域中的各种实体(例如基因、疾病、药物)及其之间的复杂关系表示为图结构,极大地促进了对异构生物医学数据的整合、高级推理和新知识的发现[41, 42]。值得一提的是,大语言模型(LLMs)虽然最初主要应用于自然语言处理领域,但通过针对特定任务进行微调以及结合领域知识库进行增强,在识别基因与表型之间的关联方面也展现出令人印象深刻的准确率[11]。这些不同的AI技术相互补充,共同推动了肿瘤基因数据分析能力的不断提升[9]。
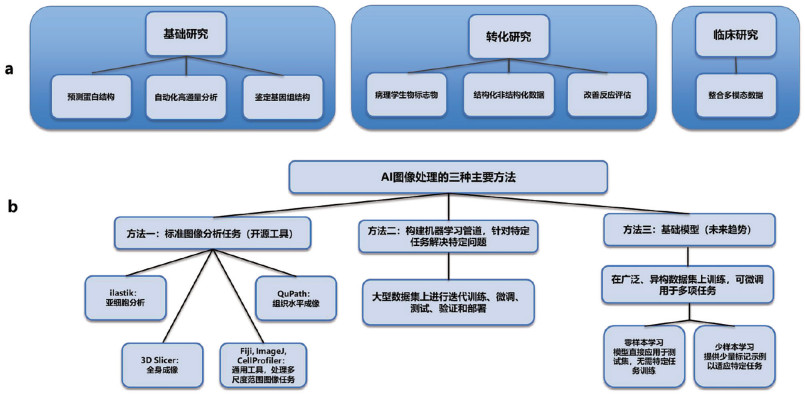
|
图 1 肿瘤研究中的AI工作流程 |
在肿瘤基因数据分析中,基因变异的识别与致病性预测是至关重要的环节。DeepVariant是一种利用深度学习算法进行基因变异识别的工具,其创新之处在于将该问题转化为一个图像分类任务。它将测序仪读段(reads)比对到参考基因组的信息转换成一种图像化张量,再利用深度卷积神经网络(CNN)进行分析。这种方法尤其擅长处理传统统计模型面临挑战的基因组区域(如高GC含量或重复序列),能有效过滤测序仪特有的错误模式。然而,DeepVariant的性能高度依赖于训练数据的质量和测序平台类型,对于训练集中未包含的罕见变异类型或新型测序技术可能表现不佳,且其运行所需的大量计算资源也对普通实验室构成了应用门槛。SpliceAI则是一款专门用于预测基因剪接位点和可能影响剪接的隐蔽性变异的AI工具,它通过一个深度残差网络(deep residual network),分析包含变异位点在内的数千个碱基对的序列上下文,以预测该变异是否会产生新的剪接位点或破坏原有位点[43]。研究表明,人工智能算法能够准确地预测基因突变对基因表达水平的影响,并对调控区域的变异进行优先排序[44],从而帮助研究人员聚焦于可能具有重要生物学意义的变异,有时结合可解释性方法能进一步估计其致病性[45]。SpliceAI的适用场景主要集中在遗传病诊断和癌症基因组学研究,特别是需要评估变异对基因表达影响的研究。然而,SpliceAI的预测准确性可能受到训练数据的影响,对于复杂的剪接事件,其预测能力可能有限,且需要结合其他实验证据进行验证。这些AI工具的应用,极大地提升了我们识别和理解肿瘤基因组中关键变异的能力,为后续的致病机制研究和临床应用奠定了基础。
家族史是评估个体患癌风险的重要因素。人工智能技术可以辅助将传统的纸质家族谱系图数字化,并利用机器学习和深度学习技术,自动预测个体罹患遗传性肿瘤的风险[46]。此外,基于大规模基因数据的风险分层模型,结合AI算法,能够更精确地识别出具有较高癌症风险的人群,从而为他们提供更有效的筛查和预防策略[47]。这种基于基因数据的风险分层方法,有助于实现更个性化的癌症预防和早期干预措施。
3.3 风险评估与个性化治疗推荐人工智能还在基于基因数据的预筛查工具和治疗推荐系统的开发中发挥关键作用。AI驱动的预筛查工具能够利用基因数据进行早期癌症检测,提高早期诊断率[13]。同时,基于患者个体基因数据的推荐系统,能够为临床医生提供更加个性化的治疗建议,从而提高治疗的精准性和有效性[48, 49]。AI算法通过分析大量的医疗数据,包括患者的病历记录、医学影像以及基因信息,能够以更高的准确性和更快的速度辅助诊断疾病[50, 51]。这些先进的预测和推荐技术正在显著改变健康预测的未来趋势,逐步实现从基于人口水平的健康管理向精准个性化健康预测的转变[52]。
3.4 多模态数据融合与整合分析近年来,多模态数据融合成为人工智能在肿瘤研究中的一个重要趋势。多模态深度学习(MDL)方法通过整合来自不同数据来源的信息,例如医学影像、基因组数据和临床文本,能够提供对肿瘤更全面、更深入的理解[14, 19]。例如,Pathology-GPTs等模型正在积极探索将病理图像、基因数据和临床文本相结合,以期显著提高癌症诊断和预后预测的准确性[53]。这种整合不同数据类型的方法,能够捕捉它们之间复杂的相互作用关系,从而为临床决策提供更丰富的信息[22]。Pathology-GPT的适用场景包括复杂的癌症病理诊断和预后预测,通过提供更全面的癌症诊断信息,帮助医生制定更个性化的治疗方案。然而,多模态模型也面临一些挑战,如对大量多模态数据的需求、模型复杂性和计算资源的高要求,以及多模态数据整合和标准化的难题。此外,模型的解释性较差,也在一定程度上限制了其在临床实践中的应用。表 2系统地梳理了人工智能在肿瘤基因数据分析中的若干典型任务及其相应的AI技术和应用案例。例如,在“基因变异识别”任务中,深度学习技术(如DeepVariant工具)得到了应用;在“致病性预测”方面,深度神经网络(如SpliceAI)展现了其能力;而“多模态数据融合”则依赖多模态深度学习方法,多模态模型(整合影像、基因与临床文本数据)便是一个代表性案例。该表格清晰地展示了AI技术在肿瘤基因分析各个环节的渗透和贡献。
| 表 2 人工智能在肿瘤基因数据分析中的典型任务与案例 |
在肿瘤研究领域,多模态模型的初步探索正成为一个引人注目的前沿方向[54]。这些研究尝试将视觉语言模型与多组学数据进行整合[19, 55],旨在从基因组学、蛋白质组学以及医学影像等多个维度理解肿瘤的复杂性[14],从而更全面地认识肿瘤的发生、发展过程以及对治疗的反应[15, 56]。例如,MULGONET框架便是一个创新的尝试,它通过构建基于基因本体论的层级网络,并结合基于注意力机制的数据融合方法,显著提高了肿瘤复发风险预测的准确性和可解释性[57]。相比传统的单模态模型,MULGONET在肿瘤复发风险预测任务上的准确率提升明显,这一提升主要得益于多模态数据的整合和注意力机制的应用,使得模型能够更全面地捕捉肿瘤的复杂性。MULGONET通过基于基因本体论的层级网络,提供了对预测结果的生物学解释。研究人员可以清晰地看到哪些基因或通路在预测肿瘤复发风险中起到了关键作用,从而有助于深入理解肿瘤的生物学机制。多模态模型通过整合来自不同层面的信息,能够捕获肿瘤更为全面的特征,这为我们理解肿瘤的生物学特性提供了比单一模态数据更为丰富的视角[16, 17]。视觉语言预训练模型在理解和关联不同类型的数据方面具有天然的优势,这使得它们成为整合多组学数据的有力工具。这些初步的探索性研究有望开发出更为精准的肿瘤诊断和预后预测模型,从而推动个性化医疗的发展[20, 23]。AI技术经历了从早期机器学习到深度学习,再到自监督学习等更高级形态的演变。更具体地,AI在肿瘤学中的应用潜力从最初依赖手工特征的机器学习模型,逐步发展到针对特定领域的单用途模型,再到当前备受关注的多模态模型,并最终朝着能够整合海量数据、预测癌症类型、解读放射影像、判断治疗反应亚型等的通用基础模型演进。
从实验室到临床的转化应用,多模态分析和AI技术正从基础研究到实际临床环境的各层面发挥作用。多组学与新型商业模式的结合也开始重塑生物医学研究与产业界的合作模式[58],领先的癌症研究中心也将多模态AI和组学整合作为其临床转化研究的核心战略领域之一[59]。
4.2 大语言模型在生物医学中的应用大语言模型(LLMs)在自动生成基因报告和辅助解读复杂的遗传信息方面也展现出巨大的潜力[12, 38]。例如,GatorTronGPT等是专门为临床应用开发的大语言模型,能够理解和生成医学文本,这有助于从繁琐的基因报告中快速提取关键信息[60]。然而,值得注意的是,LLMs在进行临床推理时,也可能会表现出与人类相似的认知偏差,例如框架效应、首因效应和后见之明偏差[61],这提醒我们在应用这些模型时需要保持谨慎,并确保其整合安全有效[35, 36]。为克服这些挑战,提升LLMs在医疗场景中的应用效果,可采取多项改进策略。首先,通过提示工程优化,设计多样化的提示模板,引导LLMs从不同角度分析问题,减少框架效应的影响。例如,在提供治疗方案建议时,同时展示生存率和死亡率的数据,鼓励模型综合考虑。其次,构建人类-AI协作框架,在医疗决策过程中结合医生的初步诊断和LLMs的分析结果,通过交互式讨论减少首因效应对最终决策的影响。表 3具体列举并解释了这些在临床推理中观察到的大语言模型可能存在的认知偏差。例如,“框架效应”指的是信息的呈现方式会影响决策,如在选择肺癌治疗方案时,强调生存率与强调死亡率可能引导出不同结论;“首因效应”则指最先获得的信息对后续判断产生更大影响;而“后见之明偏差”则体现在事后回顾时,人们倾向于认为事件的发生比实际更具可预测性。理解这些潜在偏差对于负责任地开发和应用LLMs至关重要。尽管如此,大语言模型在自动化基因报告总结过程中的应用减轻了临床医生的工作负担;以及作为辅助解读复杂遗传信息的工具,帮助临床医生更好地理解患者的基因数据。因此,在医学领域应用LLMs时,需要特别关注其可靠性和潜在的偏差[62],以确保其安全有效地服务于患者。
| 表 3 大语言模型在临床推理中观察到的认知偏差 |
医疗大模型和智能体及其衍生的AI助手也正在科研和临床辅助领域发挥着越来越重要的作用[34, 60]。例如,AI助手可以为医生提供实时的临床决策支持,基于患者的基因数据和临床信息,提供循证医学的建议[63]。在科学研究方面,大语言模型可以作为强大的工具,用于生物医学假设的生成[64],从而加速新知识的发现和科研进展。然而,将大语言模型和智能体全面整合到临床实践中,仍然面临着诸多挑战,包括高质量标注数据的缺乏、模型可能存在的偏见,以及在复杂临床场景中推理能力的不足等[34, 62]。尽管如此,医疗大型模型和智能体在为科研人员提供强大的知识挖掘和假设生成工具,以及在临床实践中辅助医生进行诊断、治疗选择和患者管理方面,都展现出巨大的潜力。未来,随着技术的不断进步和相关问题的逐步解决,我们有望看到AI在医疗领域发挥更加广泛和深入的作用[9, 10]。
5 实际应用案例与落地平台在肿瘤基因数据分析领域,国内外都涌现出一些具有代表性的平台和企业,它们正积极探索和实践人工智能技术的应用。例如,Deep Genomics是一家利用人工智能平台进行药物发现的公司,其AI平台涵盖了靶点识别、新型生物机制发现、药物分子设计以及预测分子与靶点相互作用等多个环节[65, 66]。此外,一些研究团队也开发了专门的AI模型,用于分析遗传突变并改进癌症的治疗策略[67]。这些实践案例表明,产业界和学术界都在积极拥抱人工智能,并将其应用于肿瘤研究和个性化治疗的各个方面。
同时,一些平台正在尝试将基因数据与临床数据深度融合,以便在更广泛的临床场景中应用,如基于基因数据的病历生成、临床决策辅助、遗传信息解读以及患者筛查等[33]。例如,AI驱动的临床决策支持系统正在肿瘤和心血管疾病的管理中发挥越来越重要的作用,它们通过整合患者的各项数据,包括基因数据和临床表现,为医生提供基于证据的治疗建议,从而提高医疗决策的质量和效率[63]。这些融合平台和临床试点场景的推进,有助于在真实的医疗环境中验证人工智能技术的有效性和可行性,为未来的临床转化奠定基础。
6 面临挑战与未来展望 6.1 主要挑战尽管人工智能在肿瘤基因数据分析领域取得了显著的进展,但在实际应用中仍然面临着诸多挑战。首先,由于患者的基因数据涉及高度敏感的个人隐私信息,因此数据合规和共享成为人工智能在该领域应用的重要难题[38]。这种由隐私保护需求催生的“数据孤岛”效应,又直接加剧了后续挑战,它限制了模型训练所需的数据规模和多样性,使得开发出泛化能力强的稳健模型变得异常困难。如何在严格遵守数据隐私保护法规的前提下,建立安全可靠的数据共享机制,以促进更广泛的研究合作和进展,是亟待解决的关键问题。
其次,人工智能模型的可靠性以及部分深度学习模型固有的“黑箱”问题,是其在临床应用中的主要障碍[68]。这不仅是技术层面的不透明,更是信任层面的壁垒,因为临床医生无法将性命攸关的决策托付给一个无法解释其推理过程的系统。因此,开发更具可解释性的AI模型[45],并建立完善的评估和验证机制,以确保模型的准确性和可靠性,对于推动AI在医疗领域的应用至关重要。
此外,将人工智能技术真正应用于临床实践还面临诸多落地障碍,包括缺乏统一和完善的验证机制,以及临床医生对AI技术的信任度普遍不高[35, 36, 38]。为了克服这些障碍,需要加强对AI技术的临床验证[56],通过真实世界的数据评估其性能和安全性,并积极开展针对临床医生的培训和推广工作。归根结底,这些挑战并非孤立存在,而是形成了一个恶性循环:数据壁垒导致模型性能受限,模型的不透明性又加剧了临床医生的不信任,从而进一步阻碍了数据的汇集与应用。因此,建立清晰的监管框架和行业标准对于规范和推动AI在医疗领域的健康发展至关重要[34]。
6.2 未来展望展望未来,以大型模型为核心的智能基因分析系统有望实现对肿瘤基因数据更为全面和深入的分析[12],从而为个性化治疗提供更加精准的指导[62]。这些未来的系统将能够整合来自基因组学、转录组学、蛋白质组学等多个层面的数据[16, 18],结合患者的临床信息和丰富的医学知识,实现更智能化的疾病诊断、预后预测以及治疗方案的推荐[10, 22]。大型模型有望成为未来智能基因分析系统的核心驱动力,它们将具备更强大的数据整合、分析和推理能力,从而推动个性化医疗迈向一个全新的高度。
7 结语综上所述,人工智能技术在肿瘤基因数据分析和个性化治疗领域展现出巨大的潜力[9, 34]。目前,AI在基因变异识别、癌症风险预测以及多模态数据整合[19]等方面已经取得了显著的应用进展。然而,我们也必须清醒地认识到,当前AI在肿瘤基因数据分析领域仍然存在一些不足,例如数据隐私的保护、模型可解释性的挑战[45]以及临床落地的实际障碍[35, 38]。
展望未来,以大型模型为核心的智能基因分析系统有望成为驱动肿瘤研究和个性化治疗新范式的关键力量[12, 62]。这些系统将能够更有效地整合多组学数据、自动化提取知识,并为临床医生提供更精准的决策支持。我们有理由相信,随着人工智能技术的不断发展和完善,它将在攻克癌症、实现个性化治疗的道路上发挥越来越重要的作用。
| [1] |
Proietto M, Crippa M, Damiani C, et al. Tumor heterogeneity: preclinical models, emerging technologies, and future applications. Front Oncol, 2023, 13: 1164535. DOI:10.3389/fonc.2023.1164535 |
| [2] |
Dagogo-Jack I, Shaw AT. Tumour heterogeneity and resistance to cancer therapies. Nat Rev Clin Oncol, 2018, 15: 81-94. DOI:10.1038/nrclinonc.2017.166 |
| [3] |
Tomasik B, Garbicz F, Braun M, et al. Heterogeneity in precision oncology. Camb Prism Precis Med, 2023, 2: e2. |
| [4] |
Zhu L, Jiang M, Wang H, et al. A narrative review of tumor heterogeneity and challenges to tumor drug therapy. Ann Transl Med, 2021, 9: 1351. DOI:10.21037/atm-21-1948 |
| [5] |
Sondka Z, Bamford S, Cole CG, et al. The COSMIC Cancer Gene Census: describing genetic dysfunction across all human cancers. Nat Rev Cancer, 2018, 18: 696-705. DOI:10.1038/s41568-018-0060-1 |
| [6] |
Berger MF, Mardis ER. The emerging clinical relevance of genomics in cancer medicine. Nat Rev Clin Oncol, 2018, 15: 353-65. DOI:10.1038/s41571-018-0002-6 |
| [7] |
GalićI, Habijan M, LeventićH, et al. Machine learning empowering personalized medicine: a comprehensive review of medical image analysis methods. Electronics, 2023, 12: 4411. DOI:10.3390/electronics12214411 |
| [8] |
Rezayi S, Niakan Kalhori SR, Saeedi S. Effectiveness of artificial intelligence for personalized medicine in neoplasms: a systematic review. Biomed Res Int, 2022, 2022: 7842566. DOI:10.1155/2022/7842566 |
| [9] |
Hamamoto R, Komatsu M, Yamada M, et al. Current status and future direction of cancer research using artificial intelligence for clinical application. Cancer Sci, 2024, 116: 297-307. |
| [10] |
Sun F, Zhang L, Tong Z. Application progress of artificial intelligence in tumor diagnosis and treatment. Front Artif Intell, 2024, 7: 1487207. |
| [11] |
Suhardi NM, Oktarina A, Bostrom MPG, et al. Database-augmented transformer-based large language models achieve high accuracy in mapping gene-phenotype relationships. bioRxiv, 2025. |
| [12] |
Meng X, Yan X, Zhang K, et al. The application of large language models in medicine: a scoping review. iScience, 2024, 27: 109713. DOI:10.1016/j.isci.2024.109713 |
| [13] |
Clark AJ, Lillard JW Jr. A comprehensive review of bioinformatics tools for genomic biomarker discovery driving precision oncology. Genes (Basel), 2024, 15: 1036. DOI:10.3390/genes15081036 |
| [14] |
Shao Y, Lv X, Ying S, et al. Artificial intelligence-driven precision medicine: multi-omics and spatial multi-omics approaches in diffuse large B-cell lymphoma (DLBCL). Front Biosci (Landmark Ed), 2024, 29: 404. DOI:10.31083/j.fbl2912404 |
| [15] |
Arjmand B, Hamidpour SK, Tayanloo-Beik A, et al. Machine learning: a new prospect in multi-omics data analysis of cancer. Front Genet, 2022, 13: 824451. DOI:10.3389/fgene.2022.824451 |
| [16] |
Menyhárt O, Győrffy B. Multi-omics approaches in cancer research with applications in tumor subtyping, prognosis, and diagnosis. Comput Struct Biotechnol J, 2021, 19: 949-60. DOI:10.1016/j.csbj.2021.01.009 |
| [17] |
Heo YJ, Hwa C, Lee GH, et al. Integrative multi-omics approaches in cancer research: from biological networks to clinical subtypes. Mol Cells, 2021, 44: 433-43. DOI:10.14348/molcells.2021.0042 |
| [18] |
Li L, Sun M, Wang J, et al. Multi-omics based artificial intelligence for cancer research. Adv Cancer Res, 2024, 163: 303-56. |
| [19] |
Waqas A, Tripathi A, Ramachandran RP, et al. Multimodal data integration for oncology in the era of deep neural networks: a review. Front Artif Intell, 2024, 7: 1408843. DOI:10.3389/frai.2024.1408843 |
| [20] |
Yetgin A. Revolutionizing multi-omics analysis with artificial intelligence and data processing. Quant Biol, 2025, 13: e70002. DOI:10.1002/qub2.70002 |
| [21] |
Olsen C. Integrated multi-omic data: powering precision medicine[EB/OL]. (2025-03-10)[2025-07-10]. https://www.appliedclinicaltrialsonline.com/view/multi-omic-data-precision-medicine
|
| [22] |
Ozaki Y, Broughton P, Abdollahi H, et al. Integrating omics data and ai for cancer diagnosis and prognosis. Cancers (Basel), 2024, 16: 2448. DOI:10.3390/cancers16132448 |
| [23] |
Nam Y, Kim J, Jung SH, et al. Harnessing artificial intelligence in multimodal omics data integration: paving the path for the next frontier in precision medicine. Annu Rev Biomed Data Sci, 2024, 7: 225-50. DOI:10.1146/annurev-biodatasci-102523-103801 |
| [24] |
Tomczak K, Czerwińska P, Wiznerowicz M. The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge. Contemp Oncol (Pozn), 2015, 19: A68-77. |
| [25] |
National Cancer Institute. The Cancer Genome Atlas Program (TCGA)[EB/OL]. [2025-09-19]. https://www.cancer.gov/ccg/research/genome-sequencing/tcga
|
| [26] |
AACR Project GENIE Consortium. AACR Project GENIE: powering precision medicine through an International Consortium. Cancer Discov, 2017, 7: 818-31. DOI:10.1158/2159-8290.CD-17-0151 |
| [27] |
American Association for Cancer Research (AACR). AACR Project GENIE: data[EB/OL]. (2025-07-17)[2025-09-19]. https://www.aacr.org/professionals/research/aacr-project-genie/aacr-project-genie-data/
|
| [28] |
cBioPortal for Cancer Genomics[EB/OL]. (2025-05-06)[2025-07-19]. https://www.cbioportal.org/
|
| [29] |
Yi M, Peng C, Xia B, et al. CXCL8 facilitates the survival and paclitaxel-resistance of triple-negative breast cancers. Clin Breast Cancer, 2022, 22: e191-8. DOI:10.1016/j.clbc.2021.06.009 |
| [30] |
National Cancer Institute. GDC Data Portal Homepage[EB/OL]. [2025-05-09]. https://portal.gdc.cancer.gov/
|
| [31] |
Fonseca-Montaño A, Blancas S, Herrera-Montalvo LA, et al. Cancer genomics. Arch Med Res, 2022, 53: 723-31. DOI:10.1016/j.arcmed.2022.11.011 |
| [32] |
Liu J, Cen X, Yi C, et al. Challenges in AI-driven biomedical multimodal data fusion and analysis. Genomics Proteomics Bioinformatics, 2025, 23: qzaf011. DOI:10.1093/gpbjnl/qzaf011 |
| [33] |
O'Connor O, McVeigh TP. Increasing use of artificial intelligence in genomic medicine for cancer care- the promise and potential pitfalls. BJC Rep, 2025, 3: 20. DOI:10.1038/s44276-025-00135-4 |
| [34] |
Lotter W, Hassett MJ, Schultz N, et al. Artificial intelligence in oncology: current landscape, challenges, and future directions. Cancer Discov, 2024, 14: 711-26. DOI:10.1158/2159-8290.CD-23-1199 |
| [35] |
Corti C, Celi LA. Can we ensure a safe and effective integration of language models in oncology?. Lancet Reg Health Eur, 2024, 46: 101081. DOI:10.1016/j.lanepe.2024.101081 |
| [36] |
Verlingue L, Boyer C, Olgiati L, et al. Artificial intelligence in oncology: ensuring safe and effective integration of language models in clinical practice. Lancet Reg Health Eur, 2024, 46: 101064. DOI:10.1016/j.lanepe.2024.101064 |
| [37] |
Sussman L, Garcia-Robledo JE, Ordóñez-Reyes C, et al. Integration of artificial intelligence and precision oncology in Latin America. Front Med Technol, 2022, 4: 1007822. DOI:10.3389/fmedt.2022.1007822 |
| [38] |
Al Kuwaiti A, Nazer K, Al-Reedy A, et al. A review of the role of artificial intelligence in healthcare. J Pers Med, 2023, 13: 951. DOI:10.3390/jpm13060951 |
| [39] |
Sousa RT, Paulheim H. Multi-dataset and transfer learning using gene expression knowledge graphs[C]//Curry E, Acosta M, Poveda-Villalón M, et al. The Semantic Web. Cham: Springer, 2025
|
| [40] |
Zhang XM, Liang L, Liu L, et al. Graph neural networks and their current applications in bioinformatics. Front Genet, 2021, 12: 690049. DOI:10.3389/fgene.2021.690049 |
| [41] |
Zhang Y, Sui X, Pan F, et al. A comprehensive large scale biomedical knowledge graph for AI powered data driven biomedical research. Nat Mach Intell, 2025, 7: 602-14. DOI:10.1038/s42256-025-01014-w |
| [42] |
Beasley JT, Korn DR, Tucker NN, et al. ExEmPLAR (Extracting, Exploring, and Embedding Pathways Leading to Actionable Research): a user-friendly interface for knowledge graph mining. Bioinformatics, 2024, 40: btad779. DOI:10.1093/bioinformatics/btad779 |
| [43] |
Garcia FAO, de Andrade ES, Palmero EI. Insights on variant analysis in silico tools for pathogenicity prediction. Front Genet, 2022, 13: 1010327. DOI:10.3389/fgene.2022.1010327 |
| [44] |
Infante D. AI-powered genomic analysis: revolutionizing the detection of genetic mutations[EB/OL]. (2023-11-08)[2025-05-06]. https://www.news-medical.net/health/AI-Powered-Genomic-Analysis-Revolutionizing-the-Detection-of-Genetic-Mutations.aspx
|
| [45] |
Abe S, Tago S, Yokoyama K, et al. Explainable AI for estimating pathogenicity of genetic variants using large-scale knowledge graphs. Cancers (Basel), 2023, 15: 1118. DOI:10.3390/cancers15041118 |
| [46] |
Conte L, Rizzo E, Grassi T, et al. Artificial intelligence techniques and pedigree charts in oncogenetics: towards an experimental multioutput software system for digitization and risk prediction. Computation, 2024, 12: 47. DOI:10.3390/computation12030047 |
| [47] |
PDQ Cancer Genetics Editorial Board. Genetics of Breast and Gynecologic Cancers (PDQ®): health professional version[EB/OL]. (2025-03-06)[2025-05-06]. https://www.cancer.gov/types/breast/hp/breast-ovarian-genetics-pdq
|
| [48] |
Shams A. Leveraging state-of-the-art AI algorithms in personalized oncology: from transcriptomics to treatment. Diagnostics, 2024, 14: 2174. DOI:10.3390/diagnostics14192174 |
| [49] |
Riaz IB, Harmon S, Chen Z, et al. Applications of artificial intelligence in prostate cancer care: a path to enhanced efficiency and outcomes. Am Soc Clin Oncol Educ Book, 2024, 44: e438516. DOI:10.1200/EDBK_438516 |
| [50] |
Breen J, Allen K, Zucker K, et al. Artificial intelligence in ovarian cancer histopathology: a systematic review. NPJ Precis Oncol, 2023, 7: 83. DOI:10.1038/s41698-023-00432-6 |
| [51] |
Kolla L, Parikh RB. Uses and limitations of artificial intelligence for oncology. Cancer, 2024, 130: 2101-7. DOI:10.1002/cncr.35307 |
| [52] |
Farm H. Predicting: the future of health?[R/OL]. (2024-09-11)[2025-05-06]. https://www.adalovelaceinstitute.org/report/predicting-the-future-of-health/
|
| [53] |
Philip AK, Samuel BA, Bhatia S, et al. Artificial intelligence and precision medicine: a new frontier for the treatment of brain tumors. Life (Basel), 2022, 13: 24. |
| [54] |
Yang H, Yang M, Chen J, et al. Multimodal deep learning approaches for precision oncology: a comprehensive review. Brief Bioinform, 2024, 26: bbae699. DOI:10.1093/bib/bbae699 |
| [55] |
Lee HS, Hong SH, Kim GH, et al. Generative models utilizing padding can efficiently integrate and generate multi-omics data. AI, 2024, 5: 1614-32. DOI:10.3390/ai5030078 |
| [56] |
Akhoundova D, Rubin MA. Clinical application of advanced multi-omics tumor profiling: shaping precision oncology of the future. Cancer Cell, 2022, 40: 920-38. DOI:10.1016/j.ccell.2022.08.011 |
| [57] |
Lan W, Tang Z, Liao H, et al. MULGONET: an interpretable neural network framework to integrate multi-omics data for cancer recurrence prediction and biomarker discovery. Fundam Res, 2025. |
| [58] |
Vitorino R, Ghavami S. Convergence: multi-omics and AI are reshaping the landscape biomedical research. Biochim Biophys Acta Mol Basis Dis, 2026, 1872: 168027. DOI:10.1016/j.bbadis.2025.168027 |
| [59] |
Massachusetts General Hospital Cancer Center. Krantz Family Center for Cancer Research: 2024 annual report[R/OL]. [2025-06-07]. https://www.massgeneral.org/cancer-center/clinical-trials-and-research/center-for-cancer-research
|
| [60] |
Peng C, Yang X, Chen A, et al. A study of generative large language model for medical research and healthcare. NPJ Digit Med, 2023, 6: 210. DOI:10.1038/s41746-023-00958-w |
| [61] |
Mahajan A, Obermeyer Z, Daneshjou R, et al. Cognitive bias in clinical large language models. NPJ Digit Med, 2025, 8: 428. DOI:10.1038/s41746-025-01790-0 |
| [62] |
Thirunavukarasu AJ, Ting DSJ, Elangovan K, et al. Large language models in medicine. Nat Med, 2023, 29: 1930-40. DOI:10.1038/s41591-023-02448-8 |
| [63] |
K PD, Abirami MS. AI Clinical Decision Support System (AI-CDSS) for cardiovascular diseases[C/OL]//2023 International Conference on Computer Science and Emerging Technologies (CSET). IEEE, 2023
|
| [64] |
Qi B, Zhang K, Tian K, et al. Large language models as biomedical hypothesis generators: a comprehensive evaluation. arXiv, 2024. |
| [65] |
Deep Genomics. AI platform[EB/OL]. [2025-04-25]. https://www.deepgenomics.com/AI-Platform/
|
| [66] |
Dawson C. How AI and genomics are personalizing cancer treatment[EB/OL]. (2025-02-11)[2025-05-10]. https://viterbischool.usc.edu/news/2025/02/how-ai-and-genomics-are-personalizing-cancer-treatment/
|
| [67] |
King B. Unlocking the power of AI[EB/OL]. (2024-12-11)[2025-05-10]. https://medicine.iu.edu/magazine/issues/winter-2025/unlocking-the-power-of-ai
|
| [68] |
Prevedello LM, Halabi SS, Shih G, et al. Challenges related to artificial intelligence research in medical imaging and the importance of image analysis competitions. Radiol Artif Intell, 2019, 1: e180031. DOI:10.1148/ryai.2019180031 |
 2025, Vol. 37
2025, Vol. 37 

 雷新刚,西北工业大学机电一体化学士,复旦大学–台湾大学EMBA高级工商管理硕士。现为医疗人工智能与数字健康领域专家,中国生物医学工程学会人工智能分会青年委员,国际项目管理协会高级会员,PMP国际项目管理师(PMI认证)。曾任柯达医疗集团全球研发中心医疗信息化研发总监,拥有二十余年医疗信息系统与人工智能产品研发经验。2012—2020年创立并担任上海米健医疗总裁,主导构建基于人工智能的急危重症信息化体系,其成果入选工业和信息化部新型信息消费示范项目。长期致力于人工智能在医学影像、精准医疗与智能决策支持中的研究与产业化推广,在医疗AI产品创新与临床应用转化方面具有丰富实践经验
雷新刚,西北工业大学机电一体化学士,复旦大学–台湾大学EMBA高级工商管理硕士。现为医疗人工智能与数字健康领域专家,中国生物医学工程学会人工智能分会青年委员,国际项目管理协会高级会员,PMP国际项目管理师(PMI认证)。曾任柯达医疗集团全球研发中心医疗信息化研发总监,拥有二十余年医疗信息系统与人工智能产品研发经验。2012—2020年创立并担任上海米健医疗总裁,主导构建基于人工智能的急危重症信息化体系,其成果入选工业和信息化部新型信息消费示范项目。长期致力于人工智能在医学影像、精准医疗与智能决策支持中的研究与产业化推广,在医疗AI产品创新与临床应用转化方面具有丰富实践经验