(2 南方科技大学工学院, 深圳 518055)
(3 宁波诺丁汉大学理工学院, 宁波 315000)
(2 Faculty of Engineering, Southern University of Science and Technology, Shenzhen 518055, China)
(3 Faculty of Science and Engineering, University of Nottingham Ningbo China, Ningbo 315000, China)
合成生物学自21世纪初兴起以来,已从基因线路设计拓展到基因簇、亚基因组和全基因组的合成与设计[1-3]。该领域引入了工程学设计思想,强调标准化、模块化,遵循设计-构建-测试-学习(Design-Build-Test-Learn, DBTL)原则进行研究,致力于实现生物系统的理性设计和可预测性设计。复杂生物功能的系统性构建面临关键挑战:(1)实验成本及通量限制;(2)自动化平台覆盖率不足;(3)DNA序列设计空间爆炸(4n设计空间)导致元件与模块的理性设计效率低下;(4)批次效应;(5)动态过程数据缺失(由于破坏性采样);(6)预测模块化组件间复杂、非线性的互作关系困难。
高通量测序技术和微流控技术的进步,使得DNA、RNA、蛋白质和代谢组学数据的积累速度呈指数增长,一定程度上缓解了实验通量问题[4, 5]。当前的核心挑战更聚焦于:(1)高精度、可泛化的标准化元件和模块接口序列-功能映射建模;(2)复杂调控逻辑的解析及其在模块化设计中的可靠实现;(3)生成满足特定功能与正交性要求的新型生物元件。在此背景下,近年来基于Transformer架构的大语言模型(large language models, LLMs)等人工智能算法取得显著突破,在挖掘海量生物序列数据中的深层模式、理解序列“语法”与“语义”,以及生成新颖序列方面展现出独特优势,这些能力为解决上述挑战,特别是提升元件设计精度、预测模块兼容性、优化组装策略提供了新的可能。
本综述将重点探讨LLMs如何作为一种强大的计算工具,赋能合成生物学的模块化设计与构建。为厘清其技术渊源并构建统一的分析视野,在本综述中,“LLMs”泛指基于Transformer、Diffusion等新一代架构,在海量生物序列(如DNA、RNA、蛋白质)语料上进行预训练、旨在理解或生成生物序列语言的深度学习模型,其关键在于将生物序列视为一种“语言”,通过学习其中的统计规律来解决合成生物学中的设计难题。文章将系统解析LLMs在基因元件、基因线路、基因簇重构、噬菌体基因组等具体应用场景中的最新进展,着重分析其如何辅助实现高效、可靠的正交化模块组装,并讨论其与传统模型协同应用的潜力及当前面临的技术瓶颈(图 1)。
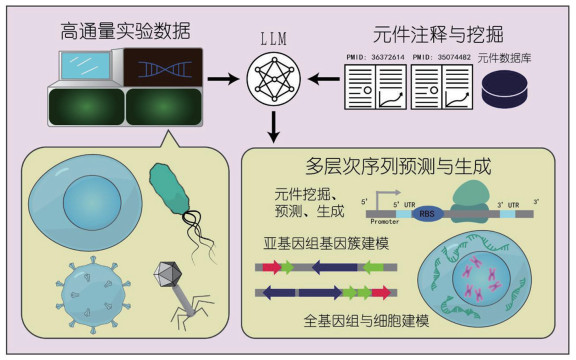
|
左侧展示合成生物学常见的改造对象(细胞、原核细菌、病毒、噬菌体)及对应的高通量实验数据,这些数据与元件注释资源、元件数据库等组合输入LLMs。LLMs作为核心计算引擎,整合多源信息后,输出从元件级的挖掘、预测与生成,亚基因组级的基因簇建模,到全基因组级细胞状态推演的多层次序列预测与生成结果。 图 1 LLMs赋能合成生物学设计的系统性框架 |
标准化、功能可预测的基因元件是构建正交化、模块化生物系统的基础单元,然而其序列-结构-功能的映射关系的高度复杂性,使得传统依赖经验模型或大规模实验筛选的设计方法成本高昂、效率低下,难以满足复杂生物系统高效构建的需求。近年来,基于Transformer架构的LLMs展现出强大的序列模式识别与知识挖掘能力,为加速基因元件的理性设计与标准化提供了新的计算工具支持,其应用主要体现在两个方面:(1)基于多源异构数据的元件特征挖掘与知识图谱构建;(2)面向功能约束的生成式序列设计与优化。
1.1 文献挖掘:基于语言模型的元数据知识图谱构建利用文献与数据库元数据挖掘元件功能关联,LLMs在生物医学领域得到应用,训练了BioBERT[6]、PubMedBERT[7]、BioGPT[8]等生物医学专用模型,显著提升了文本分类、摘要生成、命名实体识别(NER)和关系抽取(RE)等任务的效率,为从海量非结构化文本中自动化提取与标准化元件功能、互作关系及设计规则提供了关键技术支持。这种基于知识的挖掘对于构建元件知识图谱、整合分散的功能信息以支持模块化设计至关重要。同时,受机器翻译思想启发开发的模型,如TALE[9]、TransFun[10]、ProtNLM[11]等将序列功能注释视为序列到自然语言描述的翻译任务,这种方法有效提升了自动化注释的效率和覆盖度。类似地,TransformerGO[12]等模型通过分析基因本体(gene ontology, GO)语义相似性来预测蛋白质-蛋白质相互作用(protein-protein interaction, PPI),展示了从已有注释反向推断蛋白质特性的潜力,有助于理解元件间的兼容性。
在具体应用层面,合成生物学知识系统(synthetic biology knowledge system)[13]利用LLMs构建开放的知识整合平台。它通过自动化工作流挖掘文献与数据库,解析元件的功能、互作关系和设计规则,并利用本体注释关联异构数据源。该系统为研究人员提供统一接口和自然语言查询能力,加速标准化元件库的构建与更新,支撑更可靠的遗传设计工具。此外,SIMPLEX框架首先收集并整合了海量的生物医学文献文本数据,使用微调的LLMs执行知识图谱的抽取,并构建从基因到目标功能的证据链,随后通过扩展性搜索即可高效挖掘非模式微生物序列数据,成功鉴定了多样且高活性的新型加帽酶,大幅提升特定功能元件的发现效率,提供性能更优、潜在正交性更高的标准化新元件[14]。类似地,LLMs辅助标注框架也可自动化、标准化地注释元件功能及其兼容性信息,高效构建模块化设计知识库[15]。
尽管当前LLMs在元件知识挖掘方面展现出潜力,其在合成生物学专用场景的应用仍面临挑战,如领域适配性不足、专业文献动态更新滞后、实体关系推理能力有限等。为此,参数高效微调(如LoRA[16])、提示词工程(Prompt Engineering)[17]与思维链(Chang of Thought, COT)[18, 19]、知识增强架构[20]、智能体(Agent)等技术被积极采用以提升模型专业性。例如,BioRAG[21]通过整合外部搜索引擎与内建数据库系统,包括NCBI摘要及NCBI Gene、dSNP、Genome、Protein数据库标注,在生物医学问答任务中实现了较强的竞争力。类似地,Syn-BioGPT[22, 23]建立了可开放获取的PDF文献库,并训练模型,通过RAG方法搭建了问答平台。Biomni[24]是由LLMs驱动的首个通用型生物医学AI代理系统,通过集成105种软件、59个数据库、150种生物学工具,Biomni可将研究需求转换为可操作的代码,自主设计复杂任务。然而,模型可解释性与数据安全问题仍是制约其在标准化元件设计流程中深度应用的关键瓶颈,当前缺乏标准化解决方案,自动化实验平台的不足同样制约着自动化方案设计的实际应用。
1.2 生成式语言建模:基于语言模型的基因元件设计生成式语言模型正成为辅助基因元件理性设计与功能优化的强大工具。利用大规模平行报告测定方法(massively parallel reporter assay, MPRA)[25]测定活性后,通过海量数据的监督预训练,模型可学习生物序列的深层语法规则,配合任务特异性微调实现功能导向的序列设计与优化。LLMs适配多种生物序列的生成式设计,在启动子设计方面,该领域早期研究主要采用生成对抗网络(Generative Adversarial Networks, GAN)[26]和变分自编码器(Variational AutoEncoder, VAE)[27]等传统生成模型,GAN-promoter[28]框架已成功生成了具有高功能保真度的启动子。随着近年来模型能力的提高,PromoGen在Transformer架构的基础上采用了迁移学习的策略,能够在数据集相对匮乏的条件下,在多个物种中生成质量较高的原核生物启动子[29]。为提高复杂环境下的预测可靠性,Proformer[30]等混合架构模型通过融合Transformer和卷积网络,有效提升了对复杂表达图谱的建模能力。这种混合架构模仿了基因调控的多尺度特征,使得模型能更准确地预测启动子在不同序列环境下的活性,为多基因线路中的元件设计提供更可靠的依据。PromoDiff[31]框架利用在稳定性和可解释性上更有效的Diffusion模型,通过逐步“去噪”的方式,探索启动子序列空间的更广阔区域,从而直接根据用户设定的目标强度,生成大量具有不同序列特征,且表达活性传代稳定的新启动子[32]。LLMs的进步显著推进了启动子设计从经验性的试错转变为可预测的参数化过程,为复杂的代谢通路或基因线路构建提供精细可调的标准化元件。
除经典的启动子外,增强子等其他顺势调控元件的设计也逐渐成为生成式语言建模的新前沿。一些使用经典模型的工作,如Taskiran等[33]将遗传算法与GAN结合,通过模拟体外定向进化过程,揭示了通过破坏抑制子结合位点、创建激活子结合位点以系统性提升增强子强度的普适性规律,并提供了实验证据。在果蝇中,de Almeida等[34]利用大规模的单细胞ATAC-seq数据预训练深度学习模型,并利用迁移学习方法,通过在小规模体内数据上微调,实现了组织特异性增强子的针对性设计。来自清华大学的团队强调了上下文对增强子活性预测的重要性,通过Dense-LSTM建模,证明了通过操纵上下文可以有效微调增强子的活性和细胞特异性[35]。大模型时代后,新的框架,如DNA-Diffusion[36]利用Diffusion架构进一步扩展了顺式调控元件设计的能力。新一代计算框架,如Ledidi[37]可兼容多种评分模型,能够在施加最小突变的前提下,最大化所需的编辑效果。
除了传统的调控元件,LLMs在功能性核酸元件设计领域展现出显著优势,特别是在核酶(ribozyme)和核酸适配体(aptamer)的理性设计方面。通过深度挖掘核酸序列-结构-功能关联特征,模型能够预测复杂RNA分子的二级和三级结构,并基于结构约束生成具有特定功能的优化序列。在人工核酶设计中,模型能够通过学习海量RNA序列和结构数据,生成具有高度稳定性和催化效率的新序列。Angenent-Mari等[38]构建了一个基于卷积神经网络(CNN)的计算框架,用于预测和前向设计基于核酶的基因调控元件。该模型通过学习MPRA实验生成的功能数据,成功捕捉了核酶序列中与基因剪切活性相关的局部基序和组合规则,实现了对数千个核酶变体活性的高精度预测,并可通过优化核酶序列以实现所需的剪切活性,为开发新型基因治疗工具提供了可能。此外,LLMs作为“虚拟SELEX”引擎,通过学习数百万个已知适配体序列及其靶标的关联,能够直接生成针对特定靶标的适配体序列。AR-VAE[39]创新性地整合序列生成与二级结构预测,能够从头设计具有特定功能的RNA适配体,且能确保这些序列能够稳定地折叠成目标结构。与此同时,DeepAptamer[40]模型通过将CNN的局部基序识别能力与递归神经网络(RNN)的长程依赖捕捉能力相结合,在早期阶段就从海量候选序列中精准预测高亲和力适配体,大幅度缩减实验成本。
在翻译调控元件设计上,LLMs的应用同样取得了显著进展。核糖体结合位点(RBS)的调控机制高度依赖于上下文,早期基于统计热力学的RBS Calculator模型,定量分析了起始密码子邻近核苷酸与16S rRNA的结合自由能,支持可调控的RBS设计[41]。结合深度学习方法,Cambray等[42]构建了包含24万条RBS的合成文库,通过大规模文库与回归模型优化大肠杆菌关键翻译位点。Mutalik等[43]则进一步提出标准化转录-翻译起始元件的模块化组合方法,在不同构建背景下实现高一致性的表达调控,并通过贝叶斯建模框架提升表达强度的可预测性。在大模型时代,对于非翻译区(UTR),UTR-LM[44]和UTR-Insight[45]等模型通过结合多层Transformer与卷积网络,有效整合了局部与全局特征,极大地提升了对翻译效率的预测精度和鲁棒性。Tim Lu研究组开发的GEMORNA[46]使用大规模的mRNA表达数据,通过Transformer Decoder建模,同时在翻译效率、半衰期、免疫原性等多个方面进行优化,成功超越了商业基准。
除调控元件外,蛋白质作为生命功能的主要执行者,其设计构成了合成生物学理性设计的另一支柱。蛋白质结构预测领域的突破为后续的功能性蛋白质生成式设计奠定了坚实基础。其代表性方法AlphaFold2[47],利用多序列比对(multi-sequence alignment, MSA)整合进化信息并在Transformer (EvoFormer)中编码,最终通过结构模块精准预测蛋白质的三维结构。此后,研究重心逐渐从结构预测转向功能生成,如今蛋白质设计领域已形成两大核心计算范式,包括以ESM[48-50]系列为代表的掩码语言模型(masked language modeling, MLM)和以ProGen系列为代表的因果语言模型(casual languange modeling, CLM)。按照时间顺序,ESM2[51]率先证明了蛋白质语言模型内部表征可自发涌现出原子级的蛋白质结构信息,实现了无需MSA的快速、高精度3D结构预测;ProGen2[52]随后利用CLM的自回归架构,实现了高效的蛋白质从头生成与零样本预测;ESM3[50]升级为首个统一处理序列、结构和功能的多模态通用生成模型,能够进行序列到结构和结构到序列的双向预测,并通过实验证明了模型能够生成功能完整且与天然蛋白相似度较低的蛋白质序列;ProGen3[53]引入稀疏注意力机制,极大地提高了计算效率并训练了达460亿参数的模型,并通过湿实验验证了模型规模对生成能力的影响。在具体蛋白质设计上,EVOLVEpro[54]在ESM2的基础上设计了主动学习策略,让模型提出下一步实验测试的对象并指导实验,能够以极少的数据点快速收敛,显著加速了蛋白质功能优化的周期。OpenCRISPR[55]系统性挖掘了数百万个CRISPR-Cas蛋白及sgRNA并训练模型,使其深度学习CRISPR系统的序列、结构和功能规律,实现了对高功能性基因编辑器的从头、协同设计。Ivančić等[56]通过生物信息学工作流,搜索、构建了系统发育树并利用domain注释和AlphaFold筛选得到了上万个PiggyBac家族转座酶与对应的转座子元件,随后在ProGen2的基础上进行微调,生成并实验验证了所获得的具有更高活性潜力的新颖转座酶序列。
2 LLMs辅助基因线路与基因簇设计亚基因组尺度(如基因簇、质粒、噬菌体)的系统设计,是连接标准化元件与完整基因组工程的关键桥梁。相比于基因元件,亚基因组尺度的设计对跨宿主、正交性、协同型的要求更高,并需要在设计效率、鲁棒性上保持平衡。传统方法在预测大规模序列互作、优化非编码调控区以及跨尺度功能协调方面面临巨大挑战。基于Transformer架构的LLMs,通过学习海量基因组序列数据,能够挖掘元件组合规律并在保持核心功能模块正交性的前提下,辅助生成或优化非核心序列区域,从而协同推进亚基因组系统的模块化设计与组装预测(图 2)。
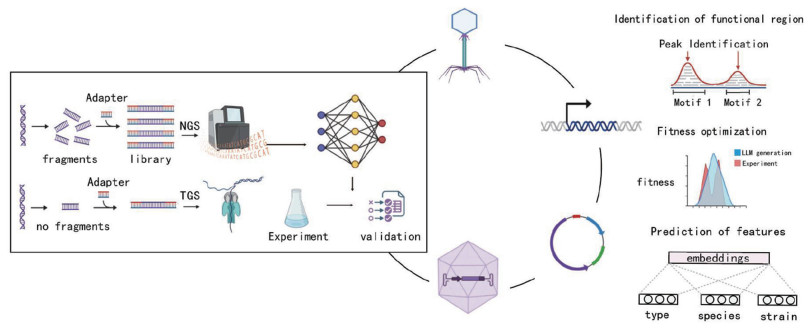
|
左侧展示利用二代测序(NGS)与三代测序(TGS)获取高通量序列功能数据,并据此得到相应的预训练基础模型;随后结合基因线路与基因簇的任务特征进行专门的微调,以支持多类亚基因组系统的设计,如基因线路、噬菌体、质粒和病毒载体等。LLMs作为核心计算引擎整合多源信息,实现功能预测、序列生成与非核心区域的优化设计,从而协同推动亚基因组层面的模块化构建、功能组装与整体性规划。 图 2 LLMs辅助基因线路与基因簇设计系统性框架 |
基因线路作为合成生物学中介于蛋白质与基因组之间的介观尺度功能单元,其理性设计面临着独特的“双重困境”:向上,它缺乏如基因组一般的进化保守性作为设计约束;向下,它又无法像单个蛋白质那样被完全精准地物化预测。当前,该领域正处于从“定性拼接”到“定量设计”的转型期,其核心挑战已从单一元件的性能转向元件间互作的系统性表征与预测。当前的基因线路设计面临的核心瓶颈在于标准生物元件的稀缺性,这直接限制了可设计线路的复杂度与可靠性。虽然MPRA技术部分缓解了元件表征的瓶颈,但基于二代测序(next generation sequencing, NGS)的方法受限于短读长(通常 < 300 bp),难以准确捕获长片段调控元件的互作关系。三代测序(third generation sequencing, TGS)虽具备长读长优势(> 10 kb),但其较高的错误率(普遍高于10%)限制了在定量表征中的应用。以CLASSIC为代表的近期突破性工作表明,通过引入半随机barcode,可以通过TGS鉴定元件组合,并使用模型预测其性能[57]。Gosai等[58]引入一个机器学习驱动的自动化设计平台,利用一个经过大规模多组学数据训练的机器学习模型,来预测并指导顺式调控元件的理性设计。该模型能够学习决定调控元件活性的复杂序列-表观遗传学特征,从而在海量的序列空间中,高效地识别和生成具有特定细胞类型偏好的顺式调控元件。机器学习与机制动力学模型的混合方法[59]以及风险感知的贝叶斯优化策略[60],则进一步提升了线路设计在噪声环境下的稳健性与可解释性。Zhou Jian实验室开发的Puffin框架则通过小型CNN来为复杂设计的CNN模型蒸馏出可解释性[61]。DeepMind的AlphaGenome框架采用受UNet启发的Transformer架构,首次成功克服了上下文长度和序列生成精度的矛盾,得以在1 Mb长度的序列上进行单碱基精度的预测,为线路设计提供更整体性的指导[62]。与此同时,Alpha-Genome在单一模型中统合了包括染色质可及性、组蛋白修饰、转录因子结合等多达11种生物学模态,为解码复杂的人类和小鼠基因组调控编码设定了新的技术标准。
针对介于基因元件与完整基因组之间的生物系统设计需求,LLMs在质粒、噬菌体、合成代谢途径模块化优化与病毒载体工程等亚基因组尺度设计中展现出独特价值[63]。在质粒设计中,PlasmidGPT[64]使用Addgene数据库的质粒序列完成训练,模型生成的序列在指定提示词引导下能够准确适配对应宿主,且相比随机序列有更高概率包含限制酶切位点,便于使用。在噬菌体基因组设计中,megaDNA[65]在未注释的噬菌体基因组上进行了预训练,展示了对必需基因、遗传变异效应、调控元件活性等下游任务的预测能力。在撰稿期间,Brian Hie团队首次利用基因组语言模型从头设计生成噬菌体,并在实验室中合成和验证了噬菌体活性,这项工作确凿地证明了模型的设计能力,是LLMs应用于合成生物学的最强有力的证据之一[66];同时,这项工作也论证了模型的性能与其训练数据的生物学目标特异性密切相关。
在病毒载体设计方面,Wu等[67]结合蛋白质语言模型与传统机器学习,精准预测AAV2衣壳突变体的包装质量,显著提高设计效率。同时,Eid等[68]开发的Fit4Function方法,通过机器学习设计多功能AAV衣壳,实现了跨物种的高效转导性能,其设计候选在多个性能指标上优于AAV9,具有很高的实验应用潜力。这些研究表明,LLMs与其他机器学习工具不仅能够处理核酸/蛋白质序列的“语法”,还能在亚基因组层面捕捉功能模块之间的依赖关系,为质粒构建、合成途径重写和病毒载体设计提供强大计算框架。
3 基因组尺度的建模与系统级设计对基因组尺度进行建模的核心价值在于系统性预测合成生物学设计的整体行为,然而传统方法难以处理高维组合复杂性和多模块耦合,且高度依赖经验参数,限制了其在大型生物系统设计中的应用。基于Transformer的LLMs为这一挑战提供了新工具:通过将DNA序列视为具有语法规则和语义特征的特殊“语言”,LLMs能够从海量生物数据中学习隐含的序列-功能关系,为复杂生物系统的可编程设计提供支撑。基因组DNA序列的超长长度和复杂上下文依赖为建模带来了独特的难点,为了应对这些挑战,早期尝试如Enformer[69]模型通过结合CNN与Transformer,并引入超大感受野设计,实现对超长DNA序列(~2 Mb)的整体语境建模能力,有助于预测大型调控模块在复杂基因组背景下的整合效应。DNABERT[70]利用BERT架构的双向注意力捕捉非编码区的局部与上下文调控信号,为设计具有特定调控功能的遗传模块提供了新的预测工具。为了进一步提升泛化能力,Nucleotide Transformer[71]等模型通过在多物种基因组数据上进行大规模预训练,展现出强大的跨物种功能零样本预测能力,允许模块设计规则从模式生物快速迁移应用于非模式生物,极大地加速了基础研究。HyenaDNA[72]和GENA-LM[73]等创新架构引入了Hyena Layer和稀疏注意力,显著降低了捕捉超长基因组范围依赖关系的计算成本,使基因组尺度下模块间相互作用的全局建模更具可行性。Evo系列模型(Evo[74]及Evo 2.0[75])是基于此前在长序列架构上的探索积累,最终实现的里程碑式基因组基础模型。Evo使用专为长序列优化的StrippedHyena架构,替代了标准的Transformer,以高效处理百万碱基级别的上下文,并在以细菌、古菌、真核病毒/质粒为主的超大型基因组数据中完成训练。Evo2进一步扩展了训练数据集到跨越生命的所有域,并在参数量和性能上有巨大提升,适配从分子到百万碱基对级的预测和生成任务。
上述基因组尺度的建模仍侧重于静态的序列设计。然而,合成生物学的终极愿景之一是构建一个能够模拟整个细胞生命活动的“虚拟细胞”。CRISPR扰动结合单细胞转录组测序[76-78](scRNA-seq)实现了高通量扰动效应检测,为系统解析基因调控网络奠定了基础,推动了scGPT[79]、scFoundation[80]、Geneformer[81]、rbio1[82]、State[83]等微扰驱动的虚拟细胞建模的发展。这些模型的进步预示着大模型对未观测扰动组合全局细胞状态的推演能力,更精确的预测模型将在未来全面降低实验成本,并为模块化、正交化的合成生物学理性设计研究赋能。
4 结论与展望随着高通量测序技术、多组学分析平台及自动化实验系统的协同发展,传统机理模型在实现高度正交化、模块化的生物系统设计时仍有局限。在本综述中,从功能元件挖掘、自动化实验方案设计、基因/蛋白质元件设计,到基因簇及基因组尺度大规模序列设计与生成,LLMs的强大能力已得到充分验证。以Transformer、Diffusion等新一代架构为核心的LLMs凭借其在长程依赖的精确捕获、跨任务知识迁移及渐进式探索连续构象空间等方面的出色能力,为增强传统建模方法提供了新的路径,赋能了从元件到线路、亚基因组乃至全基因组的智能化设计。LLMs的核心潜力在于弥合机理模型与数据驱动方法之间的鸿沟,服务于合成生物学的正交模块化设计目标。此外,通过整合多模态数据(序列、结构、互作、表型),LLMs有望构建更整体化的预测模型,通过捕捉模块间相互作用的关键规律,为模块的选择、组合与优化提供更可靠的预测依据。
为突破单一序列维度的限制,多模态嵌入技术正在整合DNA、RNA、蛋白质乃至自然语言信息,为理解和预测复杂生物系统的模块化行为提供更全面的视角。除前文所述多模态模型外,BioLangFusion[84]框架通过联合学习DNA、mRNA与蛋白质的跨模态表示,实现了序列-结构-功能的协同优化。进一步地,自然语言模态的引入显著增强了模型的生物逻辑推理能力:BIOREASON[85]通过整合自然语言标注,首次实现并在KEGG中验证了从分子到表型的准确逻辑推理;ChatNT则允许研究者直接以自然语言描述设计需求,展示了语言模型作为交互式设计接口在降低模块化设计门槛上的前景。阿里云发布的LucaOne[86]通过联合学习DNA、RNA和蛋白质序列信息,能够捕捉序列-结构-功能之间的关联,实现跨物种的功能预测和模块化优化。该模型在序列生成和功能评估中展示了高效的泛化能力,为模块化生物系统设计提供了计算支持。来自西湖大学团队的ProTrek[87]则结合蛋白质序列、结构与功能的自然语言描述,通过三模态学习实现序列-功能映射和跨模态检索。该方法可用于生成具有特定功能的蛋白质序列,并在高维功能空间中优化模块化设计,为蛋白质工程与系统级设计提供了有效工具。这些进展表明,大语言模型通过整合多源信息和学习复杂规则,为解决合成生物学中模块化系统的整体预测难题提供了新的可能性。当然,充分释放其潜力仍需克服诸多障碍(如高质量数据获取、生成序列的可靠验证等),其核心价值在于作为强大的计算工具,辅助研究者更有效地设计、理解和优化基于正交模块构建的生物系统。
模型可解释性的话题一直以来都备受关注。InterPLM[88]提出一种类似蒸馏的方法,首先将蛋白质语言模型(如ESM2)的嵌入表示输入稀疏自动编码器(sparse autoencoders, SAEs),随后从SAEs中间隐藏层分解出与已知的生物学概念(如结合位点、结构基序、功能域)高度相关的“可解释特征”。类似地,Tomaz da Silva等[89]提出核苷酸依赖性(nucleotide dependencies)的方法,通过量化一个基因组位置上的核苷酸替换如何影响模型预测的其他位置上的核苷酸概率来反映模型内部学习到的功能关联,并通过该方法分析与实验验证了RNA内部的接触碱基。
伴随模型规模与架构创新的持续迭代、多模态数据的深度融合以及可解释性方法对黑箱模型工作机理的拆解,生物大语言模型有望成为真正意义上的“生命编程引擎”。未来,我们将见证LLMs与传统模型的深度协同。这种融合不仅应提升生物设计的整体预测能力,更要在模块接口的兼容性预测、复杂线路行为的涌现特性模拟以及基因组尺度下模块化元件的优化布局方面提供支持。通过跨层级捕捉从单碱基突变到细胞网络动态的全链路信息,这类模型不仅可以实现对多层级调控网络的高精度建模,还具备可解释的因果推理能力,有望推动合成生物学从经验驱动向原理驱动的转变,并通过嵌入自动化实验平台,有望显著缩短设计验证周期,探索更广阔的基因回路、代谢通路及底盘细胞优化空间,从而能够更高效地构建功能可靠、可预测性强且高度模块化的生物系统,为生物制造、医疗等领域提供更强大的工程化底盘解决方案。
| [1] |
Groff-Vindman CS, Trump BD, Cummings CL, et al. The convergence of AI and synthetic biology: the looming deluge. NPJ Biomed Innov, 2025, 2: 20. DOI:10.1038/s44385-025-00021-1 |
| [2] |
Mirchandani I, Khandhediya Y, Chauhan K. Review on advancement of AI in synthetic biology[M]//Mandal S. Artificial Intelligence (AI) in Cell and Genetic Engineering. New York (NY): Springer US, 2025: 483-90
|
| [3] |
García Martín H, Mazurenko S, Zhao H. Special issue on artificial intelligence for synthetic biology. ACS Synth Biol, 2024, 13: 408-10. DOI:10.1021/acssynbio.3c00760 |
| [4] |
Lambert CLG, van Mierlo G, Bues JJ, et al. The evolution of DNA sequencing with microfluidics. Nat Rev Genet, 2025, 26: 1-2. |
| [5] |
Abdelaziz EH, Ismail R, Mabrouk MS, et al. Multi-omics data integration and analysis pipeline for precision medicine: systematic review. Comput Biol Chem, 2024, 113: 108254. DOI:10.1016/j.compbiolchem.2024.108254 |
| [6] |
Lee J, Yoon W, Kim S, et al. BioBERT: a pre-trained biomedical language representation model for biomedical text mining. Bioinformatics, 2020, 36: 1234-40. DOI:10.1093/bioinformatics/btz682 |
| [7] |
Gu Y, Tinn R, Cheng H, et al. Domain-specific language model pretraining for biomedical natural language processing. ACM Transac Comput Healthc, 2022, 3: 1-23. |
| [8] |
Luo R, Sun L, Xia Y, et al. BioGPT: generative pre-trained transformer for biomedical text generation and mining. Briefing Bioinform, 2022, 23: bbac409. DOI:10.1093/bib/bbac409 |
| [9] |
Han T, Fang C, Zhao S, et al. Token-budget-aware LLM reasoning. arXiv, 2025. DOI:10.48550/arXiv.2412.18547 |
| [10] |
Boadu F, Lee A, Cheng J. TransFun: a tool of integrating large language models, transformers, and equivariant graph neural networks to predict protein function. Methods Mol Biol, 2025, 2941: 101-11. |
| [11] |
Gane A, Bileschi ML, Dohan D, et al. ProtNLM: model-based natural language protein annotation[EB/OL]. (2022-10-12)[2025-09-29]. https://storage.googleapis.com/brain-genomics-public/research/proteins/protnlm/uniprot_2022_04/protnlm_preprint_draft.pdf
|
| [12] |
Ieremie I, Ewing RM, Niranjan M. TransformerGO: predicting protein-protein interactions by modelling the attention between sets of gene ontology terms. Bioinformatics, 2022, 38: 2269-77. DOI:10.1093/bioinformatics/btac104 |
| [13] |
Mante J, Hao Y, Jett J, et al. Synthetic biology knowledge system. ACS Synth Biol, 2021, 10: 2276-85. DOI:10.1021/acssynbio.1c00188 |
| [14] |
Wang T, Qin BR, Li S, et al. Discovery of diverse and high-quality mRNA capping enzymes through a language model-enabled platform. Sci Adv, 2025, 11: eadt0402. DOI:10.1126/sciadv.adt0402 |
| [15] |
Schaffer LV, Hu M, Qian G, et al. Multimodal cell maps as a foundation for structural and functional genomics. Nature, 2025, 642: 222-31. DOI:10.1038/s41586-025-08878-3 |
| [16] |
Yang M, Chen J, Zhang Y, et al. Low-rank adaptation for foundation models: a comprehensive review. arXiv, 2024. DOI:10.48550/arXiv.2501.00365 |
| [17] |
Brown TB, Mann B, Ryder N, et al. Language models are few-shot learners. arXiv, 2020. DOI:10.48550/arXiv.2005.14165 |
| [18] |
Wei J, Wang X, Schuurmans D, et al. Chain of Thought prompting elicits reasoning in large language models. arXiv, 2022. DOI:10.48550/arXiv.2201.11903 |
| [19] |
Hu M, Alkhairy S, Lee I, et al. Evaluation of large language models for discovery of gene set function. Nat Methods, 2025, 22: 82-91. DOI:10.1038/s41592-024-02525-x |
| [20] |
Lewis P, Perez E, Piktus A, et al. Retrieval-augmented generation for knowledge-intensive NLP tasks. arXiv, 2020. DOI:10.48550/arXiv.2005.11401 |
| [21] |
Wang C, Long Q, Meng X, et al. BioRAG: a RAG-LLM framework for biological question reasoning. arXiv, 2024. DOI:10.48550/arXiv.2408.01107 |
| [22] |
Li W, Mao Z, Xiao Z, et al. Large language model for knowledge synthesis and AI-enhanced biomanufacturing. Trends Biotechnol, 2025, 43: 1864-75. DOI:10.1016/j.tibtech.2025.02.008 |
| [23] |
Mao Z, Du J, Wang R, et al. SynBioGPT: a retrieval-augmented large language model platform for AI-guided microbial strain development. bioRxiv, 2025. DOI:10.1101/2025.03.23.644789 |
| [24] |
Huang K, Zhang S, Wang H, et al. Biomni: a general-purpose biomedical AI agent. bioRxiv, 2025. DOI:10.1101/2025.05.30.656746 |
| [25] |
Melnikov A, Murugan A, Zhang X, et al. Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat Biotechnol, 2012, 30: 271-7. DOI:10.1038/nbt.2137 |
| [26] |
Goodfellow IJ, Pouget-Abadie J, Mirza M, et al. Generative adversarial nets[C]//Proceedings of the 28th International Conference on Neural Information Processing Systems. Montreal, Canada: MIT Press, 2014: 2672-80
|
| [27] |
Kingma DP, Welling M. Auto-encoding variational Bayes. arXiv, 2022. DOI:10.48550/arXiv.1312.6114 |
| [28] |
Wang Y, Wang H, Wei L, et al. Synthetic promoter design in Escherichia coli based on a deep generative network. Nucleic Acids Res, 2020, 48: 6403-12. DOI:10.1093/nar/gkaa325 |
| [29] |
Xia Y, Du X, Liu B, et al. Species-specific design of artificial promoters by transfer-learning based generative deep-learning model. Nucleic Acids Res, 2024, 52: 6145-57. DOI:10.1093/nar/gkae429 |
| [30] |
Kwak IY, Kim BC, Lee J, et al. Proformer: a hybrid macaron transformer model predicts expression values from promoter sequences. BMC Bioinformatics, 2024, 25: 81. DOI:10.1186/s12859-024-05645-5 |
| [31] |
Cheng Y, Zhang X, Liu F, et al. PromoterDiff: de novo design approach for escherichia coli promoters based on a diffusion model[C]//27th International Conference on Computer Supported Cooperative Work in Design (CSCWD). Tianjin, China, 2024: 2381-7
|
| [32] |
Wang H, Xiang Y, Liu Z, et al. De novo design of insulated cis-regulatory elements based on deep learning-predicted fitness landscape. Nucleic Acids Res, 2025, 53: gkaf611. DOI:10.1093/nar/gkaf611 |
| [33] |
Taskiran Ⅱ, Spanier KI, Dickmänken H, et al. Cell-type-directed design of synthetic enhancers. Nature, 2024, 626: 212-20. DOI:10.1038/s41586-023-06936-2 |
| [34] |
de Almeida BP, Schaub C, Pagani M, et al. Targeted design of synthetic enhancers for selected tissues in the Drosophila embryo. Nature, 2024, 626: 207-11. DOI:10.1038/s41586-023-06905-9 |
| [35] |
Li J, Zhang P, Xi X, et al. Modeling and designing enhancers by introducing and harnessing transcription factor binding units. Nat Comm, 2025, 16: 1469-16. DOI:10.1038/s41467-025-56749-2 |
| [36] |
Lucas Ferreira D, Senan S, Zain Munir P, et al. DNA-Diffusion: leveraging generative models for controlling chromatin accessibility and gene expression via synthetic regulatory elements. bioRxiv, 2024. DOI:10.1101/2024.02.01.578352 |
| [37] |
Schreiber J, Lorbeer FK, Heinzl M, et al. Programmatic design and editing of cis-regulatory elements. bioRxiv, 2025. DOI:10.1101/2025.04.22.650035 |
| [38] |
Schmidt CM, Smolke CD. A convolutional neural network for the prediction and forward design of ribozyme-based gene-control elements. eLife, 2021, 10: e59697. DOI:10.7554/eLife.59697 |
| [39] |
Wong F, He D, Krishnan A, et al. Deep generative design of RNA aptamers using structural predictions. Nat Comput Sci, 2024, 4: 829-9. DOI:10.1038/s43588-024-00720-6 |
| [40] |
Yang X, Chan CH, Yao S, et al. DeepAptamer: advancing high-affinity aptamer discovery with a hybrid deep learning model. Mol Ther Nucleic Acids, 2025, 36: 102436. DOI:10.1016/j.omtn.2024.102436 |
| [41] |
Salis HM. The ribosome binding site calculator. Methods Enzymol, 2011, 498: 19-42. |
| [42] |
Cambray G, Guimaraes JC, Arkin AP. Evaluation of 244, 000 synthetic sequences reveals design principles to optimize translation in Escherichia coli. Nat Biotechnol, 2018, 36: 1005-15. DOI:10.1038/nbt.4238 |
| [43] |
Mutalik VK, Guimaraes JC, Cambray G, et al. Precise and reliable gene expression via standard transcription and translation initiation elements. Nat Methods, 2013, 10: 354-60. DOI:10.1038/nmeth.2404 |
| [44] |
Chu Y, Yu D, Li Y, et al. A 5' UTR language model for decoding untranslated regions of mRNA and function predictions. Nat Mach Intell, 2024, 6: 449-60. DOI:10.1038/s42256-024-00823-9 |
| [45] |
Pan S, Wang H, Zhang H, et al. UTR-Insight: integrating deep learning for efficient 5' UTR discovery and design. BMC Genomics, 2025, 26: 107. DOI:10.1186/s12864-025-11269-7 |
| [46] |
Zhang H, Liu H, Xu Y, et al. Deep generative models design mRNA sequences with enhanced translational capacity and stability. Science, 2025, 390: eadr8470. DOI:10.1126/science.adr8470 |
| [47] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-9. DOI:10.1038/s41586-021-03819-2 |
| [48] |
Rives A, Meier J, Sercu T, et al. Biological structure and function emerge from scaling unsupervised learning to 250 million protein sequences. Proc Natl Acad Sci U S A, 2021, 118: e2016239118. DOI:10.1073/pnas.2016239118 |
| [49] |
Lin Z, Akin H, Rao R, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [50] |
Hayes T, Rao R, Akin H, et al. Simulating 500 million years of evolution with a language model. Science, 2025, 387: 850-8. DOI:10.1126/science.ads0018 |
| [51] |
Lin Z, Akin H, Rao R, et al. Evolutionary-scale prediction of atomic level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [52] |
Nijkamp E, Ruffolo JA, Weinstein EN, et al. ProGen2:exploring the boundaries of protein language models. Cell Syst, 2023, 14: 968-78. e3. DOI:10.1016/j.cels.2023.10.002 |
| [53] |
Bhatnagar A, Jain S, Beazer J, et al. Scaling unlocks broader generation and deeper functional understanding of proteins. bioRxiv, 2025. DOI:10.1101/2025.04.15.649055 |
| [54] |
Jiang K, Yan Z, Di Bernardo M, et al. Rapid protein evolution by few-shot learning with a protein language model. bioRxiv, 2024. DOI:10.1101/2024.07.17.604015 |
| [55] |
Ruffolo JA, Nayfach S, Gallagher J, et al. Design of highly functional genome editors by modeling the universe of CRISPR-Cas sequences. bioRxiv, 2024. DOI:10.1101/2024.04.22.590591 |
| [56] |
Ivančić D, Agudelo A, Lindstrom-Vautrin J, et al. Discovery and protein language model-guided design of hyperactive transposases. Nat Biotechnol, 2025. DOI:10.1038/s41587-025-02816-4 |
| [57] |
O'Connell RW, Rai K, Piepergerdes TC, et al. Ultra-high throughput mapping of genetic design space. bioRxiv, 2023. DOI:10.1101/2023.03.16.532704 |
| [58] |
Gosai SJ, Castro RI, Fuentes N, et al. Machine-guided design of cell-type-targeting cis-regulatory elements. Nature, 2024, 634: 1211-20. DOI:10.1038/s41586-024-08070-z |
| [59] |
Palacios S, Collins JJ, Del Vecchio D. Machine learning for synthetic gene circuit engineering. Curr Opin Biotechnol, 2025, 92: 103263. DOI:10.1016/j.copbio.2025.103263 |
| [60] |
Kobiela M, Oyarzún D, Gutmann M. Risk-averse optimization of genetic circuits under uncertainty. bioRxiv, 2024. DOI:10.1101/2024.11.13.623219 |
| [61] |
Dudnyk K, Cai D, Shi C, et al. Sequence basis of transcription initiation in the human genome. Science, 2024, 384: eadj0116. DOI:10.1126/science.adj0116 |
| [62] |
Avsec Ž, Latysheva N, Cheng J, et al. AlphaGenome: advancing regulatory variant effect prediction with a unified DNA sequence model. bioRxiv, 2025. DOI:10.1101/2025.06.25.661532 |
| [63] |
Ryu G, Kim GB, Yu T, et al. Deep learning for metabolic pathway design. Metab Engin, 2023, 80: 130-41. DOI:10.1016/j.ymben.2023.09.012 |
| [64] |
Shao B. PlasmidGPT: a generative framework for plasmid design and annotation. bioRxiv, 2024. DOI:10.1101/2024.09.30.615762 |
| [65] |
Shao B, Yan J. A long-context language model for deciphering and generating bacteriophage genomes. Nat Commun, 2024, 15: 9392. DOI:10.1038/s41467-024-53759-4 |
| [66] |
King SH, Driscoll CL, Li DB, et al. Generative design of novel bacteriophages with genome language models. bioRxiv, 2025. DOI:10.1101/2025.09.12.675911 |
| [67] |
Wu J, Qiu Y, Lyashenko E, et al. Prediction of adeno-associated virus fitness with a protein language-based machine learning model. Human Gene Ther, 2025, 36: 823-9. DOI:10.1089/hum.2024.227 |
| [68] |
Eid FE, Chen AT, Chan KY, et al. Systematic multi-trait AAV capsid engineering for efficient gene delivery. Nat Commun, 2024, 15: 6602. DOI:10.1038/s41467-024-50555-y |
| [69] |
Avsec Ž, Agarwal V, Visentin D, et al. Effective gene expression prediction from sequence by integrating long-range interactions. Nat Methods, 2021, 18: 1196-203. DOI:10.1038/s41592-021-01252-x |
| [70] |
Ji Y, Zhou Z, Liu H, et al. DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome. Bioinformatics, 2021, 37: 2112-20. DOI:10.1093/bioinformatics/btab083 |
| [71] |
Dalla-Torre H, Gonzalez L, Mendoza-Revilla J, et al. Nucleotide Transformer: building and evaluating robust foundation models for human genomics. Nat Methods, 2025, 22: 287-97. DOI:10.1038/s41592-024-02523-z |
| [72] |
Nguyen E, Poli M, Faizi M, et al. HyenaDNA: long-range genomic sequence modeling at single nucleotide resolution. arXiv, 2023. DOI:10.48550/arXiv.2306.15794 |
| [73] |
Fishman V, Kuratov Y, Shmelev A, et al. GENA-LM: a family of open-source foundational DNA language models for long sequences. Nucleic Acids Res, 2025, 53: gkae1310. DOI:10.1093/nar/gkae1310 |
| [74] |
Nguyen E, Poli M, Durrant MG, et al. Sequence modeling and design from molecular to genome scale with Evo. Science, 2024, 386: ado9336. DOI:10.1126/science.ado9336 |
| [75] |
Brixi G, Durrant MG, Ku J, et al. Genome modeling and design across all domains of life with Evo 2. bioRxiv, 2025. DOI:10.1101/2025.02.18.638918 |
| [76] |
Peidli S, Green TD, Shen C, et al. scPerturb: harmonized single-cell perturbation data. Nat Methods, 2024, 21: 531-40. DOI:10.1038/s41592-023-02144-y |
| [77] |
Zhang J, Ubas AA, de Borja R, et al. Tahoe-100M: a giga-scale single-cell perturbation atlas for context-dependent gene function and cellular modeling. bioRxiv, 2025. DOI:10.1101/2025.02.20.639398 |
| [78] |
Wei Z, Si D, Duan B, et al. PerturBase: a comprehensive database for single-cell perturbation data analysis and visualization. Nucleic Acids Res, 2025, 53: D1099-111. DOI:10.1093/nar/gkae858 |
| [79] |
Cui H, Wang C, Maan H, et al. scGPT: toward building a foundation model for single-cell multi-omics using generative AI. Nat Methods, 2024, 21: 1470-80. DOI:10.1038/s41592-024-02201-0 |
| [80] |
Hao M, Gong J, Zeng X, et al. Large-scale foundation model on single-cell transcriptomics. Nat Methods, 2024, 21: 1481-91. DOI:10.1038/s41592-024-02305-7 |
| [81] |
Theodoris CV, Xiao L, Chopra A, et al. Transfer learning enables predictions in network biology. Nature, 2023, 618: 616-24. DOI:10.1038/s41586-023-06139-9 |
| [82] |
Istrate AM, Milletari F, Castrotorres F, et al. rbio1 - training scientific reasoning LLMs with biological world models as soft verifiers. bioRxiv, 2025. DOI:10.1101/2025.08.18.670981 |
| [83] |
Adduri AK, Gautam D, Bevilacqua B, et al. Predicting cellular responses to perturbation across diverse contexts with State. bioRxiv, 2025. DOI:10.1101/2025.06.26.661135 |
| [84] |
Mollaysa A, Moskale A, Pati P, et al. BioLangFusion: multimodal fusion of DNA, mRNA, and protein language models. arXiv, 2025. DOI:10.48550/arXiv.2506.08936 |
| [85] |
Fallahpour A, Magnuson A, Gupta P, et al. BioReason: incentivizing multimodal biological reasoning within a DNA-LLM model. arXiv, 2025. DOI:10.48550/arXiv.2505.23579 |
| [86] |
He Y, Fang P, Shan Y, et al. LucaOne: generalized biological foundation model with unified nucleic acid and protein language. bioRxiv, 2024. DOI:10.1101/2024.05.10.592927 |
| [87] |
Su J, He Y, You S, et al. A trimodal protein language model enables advanced protein searches. Nat Biotechnol, 2025. DOI:10.1038/s41587-025-02836-0 |
| [88] |
Simon E, Zou J. InterPLM: discovering interpretable features in protein language models via sparse autoencoders. Nat Methods, 2025, 22: 2107-17. DOI:10.1038/s41592-025-02836-7 |
| [89] |
Tomaz da Silva P, Karollus A, Hingerl J, et al. Nucleotide dependency analysis of genomic language models detects functional elements. Nat Genet, 2025, 57: 2589-602. DOI:10.1038/s41588-025-02347-3 |
 2025, Vol. 37
2025, Vol. 37 

 娄春波,博士,中国科学院深圳先进技术研究院研究员。国家高层次青年人才计划入选者,国家基金委优秀青年科学基金获得者,2003年和2009年分别毕业于武汉大学和北京大学,2009年至2013年先后在美国加州大学旧金山分校和麻省理工学院从事博士后研究,2013年加入中国科学院微生物研究所,2019年加入中国科学院深圳先进技术研究院。他长期致力于人工基因线路的理性可预测设计的理论建模与验证实验。主要研究内容可以分为基因线路可预测设计与构建及相关理论模拟,并为了实现精准基因线路的精准预测,开发了一系列绝缘化、正交化和模块化等设计原则。相关结果以第一作者或通讯作者发表在Nature Biotechnology、PNAS、Molecular Systems Biology、Nature Communications、Nucleic Acids Research、ACS Synthetic Biology等杂志上,共50余篇;申请专利10项,包括美国专利3项、国际PCT专利3项
娄春波,博士,中国科学院深圳先进技术研究院研究员。国家高层次青年人才计划入选者,国家基金委优秀青年科学基金获得者,2003年和2009年分别毕业于武汉大学和北京大学,2009年至2013年先后在美国加州大学旧金山分校和麻省理工学院从事博士后研究,2013年加入中国科学院微生物研究所,2019年加入中国科学院深圳先进技术研究院。他长期致力于人工基因线路的理性可预测设计的理论建模与验证实验。主要研究内容可以分为基因线路可预测设计与构建及相关理论模拟,并为了实现精准基因线路的精准预测,开发了一系列绝缘化、正交化和模块化等设计原则。相关结果以第一作者或通讯作者发表在Nature Biotechnology、PNAS、Molecular Systems Biology、Nature Communications、Nucleic Acids Research、ACS Synthetic Biology等杂志上,共50余篇;申请专利10项,包括美国专利3项、国际PCT专利3项