近年来,核酸药物作为一种具有革命性意义的新型治疗手段,在生物医药领域取得了显著进展,展现出广阔的临床应用前景[1]。与传统的小分子药物和抗体药物相比,核酸药物通过直接干预基因表达过程,提供了全新的治疗策略,尤其适用于那些传统方法难以靶向的疾病[2]。特别是在2019年末爆发的新型冠状病毒(COVID-19)疫情期间,信使RNA(mRNA)疫苗实现了快速研发并投入使用,充分展示了核酸技术平台在应对突发公共卫生事件中的高效性和可扩展性,极大推动了全球对核酸药物的重视和投资[1, 3]。截至2023年,美国食品药品监督管理局(FDA)已批准17种以上基于RNA的治疗药物与疫苗,涵盖反义寡核苷酸(antisense oligo-nucleotides, ASOs)、小干扰RNA (small interfering RNA, siRNA)、RNA适配体(Aptamer)以及信使RNA (mRNA)疫苗等多种形式[1]。这些药物在遗传性疾病(如脊髓性肌萎缩症、杜氏肌营养不良症)、代谢性疾病、传染病,乃至部分恶性肿瘤的治疗中表现出良好的疗效和安全性,标志着核酸药物正在从早期研究向临床成熟阶段稳步迈进[2, 3]。
1.2 核酸药物的主要类型及应用现状核酸药物根据其结构和作用机制可分为多种类型,其中研究和应用最为广泛的主要包括mRNA疫苗与治疗剂、小干扰RNA (siRNA)、反义寡核苷酸(ASO)和核酸适配体(Aptamer)。
mRNA疫苗与治疗剂利用了携带特定蛋白质遗传信息的mRNA分子。在细胞内,mRNA被核糖体翻译成蛋白质。mRNA疫苗通过递送编码病原体抗原(如病毒刺突蛋白)的mRNA,诱导人体免疫系统产生针对该抗原的免疫应答,从而预防感染[3]。此外,mRNA技术也正被开发用于蛋白质替代疗法(治疗因基因缺陷导致蛋白质缺乏的疾病)和癌症免疫疗法(编码肿瘤相关抗原或免疫刺激因子)[3, 4]。siRNA是长度约为21~23个核苷酸的双链RNA分子。它利用细胞内源性的RNA干扰(RNAi)机制,通过与目标mRNA特异性结合并引导其降解,从而实现基因表达的沉默[5]。siRNA药物已被成功用于治疗肝脏相关的遗传性疾病,如遗传性转甲状腺素蛋白淀粉样变性(hereditary transthyretin amyloidosis, hATTR),并且在其他疾病领域(如高胆固醇血症、病毒感染)的研发也在积极推进中[2, 5]。ASO是短链(通常15~25个核苷酸)单链合成核酸分子,通常经过化学修饰以提高稳定性与亲和力。ASO通过多种机制调控基因表达,例如与靶标mRNA结合诱导RNase H介导的降解、阻断核糖体翻译或调控mRNA剪接等[1]。ASO药物已在治疗神经肌肉疾病(如脊髓性肌萎缩症)、遗传性代谢性疾病等领域取得成功,是目前获批数量最多的核酸药物类型之一[1, 2]。Aptamer是通过体外筛选技术(如SELEX)得到的短链单链DNA或RNA分子,能够折叠成特定的三维结构,从而高亲和力、高特异性地结合靶标分子(如蛋白质、小分子,甚至细胞)[6]。适配体功能类似于抗体,但具有生产成本低、免疫原性低、组织穿透性好等优点。目前已有适配体药物被批准用于治疗年龄相关性黄斑变性,其在诊断、靶向递送等方面的应用也在探索中[6]。
1.3 AI技术特别是深度学习在药物研发中的潜力概述传统的药物研发过程漫长、耗资巨大且失败率高[7, 8]。人工智能(AI),尤其是其子领域深度学习(deep learning, DL),正以其强大的数据处理和模式识别能力,深刻变革着药物发现与开发的各个环节[1, 9]。深度学习模型模仿人脑神经网络结构,能够从海量、高维、复杂的生物数据(如基因组序列、蛋白质结构、高通量筛选结果)中自动学习隐藏的规律和特征,而无需预先设定规则[9, 10]。在核酸药物设计领域,深度学习展现出巨大潜力,主要体现在多个方面。首先,它可以加速靶点发现与验证,通过分析多组学数据识别新的疾病相关基因或RNA靶点[11]。其次,深度学习能够优化分子设计,在巨大的序列空间中高效搜索具有最优疗效、稳定性及安全性的核酸序列[1, 3]。再次,它有助于预测药物性质,准确预测核酸分子的二级/三级结构、稳定性、免疫原性、脱靶效应及药代动力学特性[1, 12, 13]。最后,深度学习还能改进递送系统,辅助设计和优化用于核酸药物递送的载体(如脂质纳米颗粒LNP),提高递送效率和靶向[14, 15]。通过整合深度学习,研究人员有望显著缩短核酸药物的研发周期,降低成本,提高成功率,并最终为患者带来更有效、更安全的治疗方案[1, 7]。
1.4 核酸药物设计面临的挑战尽管核酸药物前景广阔,但其设计和开发仍面临诸多挑战。序列空间巨大是首要难题,即便是短链核酸,其可能的序列组合也是天文数字。例如,编码一个典型蛋白质(如新冠病毒刺突蛋白)的mRNA,其同义密码子组合产生的潜在序列数量可达10632之巨[3],在如此庞大的空间中找到最优序列无异于大海捞针。其次,设计成本高昂,传统方法依赖实验筛选(如高通量筛选)来探索序列空间,不仅耗时,而且成本高昂,限制了可以测试的序列数量和多样性[1, 6]。脱靶效应也是一个关键问题,核酸药物(特别是siRNA和ASO)可能与非预期的RNA序列发生部分互补结合,导致非特异性基因沉默或功能调节,引发潜在的毒副作用,因此预测和最小化脱靶效应是确保药物安全性的关键[1, 5]。此外,体内稳定性问题不容忽视,核酸分子在体内易被核酸酶降解,半衰期短。虽然化学修饰可以提高稳定性,但如何平衡稳定性与药效、如何预测不同修饰的影响仍然非常复杂[1, 2]。mRNA的稳定性还与其复杂的二级/ 三级结构密切相关,难以精确预测和控制[3]。同时,免疫原性也是一个挑战,外源核酸可能被机体免疫系统识别为“非我”信号,触发固有免疫反应,导致炎症甚至过敏反应,影响药物的安全性和有效性[1]。最后,递送效率是核酸药物成功的关键瓶颈,核酸分子通常带负电荷且分子量较大,难以穿过细胞膜进入细胞质或细胞核发挥作用,因此开发高效、安全、靶向的递送系统至关重要[2, 14]。这些挑战凸显了传统设计方法的局限性,也为人工智能特别是深度学习的应用提供了广阔的舞台。
1.5 本综述的目的与内容安排面对核酸药物设计中的机遇与挑战,本综述旨在系统性地梳理和总结近年来(特别是2020—2025年)人工智能(重点是深度学习)在该领域的应用进展。我们将重点探讨人工智能如何被用于解决核酸药物设计中的核心问题。具体而言,我们将讨论核酸序列的设计与优化,涵盖mRNA、siRNA、ASO及Aptamer等主要类型。接着,我们将关注核酸药物关键特性的预测,包括稳定性、免疫原性、靶向性和表达水平。此外,我们还将探讨核酸药物递送系统的优化,特别是脂质纳米颗粒(LNP)及其他新兴载体。本综述还将介绍应用于该领域的主流深度学习模型及其技术特点,分析当前AI应用面临的挑战与局限性,并展望未来的发展趋势和潜在应用方向。通过整合现有研究成果,本文希望能为从事核酸药物研发及相关计算生物学研究的科研人员提供有价值的参考,并推动AI技术在该领域的进一步发展与应用。
2 深度学习模型、技术特点及核酸药物应用概览深度学习领域包含了多种具有不同架构和优势的神经网络模型。在核酸药物设计中,根据任务需求(如序列分析、结构预测、性质评估),研究人员会选择或组合不同的模型。以下是几类在该领域应用广泛的深度学习模型、其技术特点以及在核酸药物中的典型应用。
卷积神经网络(CNN)以其卷积层为核心,能够通过滑动滤波器(卷积核)自动学习输入数据(如核酸序列的一维表示或结构的二维/三级表示)中的局部模式或基序[16]。池化层则用于降低数据维度并增强模型的平移不变性。CNN特别擅长从序列数据中提取短程的、位置相关的特征。在核酸药物设计中,CNN被广泛应用于预测siRNA抑制效率,例如DeepSipred利用CNN检测决定siRNA抑制潜力的关键序列基序[17]。它也被用于识别功能位点,如预测mRNA剪接位点[5]、DNA/RNA结合蛋白的结合位点[18, 19],以及预测序列-结构稳定性,如NU-ResNet使用CNN处理编码序列和二级结构信息的矩阵[12]。此外,CNN还用于预测免疫原性,例如DeepImmuno-CNN直接从MHC-肽序列信息中预测免疫原性[20],以及预测药物-靶点相互作用,学习化合物-靶点对的模式[21]。
循环神经网络(RNN),包括其变种长短期记忆网络(LSTM)和门控循环单元(GRU),专为处理序列数据而设计。其内部状态(“记忆”)能够捕获序列中的时间(或位置)依赖关系[22],使其适合建模核酸序列中碱基之间的顺序关系和上下文信息。RNN的应用实例包括预测RNA的编码潜力,如mRNARNN使用门控RNN学习长程模式来区分编码和非编码RNA[22]。在密码子优化方面,研究人员利用RNN (特别是LSTM)学习基因表达数据中的密码子使用模式,以预测能增强蛋白质表达的最佳密码子序列[23]。RNN也被用于从头开始药物设计,例如GxRNN基于基因表达谱生成化学结构[24],以及预测药物-靶点相互作用,如DeepLSTM用于整合蛋白质和药物特征进行预测[25]。虽然纯RNN在处理极长序列时可能遇到梯度消失/爆炸问题,但在处理长度适中的核酸片段(如siRNA、适配体)时仍然有效。
Transformer模型的核心是自注意力(self-attention)机制,它允许模型在处理序列中的每个元素时,同时权衡序列中所有其他元素的重要性,从而能够直接捕获长距离依赖关系,且易于并行计算[10, 26]。这使其在处理长核酸序列(如mRNA、基因组区域)方面具有显著优势。Transformer在siRNA设计中有所应用,如OligoFormer利用Transformer编码器捕获siRNA-mRNA相互作用的深层序列特征[27]。在mRNA降解预测方面,RNAdegformer结合使用Transformer和CNN来预测mRNA在核苷酸分辨率上的降解[13]。它也被用于从头开始药物设计,如TransAntivirus用于抗病毒药物设计[28]和drugAI结合Transformer与强化学习生成小分子[29]。此外,Transformer还用于核酸序列分析与分类,如Nucleic Transformer结合自注意力和卷积对DNA序列进行分类[30]。受AlphaFold成功的启发,Transformer也被应用于预测RNA结构或蛋白质-核酸复合物结构[31]。Transformer架构非常适合构建大型预训练语言模型(如DNA-BERT[32]、RNA-FM[33]),这些模型在海量生物序列数据上预训练后,可以迁移到下游任务,提高性能并减少对特定任务数据的需求[34]。
图神经网络(GNN)专门设计用于处理图结构数据,能够学习节点(如原子、碱基、基因)的特征及其之间的连接关系(如化学键、碱基配对、相互作用)[35]。GNN通过聚合邻居节点的信息来更新节点表示,可以捕捉复杂的拓扑结构和关系信息。在ASO化学修饰优化中,ASOptimizer使用边缘增强图Transformer (一种GNN)将ASO分子(包含化学修饰)表示为图,学习结构-活性/毒性关系[36]。在siRNA效力预测方面,siRNADiscovery将siRNA和mRNA序列及其拓扑结构建模为图,以预测抑制效率[37]。GNN也用于RNA-配体结合预测,如GerNA-Bind使用GNN对RNA二级结构和配体分子图进行编码[38]。此外,GNN还被广泛用于预测药物-靶点相互作用,将药物和蛋白质靶标之间的关系建模为图进行预测[39],以及预测小分子(包括潜在的递送载体组分)的各种理化和生物活性[40]。
实践中,研究人员常常组合使用不同类型的深度学习架构以发挥各自优势。例如,RNAdegformer结合了CNN (捕捉局部特征)和Transformer (捕捉全局依赖)[13],而RiboDecode则结合了CNN和注意力机制[41]。图Transformer (如ASOptimizer中的EGT)则融合了GNN和Transformer的思想。除了上述主要用于预测或分类的判别模型外,生成模型(如生成对抗网络GAN、变分自编码器VAE、扩散模型)在核酸药物设计中也越来越重要。它们旨在学习数据的分布并生成新的、具有期望性质的序列或分子结构。例如,AptaDiff使用扩散模型进行适配体的从头开始设计[42],而drugAI等模型使用Transformer结合强化学习(reinforcement learning, RL)生成新的药物分子[29]。生成模型为探索广阔的化学和序列空间、发现全新设计提供了强大的工具[43]。表 1对深度学习模型在核酸药物设计中的原理、优势和应用进行了总结。
| 表 1 深度学习模型在核酸药物设计中的原理、优势和应用概览 |
人工智能,特别是深度学习技术,正在全面重塑核酸药物的序列设计流程。其应用可系统性地归纳为三个核心领域:序列与化学设计、关键性质预测和递送系统优化(图 1)。这三个领域相互关联,共同构成了AI驱动的核酸药物研发完整流程。其中,序列设计是整个流程的起点和基础,对药物活性、稳定性、翻译效率以及脱靶风险具有决定性影响。本节将重点介绍人工智能在核酸药物序列设计中的关键应用及其技术路线。
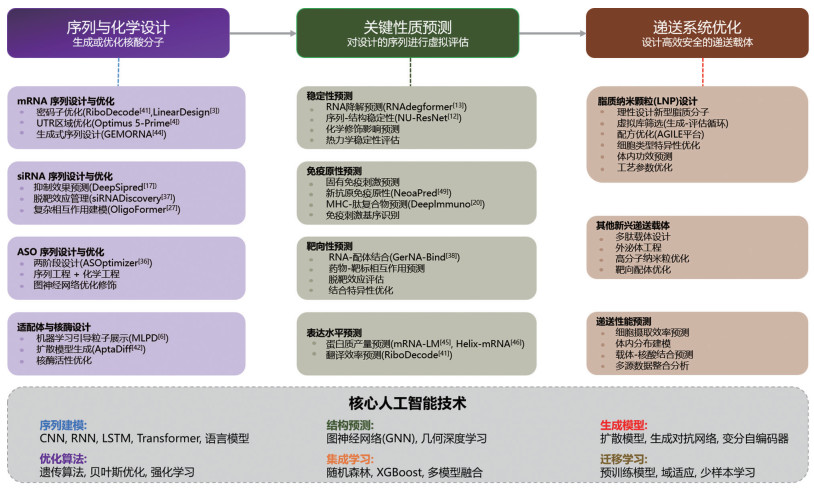
|
本图展示了人工智能技术在核酸药物开发全流程中的三大核心应用领域:序列与化学设计、关键性质预测以及递送系统优化。每个领域包含具体的应用场景和相关的人工智能模型技术。底部展示了支撑这些应用的核心人工智能技术架构。 图 1 人工智能技术在核酸药物研发中的系统性应用框架 |
深度学习凭借强大的序列建模能力,能够从大规模生物数据中自动学习复杂序列模式,为多类型核酸药物构建高效、可控且具备良好药理特性的序列方案。图 2展示了从输入数据到实验验证的完整AI序列设计工作流程,包括六个关键阶段:(1)输入数据准备与预处理,包括核酸序列、实验数据、结构信息与理化性质的收集和标准化;(2)序列编码层,通过one-hot、k-mer或预训练语言模型(如RNA-FM[33])将序列转化为数值表示,并可加入多模态信息;(3)特征提取层,根据任务进行模型选择,如CNN (捕获局部基序)、RNN/LSTM/GRU (建模顺序依赖)、Transformer (处理长程依赖)或GNN (建模拓扑结构);(4)预测/生成层,包括性质预测的判别模型与用于从头生成序列的生成模型(VAE、扩散模型、强化学习等);(5)输出与解释,包括序列评分、性质预测以及注意力可视化或SHAP值等可解释性分析;(6)实验验证,通过体外/体内实验检验模型预测的真实性与适用性。流程底部的反馈循环体现了现代AI药物研发的核心理念:实验数据不断反哺模型,通过主动学习实现预测能力的迭代提升。基于这一流程,以下各小节将介绍人工智能在不同类型核酸药物序列设计中的典型应用案例,展示其在实际研发体系中的作用。

|
流程包括数据准备、序列编码、特征提取、预测/生成、结果输出与解释,以及实验验证。底部反馈环展示了实验数据用于反哺模型、实现持续迭代优化的闭环机制。 图 2 深度学习模型在核酸药物设计中的典型工作流程 |
mRNA分子的序列设计对其作为疫苗或治疗药物的最终效力至关重要,影响着蛋白质表达水平(即翻译效率)、分子稳定性、半衰期以及免疫原性等多个关键环节[41]。优化mRNA序列主要涉及编码区(coding sequence, CDS)和非翻译区(untranslated regions, UTRs)的设计,这两者都直接关系到mRNA翻译成蛋白质的效率。
CDS区域的密码子优化是提升mRNA表达效率(即翻译效率)的常用手段,即在不改变编码氨基酸序列的前提下,选择更“偏好”的同义密码子。这种偏好性可能与tRNA丰度、mRNA二级结构(尤其是在起始密码子附近)、翻译速度等多种因素相关,并且可能存在组织或细胞类型的特异性[41]。传统的密码子优化通常基于密码子使用频率表,但这种方法可能过于简化,未能捕捉到影响翻译效率的复杂序列上下文依赖性。LinearDesign算法[3]虽然不完全是标准的深度学习模型,但其利用计算语言学中的格点解析思想,能够在极短时间内(如11分钟找到新冠刺突蛋白mRNA的最优设计)同时优化mRNA的二级结构稳定性和密码子使用,旨在提升整体表达水平。实验证明,LinearDesign设计的mRNA疫苗相比传统密码子优化的序列,具有更长的半衰期、更高的蛋白质表达量(表明翻译效率提升),并在小鼠中诱导了显著更高(最多128倍)的抗体滴度[3]。
深度学习模型,如基于CNN的工具,可以通过学习不同组织或细胞类型的大规模基因表达数据,揭示更精细的、细胞类型依赖性的密码子偏好规律,预测能够最大化蛋白质表达(即最大化翻译效率)的最佳密码子使用策略[23]。例如,Ribo-Decode框架利用深度学习模型直接从核糖体谱数据中学习翻译效率的关键决定因素,并结合最小自由能(minimum free energy, MFE)预测模型来评估mRNA稳定性,通过上下文感知优化生成具有更高蛋白质表达潜力的密码子序列[41]。近期开发的GEMORNA等生成式模型,通过学习天然mRNA序列的分布特征,能够从头生成具有优化特性的全长mRNA序列。该模型结合变分自编码器(variational autoencoder, VAE)架构和强化学习策略,在保持序列可翻译性的同时优化多个目标(如表达量、稳定性、免疫原性),在序列设计方面展现出创新潜力[44]。与传统的基于规则或频率表的方法相比,GEMORNA能够在更广阔的序列空间中探索,发现兼顾多种性能指标的新颖序列组合。这些例子说明,AI/DL通过综合考虑序列、结构和细胞环境因素,能够更有效地优化密码子选择,从而显著提高mRNA的翻译效率。
对于UTR优化,5'UTR和3'UTR在mRNA的稳定性、亚细胞定位和翻译起始/调控中扮演关键角色,对翻译效率有直接影响[45]。优化UTR序列是提高mRNA药物性能,特别是翻译效率的另一个重要策略。Optimus 5-Prime模型是一个利用CNN分析大规模(28万条) 5'UTR序列数据的工具,能够准确预测5'UTR对翻译效率的影响(相关系数达0.93),并结合遗传算法或基于梯度的Fast SeqProp方法来设计具有特定表达水平(即特定翻译效率)的优化5'UTR序列[4]。在此基础上,近期提出的Helix-mRNA模型通过将UTR、编码区和多种调控元件纳入统一的全序列建模框架,实现了对mRNA全序列功能的联合建模与优化,为UTR序列在整体mRNA治疗分子设计中的系统性优化提供了新的思路和技术路径[46]。
3.2 siRNA序列设计与优化siRNA通过RNAi机制沉默目标基因,其设计的核心在于确保高效的抑制活性(efficacy)和高度的特异性,同时最大限度地减少脱靶效应(off-target effects)。深度学习模型正被用于解决siRNA有效性预测与脱靶效应管理的挑战。尽管RNAi通路已被广泛研究,但预测哪些siRNA序列能够高效地抑制目标基因仍然是一个挑战[17]。目前主流的siRNA设计模型包括基于CNN的DeepSipred[17]、基于GNN的siRNADiscovery[37]以及基于Transformer的Oligo-Former[27],它们分别采用不同的技术路线来预测siRNA的抑制效率和特异性。除了预测有效性,机器学习(包括深度学习)也被用于预测和管理siRNA的脱靶效应。例如,通过分析siRNA子区域的热力学性质或利用GNN建模siRNA与全基因组转录本的相互作用网络,可以评估潜在的非特异性结合风险,从而指导设计更安全的siRNA[5, 47]。为了更系统地比较这些主流siRNA设计模型的优劣,我们在表 2中总结了它们的核心技术、主要优势以及局限性。
| 表 2 主流siRNA设计模型的系统对比 |
基于表 2的对比分析,三种主流siRNA设计模型各有特色,适用于不同的应用场景。Deep-Sipred的优势在于通过CNN卷积核直接学习关键抑制基序(如seed region的碱基偏好、位置依赖的序列模式),其预测结果具有相对较强的可解释性,研究人员可以通过分析卷积核的权重来理解模型关注的序列特征[17]。然而,该模型需要人工设计热力学等特征(如自由能、GC含量等),这种特征工程过程依赖于领域专家知识。当应用于新的物种或细胞类型时,可能需要重新设计和筛选特征,限制了其通用性。siRNADiscovery的创新在于将siRNA和靶mRNA的相互作用建模为图网络,能够同时捕获序列信息、热力学性质(如结合自由能)以及siRNA-mRNA配对后形成的拓扑结构信息[37]。这种多层次信息的协同建模使其在处理复杂靶标(如长mRNA、存在多个剪接亚型的基因)时表现更优,因为图结构天然适合表示分子间的复杂相互作用关系。但是,图的构建和GNN的训练计算成本较高,且模型需要准确的RNA二级结构预测作为输入,而结构预测本身的不确定性可能会影响最终性能。OligoFormer的强项是利用预训练RNA语言模型(RNA-FM)的强大序列表示能力。通过在海量RNA序列上的无监督预训练,RNA-FM已经学习到了丰富的序列-功能关系知识[33]。OligoFormer在此基础上进行微调,无需显式的RNA二级结构预测即可捕获序列的深层语义特征和长程依赖关系,这使其在处理长siRNA或复杂序列上下文时具有优势[27]。此外,在小样本场景下(例如针对新靶标仅有少量实验数据时),OligoFormer可以通过迁移学习仍然保持较好的预测性能。但是,Transformer架构的“黑箱”特性使研究人员难以理解模型的决策依据,也难以从中提取可直接用于实验设计的生物学规则,这在一定程度上限制了其在需要机制解释的场景中的应用。在选择siRNA设计工具时,应根据具体需求进行权衡。对于数据充足且需要较强可解释性的项目(例如需要向监管机构解释设计原理),DeepSipred是稳健的选择;在处理结构复杂或具有多种相互作用模式的靶标时,siRNADiscovery的图网络建模能力更具优势;而在数据受限、需要快速迭代或希望利用大规模预训练知识的场景下,OligoFormer的迁移学习能力更具实用价值。未来的研究方向可能是将这些方法的优势结合起来,例如开发可解释的Transformer模型或将图网络与预训练语言模型相融合。
3.3 ASO序列设计与优化ASO的设计需要精确选择与目标RNA结合的位点,并优化序列以获得高亲和力和特异性,同时常需要进行化学修饰以提高稳定性、降低毒性并改善药代动力学。人工设计最优ASO序列费时费力[1],因此深度学习平台被开发出来以加速这一过程,重点关注ASO序列的特异性和结合效率优化策略。ASOptimizer是一个典型的例子,它是一个两阶段的深度学习框架[36]。第一阶段是序列工程,利用机器学习模型(如线性因子模型)分析大规模实验数据,学习序列特征(如结合热力学、二级结构)与ASO效力之间的关系,预测候选ASO序列对目标mRNA的抑制效果,从而筛选出潜在的高效靶位点和序列[36]。第二阶段是化学工程,利用先进的深度图神经网络架构,如边缘增强图Transformer (edge-augmented graph transformer, EGT),学习不同化学修饰组合对ASO性能(活性、毒性)的影响。模型将ASO的序列和化学修饰信息表示为图结构,通过学习已知修饰模式,优化新ASO序列的化学修饰方案,以进一步提升活性并降低风险[36]。
相较于此,传统ASO设计主要依赖研究人员根据经验采用启发式规则(如适度的GC含量、避免复杂二级结构区域、排除重复序列)挑选有限数量的候选靶位点;随后结合常用的、文献中已验证的化学修饰(如全硫代磷酸酯骨架、局部2′-O-甲基修饰等)进行小规模实验筛选。这种方法虽然具有直观、可解释性强等优点,但其根本局限在于:(1)搜索范围受限,仅能测试数个至数十个序列,极易错过最优解;(2)实验成本高且迭代周期长;(3)化学修饰组合空间过大(例如20-mer在仅含5种修饰类型的条件下理论组合超过1014),人工难以系统探索;(4)难以整合已有大规模ASO实验数据和结构信息[1]。因此,传统流程在高通量筛选能力和化学空间探索能力上均显不足。
基于此,多阶段深度学习方法(如ASOptimizer)在多个方面展示了相较传统经验流程的系统性优势[36]。其序列工程模块能够在全转录本范围内进行高通量预测式筛查,从“盲目试错”转向“定量预测驱动”,显著提高初筛命中率;而化学工程模块通过图神经网络整合位置效应、协同修饰效应及毒性风险等因素,避免了传统方法中对固定修饰模式的依赖,使得修饰优化从经验式选择跃迁为可计算探索。在IDO1基因的实验验证中,ASOptimizer输出的ASO在体外对目标mRNA表达的抑制效率显著优于传统经验设计(提升幅度达40%~60%),并且毒性与脱靶效应更低[36],进一步证明了这种多阶段优化策略的应用价值。
尽管如此,ASOptimizer仍存在一定局限性,主要包括:训练数据主要覆盖常用修饰类型,对新兴非经典修饰(如PMO、tricyclo-DNA、2′-F等)的预测能力仍需验证;体内药代与组织分布等复杂因素尚未系统整合,导致体外到体内转化的预测能力有限;图神经网络的可解释性不足,使研究人员难以直接从模型中提炼机制性设计规则;模型的跨基因泛化能力也需要更广泛的数据验证[36]。总体而言,以ASOptimizer为代表的多阶段深度学习框架在序列与化学修饰双维度的系统优化方面展现出传统经验流程无法比拟的能力,是ASO设计从经验驱动走向理性设计的重要方向,但其进一步成熟仍依赖于更大规模的实验数据积累与可解释性增强技术的发展。
3.4 核酸适配体与核酶设计适配体和核酶是具有特定结构和功能的RNA或DNA分子,其设计也受益于深度学习。在核酸适配体设计与优化方面,目标是找到能高亲和力、高特异性结合靶标的序列。传统SELEX方法效率有限,且难以覆盖巨大的理论序列空间[6]。机器学习,特别是深度学习,被引入以指导适配体的发现和优化。机器学习引导的粒子展示(machine learning guided particle display, MLPD)方法是一个代表性实例[6]。该方法首先通过高通量实验(粒子展示)获取初始文库中大量序列与其靶标(如NGAL蛋白)的相对结合亲和力数据。然后,利用这些数据训练深度神经网络模型(如全连接网络和CNN)来学习序列-亲和力关系。训练好的模型随后被用于“计算进化”:一方面,模型可以预测对已有高亲和力序列进行智能突变后的效果;另一方面,模型可以直接生成全新的序列并预测其亲和力。通过这种模型预测与实验验证相结合的迭代优化循环,仅需评估相对较少(约18.7万)的序列,就能发现比初始文库中最优序列亲和力更高的新适配体。值得注意的是,该方法还能指导序列截短,得到更短但结合力相当或更优的适配体,提高了临床应用潜力[6]。除了判别模型,生成模型也被用于适配体的从头开始设计。例如,AptaDiff框架利用扩散模型(diffusion model)在离散的适配体序列空间中生成具有高亲和力和新颖性的新序列[42]。
进一步来看,当前适配体设计中常见的生成模型包括扩散模型、变分自编码器(VAE)和生成对抗网络(generative adversarial network, GAN),它们各具优势与局限。以AptaDiff为代表的扩散模型通过逐步去噪的方式生成序列,具有生成多样性高、可控性强和训练稳定性好的特点,因此能够有效探索更加广阔的序列空间。例如,AptaDiff生成序列的新颖性比例可达到68%左右[42]。不过,由于生成过程需经历多步迭代,其计算成本较高,且对预测结构稳定性的信心相对有限,可能影响部分序列的折叠正确性。相比之下,VAE通过构建连续潜在空间来生成序列,更容易保持与训练数据一致的统计规律,其结构预测置信度较高(约0.87),生成速度快,也便于在潜在空间进行插值探索[48]。然而,VAE生成序列的多样性不足,新颖性仅约45%,可能限制模型发现完全新型序列的能力。GAN及其变体(如cGAN)亦被应用于适配体序列生成[48],但由于其训练不稳定、容易出现模式崩溃,且难以处理离散类型数据,因此目前更适合用于辅助任务而非直接承担从头生成的核心角色。相比纯生成式方法,MLPD将深度学习预测与高通量实验筛选结合起来,其优势在于能够依靠真实实验反馈不断提升模型精度,同时以远低于SELEX的规模筛选出高亲和力序列[6],因此具有极高的实际应用价值。
尽管深度学习为适配体设计带来显著进展,但仍面临若干核心挑战。例如,适配体功能依赖其精细的三维结构,但目前RNA/DNA结构预测的准确性仍明显落后于蛋白质。尽管AlphaFold 3在蛋白质结构预测中可达到TM-score > 0.9,但对RNA的预测TM-score仅为0.6~0.7[49],这一偏差会直接影响基于结构的设计与虚拟筛选。另一方面,不同靶标的结合机制差异显著,而现有数据库仍然集中于少数靶标,使得深度模型在面对结构未知的新靶标时缺乏足够的训练数据[6]。此外,高亲和力结构往往复杂,可能包含稳定性较差的区域,与体内半衰期需求相冲突;虽然化学修饰能够提升稳定性,却可能反向影响折叠与靶标识别。如何通过深度学习在多目标之间取得平衡,例如同时优化亲和力与稳定性,仍是需要重点攻克的问题。未来研究可进一步发展多目标优化算法、构建可预测修饰效果的模型,或探索更稳定的适配体骨架。总体而言,随着大规模实验数据的累积、核酸结构预测技术的突破以及生成模型不断成熟,AI有望将适配体开发周期从数月压缩至数周,并推动性能超越传统SELEX的新型适配体分子的诞生。
相比于适配体,核酶的设计主要聚焦于催化活性的优化,其序列-功能关系通常更加复杂。为了高效探索核酶在序列空间中的可行变体,深度学习同样被引入核酶进化策略中。例如,Rotrattanadumrong等[50]将深度学习嵌入进化算法,用于研究RNA连接酶核酶的“中性突变网络”。在该研究中,深度神经网络首先被训练用于预测核酶序列的催化活性,随后进化算法依赖模型的预测结果,引导搜索那些在保持活性前提下具有不同序列的“中性”变体。通过深度模型与进化策略的结合,研究者能够在高维序列空间中高效识别功能等效但序列多样的核酶,为理解核酶的鲁棒性和可进化性提供了重要证据[50]。这一思路展示了深度学习在解析复杂序列-功能景观方面的潜力,也为未来核酶的定向设计与优化提供了可扩展的技术路径。
4 人工智能在核酸药物特性预测中的应用除了直接设计序列,人工智能(特别是深度学习)在预测核酸药物的关键理化和生物学特性方面也发挥着重要作用,这些预测结果可以反馈到设计环节,指导序列的进一步优化。
4.1 稳定性预测核酸序列的稳定性对药效起着关键作用。核酸药物在体内的稳定性直接影响其作用时间和最终疗效,RNA分子尤其容易被内源性核酸酶降解[1]。因此,提高分子的稳定性是核酸药物设计中的一个核心目标,这不仅关系到药物在体内的半衰期,也影响其储存和运输条件[1, 3]。
人工智能技术,尤其是深度学习模型,为预测序列稳定性提供了更精细和可扩展的工具。首先是在预测RNA降解方面,mRNA的降解速率与其序列和结构密切相关。RNAdegformer模型结合了卷积(CNN)和Transformer中的自注意力机制(self-attention),能够捕获mRNA序列中的局部和全局依赖性,以核苷酸级别的分辨率精确预测RNA的降解速率。这类预测有助于在设计早期就识别和修饰不稳定的序列区域,从而生产更稳定的mRNA疫苗和治疗药物[13]。其次是预测序列-结构稳定性,RNA的二级和三级结构对其稳定性至关重要。NU-ResNet和NUMO-ResNet是基于CNN (ResNet架构)的深度学习模型,它们将RNA序列和预测的二级结构信息编码为3D矩阵,用于评估特定RNA序列形成稳定结构的可能性,这间接反映了其热力学稳定性[12]。最后,深度学习还用于预测化学修饰对稳定性的影响。在ASO和siRNA中,引入化学修饰(如硫代磷酸酯骨架、2'-O-甲基修饰等)是提高核酸酶抗性的常用策略。深度学习模型(如ASOptimizer中使用的图神经网络)可以学习不同化学修饰组合对分子稳定性和活性的影响,从而推荐最优的修饰方案[36]。此外,一些预测siRNA有效性的模型也会将热力学稳定性作为输入特征或预测目标之一[17]。
4.2 免疫原性预测控制核酸药物的免疫原性风险至关重要。外源核酸分子,特别是未经修饰的RNA或含有特定基序(如CpG)的DNA,可能被细胞内的模式识别受体(PRRs,如TLR7/8、RIG-I、MDA5)识别,触发固有免疫反应,导致细胞因子释放和炎症[1]。虽然在疫苗设计中有时需要适度的免疫刺激,但在治疗性应用中,过度或非预期的免疫原性通常是有害的,可能导致副作用、降低疗效甚至危及患者安全。因此,预测和控制免疫原性风险是核酸药物设计中的关键一环。
人工智能技术,尤其是深度学习模型,在核酸药物免疫原性预测领域也展现出显著潜力。一方面,模型可以用于预测序列的固有免疫刺激性。通过学习已知具有高或低免疫刺激活性的核酸序列特征,基于RNN或Transformer的模型可以扫描候选序列,识别是否存在已知的免疫刺激基序(如富含UG的序列),并评估其激活PRRs的可能性。这种预测可以指导通过同义突变或化学修饰来移除或掩盖这些基序[1]。另一方面,在癌症疫苗等应用中,需要预测编码产物的免疫原性(如新抗原),即由mRNA编码的多肽(特别是源自体细胞突变的新抗原)能否有效激活T细胞免疫应答。NeoaPred是一个深度学习框架,它通过预测肽-HLA复合物的结构,并整合表面和结构特征来计算指示免疫原性的“异物评分”[51]。DeepImmuno-CNN则直接使用CNN预测MHC-肽复合物的免疫原性,而非仅仅预测结合亲和力,旨在更直接地评估免疫激活潜力[20]。此外,机器学习算法如随机森林、多层感知器(MLP)和XGBoost也被用于构建预测病毒来源保护性免疫原的模型[52]。这些模型有助于筛选最有可能诱导有效免疫反应的抗原序列,为疫苗设计提供指导。这类新抗原预测工具通常需要准确预测肽段与HLA分子的结合亲和力,基于此目的已有大量生物信息学工具被开发[53],但这些工具的性能和适用范围仍在不断优化中。此外,预测免疫原性不仅需要考虑肽-HLA结合,还需要考虑T细胞受体(TCR)识别以及肽-HLA复合物与免疫细胞表面受体的相互作用[54]。
尽管已有诸多方法被用于免疫原性预测,但相关研究目前仍然面临根本性的困难,而最核心的挑战在于用于训练模型的数据本身缺乏明确的生物学机制依据。现有研究主要依赖两类数据:一类来自体外细胞实验中对核酸序列诱导的TLR激活或细胞因子释放的检测;另一类则来自临床试验中的免疫不良事件统计。然而,体外实验往往只涉及单一通路(通常是TLR7/8/9),无法全面反映体内复杂的PRR网络;同时实验中使用的核酸浓度通常远超临床水平,因此难以反映真实免疫反应[1, 55]。而临床数据虽然更贴近真实应用,却受到患者个体差异、剂量、递送系统、疾病状态等多重因素干扰,使得“序列-免疫反应”之间的因果链条难以建立。此外,不同研究对免疫原性的定义并不统一,有基于细胞因子浓度阈值的,也有基于不良事件发生率或抗体滴度的,使得标签本身具有主观性和不一致性。这不仅导致不同来源的数据难以整合,也使监督学习模型难以形成稳定可靠的预测能力。更重要的是,免疫原性本身是一个高度多维度、个体化的复杂生物学性质,与HLA型别、既往免疫史、微生物组甚至mRNA的化学修饰和二级结构等因素均密切相关[1, 55]。在免疫机制尚未完全阐明的前提下,任何模型都不可避免地只能学习到数据的统计相关性,而无法真正捕捉免疫激活的因果规律,这也限制了模型在新类型序列、新修饰方式或新临床情境中的泛化性能。
现有免疫原性预测方法在建模策略上也存在显著差异。基于序列基序(motif)的规则模型通常通过扫描CpG、UG-rich片段或poly(U)等已知高风险模式来评估免疫刺激倾向[1],其优势是直观可解释,但无法识别此前未报道的新型免疫刺激序列,也无法处理复杂结构依赖的激活机制。基于结构的模型尝试预测RNA的二级结构并评估其激活RIG-I或MDA5的可能性,但RNA结构预测在长序列上的不确定性较高,且目前缺乏明确的定量规律来描述特定结构触发免疫激活的阈值,因此仍只能提供粗略的定性评估[55, 56]。端到端的深度学习模型(如DeepImmuno-CNN)能够从序列中自动学习复杂模式,在现有数据集上通常可以获得较高的预测性能[20, 57],但其可解释性差、对训练数据质量高度敏感,且难以应对全新的序列模式或化学修饰。某些模型如NeoaPred[51]则专注于新抗原免疫原性预测,其适用于癌症免疫应用,但无法用于siRNA、ASO等固有免疫激活风险评估。总体来看,不同方法各有优劣,没有任何单一策略能够全面准确地预测核酸药物的免疫原性,因此实际设计中更常见的做法是结合多种模型,将基序扫描、结构预测和深度学习风险评估联合使用,并辅以实验验证以确保可靠性。
未来提升免疫原性预测的关键在于从数据和方法两个层面同时改进。首先,需要构建具有机制标注的高质量数据集,不仅记录免疫反应的整体强弱,也应包含PRR激活谱、细胞因子类型、剂量-反应曲线、时间动力学及不同免疫细胞亚群的反应特征,并结合单细胞转录组、细胞因子多重检测和体内成像等多维数据[55]。这样的数据将使模型能够学习免疫激活的机制逻辑,而不仅仅是序列特征的统计共现关系。其次,在模型方法上,可解释的深度学习架构是未来方向,通过注意力机制、GNN或基于SHAP的解释工具,使模型能够指出导致免疫激活的关键序列或结构特征[58]。此外,将免疫学知识构建为知识图谱并引入因果推断框架,可以帮助模型学习更接近因果机理的规律,例如利用干预实验(如定点突变去除某一基序)的数据来推断“去除某基序导致免疫原性下降”这一因果关系,而非单纯统计相关性。进一步地,通过预测-实验迭代的主动学习策略,使模型能够主动选择信息量最大的序列进行验证,在数据稀缺的情况下更高效地提升模型性能。
总体而言,免疫原性预测仍是核酸药物设计中最具挑战性的环节之一。当前的工具大多仍停留在相关性建模阶段,准确性和可推广性受到数据、机制认知及应用场景差异的多重限制。要实现真正可靠、可推广的免疫原性预测,需要计算模型与免疫机制研究的深度结合,通过多学科协作推动形成机制驱动的预测框架,从而在未来的核酸药物开发中发挥更为关键的作用。
4.3 靶向性预测确保核酸药物(尤其是siRNA、ASO、适配体)精确地与其预定靶标(通常是特定的RNA或蛋白质)结合,同时最大限度地减少与非目标分子的相互作用(脱靶效应),对于保证疗效和安全性至关重要[1]。人工智能技术已被开发用于辅助预测核酸药物的靶向特异性。
在预测RNA-配体结合特异性方面,对于靶向RNA的小分子或适配体,预测其结合特异性具有挑战性。GerNA-Bind是一个几何深度学习(geometric deep learning)框架,它利用GNN对RNA的多种构象状态和配体的结构进行编码,并整合两者之间的相互作用信息,以预测小分子选择性结合特定RNA构象的能力。该模型在相关任务上取得了领先性能[38]。
更广义地,深度学习模型被用于预测药物-靶标相互作用(DTI),这包括核酸类药物的潜在靶标与蛋白质靶点之间的相互作用。例如,有研究使用DeepLSTM (长短期记忆网络)结合蛋白质的进化特征和药物分子的亚结构指纹来预测DTI[25]。
对于预测siRNA/ASO脱靶效应,如前所述(第3.2节),机器学习和深度学习模型可以通过分析序列特征、热力学性质或构建相互作用网络来预测siRNA或ASO与其潜在脱靶转录本的结合可能性,从而指导设计具有更高特异性的序列[5]。
4.4 表达水平预测对于以产生功能性蛋白质为目标的核酸药物(主要是mRNA疗法和疫苗),其最终的蛋白质表达水平是衡量其有效性的核心指标。准确预测给定核酸序列能够产生的蛋白质总量,对于药物筛选和优化至关重要。蛋白质的表达水平是一个复杂过程的综合结果,受到多种因素的调控,包括mRNA的转录后修饰、稳定性(如第4.1节所述)、细胞内运输、翻译起始和延伸的效率(这与序列设计紧密相关,如第3.1节讨论的密码子和UTR优化),以及可能的免疫反应(如第4.2节所述)对翻译过程的影响。
人工智能技术,尤其是深度学习模型,因其能够整合多维度信息并捕捉复杂非线性关系的能力,在预测蛋白质表达水平方面显示出巨大潜力。这些模型通常利用大规模的功能性实验数据进行训练,例如包含大量不同mRNA序列(特别是UTR和编码区变体)及其对应蛋白质产量的报告基因检测数据,或者利用核糖体图谱(ribosome profiling)数据来推断翻译效率。通过学习序列特征(如密码子使用偏好、特定序列基序、预测的二级结构)与观察到的蛋白质产量之间的关联,深度学习模型能够对新的mRNA序列的表达潜力进行预测。
例如,前文提到的Optimus 5-Prime模型[4],虽然主要用于设计优化的5'UTR,但其核心能力在于准确预测5'UTR序列对翻译效率(进而影响总表达量)的影响。同样,RiboDecode框架[41]结合深度学习模型和核糖体谱数据,旨在直接优化并预测具有更高蛋白表达潜力的密码子序列。此外,像LinearDesign[3]这样能够同时优化稳定性和密码子使用的算法,其设计的序列在实验中表现出更高的蛋白质表达量,也间接验证了其底层模型对影响表达水平因素的预测能力。这些基于人工智能技术的表达水平预测工具,能够有效地指导研究人员在庞大的序列空间中筛选和设计出具有理想蛋白质产量的mRNA候选药物,从而加速研发进程并提高成功率。
5 人工智能技术驱动的核酸药物递送系统优化有效的递送是将核酸药物送达作用部位的关键环节。由于核酸分子本身的特性(大分子量、负电荷、易降解),通常需要借助递送载体来保护核酸、促进细胞摄取并实现靶向递送[2]。人工智能技术正被越来越多地应用于优化这些递送系统,特别是脂质纳米颗粒(LNP)。
5.1 LNP递送系统LNP是目前临床上最成功、应用最广泛的核酸递送载体,其成功应用是siRNA药物(如Onpattro)和mRNA疫苗(如辉瑞/BioNTech和Moderna的COVID-19疫苗)得以实现的关键因素之一[15]。LNP通常由四种主要成分组成:可电离阳离子脂质(ionizable cationic lipid)、胆固醇、辅助脂质(helper lipid)和聚乙二醇化脂质(PEGylated lipid)。其中,可电离脂质是核心成分,负责在酸性内涵体中质子化以包裹核酸,并在细胞质中帮助核酸释放[15]。
传统LNP的开发依赖于经验和大量的试错性化学合成与生物测试,而AI,特别是深度学习,正在加速这一过程,尤其体现在脂质纳米颗粒设计与筛选的AI应用中。一个重要的方向是新型脂质分子的理性设计。Wang等[14]报道了一个成功案例,他们首先训练深度学习模型分别预测候选脂质分子的关键物理化学性质(如表观pKa)和mRNA递送效率。然后,利用生成模型创建了一个包含近2 000万种潜在脂质结构的虚拟库。通过AI驱动的“生成-评估”迭代循环,研究人员仅需合成和测试少量(几十种)由AI筛选出的顶尖候选分子。结果显示,AI设计的多种新型脂质在小鼠模型中的mRNA递送效率显著优于经典的MC3脂质,甚至媲美更先进的SM-102脂质。这项工作证明了AI在大规模虚拟筛选和发现全新高性能递送材料方面的强大能力[14]。重要的是,模型还提供了一定的可解释性,揭示了如包含芳香环等结构特征与高递送效率之间的关联,有助于指导后续设计[14]。
另一个关键应用是LNP配方和性能的预测与优化。AGILE (accelerated generative inverse design of lipid nanoparticles with experiments)平台是这方面的一个例子,它将深度学习与高通量的组合化学合成及体外筛选相结合[15]。AGILE利用深度神经网络对包含不同结构脂质的LNP配方进行体外递送效率评分预测。该平台的一个关键特点是能够根据不同的目标细胞类型调整模型,从而实现“细胞类型定制化”的LNP设计。研究团队利用AGILE快速设计并评估了一系列用于mRNA递送的LNP,发现不同细胞系对脂质结构确实存在偏好性差异,证明了为特定组织或细胞类型优化递送载体的可行性,这对于将核酸疗法拓展到肝脏以外的组织(如肺、免疫细胞等)具有重要意义[15]。
此外,AI也被用于预测LNP的体内功效。有研究利用机器学习模型(如随机森林、XGBoost、贝叶斯优化)结合LNP的配方组成(脂质比例)、制备参数(如微流控条件)以及分子的理化性质描述符,来预测siRNA-LNP或mRNA-LNP在体内的基因沉默效率或蛋白质表达水平[5, 59, 60]。这类模型可以指导研究人员优化LNP的配方和生产工艺,以获得最佳的体内药效和安全性。
5.2 其他新兴递送载体的AI优化方法除了LNP,研究人员还在探索其他类型的核酸递送载体,如多肽载体、高分子纳米粒、外泌体、病毒样颗粒(virus-like particles, VLPs)等。虽然目前AI在这些载体优化中的应用相较于LNP可能还不够深入,但其潜力同样巨大。机器学习方法可以被应用于这些基于纳米颗粒的其他核酸药物递送策略优化中。首先,类似于LNP中的脂质设计,AI可以用于设计新型载体材料,例如设计具有特定性质(如生物降解性、靶向性、核酸结合能力)的新型聚合物或多肽。其次,AI可以预测载体性能,通过建立模型预测不同载体(如不同序列的多肽、不同结构的高分子)与核酸的结合效率、形成的纳米复合物的尺寸和稳定性、细胞摄取效率以及体内分布等。再次,如果载体需要通过连接靶向配体(如抗体片段、适配体、小分子)来实现组织或细胞特异性递送,AI可以辅助优化靶向配体,包括设计或筛选最优的配体及其与载体的连接方式。最后,AI有助于整合多源数据,将来自不同实验(体外、体内)、不同载体类型的数据整合起来,训练更通用的预测模型,或者利用迁移学习将在数据较丰富的载体系统(如LNP)上学到的知识应用于数据较少的新兴载体系统。虽然针对这些其他载体的具体AI应用案例在现有文献中细节不多,但基本原理与LNP的应用是相通的。随着这些新兴递送技术的发展和相关数据的积累,可以预见AI将在其设计和优化中扮演越来越重要的角色。
6 人工智能在核酸药物设计中的挑战与局限性尽管人工智能技术在核酸药物设计中取得了显著进展,但其应用仍面临一系列挑战和固有限制,需要在未来的研究中加以解决。值得注意的是,虽然人工智能技术包含多种方法(如传统机器学习、深度学习、进化算法等),但当前该领域面临的主要挑战集中在深度学习模型的应用上,这主要是因为深度学习已成为核酸药物设计中最主流和最具潜力的AI技术。
6.1 数据稀疏与高质量数据不足的瓶颈深度学习模型的性能在很大程度上依赖于大规模、高质量的标注数据进行训练。然而,在核酸药物领域,获取这样的数据往往成本高昂且耗时[61]。一个主要问题是数据量不足。相比于小分子药物或蛋白质,许多核酸药物相关的实验数据集规模仍然较小。例如,用于训练ASO效力预测模型的数据量远不如siRNA,且常常局限于特定基因或细胞类型[1]。高质量的RNA三维结构数据、RNA-小分子相互作用数据也相对匮乏[56, 62]。另一个相关问题是数据质量与偏倚。生物实验数据往往存在异质性(来自不同实验室、不同实验条件)、噪声和批次效应。此外,已发表的数据可能存在“报告偏倚”,即更倾向于报告阳性结果(有效的序列或化合物),而阴性结果数据较少,这可能导致模型产生过于乐观的预测[10]。数据标注的准确性和一致性也是关键问题。这些数据问题限制了模型的泛化能力和可靠性,是当前AI应用于核酸药物设计的主要瓶颈之一[5]。
6.2 模型的可解释性问题与临床接受度许多深度学习模型,特别是结构复杂的模型(如深度神经网络、Transformer),常被批评为“黑箱”,即其做出预测的内部决策过程不透明,难以解释[10, 63]。这种缺乏生物学洞见的特性是一个主要障碍。如果模型仅给出预测结果(如某个序列高效或有毒),而不能解释其判断依据(如哪些序列特征或结构模式是关键),研究人员就难以从中获得新的生物学知识或设计规则,也难以信任模型的预测。这进一步影响临床转化。在药物研发这一高风险领域,理解药物的作用机制和模型预测的可靠性至关重要。缺乏可解释性可能会阻碍AI设计的候选药物获得监管机构(如FDA)的批准和临床医生的接受[58]。虽然近年来可解释人工智能(explainable AI, XAI)技术(如注意力机制可视化、特征重要性评分、SHAP值分析、反事实解释等)有所发展[64],但在生物序列和结构等复杂应用场景下,实现真正有意义且可靠的解释仍然是一个挑战[65]。
6.3 计算成本、训练效率与优化难题训练大型深度学习模型(尤其是基于Transformer或GNN的模型,以及处理大规模数据集的模型)通常需要强大的计算资源,如图形处理单元(GPU)或张量处理单元(TPU)集群,这带来了显著的硬件成本和能耗[63]。高昂的计算成本限制了模型的可及性。同时,训练时间可能非常耗时,从几小时到几天甚至几周不等,这减慢了模型开发和迭代的速度。此外,超参数优化本身就是一个复杂的优化问题。寻找最佳的模型架构和超参数(如学习率、层数、节点数等)往往需要大量的实验和调整,进一步增加了时间和计算开销[63]。
6.4 模型泛化与过拟合问题过拟合是深度学习中常见的问题。当训练数据有限或模型过于复杂时,模型容易“记住”训练数据中的噪声或特有模式,而不是学习到底层的普适规律,导致模型在训练集上表现很好,但在新的、未见过的数据(测试集或实际应用场景)上表现较差[10]。与之相关的是泛化能力的挑战。开发出能够在不同数据集、不同生物条件(如不同细胞类型、物种)、不同分子类型或不同实验设置下都能稳健工作的模型(即具有良好泛化能力)是一个重要的目标[27]。目前许多模型可能只在特定的数据集或任务上表现良好。为了缓解过拟合和提高泛化能力,需要采用正则化技术、交叉验证、数据增强以及设计更鲁棒的模型架构。
6.5 生物系统复杂性当前的AI模型往往侧重于预测核酸分子本身的性质或在简化系统(如体外实验)中的行为。然而,核酸药物在体内的最终效果受到极其复杂的生物系统因素影响,包括吸收、分布、代谢、排泄(absorption, distribution, metabolism and excretion, ADME),以及与体内分子的相互作用、细胞内运输、免疫系统应答、个体遗传背景差异等[1]。将这些系统层面的因素有效整合到模型中非常困难,主要是因为缺乏足够的跨尺度、高质量的体内数据。这导致模型预测与体内实际药效之间可能存在差距,是当前AI应用于核酸药物设计面临的又一重大挑战。
7 未来趋势与潜在应用方向尽管存在挑战,人工智能(特别是深度学习)与核酸药物设计的融合仍是大势所趋,未来几年有望在以下方向取得重要进展。
7.1 多组学数据与深度学习整合以推动精准医疗未来的研究将更加注重整合来自基因组学、转录组学、蛋白质组学、表观遗传组学、代谢组学等多维度的数据(multi-omics)[66]。深度学习模型具备处理这种高维、异构数据的能力,能够从中挖掘更深层次的疾病机制、识别更精准的药物靶点,并预测个体对药物的反应[67]。结合单细胞组学技术,AI有望在单细胞分辨率上理解疾病异质性,为开发高度个性化的核酸药物(例如,针对特定肿瘤突变或患者遗传背景的药物)提供前所未有的机遇[68]。
7.2 复杂且可解释的混合AI模型开发前景为了克服“黑箱”问题,开发兼具高性能和高可解释性的模型将是关键研究方向[58]。这可能涉及设计内生可解释的模型架构,例如结合注意力机制或引入生物学先验知识。同时,需要开发更先进的XAI技术,提供更可靠、更有意义的解释。探索混合AI模型,例如将基于知识或规则的系统与数据驱动的深度学习相结合,也是一个有前景的方向[58]。随着模型复杂性的增加,例如更大规模的预训练模型和更复杂的图网络,保持和提升可解释性将面临新的挑战和机遇[26]。
7.3 个性化核酸药物的精准设计与预测方法AI将驱动核酸药物从“通用型”向“个体化”转变。基于患者的特定基因型、转录谱、疾病状态或其他生物标志物,AI模型有望实现多个目标。首先,它可以设计针对个体突变或异常表达基因的siRNA/ASO/gRNA序列[69]。其次,模型能够预测个体对特定核酸药物的敏感性、疗效和潜在副作用[65]。再次,AI可以优化针对个体患者的给药剂量和递送策略,这将极大推动精准医疗在核酸治疗领域的实现[70, 71]。
7.4 自动化实验与计算平台的集成趋势未来的药物发现将更加依赖于自动化。AI/ML模型将与机器人自动化平台(用于高通量合成、筛选和测试)紧密集成,形成快速迭代的“设计-构建-测试-学习”(design-build-test-learn, DBTL)闭环系统[72, 73]。这种自动化平台能够以远超人力的速度和规模进行实验和数据分析,极大加速核酸药物及其递送系统的发现和优化过程[6]。
7.5 RNA靶向小分子药物及基因编辑应用的AI支持AI的应用将扩展到核酸相关的其他领域。在RNA靶向小分子药物方面,RNA正成为越来越重要的小分子药物靶点。AI,特别是基于结构的药物设计和GNN,将在预测RNA结构、识别潜在的小分子结合位点、虚拟筛选和优化RNA靶向小分子方面发挥关键作用[10, 38]。同时,AI在基因编辑系统(如CRISPR-Cas)中的应用已相当广泛。其主要应用包括设计高效、特异性的向导RNA (gRNA)[11],预测基因编辑的效率和结果(如同源重组修复vs非同源末端连接)[74],评估和预测脱靶效应[11],以及优化基因编辑系统的递送载体[75]。随着基因编辑疗法进入临床,AI在其精准性和安全性优化方面的作用将更加凸显[76]。
8 结论人工智能,尤其是深度学习技术,正以前所未有的方式渗透并重塑着核酸药物设计的范式。通过利用强大的数据处理、模式识别和预测能力,深度学习正在帮助研究人员克服传统药物研发方法面临的诸多瓶颈,推动该领域从经验驱动的试错模式向数据驱动的理性设计模式转变。本综述系统总结了人工智能技术在核酸药物(包括mRNA、siRNA、ASO、适配体等)序列设计与优化、关键性质(稳定性、免疫原性、靶向性、表达水平)预测以及递送系统(特别是LNP)优化等方面的应用进展。大量研究案例表明,人工智能技术不仅能够显著提高设计效率、降低研发成本,还能发现性能更优、更安全的候选药物,甚至揭示新的生物学机制。
然而,我们也必须清醒地认识到当前深度学习应用所面临的挑战与局限性。数据稀疏性与质量问题、模型的可解释性不足、高昂的计算成本、模型的泛化能力以及如何有效整合复杂的生物系统因素等,都是亟待解决的关键问题。克服这些挑战需要跨学科的紧密合作,包括开发更先进的算法、构建更大规模和更高质量的数据库、发展更可靠的可解释性方法,以及将AI模型更紧密地融入实验验证流程。
展望未来,AI与核酸药物领域的结合将更加深入和广泛。多组学数据的整合将推动个性化精准医疗的发展;更复杂且可解释的AI模型将成为研究人员的得力助手;自动化实验与计算平台的集成将极大加速研发进程;同时,AI也将在RNA靶向小分子药物、基因编辑等新兴领域扮演关键角色。可以预见,深度学习将持续作为核心驱动力,引领核酸药物研发进入一个更加高效、精准和富有创造力的新时代。未来将有更多由AI辅助设计的创新核酸疗法问世,为攻克遗传病、传染病、癌症等重大疾病带来新的希望,这无疑将是人工智能赋能生物医药领域所描绘的最激动人心的前景之一。
| [1] |
Lin S, Hong L, Wei DQ, et al. Deep learning facilitates efficient optimization of antisense oligonucleotide drugs. Mol Ther Nucleic Acids, 2024, 35: 102208. DOI:10.1016/j.omtn.2024.102208 |
| [2] |
Roberts TC, Langer R, Wood MJA. Advances in oligonucleotide drug delivery. Nat Rev Drug Discov, 2020, 19: 673-94. DOI:10.1038/s41573-020-0075-7 |
| [3] |
Zhang H, Zhang L, Lin A, et al. Algorithm for optimized mRNA design improves stability and immunogenicity. Nature, 2023, 621: 396-403. DOI:10.1038/s41586-023-06127-z |
| [4] |
Castillo-Hair SM, Seelig G. Machine learning for designing next-generation mRNA therapeutics. Acc Chem Res, 2022, 55: 24-34. DOI:10.1021/acs.accounts.1c00621 |
| [5] |
Lee M. Machine learning for small interfering RNAs: a concise review of recent developments. Front Genet, 2023, 14: 1226336. DOI:10.3389/fgene.2023.1226336 |
| [6] |
Bashir A, Yang Q, Wang J, et al. Machine learning guided aptamer refinement and discovery. Nat Commun, 2021, 12: 2366. DOI:10.1038/s41467-021-22555-9 |
| [7] |
Foley & Lardner LLP. Artificial intelligence in drug discovery: 2025 outlook[EB/OL]. (2024-12-18). https://www.foley.com/insights/publications/2024/12/ai-drug-discovery-2025-outlook/
|
| [8] |
Santa Maria JP Jr, Wang Y, Camargo LM. Perspective on the challenges and opportunities of accelerating drug discovery with artificial intelligence. Front Bioinform, 2023, 3: 1121591. DOI:10.3389/fbinf.2023.1121591 |
| [9] |
Li B, Tan K, Lao AR, et al. A comprehensive review of artificial intelligence for pharmacology research. Front Genet, 2024, 15: 1450529. DOI:10.3389/fgene.2024.1450529 |
| [10] |
Morishita EC, Nakamura S. Recent applications of artificial intelligence in RNA-targeted small molecule drug discovery. Expert Opin Drug Discov, 2024, 19: 415-31. DOI:10.1080/17460441.2024.2313455 |
| [11] |
Lee M. Deep learning in CRISPR-Cas systems: a review of recent studies. Front Bioeng Biotechnol, 2023, 11: 1226182. DOI:10.3389/fbioe.2023.1226182 |
| [12] |
Zhou Y, Pedrielli G, Zhang F, et al. Predicting RNA sequence-structure likelihood via structure-aware deep learning. BMC Bioinformatics, 2024, 25: 316. DOI:10.1186/s12859-024-05916-1 |
| [13] |
He S, Gao B, Sabnis R, et al. RNAdegformer: accurate prediction of mRNA degradation at nucleotide resolution with deep learning. Brief Bioinform, 2023, 24: bbac581. DOI:10.1093/bib/bbac581 |
| [14] |
Wang W, Chen K, Jiang T, et al. Artificial intelligence-driven rational design of ionizable lipids for mRNA delivery. Nat Commun, 2024, 15: 10804. DOI:10.1038/s41467-024-55072-6 |
| [15] |
Xu Y, Ma S, Cui H, et al. AGILE platform: a deep learning powered approach to accelerate LNP development for mRNA delivery. Nat Commun, 2024, 15: 6305. DOI:10.1038/s41467-024-50619-z |
| [16] |
Vaz JM, Balaji S. Convolutional neural networks (CNNs): concepts and applications in pharmacogenomics. Mol Divers, 2021, 25: 1569-84. DOI:10.1007/s11030-021-10225-3 |
| [17] |
Liu B, Huang H, Liao W, et al. DeepSipred: a deep-learning-based approach on siRNA inhibition prediction[C]. Beijing: Proceedings of the 2024 4th International Conference on Bioinformatics and Intelligent Computing, 2024: 430-6
|
| [18] |
Zeng H, Edwards MD, Liu G, et al. Convolutional neural network architectures for predicting DNA-protein binding. Bioinformatics, 2016, 32: i121-7. DOI:10.1093/bioinformatics/btw255 |
| [19] |
Patiyal S, Dhall A, Bajaj K, et al. Prediction of RNA-interacting residues in a protein using CNN and evolutionary profile. Brief Bioinform, 2023, 24: bbac538. DOI:10.1093/bib/bbac538 |
| [20] |
Li G, Iyer B, Prasath VBS, et al. DeepImmuno: deep learning-empowered prediction and generation of immunogenic peptides for T-cell immunity. Brief Bioinform, 2021, 22: bbab160. DOI:10.1093/bib/bbab160 |
| [21] |
Zhu MX, Meng ZG, Wang JY. Drug response prediction based on 1D convolutional neural network and attention mechanism. Comput Math Methods Med, 2022, 2022: 8671348. |
| [22] |
Hill ST, Kuintzle R, Teegarden A, et al. A deep recurrent neural network discovers complex biological rules to decipher RNA protein-coding potential. Nucleic Acids Res, 2018, 46: 8105-13. DOI:10.1093/nar/gky567 |
| [23] |
Jain R, Jain A, Mauro E, et al. ICOR: improving codon optimization with recurrent neural networks. BMC Bioinformatics, 2023, 24: 132. DOI:10.1186/s12859-023-05246-8 |
| [24] |
Matsukiyo Y, Tengeiji A, Li C, et al. Transcriptionally conditional recurrent neural network for de novo drug design. J Chem Inf Model, 2024, 64: 5844-52. DOI:10.1021/acs.jcim.4c00531 |
| [25] |
Wang YB, You ZH, Yang S, et al. A deep learning-based method for drug-target interaction prediction based on long short-term memory neural network. BMC Med Inform Decis Mak, 2020, 20: 49. DOI:10.1186/s12911-020-1052-0 |
| [26] |
Choi SR, Lee M. Transformer architecture and attention mechanisms in genome data analysis: a comprehensive review. Biology (Basel), 2023, 12: 1033. |
| [27] |
Bai Y, Zhong H, Wang T, et al. OligoFormer: an accurate and robust prediction method for siRNA design. Bioinformatics, 2024, 40: btae577. DOI:10.1093/bioinformatics/btae577 |
| [28] |
Mao J, Wang J, Zeb A, et al. Transformer-based molecular generative model for antiviral drug design. J Chem Inf Model, 2024, 64: 2733-45. DOI:10.1021/acs.jcim.3c00536 |
| [29] |
Ang D, Rakovski C, Atamian HS. De novo drug design using transformer-based machine translation and reinforcement learning of an adaptive Monte Carlo Tree Search. Pharmaceuticals (Basel), 2024, 17: 161. DOI:10.3390/ph17020161 |
| [30] |
He S, Gao B, Sabnis R, et al. Nucleic transformer: classifying DNA sequences with self-attention and convolutions. ACS Synth Biol, 2023, 12: 3205-14. DOI:10.1021/acssynbio.3c00154 |
| [31] |
Huang T, Song Z, Ying R, et al. Protein-nucleic acid complex modeling with frame averaging transformer. Adv Neural Inf Process Syst, 2024. DOI:10.52202/079017-4038 |
| [32] |
Ji Y, Zhou Z, Liu H, et al. DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome. Bioinformatics, 2021, 37: 2112-20. DOI:10.1093/bioinformatics/btab083 |
| [33] |
Chen J, Hu Z, Sun S, et al. Interpretable RNA foundation model from unannotated data for highly accurate RNA structure and function predictions. arXiv, 2022. DOI:10.48550/arXiv.2204.00300 |
| [34] |
Shulgina Y, Trinidad MI, Langeberg CJ, et al. RNA language models predict mutations that improve RNA function. Nat Commun, 2024, 15: 10627. DOI:10.1038/s41467-024-54812-y |
| [35] |
Zhang XM, Liang L, Liu L, et al. Graph neural networks and their current applications in bioinformatics. Front Genet, 2021, 12: 690049. DOI:10.3389/fgene.2021.690049 |
| [36] |
Hwang G, Kwon M, Seo D, et al. ASOptimizer: optimizing antisense oligonucleotides through deep learning for IDO1 gene regulation. Mol Ther Nucleic Acids, 2024, 35: 102186. DOI:10.1016/j.omtn.2024.102186 |
| [37] |
Long R, Guo Z, Han D, et al. siRNADiscovery: a graph neural network for siRNA efficacy prediction via deep RNA sequence analysis. Brief Bioinform, 2024, 25: bbae563. DOI:10.1093/bib/bbae563 |
| [38] |
Xia Y, Li J, Chu YT, et al. GerNA-Bind: geometric-enhanced RNA-ligand binding specificity prediction with deep learning. arXiv, 2025. DOI:10.1101/2025.02.15.638393 |
| [39] |
Gaudelet T, Day B, Jamasb AR, et al. Utilizing graph machine learning within drug discovery and development. Brief Bioinform, 2021, 22: bbab159. DOI:10.1093/bib/bbab159 |
| [40] |
Wieder O, Kohlbacher S, Kuenemann M, et al. A compact review of molecular property prediction with graph neural networks. Drug Discov Today Technol, 2020, 37: 1-12. DOI:10.1016/j.ddtec.2020.11.009 |
| [41] |
Li Y, Wang F, Yang J, et al. Deep generative optimization of mRNA codon sequences for enhanced mRNA translation and therapeutic efficacy. Nat Commun, 2025, 16: 9957. DOI:10.1038/s41467-025-64894-x |
| [42] |
Wang Z, Liu Z, Zhang W, et al. AptaDiff: de novo design and optimization of aptamers based on diffusion models. Brief Bioinform, 2024, 25: bbae517. DOI:10.1093/bib/bbae517 |
| [43] |
Tang X, Dai H, Knight E, et al. A survey of generative AI for de novo drug design: new frontiers in molecule and protein generation. Brief Bioinform, 2024, 25: bbae338. DOI:10.1093/bib/bbae338 |
| [44] |
Linder J, Srivastava D, Yuan H, et al. Predicting RNA-seq coverage from DNA sequence as a unifying model of gene regulation. Nat Genet, 2025, 57: 949-61. DOI:10.1038/s41588-024-02053-6 |
| [45] |
Li S, Noroozizadeh S, Moayedpour S, et al. mRNA-LM: full-length integrated SLM for mRNA analysis. Nucleic Acids Res, 2025, 53: gkaf044. DOI:10.1093/nar/gkaf044 |
| [46] |
Wood M KM, Allard M. Helix-mRNA: a hybrid foundation model for full sequence mRNA therapeutics. arXiv, 2025, 13785. DOI:10.48550/arXiv.2502.13785 |
| [47] |
La Rosa M, Fiannaca A, La Paglia L, et al. A graph neural network approach for the analysis of siRNA-target biological networks. Int J Mol Sci, 2022, 23: 14211. DOI:10.3390/ijms232214211 |
| [48] |
Xing W, Zhang J, Li C, et al. iAMP-Attenpred: a novel antimicrobial peptide predictor based on BERT feature extraction method and CNN-BiLSTM-Attention combination model. Brief Bioinform, 2023, 25: bbad443. DOI:10.1093/bib/bbad443 |
| [49] |
Townshend RJL, Eismann S, Watkins AM, et al. Geometric deep learning of RNA structure. Science, 2021, 373: 1047-51. DOI:10.1126/science.abe5650 |
| [50] |
Rotrattanadumrong R, Yokobayashi Y. Experimental exploration of a ribozyme neutral network using evolutionary algorithm and deep learning. Nat Commun, 2022, 13: 4847. DOI:10.1038/s41467-022-32538-z |
| [51] |
Jiang D, Xi B, Tan W, et al. NeoaPred: a deep-learning framework for predicting immunogenic neoantigen based on surface and structural features of peptide-human leukocyte antigen complexes. Bioinformatics, 2024, 40: btae547. DOI:10.1093/bioinformatics/btae547 |
| [52] |
Doneva N, Dimitrov I. Viral immunogenicity prediction by machine learning methods. Int J Mol Sci, 2024, 25: 2949. DOI:10.3390/ijms25052949 |
| [53] |
Mei S, Li F, Leier A, et al. A comprehensive review and performance evaluation of bioinformatics tools for HLA class I peptide-binding prediction. Brief Bioinform, 2020, 21: 1119-35. DOI:10.1093/bib/bbz051 |
| [54] |
Xue LC, Dobbs D, Honavar V. HomPPI: a class of sequence homology based protein-protein interface prediction methods. BMC Bioinformatics, 2011, 12: 244. DOI:10.1186/1471-2105-12-244 |
| [55] |
Abanades B, Wong WK, Boyles F, et al. ImmuneBuilder: Deep-Learning models for predicting the structures of immune proteins. Commun Biol, 2023, 6: 575. DOI:10.1038/s42003-023-04927-7 |
| [56] |
Vamathevan J, Clark D, Czodrowski P, et al. Applications of machine learning in drug discovery and development. Nat Rev Drug Discov, 2019, 18: 463-77. DOI:10.1038/s41573-019-0024-5 |
| [57] |
Wu J, Wang W, Zhang J, et al. DeepHLApan: a deep learning approach for neoantigen prediction considering both HLA-peptide binding and immunogenicity. Front Immunol, 2019, 10: 2559. DOI:10.3389/fimmu.2019.02559 |
| [58] |
Jimenez-Luna J, Grisoni F, Weskamp N, et al. Artificial intelligence in drug discovery: recent advances and future perspectives. Expert Opin Drug Discov, 2021, 16: 949-59. DOI:10.1080/17460441.2021.1909567 |
| [59] |
Kumar G, Ardekani AM. Machine-learning framework to predict the performance of lipid nanoparticles for nucleic acid delivery. ACS Appl Bio Mater, 2025, 8: 3717-27. DOI:10.1021/acsabm.4c01716 |
| [60] |
Maharjan R, Kim KH, Lee K, et al. Machine learning-driven optimization of mRNA-lipid nanoparticle vaccine quality with XGBoost/Bayesian method and ensemble model approaches. J Pharm Anal, 2024, 14: 100996. DOI:10.1016/j.jpha.2024.100996 |
| [61] |
Tang Y. Deep learning in drug discovery: applications and limitations. Front Comput Intell Syst, 2023, 3: 118-23. |
| [62] |
Flamm C, Wielach J, Wolfinger MT, et al. Caveats to deep learning approaches to RNA secondary structure prediction. Front Bioinform, 2022, 2: 835422. DOI:10.3389/fbinf.2022.835422 |
| [63] |
Masoomkhah SS, Rezaee K, Ansari M, et al. Deep learning in drug design——progress, methods, and challenges. Front Biomed Technol, 2024. DOI:10.18502/fbt.v11i3.15893 |
| [64] |
Clauwaert J, Menschaert G, Waegeman W. Explainability in transformer models for functional genomics. Brief Bioinform, 2021, 22: bbab060. DOI:10.1093/bib/bbab060 |
| [65] |
Samal BR, Loers JU, Vermeirssen V, et al. Opportunities and challenges in interpretable deep learning for drug sensitivity prediction of cancer cells. Front Bioinform, 2022, 2: 1036963. DOI:10.3389/fbinf.2022.1036963 |
| [66] |
Molla G, Bitew M. Revolutionizing personalized medicine: synergy with multi-omics data generation, main hurdles, and future perspectives. Biomedicines, 2024, 12: 2750. DOI:10.3390/biomedicines12122750 |
| [67] |
MacEachern SJ, Forkert ND. Machine learning for precision medicine. Genome, 2021, 64: 416-25. DOI:10.1139/gen-2020-0131 |
| [68] |
Li S, Hua H, Chen S. Graph neural networks for single-cell omics data: a review of approaches and applications. Brief Bioinform, 2025, 26: bbaf109. DOI:10.1093/bib/bbaf109 |
| [69] |
Leckie J, Yokota T. Integrating machine learning-based approaches into the design of ASO therapies. Genes (Basel), 2025, 16: 185. DOI:10.3390/genes16020185 |
| [70] |
Chen W, Liu X, Zhang S, et al. Artificial intelligence for drug discovery: resources, methods, and applications. Mol Ther Nucleic Acids, 2023, 31: 691-702. DOI:10.1016/j.omtn.2023.02.019 |
| [71] |
Thakur S, Sinhari A, Jain P, et al. A perspective on oligonucleotide therapy: approaches to patient customization. Front Pharmacol, 2022, 13: 1006304. DOI:10.3389/fphar.2022.1006304 |
| [72] |
Duffy J. Revolutionizing biologics: unveiling the challenges and innovations in large-scale oligonucleotide synthesis[EB/OL]. (2023-09-19). https://oxfordglobal.com/nextgen-biomed/resources/revolutionizing-biologics-challenges-innovations-oligonucleotide-synthesis
|
| [73] |
Xu Y, Li X, Yao H, et al. Neural networks in drug discovery: current insights from medicinal chemists. Future Med Chem, 2019, 11: 1669-72. DOI:10.4155/fmc-2019-0118 |
| [74] |
Li VR, Zhang Z, Troyanskaya OG. CROTON: an automated and variant-aware deep learning framework for predicting CRISPR/Cas9 editing outcomes. Bioinformatics, 2021, 37: i342-8. DOI:10.1093/bioinformatics/btab268 |
| [75] |
Dixit S, Kumar A, Srinivasan K, et al. Advancing genome editing with artificial intelligence: opportunities, challenges, and future directions. Front Bioeng Biotechnol, 2023, 11: 1335901. |
| [76] |
Hunt C, Montgomery S, Berkenpas JW, et al. Recent progress of machine learning in gene therapy. Curr Gene Ther, 2022, 22: 132-43. DOI:10.2174/1566523221666210622164133 |
 2025, Vol. 37
2025, Vol. 37 

 谢志,中山大学中山眼科中心教授、博士生导师,广东省生物信息学会理事长,临床医学和计算机科学本科,在美国约翰霍普金斯大学及美国国立卫生研究院(NIH)等机构任职多年。2013年加入中山大学,曾参与建设中山大学精准医学中心大数据中心、中山大学健康医疗大数据国家研究院、国家遗传资源服务和共享平台粤港澳大湾区创新中心。研究兴趣包括利用AI构建虚拟细胞以赋能新药发现,以及AI驱动的mRNA药物设计等领域。截至2025年12月,发表论文92篇,影响因子超过1 000分,H index 39
谢志,中山大学中山眼科中心教授、博士生导师,广东省生物信息学会理事长,临床医学和计算机科学本科,在美国约翰霍普金斯大学及美国国立卫生研究院(NIH)等机构任职多年。2013年加入中山大学,曾参与建设中山大学精准医学中心大数据中心、中山大学健康医疗大数据国家研究院、国家遗传资源服务和共享平台粤港澳大湾区创新中心。研究兴趣包括利用AI构建虚拟细胞以赋能新药发现,以及AI驱动的mRNA药物设计等领域。截至2025年12月,发表论文92篇,影响因子超过1 000分,H index 39