(2 中国科学院上海有机化学研究所, 上海 200032)
(2 Shanghai Institute of Organic Chemistry, Chinese Academy of Sciences, Shanghai 200032, China)
小分子(Small molecules)具有分子量较小的优势,凭借其可以快速穿透细胞膜并与细胞内特定靶标精准互作的能力成为药物开发的重要来源[1]; 在材料领域同样展现出巨大的应用潜力,特别在水凝胶等材料的研发中发挥关键作用[2]; 此外,小分子农药对环境友好,是农药创新的重要方向,具有广阔的应用前景[3]。因此,深入挖掘更多新颖且满足特定需求的小分子对于推动相关领域的发展有着重要意义。但是,传统的小分子发现流程依赖于大量实验设计和化合物合成,成本高昂且效率低下,无法高效探索庞大的化合物空间并生成结构新颖的有效分子,难以满足现代药物、材料等研发对精准性和高效性的需求。
人工智能(Artificial intelligence, AI)在有效探索和理解化学空间方面的能力日益凸显,正逐渐成为小分子发现领域的革新力量,推动分子发现过程的精准化和高效化。其中,生成式深度学习模型被广泛用于小分子发现,主要包括:由将分子映射为潜在空间中概率分布的编码器和将潜在空间中的采样点映射回原始数据空间的解码器组成的变分自编码器(Variational autoencoder, VAE); 包含相互竞争的生成新数据的生成器和判断生成数据与真实数据的判别器以实现生成高度逼真分子的生成对抗网络(Generative adversarial network, GAN); 具有循环连接神经元用于保持状态并考虑先前的信息从而处理序列数据的递归神经网络(Recursive neural network, RNN)等。尤其是2017年提出的Transformer架构[4],改变了包括分子发现在内的多个领域。其最大的特点是自注意机制(Self-attention mechanisms)和前馈网络(Feed-forward networks),能够有效地捕获长期依赖关系和全局上下文信息,并且大幅提升了并行计算能力。基于此架构发展的大语言模型(Large language models, LLMs)通过学习海量的分子数据,掌握了丰富的知识与逻辑推理能力,相较于传统的深度学习方法有着更强大的理解和预测复杂分子语言的能力。
本文主要回顾了以LLM为代表的AI技术在小分子生成、优化、表征等小分子发现领域的研究进展,探讨了当前这些模型面临的挑战,并展望了基于LLM实现自主小分子合成以及加速合成生物学改造天然产物的应用前景。
1 人工智能大语言模型在小分子发现中的应用人工智能大语言模型在小分子生成、小分子优化和小分子表征等复杂任务中有着出色的表现,它们不仅可以从美国信息互换标准代码(American Standard Code for Information Interchange, ASCII)字符串描述分子结构的简化分子线性输入规范字符串(Simplified molecular-input line-entry system, SMILES)、国际纯粹与应用化学联合会(International Union of Pure and Applied Chemistry, IUPAC)制定的标准化命名规则等序列形式,以及分子图结构等多种分子表征方式中高效学习分子信息,还具有深入挖掘基因组以及代谢组数据中隐含小分子的能力,从而助力高效的兼具新颖性与有效性的目标小分子发现(图 1)。
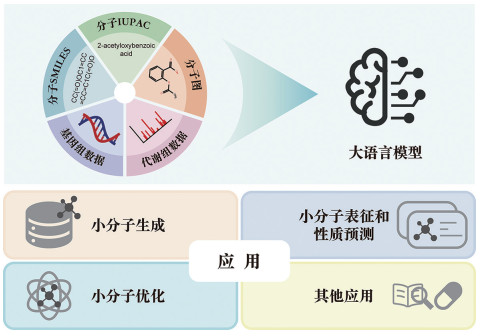
|
图 1 大语言模型在小分子发现中的应用概览 |
小分子库作为药物发现和设计的重要基础,是药物研发过程中必不可少的一部分。通用的化合物数据库,如ZINC[5]、ChEMBL[6]、DrugBank[7]等,以及某些特定领域的小分子库,例如用于脂肪肝研究的NAFLDkb[8]、环肽综合知识库CyclicPepedia[9]、药物抗纤维化知识库Dr AFC[10]等均为药物研发提供了重要的起点,为药物筛选、靶点识别以及药物设计提供了丰富的数据资源。
在传统计算机辅助药物研发流程中,小分子库的构建通常依赖于组合已有化合物的策略实现新分子构建,包括基于分子片段的直接枚举、基于反合成分析原理的片段化及重组、基于分子构象叠合和遗传算法中杂交原理的分子重组实现等,在探索新颖化学空间方面存在局限。
人工智能的发展为小分子库构建提供了新的视角和途径。利用ZINC以及QM9[11]数据库分子SMILES训练得到的VAE相较于基于手工设计规则的遗传算法,生成了更好地保持训练数据分子特征且新颖度较高的分子[12]。JT-VAE实现了将分子图结构作为VAE输入,生成以有效子图为组件的联合树(Junction tree),与图结构组合编码成为两部分的潜在表示,然后分两个阶段将潜在的表示解码回分子图,但生成分子的新颖性有待进一步提高[13]。LatentGAN结合了自编码器(Autoencoder)和生成对抗网络,利用自编码器得到ChEMBL数据库分子SMILES字符串的潜在向量训练GAN,成功实现了从头分子生成,且可以产生较高比例的新型化合物[14]。由ChEMBL中分子SMILES训练得到的基于长短期记忆(Long short-term memory, LSTM)的RNN——CharRNN能够生成大量多样且具有化学合理性的新分子[15]。Xu等[16]分别利用卷积神经网络(Convolutional neural network, CNN)和LSTM构建了用于化学命名翻译的语言模型,相比于传统基于规则的翻译工具有着明显的性能优势,其作为化学语言模型的早期尝试,扩展了中文IUPAC命名等化合物名称数据的应用场景。
利用SMILES字符串训练LLM的早期尝试是smilesGPT,对PubChem[17]来源的SMILES字符串进行了字符级的字节对编码(Byte-pair encoding, BPE),保留了72个字符作为初始词汇,用于训练类似Generative pre-trained transformer (GPT)-2的模型,展示了解码器模型在分子生成中的强大作用[18]。基于Transformer解码器的分子生成模型MolGPT,由190万个来源于MOSES数据集[19]的分子以及160万个来源于GuacaMol[20]的分子训练得到,该模型生成的分子在有效性方面优于VAE等传统模型,且生成过程可以利用显著性图(Saliency maps)得到一定的解释[21]。FragGPT引入了新型分子表示Unordered simplified molecular-input line-entry system based on fragments (FU-SMILES),基于此利用7 800万个来自PubChem以及ChEMBL的分子以GPT2为骨干模型训练得到,并根据特定的下游任务采用低秩适应(Low-rank adaptation, LoRA)进行了微调[22]。FU-SMILES利用Breaking of retrosynthetically interesting chemical substructures (BRICS)算法分割分子,标记分子片段之间的断开点,并通过对这些断开点随机编号和分子片段随机重组进行数据增强。与传统的从左到右逐个原子依次生成分子的SMILES或自引用嵌入式字符串(SELF-referencing embedded strings, SELFIES)相比,FU-SMILES可以实现将片段细节整合到上下文中,缓解基于前文生成原子的错误累积问题。该模型在从头设计、连接体设计、R基团探索、骨架跳跃、侧链优化任务中表现出色,新颖性等指标超越包括MolGPT在内的其他模型。3D SMILES-GPT创新地结合了分子的SMILES字符串和原子坐标信息,将分子的二维结构和三维坐标编码为字符,实现了对分子完整信息的统一表示,使模型能够全面了解分子的二维和三维特征,从而显著提升了生成分子的结合亲和力、类药性(Quantitative estimate of drug-likeness, QED)和可合成性(Synthetic accessibility, SA)[23]。该架构包括了具有12个注意力头的8层Transformer解码层,能够捕获更丰富的分子二维和三维信息。
化学物质的系统名称是更接近自然语言的分子表示形式,包含了化合物中原子、化学基团和空间定向信息,例如IUPAC命名法。值得注意的是,同一种化学物质被规范化之前可能会有多种不同的SMILES表示方式,但其IUPAC命名是唯一确定的。基于GPT-2的iupacGPT由通过分子量、分配系数等基本性质筛选得到9 000万个PubChem数据库的分子的IUPAC命名训练得到,利用构建的规则将IUPAC命名标记为已知官能团和基团[24]。与smiles- GPT相比,该模型在新颖性、Frechet ChemNet Distance (FCD)、多样性等多个指标上有着更出色的表现,表明其通过IUPAC命名学习到的原子和化学基团之间的层次关系以及内部序列关系,相较于利用SMILES字符串学习到的原子和键之间的简单关系更具优势。
相较于序列表示的分子结构,图结构更加完整和直观,包含更丰富的化学信息; 此外,相似化学结构的分子可能被编码为完全不同的序列表示,不利于模型学习。因此,基于图结构的表示方法能够帮助模型更好地捕捉分子的拓扑信息,使模型能够学习和理解分子内部的复杂关系,从而提高性能。Graph Transformer VAE模型结合了VAE和Transformer,利用图卷积和图池化层直接在分子图上运行,通过使用QM9数据库的分子图训练了用于输出节点特征的VAE和用于输出边特征的Transformer[25]。与传统使用位置编码的Transformer模型不同的是,该模型引入了一种新颖的节点编码层,将相邻节点的信息编码到边生成过程中; 与基于VAE或GAN的方法相比,具有更好的有效性、唯一性和新颖性的综合表现。
1.2 小分子优化高效发现具有特定物理化学性质或功能的小分子,对于加速新型药物、材料以及其他创新化合物的研发进程及提高成功率意义重大,是当前分子发现领域亟待攻克的挑战之一。生成模型构建的候选小分子库为后续优化任务提供了丰富的结构基础,但这些分子在活性、选择性或成药性上往往存在不足。小分子优化旨在通过改进小分子的结构,增强其药理特性、选择性、稳定性及生物利用度。借助AI和LLM的强大计算能力,可以在满足特定生物学目标的前提下,探索全新的分子结构; 或者通过发掘天然活性小分子的潜力,从自然界中具有生物活性的分子结构中获取创新灵感,有效减少合成过程中的时间和成本。
1.2.1 知识约束的小分子优化AI的发展使基于目标属性直接优化候选分子成为可能,基于条件的各类分子生成模型大大节省了传统实验筛选以及迭代优化所耗费的时间和资源,其中,LLM更擅长从海量化合物数据集中捕捉分子序列和性质之间的关联,从而更好地优化出符合要求的新颖小分子结构。
MolGPT除了非条件分子生成,也能利用RDKit提取的分子性质以及骨架等条件信息进行训练,实现具有特定性质的分子优化。单一属性和多属性条件优化任务均可以产生有效的新颖分子,还具有在保持骨架结构的同时产生具有特定属性分子的能力。Taiga在MolGPT的基础上进一步使用基于策略梯度(Policy gradient, PG)的强化学习(Reinforcement learning, RL)算法迭代训练模型,虽然在多数指标上表现优于MolGPT,但部分数据集的新颖性分数略低[26]。多目标优化分子的新方法MCMG (Multi-constraint molecular generation)高效探索了化学空间,得到满足约束条件且具有更高结构多样性及更多类型支架的分子[27]。具体来说,该方法首先利用了Transformer在自然语言处理(Natural language processing, NLP)领域的优越性能,使用来源于ChEMBL数据库分子的SMILES字符串训练条件Transformer (c-Transformer),在成功率(生成符合条件的分子)、有效性等方面整体表现优于RNN和条件RNN (c-RNN)。进一步将c-Transformer模型通过表现较好的知识蒸馏方式——蒸馏分子(Distilled molecules, DM)提炼为RNN用于RL微调,在减轻RL训练负担的同时提升了分子的结构多样性。TransPharmer模型结合了分子的SMILES字符串和药效团指纹作为输入,首先使用RDKit获得氢键受体供体、芳香环等药效团特征,进一步提取指纹编码特征间的拓扑距离组合[28]。利用约160万个来源于GuacaMol数据库的分子进行训练,该模型在药效团约束的从头分子生成以及基于骨架的取代基设计任务中成功生成了具有更高药物相似性的分子,而且凭借其独特的局部化学空间探索能力生成了结构独特的药物活性分子,助力骨架跃迁。
TransAntivirus实现了操纵IUPAC得到相应的新抗病毒分子SMILES[29]。该模型的预训练主要利用了筛选得到的3 000万个PubChem数据库分子的IUPAC名称和SMILES字符串,分别根据官能团、基团以及原子和键进行分子的双重标记,并结合了分子油水分布系数的对数值(LogP)等性质。Trans-Antivirus主要由两个Transformer构成,首先学习IUPAC名称的内部关系,进一步利用这些信息实现可控的SMILES字符串生成和优化,侧重于学习两种分子表示方法之间的化学语义关系。该模型不仅生成了在新颖性、有效性、FCD等指标上均优于传统方法的分子,还在针对抗病毒任务进行微调后,成功设计出COVID-19两个不同类型靶点抑制剂的候选先导化合物,在设计抗病毒候选化合物方面展现出巨大的潜力。
可用于基于性质或骨架有条件地优化分子的GraphGPT将分子图结构与SMILES表示相结合[30]。首先采用基于图形的编码器对分子结构进行编码捕获图形信息,采用NLP技术处理SMILES字符串提取分子特性和序列之间的关系以及SMILES的句法格式,之后利用损失函数将分子结构无缝集成到序列数据中,从而完成图结构增强的序列表示。该模型在GuacaMol和MOSES测试集上,产生的所需单一属性和多属性分子的有效性、新颖性、唯一性优于MolGPT。
LLM也具有根据给定指令进行分子优化的能力。Claude 3 Opus能根据指令生成高达97%的有效且唯一的分子[31]。用于分子优化的母体分子共64个,源自ZINC数据库130万个分子量小于200道尔顿、含氮并至少包含一个氢键供体或受体的分子,使用Morgan Fingerprint方法表征后利用主成分分析(Principal component analysis, PCA)生成三维潜在嵌入,进一步通过k-means聚类选取代表性的母体分子。分子优化的提示指令包括对母体分子SMILES表征不同修饰程度的8种基础指令、针对添加供电子基团(Electron donating groups, EDGs)和吸电子基团(Electronwithdrawing groups, EWGs)的8种引导指令,以及控制生成分子与母体分子Tanimoto相似程度的3种控制指令。
1.2.2 天然活性小分子挖掘知识驱动的小分子优化聚焦于理性设计,天然产物挖掘则从自然界中获取目标分子,两者共同拓展了小分子发现的化学空间。天然产物源自细菌、真菌、植物、动物等生物体的代谢过程,是结构多样复杂、具有生物活性的小分子化合物。它们不仅覆盖了广泛的化学空间,还通过在结构上优化以服务于特定的生物学功能,且生物相容性较高、毒性相对可控。鉴于这些优势,天然产物及其衍生物已成为癌症、感染性疾病、心脑血管疾病等多种重大疾病药物研发以及生物活性材料的关键来源,广泛应用于抗生素[32]、抗肿瘤药物[33]、免疫调节剂[34]、药物递送载体[35]、小分子凝胶剂[36]的开发。此外,在农业领域,天然产物还是杀菌剂、除草剂[37]、杀虫剂[38]等作物保护化合物的重要来源。LOTUS[39]、COCONUT[40]、Natural Products Atlas[41]、NPBS[42]等大型天然产物数据库提供了海量的天然产物相关数据,涵盖化学式、结构信息、分布及来源等多维度内容。
AI技术的引入为天然产物的挖掘与利用开辟了新途径,由传统的活性筛选方法转变为探索以前未知的化学空间,不仅提高了天然产物筛选效率以及准确率,还能实现更为精准的结构预测分析。通过进一步利用AI技术结合酶改造等手段,可以优化天然产物的合成路径,提高生产效率,确保产物的质量与稳定性,拓宽天然产物的应用范围,为新分子研发提供了强有力的支持。
1.2.2.1 基于基因组学数据的天然产物挖掘基于基因组数据挖掘天然产物的常用手段是寻找基因组中成簇存在的负责次级代谢产物(Secondary metabolite, SM)生物合成的基因——生物合成基因簇(Biosynthetic gene cluster, BGC),包括主要合成基因、次要合成基因、调控基因、转运基因和其他基因等。一个BGC可以由数个至上百个基因构成,这些基因协作编码生物合成酶形成生物合成通路,共同完成一种或多种小分子代谢物的生成。
传统的BGC鉴定工具以antiSMASH[43]和PRISM[44]为代表,这些方法利用预构建的隐马尔可夫模型(Hidden Markov model, HMM)等识别次级代谢酶的结构域,进一步基于已有的规则判断是否符合BGC的组成。因此,往往受限于已知的BGC类型,最新版本的antiSMASH 7.0也仅支持81种基因簇的鉴定,识别新型BGC表现往往不佳。深度学习的应用在新型BGC的预测方面有着显著的优势。基于深度学习和NLP的BGC预测工具DeepBGC利用了双向长短期记忆(Bidirectional long short-term memory, BiLSTM)神经网络以及基于word2vec算法的Pfam2vec,通过Pfam2vec处理Pfam预测的结果输入BiLSTM计算BGC的预测分数,实现BGC预测,并使用随机森林对BGC进行分类[45]。该方法不仅降低了BGC识别的假阳性率,还提高了推断和识别新BGC类别的能力。
利用BGC的信息不仅可以获得天然产物的类型,还可以用于天然产物结构的预测,因为代谢产物的主要化学结构特征或骨架特征往往已经隐含在BGC中。PRISM采用基于图的组合方法,将BGC中核心生物合成酶对应的小分子底物作为化学图,根据618个虚拟剪裁反应的理论知识库对化学图进行转换,实现SM结构的预测。antiSMASH 7.0则是利用生物合成酶底物特异性预测聚酮合酶(Polyketide synthase, PKS)和非核糖体肽合酶(Non-ribosomal peptide synthetase, NRPS)模块所对应线性组装产物的化学结构。但这些方法受限于已知类型的酶和对应催化反应的规则,无法实现代谢产物结构的高效准确预测。LLM有着强大的生成能力,有望类似于自然语言翻译一样转换BGC和代谢产物结构的化学序列,从而实现新的天然产物挖掘和预测。基于Transformer构建的从微生物BGC序列到SM化学结构的大语言模型DeepSeMS,采用生物合成酶功能域表征BGC序列,并且设计了结构特征对齐的数据增强策略,使得模型在预测精度和预测能力上相比基于规则的模型(antiSMASH 7.0, PRISM 4)有显著提升[46]。利用DeepSeMS模型大规模挖掘了全球海洋微生物基因组,发现了65 000多个新颖的SM,首次系统性揭示了海洋微生物SM的地理环境分布特征和生物医药应用潜力。
1.2.2.2 基于代谢组学数据的天然产物挖掘基因组学数据揭示了天然产物的合成潜能,从代谢组学出发的挖掘策略可以更加直观地反映天然产物的结构、性质等特征。但是,传统的注释技术高度依赖与已有数据库的比对,对于很大一部分新颖的小分子来说,从复杂的质谱数据混合物中进行识别以及预测属性仍具有很大挑战性。
深度学习的方法在一定程度上促进了新颖小分子的鉴定,例如,CSI: Finger ID将串联质谱(Tandem MS, MS/MS)谱图转化为碎片树,结合分子指纹信息,训练支持向量机模型建立碎片树和分子指纹之间的转化关系,准确性优于检索分子结构数据库的工具[47]。基于CNN的SMART (Small molecule accurate recognition technology)由53 076个天然产物训练得到,实现了从核磁共振波谱(Nuclear magnetic resonance, NMR)中快速注释新型天然产物,已成功应用于蓝藻天然产物以及复杂结构的新颖环肽的表征[48]。
LLM进一步提高了代谢物注释的准确度与效率。基于Transformer的串联质谱峰注释模型MIST (Metabolite inference with spectrum transformers)突破传统对质谱进行分箱的模式,首先利用SIRIUS工具[49]将质谱表示为所有峰的化学式集,然后通过浅层前馈多层感知器将这些化学式和强度转换为特征向量用于表示峰[50]。MIST不仅学习了峰和对应化学式之间的关系,还学习了片段之间的中性损失关系,从而更好地进行注释。为了提高预测分子指纹的分辨率,引入一种新颖的“unfolding”layer,首先学习粗粒度的分子特性,逐渐以越来越高的分辨率预测分子指纹。该模型在准确性方面优于CSI: Finger,且有着很好的可重复性和泛化能力[51]。
LLM不仅可以实现质谱数据中新分子结构的预测,在属性预测任务中也有着出色的表现。MS2Prop通过聚合跨片段峰的信息实现了从MS/MS数据出发直接预测化合物的LogP、QED、SA等10个药物发现的关键属性[52]。该模型利用Transformer进行动态加权处理将输入转化为代表整个谱图的向量,用于进一步利用前馈神经网络模块输出预测的化学性质,无须依赖实际或预测的结构,其在已知和新颖分子的属性预测任务中表现均优于CSI: Finger。
1.3 小分子表征和性质预测经过生成与优化的候选小分子需进一步验证其理化与生物学特性。LLM因其强大的数据学习能力和上下文理解能力,已经成为小分子表征学习的重要工具。不同于传统的小分子描述符和指纹,通过大规模的无监督学习,LLM能够涵盖更广泛的化学空间,提取分子的潜在规律并生成高质量的小分子嵌入表征(Embeddings)。这些嵌入表征不仅能够全面编码分子的结构、功能和性质信息,还为小分子生成与优化任务提供了关键支持。此外,由于其蕴含的丰富的分子特征信息,LLM驱动的嵌入表征显著提升了下游预测任务,如活性预测、毒性评估以及药物的吸收、分配、代谢、排泄和毒性(Absorption Distribution Metabolism Excretion Toxicity, ADMET)性质分析的准确性、通用性和泛化能力。
目前,已有多种基于LLM的小分子表征模型取得了显著进展。Chemformer是基于Transformer架构的小分子预训练模型,其通过掩蔽技术(Masking)对ZINC-15数据库中的约1亿条SMILES表示进行表征学习[53]。类似地,MolBERT采用了BERT (Bidirectional encoder representations from transformers)架构,从SMILES中提取出复杂的分子特征,生成灵活且高质量的分子表征[54]。除此之外,其他基于Transformer架构的预训练模型还包括ChemBERTa-2[55]、Molformer[56]、MolT5[57]和MegaMolBART (https://github.com/NVIDIA/MegaMolBART)等。
这些预训练LLM小分子表征模型通过对大规模化学数据的学习,可以自动提取重要的分子特征,不受噪音或无关特征影响,也避免了传统方法中需要人工设计特征的复杂性。同时,LLM不仅能够捕捉分子内在的结构规律和化学性质等丰富信息,还能捕捉分子之间的复杂关系,生成具有高度表达能力的分子表征。这些通用的分子表征可以应用于多种下游任务(如分子分类、性质预测、药物筛选等),具有更好的跨任务泛化能力。此外,LLM的生成能力也为分子设计带来了前所未有的创新机遇。无监督预训练深度学习框架ImageMol通过在1 000万个未标记的类药生物活性分子上进行预训练,从分子图像中学习分子表征[58]。通过在ImageMol的编码器上附加全连接层进行微调,该模型成功实现了抗SARS-CoV-2分子的虚拟筛选,验证了其分子表征的有效性,能够高效识别具有抗病毒活性的候选分子。多模态分子结构-文本模型MoleculeSTM应用MegaMolBART将SMILES字符串转化为分子表征,并融合了PubChem数据库的功能描述文本知识[59]。通过EBM-NCE和InfoNCE比对学习策略,在共享表征空间对齐分子结构与文本描述,能够高效地执行多种基于文本指令的下游任务,包括结构-文本检索、分子编辑和优化,以及小分子性质预测(如结合亲和力和毒性)。这些功能使MoleculeSTM能够提取新的疾病-药物关系,并成功地修改分子结构以满足多个所需属性,进而有望加速药物重定位和先导化合物优化等药物发现过程。LLM4SD框架利用预训练的LLM对文献中的知识进行综合,并结合从SMILES等数据中推断识别的知识,将其转化为可解释的分子向量特征用于预测模型训练,在生理学、生物物理学、物理化学和量子力学四个领域的分子性质预测任务中超越现有技术,特别在回归任务中实现了显著的性能提升[60]。
1.4 其他应用LLM在小分子发现中的应用已扩展到化学知识提取、药物组合设计、个性化药物设计以及教育和研究辅助工具等新兴领域。这些方向不仅丰富了LLM的应用场景,还为药物研发和化学研究提供了创新工具。
化学文献和数据库中的知识通常以非结构化文本形式存在,LLM能够通过NLP技术高效提取结构化信息,并构建知识图谱以支持药物发现。LLM除了可以识别化学文本中的分子、反应、靶点等实体,还可从文献中提取药物-靶点相互作用、反应机理等关系。例如,国际商业机器公司(International Business Machines Corporation, IBM)的RXN for Chemistry平台利用LLM自动提取反应规则并构建合成路径图谱,显著加速了逆合成分析[61]。
药物组合疗法在癌症等复杂疾病治疗中具有重要价值,LLM可通过多模态数据分析预测协同效应和优化组合策略。CancerGPT能够解决罕见组织中缺乏结构化数据和特征的药物对的协同预测问题,推进LLM在生物和医学推理任务中的应用[62]。基于Transformer的模型MeTDDI能够挖掘药物相互作用,有助于解析药物副作用与分子结构关联[63]。对于个性化医疗领域,LLM在整合多组学数据和优化个体化方案中展现出潜力,可解析患者基因组变异与药物响应的关联。例如,PrimeKG知识图谱结合LLM预测了肿瘤突变对靶向药物的敏感性[64]。
此外,LLM正在变革化学教育和科研范式,其自动化与智能化能力显著提升研究效率。ChatGPT被证明可以帮助加速药物发现过程,包括化学文件格式转换、化合物分析、理解药物ADMET特性等,对于早期药物发现具有重要意义[65, 66]。研究者已经尝试将LLM与机器人实验平台结合,实现闭环式药物发现。例如,Coscientist平台利用GPT-4驱动的人工智能系统自动生成合成方案并指导机器人自主设计、规划和执行复杂的化学实验[67]。基于LLM开发的智能代理ChemCrow帮助化学研究,降低了专家和非专家研究人员使用化学工具的门槛,加强实验化学和计算化学的融合[68]。基于Llama-3.1-70B LLM的多级多智能体系统ChemAgents,可以满足文献阅读、实验设计、计算执行、操控机器人等不同实验需求[69]。表 1整理了上述各类应用场景中经典的LLM。
| 表 1 用于小分子发现的经典LLM |
LLM不仅能够高效处理并分析分子结构、反应、活性以及基因组学信息、临床数据等海量的化学与生物数据,更具备强大的多源数据整合能力,对不同类型或实验平台的数据进行统一标准化处理,从而深入挖掘数据中的复杂模式和隐藏关系,实现跨领域知识迁移。利用其强大的数据处理能力,LLM将这些知识应用于小分子的发现,大大增强了对分子结构、理化性质、生物活性之间关系的理解[70]。
2.2 高效的分子生成能力LLM凭借其高效的分子生成能力,在小分子的多样性、新颖性以及化学空间拓展方面展现出显著优势。这些模型能够突破传统分子设计方法的局限,充分探索化学空间中低概率但合理的区域,快速生成大量结构多样且具有化学合理性的候选分子,广泛覆盖化学空间,并实现新颖方式组合化学片段,生成具有新颖结构的分子,极大地扩展了化学空间的探索范围,大大加速了先导化合物的发现进程。
2.3 定向优化及自动化设计LLM通过海量数据学习到的分子性质信息以及跨领域知识有助于针对特定需求快速优化分子性质,并支持多目标优化,能够在多个性质中找到最佳平衡,提高候选物的成功率。此外,这些模型可以基于特定的靶点结构或已知活性数据,自动化设计满足目标性质的分子,进一步筛选出最优候选分子。AutoMolDesigner是一款专用于小分子抗生素自动设计的AI开源软件,可以实现生物活性、可合成性和成药性等多目标协同优化[71]。相较于传统方法,LLM在定向优化及自动化设计中的优势显著减少了人工干预,缩短了研发周期,推动了小分子发现的智能化与精准化发展。AI驱动的集成平台Chemistry42包含40多个先进的生成式模型,融合专业的医学、计算化学知识,加速了具有用户所需性质的新型分子设计,已服务于20多家制药公司[72]。
2.4 易扩展、产业化对接相较于传统模型因依赖特定领域数据和规则所导致的有限拓展性,LLM凭借其高度通用的架构实现了快速适应新的研究需求或跨领域应用。通过使用特定领域或任务的数据对预训练模型进行微调,可使其更好地适配新应用场景,并快速提升模型在新任务上的性能。这种易拓展的优势进一步支持其与高通量筛选系统、自动化实验平台的对接,显著提高了研发效率,大幅降低了产业化应用的技术门槛,为小分子研发的规模化与产业化提供了强有力的技术支持。
2.5 加速药物发现流程药物开发是一项复杂且耗时的工作,传统方法虽然可以在一定程度上辅助药物开发,但受限于其算法框架和数据处理能力,往往难以在短时间内快速识别出具有潜力的候选小分子,导致药物开发周期较长、成本高昂,且成功率相对较低[73]。LLM有着高效的分子生成能力,能够快速生成大量候选分子,并进一步依托其在跨领域知识整合和多目标优化方面的出色表现,筛选得到最优的先导化合物用于进一步研究,加速药物从实验室到临床应用的转化过程[74]。石药集团用于成人2型糖尿病患者血糖控制的新型口服二肽基肽酶-4 (Dipeptidyl peptidase 4, DPP-4)抑制剂普卢格列汀片已在中国获批上市,在该药物的研发过程中,AI技术被应用于优化晶型筛选,成功预测所有潜在晶型,有效规避传统方法造成的重要晶型遗漏,显著缩短了药物研发周期。
2.6 个性化药物设计传统模型受限于模型规模、数据处理能力和泛化性能,难以充分利用个体患者的基因组学、蛋白质组学等特异性数据实现药物精准设计,且优化过程效率较低。LLM则有着深入挖掘个体化特征的优势,可以实现针对特定患者或疾病亚型生成高度定制化的候选分子,精准靶向疾病相关的分子机制,提高治疗效果,并通过药物安全性等重要指标进行优化,以满足个性化治疗的需求。例如,基于LLM的药物靶点发现平台PandaOmics成功鉴定出抗纤维化的新药物靶点——TRAF2/NCK相互作用激酶(TRAF2- and NCK-interacting kinase, TNIK),并进一步开发出其小分子抑制剂INS018_05[75]。
3 人工智能大语言模型在小分子发现中面临的挑战 3.1 数据质量和数量大量高质量的数据是LLM有效训练和精准预测的基础,虽然当前化学和生物数据的规模在不断扩大,但仍面临着数据质量和数量的挑战。数据质量的高低关系着模型的准确性和可靠性[76],但目前的数据普遍存在噪声、不一致或标注不完整等问题,极易导致模型学习到错误的模式,进而对预测结果产生误导。为了提升数据质量,可综合利用Z-score、Grubbs检验等统计方法或深度孤立森林(Deep isolation forest)[77]等AI模型检测数据集中的异常值,进一步以权威数据库为基准,结合多种工具的预测结果进行数据验证与矫正。数据数量则是推动模型通用能力发展的关键因素,在小分子发现领域,由于化学空间的巨大性和复杂性,收集足够数量且具有代表性的数据是一项艰巨的任务,尤其是在特殊结构小分子等数据稀缺问题尤为突出的特定领域,严重限制了模型的表现和应用范围。因此,需要在SMILES枚举、原子掩码、标记(Token)删除、键删除、子图删除等现有的数据增强策略[78, 79]的基础上,结合生成式AI产生的高质量数据,进一步开发更高效的算法以满足LLM训练的数据需求。
3.2 模型可解释性虽然LLM在小分子发现中展现出巨大的潜力,但其面临着对小分子的可合成性和有效性产生重要影响的可解释性挑战。LLM通常被视为“黑箱”模型,其决策过程和生成结果的逻辑缺乏透明性,还会掩盖模型预测中的潜在偏差或错误。
尽管当前LLM已经在分子的逆合成预测领域取得了初步成就,推动了对复杂分子合成的探索,并提高了设计合成实验的效率。例如,基于Transformer的SCROP[80]、EditRetro[81]等工具可用于预测反应物; BioNavi-NP结合Transformer和AND-OR树,可用于预测天然产物及其类似化合物的生物合成途径[82]。但是这距离实现逆合成预测的完全自动化和智能化仍有一段距离,亟需解决现有模型在预测产物-反应物对时可能会忽略实际化学反应的合理性,导致预测出不可行反应的问题。此外,预测过程未充分考虑反应条件等实际的合成路径复杂性以及实验安全性、所需成本、产率等其他因素。
此外,AI技术驱动了众多新颖且初步满足所需理化性质分子的发现,贡献了一系列进入临床试验阶段的候选药物。然而,值得注意的是,部分药物出现了临床失败的案例,例如Exscientia的EXS-21546因疗效未达到预期止步于Ⅰ/Ⅱ期研究,BenevolentAI的BEN-2293也在Ⅱ期试验后终止。这些失败的案例警示我们,AI发现的小分子面临着从实验室到实际应用场景的转化挑战,其有效性仍需要通过严格的实验、临床等积累经验和数据。AI能否深入理解复杂的生物学机制,从而生成真正安全有效的分子,是当前分子发现领域需要解决的关键难题。
增强模型可解释性可以帮助我们更好地理解LLM内部的运行机制,提升模型性能。其中一个重要思路是,通过分析执行特定任务的可解释模型子图——知识回路(Knowledge circuits)揭示LLM的知识编辑机制,并计算回路中节点和边的贡献,从而指导针对知识回路中关键组件的模型优化[83]。Rank-One Model Editing (ROME)则通过因果干预识别对事实预测至关重要的神经元激活,从而实现对模型权重的精准调整,修改后的模型在特异性和泛化性方面均表现优异[84]。其他常用方法包括运用注意力机制可视化方法直观地展示模型的注意力分配机制,增强模型决策过程的透明度[85, 86]; 融合先验知识图谱构建可解释性网络架构,揭示模型推理路径[87]; 此外,还可以引入对抗样本生成技术,通过分析模型在对抗样本上的表现,理解模型的决策机制,揭示模型的潜在弱点,为其鲁棒性优化提供依据[88]。
3.3 计算资源LLM通常具有庞大的参数量和复杂的网络结构,使得其训练和推理成为计算密集型过程,需要消耗大量的计算资源,当前很多模型训练明显不足[89]。特别是在小分子发现领域,由于化学空间的广阔性和分子结构的复杂性,模型所需处理的数据量极为庞大,进一步加剧了对计算资源的需求。高昂的计算成本限制了其广泛应用,因此,如何优化模型架构、开发高效的训练算法、提高模型的训练和推理效率,成为了LLM在小分子发现领域面临的重要挑战。
3.4 伦理及安全LLM在小分子发现领域的应用伴随着一系列潜在的伦理和安全挑战,需引起高度重视[90]。首先,目前缺乏对模型训练所使用数据的有效保护措施,部分数据可能涉及患者隐私或商业机密,一旦泄露或被滥用,将对知识产权和个人隐私构成严重威胁。针对此类问题,无需数据共享的联邦学习(Federated learning)、删除训练数据的机器遗忘(Machine unlearning)、添加随机噪声的差分隐私(Differential privacy)等算法的开发在一定程度上保护了隐私数据,但仍需建立相关风险评估机制并完善法律法规。此外,LLM生成的新分子可能具有未知的生物活性或毒性,由于缺乏系统的风险和安全评估,若未充分验证便投入实验或临床使用,可能对人类健康或生态环境造成潜在风险,甚至可能被滥用于合成危险或非法药物,引发严重的社会安全问题。Collaborations Pharmaceutical公司发现反向使用其用于生成人类疾病靶点新抑制剂的商用模型MegaSyn,即奖励毒性行为,可在6小时内生成40 000个包括VX神经毒剂、新型高毒性分子在内的分子[91]。值得注意的是,LLM的广泛应用可能进一步加剧药物研发中的不平等问题,使得资源匮乏的地区难以获得先进技术,拉大全球健康差距。因此,亟需开发能够检测分子毒性与环境风险的分子过滤器,以限制有害分子的生成,同时联合毒理学会等有关部门的监管,防止模型滥用。
4 展望LLM在小分子发现领域有着巨大的应用潜力,不仅可以实现小分子生成、优化、表征等,更有着整合现有知识和资源、协调计算和实验工具的能力。针对其目前在小分子发现领域存在的可合成性以及在实际应用场景中的有效性问题,强大的LLM可以通过与实验室自动化技术相结合完成复杂分子合成任务的精心规划与精准执行,利用专业知识进行高效的天然产物改造以提高有效性(图 2)。
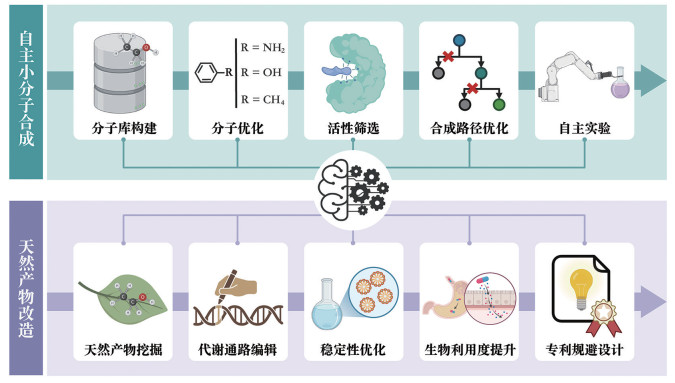
|
图 2 LLM在小分子发现中的发展趋势(利用BioRender.com绘制) |
基于GPT-4的自主设计、规划和执行复杂实验的人工智能系统取得了自主小分子合成的初步进展,例如Coscientist成功设计和执行了Suzuki-Miyaura和Sonogashira偶联反应的方案; ChemCrow成功完成了避蚊胺(DEET),已知硫脲有机催化剂Schreiner's、Ricci's和Takemoto's的合成规划,并进一步通过IBM Research的RoboRXN平台执行了合成过程; 此外,该工具通过与人类合作指导了新发色团的筛选和合成。
LLM驱动自动实验室实现分子合成,为科研领域带来了前所未有的变革,通过优化实验流程显著提高了实验的效率和准确性,加速了分子发现的进程。未来,通过提高训练模型数据的质量以及更新集成的各类工具和技术,LLM可以更加高性能地执行各类复杂的合成任务,增强LLM对小分子发现领域的贡献。
4.2 LLM助力通过合成生物学改造天然产物LLM具有完成复杂生物任务的巨大潜力,能够提供天然产物的优化改造思路。综合CNN、Attention、LSTM和Transformer的模型AMP-CLIP和AMP-READ可用于抗菌肽分类和最低抑菌浓度预测; 基于AMP-READ的可解释性,开发了用于抗原肽虚拟定向进化的EvoGradient,通过迭代的梯度下降过程,逐步优化肽的氨基酸序列。利用该模型成功实现了对人类口腔微生物抗菌肽的改造,得到了32种效力显著增强的进化抗菌肽,并进一步合成了6种对多重耐药病原体有效的药物[92]。此外,LLM通过提高合成生物学重要工具——基因编辑的效率和准确性,加速了天然产物改造。CRISPR-GPT可以引导用户完成选择CRISPR系统、设计向导RNA、推荐细胞递送方法、起草实验方案、规划验证实验以确认编辑结果等常规流程步骤的设计,支持根据用户需求定制必要任务,并能够解答基因编辑的相关问题[93]。
LLM不仅为天然产物的优化指明了方向,还大大加速了其改造进程。随着跨学科知识的融合以及引入更加专业的领域知识和人类智慧用于模型训练,LLM能够深入理解复杂的生物活性信息,有望在未来实现直接根据目标分子的预期性质或特定功能设计天然产物的改造方案,为天然产物的开发和利用提供更多创新和可能性。
5 总结综上所述,LLM凭借其强大的数据处理能力、高效的分子生成能力、定向优化策略以及个性化药物设计和产业化对接方面表现出色,在小分子发现领域展现了巨大的潜力,其应用涵盖小分子生成、优化、表征及其他相关任务,不仅加速了新颖先导化合物的发现,也推动小分子研发的规模化与商业化发展。然而,LLM在小分子发现中仍面临数据质量与数量、模型可解释性、计算资源及伦理安全等方面的挑战,这些问题需要通过技术创新和跨学科合作加以解决。展望未来,LLM驱动的自主小分子合成和通过合成生物学改造天然产物将成为重要方向,重塑小分子发现的范式,为小分子研发的智能化与高效化发展提供强有力的支持。
| [1] |
Wu Q, Qian W, Sun X, et al. Small-molecule inhibitors, immune checkpoint inhibitors, and more: FDA-approved novel therapeutic drugs for solid tumors from 1991 to 2021. J Hematol Oncol, 2022, 15: 143. DOI:10.1186/s13045-022-01362-9 |
| [2] |
Luo W, Yang Z, Zheng J, et al. Small molecule hydrogels loading small molecule drugs from Chinese medicine for the enhanced treatment of traumatic brain injury. ACS Nano, 2024, 18: 28894-909. DOI:10.1021/acsnano.4c09097 |
| [3] |
Miao P, Wang H, Wang W, et al. A widespread plant defense compound disarms bacterial type Ⅲ injectisome assembly. Science, 2025, 387: eads0377. DOI:10.1126/science.ads0377 |
| [4] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. arXiv, 2017. DOI:10.48550/arXiv.1706.03762 |
| [5] |
Tingle BI, Tang KG, Castanon M, et al. ZINC-22 horizontal line A free multi-billion-scale database of tangible compounds for ligand discovery. J Chem Inf Model, 2023, 63: 1166-76. DOI:10.1021/acs.jcim.2c01253 |
| [6] |
Mendez D, Gaulton A, Bento AP, et al. ChEMBL: towards direct deposition of bioassay data. Nucleic Acids Res, 2019, 47: D930-40. |
| [7] |
Knox C, Wilson M, Klinger CM, et al. DrugBank 6.0: the DrugBank Knowledgebase for 2024. Nucleic Acids Res, 2024, 52: D1265-75. |
| [8] |
Xu T, Gao W, Zhu L, et al. NAFLDkb: a knowledge base and platform for drug development against nonalcoholic fatty liver disease. J Chem Inf Model, 2024, 64: 2817-28. DOI:10.1021/acs.jcim.3c00395 |
| [9] |
Liu L, Yang L, Cao S, et al. CyclicPepedia: a knowledge base of natural and synthetic cyclic peptides. Brief Bioinform, 2024, 25: bbae190. DOI:10.1093/bib/bbae190 |
| [10] |
Wu D, Gao W, Li X, et al. Dr AFC: drug repositioning through anti-fibrosis characteristic. Brief Bioinform, 2021, 22: bbaa115. DOI:10.1093/bib/bbaa115 |
| [11] |
Ramakrishnan R, Dral PO, Rupp M, et al. Quantum chemistry structures and properties of 134 kilo molecules. Sci Data, 2014, 1: 140022. DOI:10.1038/sdata.2014.22 |
| [12] |
Gomez-Bombarelli R, Wei JN, Duvenaud D, et al. Automatic chemical design using a data-driven continuous representation of molecules. ACS Cent Sci, 2018, 4: 268-76. DOI:10.1021/acscentsci.7b00572 |
| [13] |
Jin W, Barzilay R, Jaakkola T. Junction tree variational autoencoder for molecular graph generation. arXiv, 2018. DOI:10.48550/arXiv.1802.04364 |
| [14] |
Prykhodko O, Johansson SV, Kotsias PC, et al. A de novo molecular generation method using latent vector based generative adversarial network. J Cheminform, 2019, 11: 74. DOI:10.1186/s13321-019-0397-9 |
| [15] |
Segler MHS, Kogej T, Tyrchan C, et al. Generating focused molecule libraries for drug discovery with recurrent neural networks. ACS Cent Sci, 2018, 4: 120-31. DOI:10.1021/acscentsci.7b00512 |
| [16] |
Xu T, Chen W, Zhou J, et al. Neural machine translation of chemical nomenclature between English and Chinese. J Cheminform, 2020, 12: 50. DOI:10.1186/s13321-020-00457-0 |
| [17] |
Kim S, Thiessen PA, Bolton EE, et al. PubChem substance and compound databases. Nucleic Acids Res, 2016, 44: D1202-13. |
| [18] |
Adilov S. Generative pre-training from molecules. ChemRxiv, 2021. DOI:10.26434/chemrxiv-2021-5fwjd |
| [19] |
Polykovskiy D, Zhebrak A, Sanchez-Lengeling B, et al. Molecular Sets (MOSES): a benchmarking platform for molecular generation models. Front Pharmacol, 2020, 11: 565644. DOI:10.3389/fphar.2020.565644 |
| [20] |
Brown N, Fiscato M, Segler MHS, et al. GuacaMol: benchmarking models for de novo molecular design. J Chem Inf Model, 2019, 59: 1096-108. DOI:10.1021/acs.jcim.8b00839 |
| [21] |
Bagal V, Aggarwal R, Vinod PK, et al. MolGPT: molecular generation using a transformer-decoder model. J Chem Inf Model, 2022, 62: 2064-76. DOI:10.1021/acs.jcim.1c00600 |
| [22] |
Yue J, Peng B, Chen Y, et al. Unlocking comprehensive molecular design across all scenarios with large language model and unordered chemical language. Chem Sci, 2024, 15: 13727-40. DOI:10.1039/D4SC03744H |
| [23] |
Wang J, Luo H, Qin R, et al. 3DSMILES-GPT: 3D molecular pocket-based generation with token-only large language model. Chem Sci, 2025, 16: 637-48. DOI:10.1039/D4SC06864E |
| [24] |
Mao J, Wang J, Cho KH, et al. iupacGPT: IUPAC-based large-scale molecular pre-trained model for property prediction and molecule generation. ChemRxiv, 2023. DOI:10.26434/chemrxiv-2023-5kjvh |
| [25] |
Mitton J, Senn HM, Wynne K, et al. A graph VAE and graph transformer approach to generating molecular graphs. arXiv, 2021. DOI:10.48550/arXiv.2104.04345 |
| [26] |
Mazuz E, Shtar G, Shapira B, et al. Molecule generation using transformers and policy gradient reinforcement learning. Sci Rep, 2023, 13: 8799. DOI:10.1038/s41598-023-35648-w |
| [27] |
Wang J, Hsieh CY, Wang M, et al. Multi-constraint molecular generation based on conditional transformer, knowledge distillation and reinforcement learning. Nat Mach Intell, 2021, 3: 914-22. DOI:10.1038/s42256-021-00403-1 |
| [28] |
Xie W, Zhang J, Xie Q, et al. Accelerating discovery of bioactive ligands with pharmacophore-informed generative models. Nat Commun, 2025, 16: 2391. DOI:10.1038/s41467-025-56349-0 |
| [29] |
Mao J, Wang J, Zeb A, et al. Transformer-based molecular generative model for antiviral drug design. J Chem Inf Model, 2024, 64: 2733-45. DOI:10.1021/acs.jcim.3c00536 |
| [30] |
Lu H, Wei Z, Wang X, et al. GraphGPT: a graph enhanced generative pretrained transformer for conditioned molecular generation. Int J Mol Sci, 2023, 24: 16761. DOI:10.3390/ijms242316761 |
| [31] |
Bhattacharya D, Cassady HJ, Hickner MA, et al. Large language models as molecular design engines. J Chem Inf Model, 2024, 64: 7086-96. DOI:10.1021/acs.jcim.4c01396 |
| [32] |
Lewis K, Lee RE, Brotz-Oesterhelt H, et al. Sophisticated natural products as antibiotics. Nature, 2024, 632: 39-49. DOI:10.1038/s41586-024-07530-w |
| [33] |
Luesch H, Ellis EK, Chen QY, et al. Progress in the discovery and development of anticancer agents from marine cyanobacteria. Nat Prod Rep, 2025, 42: 208-56. DOI:10.1039/D4NP00019F |
| [34] |
Beigoli S, Boskabady MH. The molecular basis of the immunomodulatory effects of natural products: a comprehensive review. Phytomedicine, 2024, 135: 156028. DOI:10.1016/j.phymed.2024.156028 |
| [35] |
Zhi K, Wang J, Zhao H, et al. Self-assembled small molecule natural product gel for drug delivery: a breakthrough in new application of small molecule natural products. Acta Pharm Sin B, 2020, 10: 913-27. DOI:10.1016/j.apsb.2019.09.009 |
| [36] |
Huang X, Li T, Jiang X, et al. Co-assembled supramolecular hydrogel of salvianolic acid B and a phosphopeptide for enhanced wound healing. ACS Appl Mater Interfaces, 2023, 15: 45606-15. DOI:10.1021/acsami.3c09219 |
| [37] |
Yan Y, Liu Q, Zang X, et al. Resistance-gene-directed discovery of a natural-product herbicide with a new mode of action. Nature, 2018, 559: 415-8. DOI:10.1038/s41586-018-0319-4 |
| [38] |
Yuan XL, Zhao DL, Zhang ZF, et al. Characterization of a new insecticidal benzothiazole derivative from Aspergillus sp. 1022LEF against the fall armyworm, Spodoptera frugiperda (Lepidoptera: Noctuidae). J Agric Food Chem, 2024, 72: 27939-52. |
| [39] |
Rutz A, Sorokina M, Galgonek J, et al. The LOTUS initiative for open knowledge management in natural products research. Elife, 2022, 11: e70780. DOI:10.7554/eLife.70780 |
| [40] |
Sorokina M, Merseburger P, Rajan K, et al. COCONUT online: collection of open natural products database. J Cheminform, 2021, 13: 2. DOI:10.1186/s13321-020-00478-9 |
| [41] |
Poynton EF, van Santen JA, Pin M, et al. The Natural Products Atlas 3.0: extending the database of microbially derived natural products. Nucleic Acids Res, 2025, 53: D691-9. |
| [42] |
Xu T, Chen W, Zhou J, et al. NPBS database: a chemical data resource with relational data between natural products and biological sources. Database (Oxford), 2020, 2020: baaa102. DOI:10.1093/database/baaa102 |
| [43] |
Blin K, Shaw S, Augustijn HE, et al. antiSMASH 7.0: new and improved predictions for detection, regulation, chemical structures and visualisation. Nucleic Acids Res, 2023, 51: W46-50. DOI:10.1093/nar/gkad234 |
| [44] |
Skinnider MA, Johnston CW, Gunabalasingam M, et al. Comprehensive prediction of secondary metabolite structure and biological activity from microbial genome sequences. Nat Commun, 2020, 11: 6058. DOI:10.1038/s41467-020-19986-1 |
| [45] |
Hannigan GD, Prihoda D, Palicka A, et al. A deep learning genome-mining strategy for biosynthetic gene cluster prediction. Nucleic Acids Res, 2019, 47: e110. DOI:10.1093/nar/gkz654 |
| [46] |
Xu T, Yang Y, Zhu R, et al. DeepSeMS: a large language model reveals hidden biosynthetic potential of the global ocean microbiome. bioRxiv, 2025. DOI:10.1101/2025.03.02.641084 |
| [47] |
Duhrkop K, Shen H, Meusel M, et al. Searching molecular structure databases with tandem mass spectra using CSI: FingerID. Proc Natl Acad Sci U S A, 2015, 112: 12580-5. DOI:10.1073/pnas.1509788112 |
| [48] |
Zhang C, Idelbayev Y, Roberts N, et al. Small molecule accurate recognition technology (SMART) to enhance natural products research. Sci Rep, 2017, 7: 14243. DOI:10.1038/s41598-017-13923-x |
| [49] |
Duhrkop K, Fleischauer M, Ludwig M, et al. SIRIUS 4: a rapid tool for turning tandem mass spectra into metabolite structure information. Nat Methods, 2019, 16: 299-302. DOI:10.1038/s41592-019-0344-8 |
| [50] |
Goldman S, Wohlwend J, Stražar M, et al. Annotating metabolite mass spectra with domain-inspired chemical formula transformers. Nat Mach Intell, 2023, 5: 965-79. DOI:10.1038/s42256-023-00708-3 |
| [51] |
Heirman J, Bittremieux W. Reusability report: annotating metabolite mass spectra with domain-inspired chemical formula transformers. Nat Mach Intell, 2024, 6: 1296-302. DOI:10.1038/s42256-024-00909-4 |
| [52] |
Voronov G, Frandsen A, Bargh B, et al. MS2Prop: a machine learning model that directly predicts chemical properties from mass spectrometry data for novel compounds. bioRxiv, 2022. DOI:10.1101/2022.10.09.511482 |
| [53] |
Irwin R, Dimitriadis S, He J, et al. Chemformer: a pre-trained transformer for computational chemistry. Mach Learn Sci Technol, 2022, 3: 015022. DOI:10.1088/2632-2153/ac3ffb |
| [54] |
Fabian B, Edlich T, Gaspar H, et al. Molecular representation learning with language models and domain-relevant auxiliary tasks. arXiv, 2020. DOI:10.48550/arXiv.2011.13230 |
| [55] |
Ahmad W, Simon E, Chithrananda S, et al. ChemBERTa-2: towards chemical foundation models. arXiv, 2022. DOI:10.48550/arXiv.2209.01712 |
| [56] |
Ross J, Belgodere B, Chenthamarakshan V, et al. Large-scale chemical language representations capture molecular structure and properties. Nat Mach Intell, 2022, 4: 1256-64. DOI:10.1038/s42256-022-00580-7 |
| [57] |
Edwards C, Lai T, Ros K, et al. Translation between molecules and natural language. arXiv, 2022. DOI:10.48550/arXiv.2204.11817 |
| [58] |
Zeng X, Xiang H, Yu L, et al. Accurate prediction of molecular properties and drug targets using a self-supervised image representation learning framework. Nat Mach Intell, 2022, 4: 1004-16. DOI:10.1038/s42256-022-00557-6 |
| [59] |
Liu S, Nie W, Wang C, et al. Multi-modal molecule structure – text model for text-based retrieval and editing. Nat Mach Intell, 2023, 5: 1447-57. DOI:10.1038/s42256-023-00759-6 |
| [60] |
Zheng Y, Koh HY, Ju J, et al. Large language models for scientific discovery in molecular property prediction. Nat Mach Intell, 2025, 7: 437-47. DOI:10.1038/s42256-025-00994-z |
| [61] |
Schwaller P, Probst D, Vaucher AC, et al. Mapping the space of chemical reactions using attention-based neural networks. Nat Mach Intell, 2021, 3: 144-52. DOI:10.1038/s42256-020-00284-w |
| [62] |
Li T, Shetty S, Kamath A, et al. CancerGPT for few shot drug pair synergy prediction using large pretrained language models. NPJ Digit Med, 2024, 7: 40. DOI:10.1038/s41746-024-01024-9 |
| [63] |
Zhong Y, Li G, Yang J, et al. Learning motif-based graphs for drug-drug interaction prediction via local-global self-attention. Nat Mach Intell, 2024, 6: 1094-105. DOI:10.1038/s42256-024-00888-6 |
| [64] |
Chandak P, Huang K, Zitnik M. Building a knowledge graph to enable precision medicine. Sci Data, 2023, 10: 67. DOI:10.1038/s41597-023-01960-3 |
| [65] |
Savage N. Drug discovery companies are customizing ChatGPT: here's how. Nat Biotechnol, 2023, 41: 585-6. DOI:10.1038/s41587-023-01788-7 |
| [66] |
Sharma G, Thakur A. ChatGPT in drug discovery. ChemRxiv, 2023. DOI:10.26434/chemrxiv-2023-qgs3k |
| [67] |
Boiko DA, MacKnight R, Kline B, et al. Autonomous chemical research with large language models. Nature, 2023, 624: 570-8. DOI:10.1038/s41586-023-06792-0 |
| [68] |
A MB, Cox S, Schilter O, et al. Augmenting large language models with chemistry tools. Nat Mach Intell, 2024, 6: 525-5. DOI:10.1038/s42256-024-00832-8 |
| [69] |
Song T, Luo M, Zhang X, et al. A multiagent-driven robotic AI chemist enabling autonomous chemical research on demand. J Am Chem Soc, 2025, 147: 12534-45. DOI:10.1021/jacs.4c17738 |
| [70] |
Aryal S, Do T, Heyojoo B, et al. Leveraging multi-AI agents for cross-domain knowledge discovery. arXiv, 2024. DOI:10.48550/arXiv.2404.08511 |
| [71] |
Shen T, Guo J, Han Z, et al. AutoMolDesigner for antibiotic discovery: an AI-based open-source software for automated design of small-molecule antibiotics. J Chem Inf Model, 2024, 64: 575-83. DOI:10.1021/acs.jcim.3c01562 |
| [72] |
Ivanenkov YA, Polykovskiy D, Bezrukov D, et al. Chemistry42: an AI-driven platform for molecular design and optimization. J Chem Inf Model, 2023, 63: 695-701. DOI:10.1021/acs.jcim.2c01191 |
| [73] |
Paul SM, Mytelka DS, Dunwiddie CT, et al. How to improve R&D productivity: the pharmaceutical industry's grand challenge. Nat Rev Drug Discov, 2010, 9: 203-14. DOI:10.1038/nrd3078 |
| [74] |
Zhang K, Yang X, Wang Y, et al. Artificial intelligence in drug development. Nat Med, 2025, 31: 45-59. DOI:10.1038/s41591-024-03434-4 |
| [75] |
Ren F, Aliper A, Chen J, et al. A small-molecule TNIK inhibitor targets fibrosis in preclinical and clinical models. Nat Biotechnol, 2025, 43: 63-75. DOI:10.1038/s41587-024-02143-0 |
| [76] |
Kaplan J, McCandlish S, Henighan T, et al. Scaling laws for neural language models. arXiv, 2020. DOI:10.48550/arXiv.2001.08361 |
| [77] |
Xu H, Pang G, Wang Y, et al. Deep isolation forest for anomaly detection. arXiv, 2022. DOI:10.48550/arXiv.2206.06602 |
| [78] |
Wang Y, Wang J, Cao Z, et al. Molecular contrastive learning of representations via graph neural networks. Nat Mach Intell, 2022, 4: 279-87. DOI:10.1038/s42256-022-00447-x |
| [79] |
Wu CK, Zhang XC, Yang ZJ, et al. Learning to SMILES: BAN-based strategies to improve latent representation learning from molecules. Brief Bioinform, 2021, 22: bbab327. DOI:10.1093/bib/bbab327 |
| [80] |
Zheng S, Rao J, Zhang Z, et al. Predicting retrosynthetic reactions using self-corrected transformer neural networks. J Chem Inf Model, 2020, 60: 47-55. DOI:10.1021/acs.jcim.9b00949 |
| [81] |
Han Y, Xu X, Hsieh CY, et al. Retrosynthesis prediction with an iterative string editing model. Nat Commun, 2024, 15: 6404. DOI:10.1038/s41467-024-50617-1 |
| [82] |
Zheng S, Zeng T, Li C, et al. Deep learning driven biosynthetic pathways navigation for natural products with BioNavi-NP. Nat Commun, 2022, 13: 3342. DOI:10.1038/s41467-022-30970-9 |
| [83] |
Yao Y, Zhang N, Xi Z, et al. Knowledge circuits in pretrained transformers. arXiv, 2024. DOI:10.48550/arXiv.2405.17969 |
| [84] |
Meng K, Bau D, Andonian A, et al. Locating and editing factual associations in GPT. arXiv, 2022. DOI:10.48550/arXiv.2202.05262 |
| [85] |
Miao S, Liu M, Li P. Interpretable and generalizable graph learning via stochastic attention mechanism. arXiv, 2022. DOI:10.48550/arXiv.2201.12987 |
| [86] |
Hao Y, Liu X, Fu H, et al. PGAT-ABPp: harnessing protein language models and graph attention networks for antibacterial peptide identification with remarkable accuracy. Bioinformatics, 2024, 40: btae497. DOI:10.1093/bioinformatics/btae497 |
| [87] |
Sudhahar S, Ozer B, Chang J, et al. An experimentally validated approach to automated biological evidence generation in drug discovery using knowledge graphs. Nat Commun, 2024, 15: 5703. DOI:10.1038/s41467-024-50024-6 |
| [88] |
Choudhury S, Moret M, Salvy P, et al. Reconstructing kinetic models for dynamical studies of metabolism using generative adversarial networks. Nat Mach Intell, 2022, 4: 710-9. DOI:10.1038/s42256-022-00519-y |
| [89] |
Hoffmann J, Borgeaud S, Mensch A, et al. Training compute-optimal large language models. arXiv, 2022. DOI:10.48550/arXiv.2203.15556 |
| [90] |
Tang X, Jin Q, Zhu K, et al. Prioritizing safeguarding over autonomy: risks of LLM agents for science. arXiv, 2024. DOI:10.48550/arXiv.2402.04247 |
| [91] |
Urbina F, Lentzos F, Invernizzi C, et al. Dual use of artificial intelligence-powered drug discovery. Nat Mach Intell, 2022, 4: 189-91. DOI:10.1038/s42256-022-00465-9 |
| [92] |
Wang B, Lin P, Zhong Y, et al. Explainable deep learning and virtual evolution identifies antimicrobial peptides with activity against multidrug-resistant human pathogens. Nat Microbiol, 2025, 10: 332-47. DOI:10.1038/s41564-024-01907-3 |
| [93] |
Huang K, Qu Y, Cousins H, et al. CRISPR-GPT: an LLM agent for automated design of gene-editing experiments. arXiv, 2024. DOI:10.48550/arXiv.2404.18021 |
 2025, Vol. 37
2025, Vol. 37 

 朱瑞新博士现任同济大学生命科学与技术学院生物信息系,上海市第一妇婴保健院转化医学中心教授、博士生导师。2007年毕业于中国科学院上海有机化学研究所,获得计算机化学博士学位。朱瑞新博士领导的课题组研究方向聚焦人工智能与药物设计、人工智能与微生物组及数据密集型算法与平台开发。以第一或通讯作者身份(含并列)在包括Nature Microbiology、Nature Ecology & Evolution、Nature Protocols、Nature Communications和Cell Reports Medicine等知名期刊发表SCI论文82篇(截至2025年10月)。主编出版了国内“计算机辅助药物设计”第一本本科生专业教材。任期刊Biomedical Informatics副主编,任期刊Microbiology Spectrum和iMetaMed编委。为以下期刊担任审稿:Nature Medicine、Nature Microbiology、Signal Transduction and Targeted Therapy、Protein & Cell、Briefings in Bioinformatics和Bioinformatics等
朱瑞新博士现任同济大学生命科学与技术学院生物信息系,上海市第一妇婴保健院转化医学中心教授、博士生导师。2007年毕业于中国科学院上海有机化学研究所,获得计算机化学博士学位。朱瑞新博士领导的课题组研究方向聚焦人工智能与药物设计、人工智能与微生物组及数据密集型算法与平台开发。以第一或通讯作者身份(含并列)在包括Nature Microbiology、Nature Ecology & Evolution、Nature Protocols、Nature Communications和Cell Reports Medicine等知名期刊发表SCI论文82篇(截至2025年10月)。主编出版了国内“计算机辅助药物设计”第一本本科生专业教材。任期刊Biomedical Informatics副主编,任期刊Microbiology Spectrum和iMetaMed编委。为以下期刊担任审稿:Nature Medicine、Nature Microbiology、Signal Transduction and Targeted Therapy、Protein & Cell、Briefings in Bioinformatics和Bioinformatics等