药物研发是一个耗时、昂贵且高风险的过程,从初期研究到新药上市通常需要约10年时间和20亿美元投入[1]。这一过程包括靶点发现、候选药物筛选与优化、临床前研究、临床试验及市场化等多个阶段,而每一阶段不仅耗费大量时间和资源,成功率也很低,使得药物研发面临严峻挑战。药物靶点发现作为整个过程的第一步,决定着整个药物研发的命运,其目标是通过识别和验证那些在疾病发展过程中起关键作用的生物分子或细胞途径,寻找潜在的干预点。这些靶点通常包括基因位点、受体、酶、离子通道、核酸等生物大分子。新颖且有效的药物靶点的发现是现代药物研发的基石与核心,有助于提高药物的疗效和降低副作用。
药物靶点的发现因其高难度、高成本和疾病关联复杂性而面临重大挑战。截至2022年,全球成功开发的药物靶点不足500个[2],这一瓶颈凸显了提升有效靶点发现效率的迫切性,加速技术创新将成为解决这一问题的关键。近年来,药物靶点发现方法随着技术进步逐渐形成三大主要策略:实验方法、多组学方法和计算方法[3]。其中,实验方法如小分子亲和探针标记、氨基酸稳定同位素标记比较分析、RNA干扰和CRISPR干扰筛选等技术,已在靶点鉴定和验证中展现了显著成效。多组学方法主要通过对基因组、蛋白质组、代谢组等组学数据进行差异性分析,提取出可能致病的生物分子靶点。然而,这些方法往往依赖高质量的生物样本,并消耗大量资源。计算方法作为辅助或替代方案,利用目标化合物的化学结构信息,通过药效团筛选、反向对接和结构相似性评估等技术来预测小分子的新生物靶点[3],虽然展现潜力,但仍面临对蛋白质结构强依赖性的限制,制约其广泛应用。据最新数据统计,2013-2022年创新药的平均研发成本和研发周期总体呈现增长趋势,中位平均研发成本约为24亿美元,比十年前增加了约20%,研发周期延长了1~2年。这反映了药物研发复杂性日益增加的现状,进一步强调了加速靶点发现和技术革新的必要性,以推动药物研发效率的提升。
人工智能(Artificial Intelligence, AI)作为21世纪最具变革意义的技术,在计算机视觉与自然语言处理领域取得了突破性进展,也推动了药物发现过程的全面革新[4]。Insilico Medicine是一家致力于利用AI加速药物研发的创新企业,在药物靶点发现和临床前候选药物筛选领域取得了显著成果。例如,在特发性肺纤维化研究中,该公司通过其AI平台在18个月内完成了新靶点发现,推出第一个AI生成和发现的药物[5]; 在肌萎缩侧索硬化症研究中,其PandaOmics平台识别了20多个相关高置信度基因靶点,包括11个全新治疗靶点[6]。这些成果充分展现了AI在提高靶点发现效率、加速药物开发进程以及降低研发成本方面的巨大潜力,为多种复杂疾病的治疗开辟了新路径。
ChatGPT的全球风靡卷起了人工智能大语言模型应用风暴。大语言模型(Large Language Model, LLM)是一类具有大量参数的复杂深度学习模型,它们在自然语言处理领域中,通过挖掘大量的文本数据来学习语言模式、语法和语义,理解并生成人类语言。大语言模型通常基于Transformer架构,这是一种由Vaswani等在2017年提出的神经网络模型[7]。其核心特点是自注意力机制,能够让模型在处理文本时关注到句子中不同部分之间的关系,从而有效捕捉长距离依赖,对自然语言处理和其他序列到序列的任务产生了革命性的影响。大语言模型与药物发现领域的整合更标志着重大范式转变[8]。在挖掘药物靶点过程中,大语言模型可以进行全面的文献综述和专利分析,以探索疾病所涉及的生物学途径及关键靶点。此外,通过对生物“语言”的学习,专业模型还可以对基因组学、代谢组学、蛋白质组学、多组学等生物领域进行分析和预测,以筛选具有强大潜力的候选药物靶点(图 1)。以ESMFold[9]为代表的一系列蛋白质语言模型,可以进行靶点结构预测,从而打破结构相似性分析等技术对结构输入的限制。在下文中我们将进一步总结人工智能大语言模型在药物靶点发现过程中的应用,并阐述目前存在的问题与挑战。
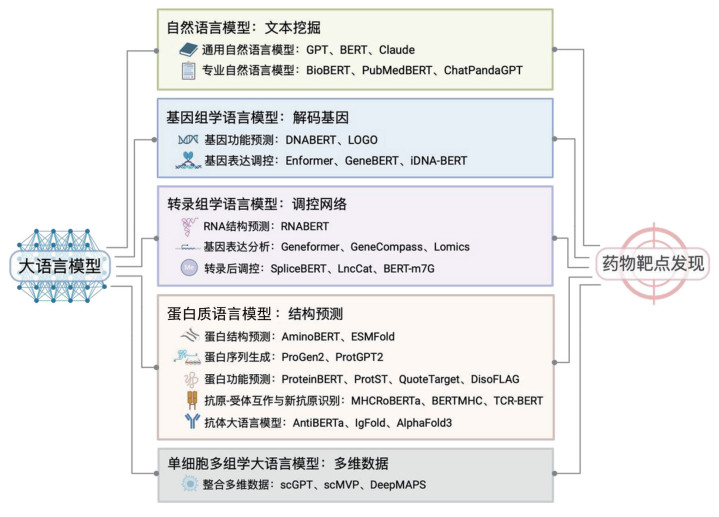
|
注:该图总结了人工智能大语言模型在药物靶点发现中的应用及其代表模型,包括自然语言模型及应用于基因组学、转录组学、蛋白质和多组学的语言模型。 图 1 人工智能大语言模型在药物靶点发现中的应用 |
GPT和BERT等自然语言模型近年来广受关注,这些基于Transformer架构的预训练模型因其强大的语言理解和生成能力,在自然语言处理领域得到广泛应用。在生物医学领域,它们通过文献分析、术语提取等方式,为疾病机制研究和靶点发现提供了全新工具。通用自然语言模型在大规模文本上进行预训练,能够处理多种语言现象和模式,通过学习语言的共性规律,这些模型可以对多种下游任务产生质的提升[8]。专用自然语言模型在这里指专门为生物学领域设计和训练的模型,它们能够理解和生成相关专业术语,处理生物医学文献中的复杂句子结构和专业概念。在药物靶点设计的应用中,通用自然语言模型和专用自然语言模型都发挥着重要作用。
1.1 通用自然语言模型通用自然语言模型接受包括科学论文、教科书和一般文献在内的各种文本信息的训练,海量的训练文本使他们能够广泛理解人类语言,对科学背景有着深刻认识。在用于如药物靶点发现等科学任务时,GPT-4[10]、BERT[11]、Claude等通用语言模型可以遍历大量文献,将提取的数据整合为知识图谱,揭示基因和疾病之间的联系,提高靶点的可解释性,帮助科学家揭示疾病背后机制[12]。这些通用语言模型可以同时熟练掌握复杂形式的科学描述语言以及通用性知识,这使其在知识广度和跨主题建立联系的能力方面具有优势。
1.2 专用自然语言模型通用自然语言模型有效促进了生物医学文本的挖掘,然而,通用模型将自然语言词语分布从一般语料库直接转移到生物医学语料库,这使得针对于专业性较强的生物医学文本的挖掘较为困难。在生物医学飞速发展的需求驱动下,专用自然语言模型应运而生。PubMed、PubMed Central (PMC)文献是生物医学专用语言模型预训练中常用的医学语料库[13]。BERT系列衍生模型如BioBERT[14]、PubMed- BERT[15],GPT系列衍生模型如BioGPT[16]、Chat-PandaGPT等专用自然语言模型提高了生物医学自然语言处理任务的准确性和效率。BioBERT使用人类蛋白质图谱的数据对其模型进行微调,可实现生物医学命名实体识别、生物医学关系提取、生物医学问题回答等功能,可以从科学文献中提取信息并识别新的药物靶点[14]。ChatPandaGPT是由Insilico Medicine公司在其生物靶点发现平台PandaOmics上集成的一项新功能,研究人员通过与该平台进行自然语言对话,可以更轻松地浏览复杂数据并识别潜在的治疗靶点和生物标志物。此外,Galactica[17]可以从科学文献中自动提取分子相互作用和途径信息,改善对复杂生物过程的理解,从而促进潜在药物靶点的发现。
通过理解自然语言和解释复杂科学概念,自然语言模型成为了加速药物靶点发现的宝贵工具。通用自然语言模型在处理多样化任务时更加灵活,然而,为了适应专业术语和语境,通用模型在应用于特定领域时可能需要额外的微调。专用自然语言模型的优势在于它们能够更加深入地理解特定领域的知识,但由于对领域的强依赖性,专用模型可能在处理领域外的任务时遇到挑战。未来结合通用性和专用性优势的,平衡专业性与泛化性的混合模型可能会更好地服务于生物领域以及其他专业领域的研究和应用。与此同时,现有模型依赖现有的文献资料进行训练,这难以避免地会在训练中延续人类的偏见和固化观念。此外,由于这些模型严重依赖已发表的数据,它们识别真正新颖药物靶点的潜力可能有限。因此,自然语言模型与其他模型协同使用也许对新颖且有效药物靶点的发现有更大的价值[3, 18]。
2 基因组学大语言模型:揭示药靶基因密码随着药物靶点发现与新药开发对生物数据挖掘需求的增加,研究开始将自然语言处理技术的优势延伸至更大规模、更复杂、更具专业性的生物数据领域,基因组学大语言模型由此应运而生。基因组学研究生物体完整的DNA,重点探究基因组的结构、功能、进化、映射和编辑。新一代基因组技术的发展使研究人员能够获得大量的基因组数据[19]。如今,大语言模型与基因组学分析的融合正在开辟新的研究方向和应用场景,基因组大语言模型在大量基因组数据上进行训练,可以对基因功能、调节及相互作用提出更深的见解,具备预测致病变异和基因表达等能力,为药物靶点发现提供理论基础,为新药的开发提供依据。
2.1 基因功能预测基因组学大语言模型通过分析DNA序列信息,识别功能区域、变异及结构特征,为药物靶点发现提供了理论支持。例如,DNABERT[20]将DNA序列语言化,通过k-mers捕获复杂模式,精准预测与疾病相关的突变及DNA-蛋白质相互作用。LOGO[21]是一个轻量级人类基因组语言模型,作者将LOGO预训练模型作为起始模型权重,通过微调模型用于启动子识别、增强子-启动子相互作用预测、染色质特征预测以及疾病相关的变异优先排序等任务。Arc研究所团队提出的多模态基因组基础模型Evo[22],该模型能够解码自然基因组,预测微小DNA变化如何影响生物体的适应性,在理解和设计跨模态及多复杂度的生物学方面,Evo实现了重大进步,为靶点筛选奠定基础。
2.2 基因表达调控大语言模型有助于对于基因表达调控关键调控因子的识别,预测基因相互作用,从而更深入地了解基因调节网络[23]。由DeepMind开发的Enformer模型能够整合基因组中跨度超过以往方法5倍(约200 kb)的长程相互作用信息,从而更准确地建模增强子对基因表达的调控作用。此外,肿瘤等诸多疾病的发生和发展往往伴随着表观遗传学的异常,如DNA甲基化水平的改变、组蛋白修饰的异常等。这些异常可以通过表观遗传药物进行干预。GeneBERT是一种基于BERT的变体,聚焦于基因组数据的功能预测,预测组蛋白修饰的差异基因表达,分析基因表达与调控。BERT6mA[25]、iDNA-ABT[26]、MuLan-Methyl[27]等模型可以分析DNA序列中的甲基化模式,预测其在基因调控中的潜在功能。这些模型为人们对表观遗传修饰对基因表达的影响提供了更深入的见解[18],为药物开发提供新的靶点与思路。
3 转录组学大语言模型:构建药靶相关调控网络在深入解析基因组数据的基础上,科学家们逐步转向更复杂的动态数据研究,探索基因表达和调控网络。转录组学是研究生物体内所有转录本的学科,旨在全面分析基因表达的变化及其在不同生物过程和条件下的作用。转录组学在疾病研究中具有重要作用,为疾病的诊治和个体化治疗提供重要信息。转录组学大语言模型可用于研究疾病相关基因表达模式的表达谱分析、构建调控网络、了解疾病机制等多个方面,为药物靶点发现提供了丰富的生物学信息。
3.1 RNA结构预测RNA的结构变化往往与其功能密切相关,通过预测RNA的二级和三级结构,研究者可以理解其在生物过程中的具体角色,进而发现新的靶点。RNABERT[28]是一种基于BERT架构的预训练模型,专门为二级结构预测和RNA聚类构建。RNABERT可以解决未知序列与现有RNA家族快速精确的结构对齐的实际需求,成为注释新转录物的宝贵工具。RhoFold+通过大规模预训练的RNA语言模型RNA-FM提取序列特征,并结合深度学习模块,采用端到端的方式实现RNA三维结构的预测,解决数据稀缺性[29]。RNA结构预测还可以通过揭示RNA的功能和结合位点,为药物靶点发现及RNA靶向药物的开发提供重要的结构基础。
3.2 基因表达分析2023年5月,Theodoris等发布Geneformer[30]模型,这是转录组计算生物学领域的第一个大模型。Geneformer基于约3 000万个单细胞转录组的大规模语料库进行预训练,在有限的数据下预测基因网络动力学、绘制基因网络图谱、加快发现疾病治疗候选靶点[30]。研究者应用Geneformer针对肥厚型心脏病和扩张型心脏病分别鉴定出了400多个相关基因,筛选出了肥厚型心脏病的候选心肌细胞特异性治疗靶点及可药用靶点,确定了抑制Geneformer预测的扩张型心肌病候选治疗基因在该疾病的实验模型中改善了心肌细胞功能。这些实例验证支持了Geneformer作为发现人类疾病候选治疗靶点的工具的效用。
此外,中国科学院团队发布的GeneCompass[31]是一个多物种生命基础大模型,它能够解析基因调控密码,并显示出加速发现关键细胞命运调节剂和候选药物靶点的巨大潜力。Lomics[32]显著加强转录组研究中生物相关途径和基因集的准确性和深度,同时将转录组数据与其他组学层集成,促进对复杂基因相互作用的理解。此外,还有如scBERT[33]和scFoundation[34]等大型细胞模型,它们也都在单细胞转录组学等领域展现出了强大的应用潜力。
3.3 转录后调控研究转录后调控涉及RNA剪接、编辑、稳定性、转运和翻译等多种机制,对基因表达的精细调控至关重要。SpliceBERT[35]提升了对剪接位点预测的准确性,帮助研究者更好地理解基因表达和剪接变异在生物过程中的作用。长非编码RNA (long non-coding RNA, lncRNA)是一种关键的转录形式,在癌症和疾病的发展中发挥重要的调节作用。有研究发现lncRNA中的小开放阅读框架(small Open Reading Frames, sORFs)可以编码肽,LncCat[36]旨在识别含sORFs的lncRNA,有助于发现新的调节因子。RNA修饰参与多种生物过程和疾病的发生发展,BERT-m7G[37]从RNA序列中有效识别m7G位点,有助于更好地了解m7G对基因功能的影响。转录后调控通过揭示基因表达的动态变化和调控机制,为药物靶点的发现提供了新的视角和方向,推动了新药研发和精准医疗的进程。
4 蛋白质组学大语言模型:加速药靶结构与功能预测在研究基因间调控网络的同时,药物靶点发掘还可以直接学习蛋白质层面的特性。蛋白质在生命过程的构建和生成中起着关键作用,是细胞内大多数生物学过程的执行者,许多疾病的发生与特定蛋白质的功能异常密切相关。通过研究蛋白质的结构、功能和相互作用,可以识别出与疾病相关的靶点,从而开发具有高特异性和疗效的药物。大语言模型通过对蛋白质序列、结构以及组学数据的学习挖掘,在加速数据分析、药物靶点筛选与设计、结构预测等方面展现出强大的应用潜力,提高研究效率、降低成本。
4.1 蛋白质结构预测蛋白质结构在药物靶点发现中至关重要,药物与靶蛋白的结合通常依赖于其三维结构的精确匹配。目前很多已知序列的蛋白质的三维结构仍然未知,这在药物靶点发现和药物设计中是一个重要的挑战。使用X射线晶体学、冷冻电镜等传统实验解析这些蛋白质的三维结构往往投入极大,耗时极长。因此,结构预测技术成为了解决这一问题的重要工具。近年来,深度学习和人工智能技术在蛋白质结构预测中取得了突破性进展。2020年,DeepMind公司推出的AlphaFold2[38]惊艳亮相,AlphaFold2基于同源序列比对方法,能够达到实验手段获取的结构精度。在超过2亿个蛋白质结构预测中,约有35%的结构具有高精度,80%的结构的可靠性足以用于多项后续分析,极大提高了蛋白质结构解析的效率。几乎与之同时发表的RoseTTAFold[39]采用了3轨注意力机制,使整个神经网络能够同时学习3个维度层次的信息,在结构解析方面的表现与AlphaFold2的水平几乎相当。2023年10月31日,Deepmind联合Isomorphic Labs共同发布了AlphaFold3模型[40],引入了扩散模块取代AlphaFold2中的结构模块,减少对同源序列信息的依赖。AlphaFold3能够高准确性预测蛋白质与各种生物分子相互作用的结构,精确度相比前代模型提高了至少50%,并且在一些关键领域甚至提高了一倍。近日,研究者将AlphaFold3和孟德尔随机化相结合,成功确定了七种蛋白质因误义突变而发生结构改变,为阿尔茨海默病的病因解析和潜在药物靶点发现提供了见解[41]。
直接从序列进行结构预测的蛋白质语言模型为蛋白质的三维结构预测提供了一个新思路,并在计算速度和预测准确性方面逐渐显示出优势。2022年,一种端到端可微循环几何网络RGN2[42]被提出,该网络使用AminoBERT蛋白质语言模型从未对齐的蛋白质中学习潜在的结构信息,证明了蛋白质语言模型在结构预测中相对于同源序列比对的实践和理论优势。与此同时,社交网络巨头Meta也正式推出了蛋白质预测模型ESMFold[9],ESMFold是一个基于Transformer的150亿参数语言模型,可以由氨基酸序列直接进行高准确度原子层级的结构预测,在保证准确度的同时,推理速度比AlphaFold2快一个数量级,从而能够在实际时间尺度上探索宏基因组蛋白的结构空间。这些新方法展现出了语言模型从海量蛋白质序列数据库中识别进化模式、结构模式的强大能力[43],为反向对接、结合位点相似性研究提供结构基础。
4.2 蛋白质序列生成随着大数据分析和AI技术的发展,蛋白质序列生成成为了靶点发现的新途径。ProGen2[44]模型具有捕捉复杂序列模式和关系的能力,能够生成表现出预期结构和功能特征的新型蛋白质序列。ProtGPT2[45]建立在GPT-2的自回归性质上,针对蛋白质设计、蛋白质功能预测和了解蛋白质的序列结构关系进行了优化,模型产生的蛋白质表现出符合天然氨基酸原理的氨基酸倾向。模型生成的蛋白质序列不仅遵循生物学规律,而且具备特定的功能,这有助于挖掘尚未被人类生物学研究发现的靶点。同时,可以通过虚拟筛选与已知药物分子进行结合,筛选出可能与药物有良好结合亲和力的蛋白质,进而验证其作为靶点的潜力。
4.3 蛋白质功能预测蛋白质在生物体的细胞代谢、信号转导和结构支持的各个方面都发挥着至关重要的作用,深入了解蛋白质在生物体中的功能对药物靶点发现和疾病机制分析具有重要意义[46]。ProteinBERT[47]在庞大的蛋白质序列数据上捕获复杂的序列模式和生物特征,且该模型展示了广泛用于蛋白质相关任务的多功能性。ProtST[48]是一个面向蛋白质序列与生物医学文本的多模态学习框架,通过融合序列信息与文本描述来提升蛋白质表征质量,从而更有效地推断蛋白质功能。即使在缺乏功能注释的情况下,该模型也能够支持从大规模数据库中识别与功能相关的蛋白质。ESM-1b通过自监督学习的方式,利用大量的未标注蛋白质序列数据进行训练,学习蛋白质序列中的进化信息和氨基酸残基之间的相互作用模式。QuoteTarget[49]是一种改进的基于序列的药物靶蛋白识别方法,它将ESM-1b与图卷神经网络分类器相结合,仅基于序列信息有效地编码蛋白质,并在为本研究构建的非冗余药物靶点和非药物靶点数据集上实现95%的准确率,在应用于人类所有蛋白质时识别出了1 213个潜在的未开发药物靶点蛋白质。
无序蛋白区域(intrinsically disordered region, IDR)是蛋白质序列中没有稳定的三维结构、在常规条件下表现为无序状态的区域。IDR结构的灵活性使它们能够结合许多分子配体,使得它们成为有效的药物靶点。因此,识别蛋白质中的IDR并了解其功能作用将有助于合理的药物设计,并提高新药开发的效率[50]。DisoFLAG [50]是一个用于识别和注释IDR的蛋白质语言模型,它采用了基于序列的预测方法,旨在准确地标定蛋白质中的无序区域及其功能特征,靶向IDR可能成为发现新型药物靶点的有效策略。
4.4 抗原-受体互作与新抗原识别在癌症、免疫疾病和传染病等领域,对抗原与受体相互作用的深刻理解,可能间接推动药物靶点的发现和优化,也为实现个性化治疗提出了新思路。在中国新药研发中,肿瘤是目前最活跃的研究领域[51]。肿瘤新抗原是在癌细胞中由于突变或其他遗传改变而出现的、在正常细胞中不存在的抗原。这些抗原是肿瘤特有的并可触发免疫反应,是癌症免疫疗法的潜在靶点[52]。主要组织相容性复合体(major histocompatibility complex,MHC)分子可以通过将抗原肽与T细胞受体(T cell receptor, TCR)结合来启动免疫反应,TCR是T细胞识别并应答外来病原或肿瘤相关抗原的关键受体分子,在免疫系统中扮演了重要角色。MHCRoBERTa[53]、BERTMHC[54]可以分别用于预测MHC-I和MHC-II分子与肽段的结合亲和力,预测免疫系统中重要的分子交互作用。TCR-BERT [55]利用BERT架构来理解和预测TCR-抗原相互作用,实现更灵活、更准确的抗原结合分析,促进了抗原识别。此外,TCR的互补决定区3 (complementarity determining region 3, CDR3)是抗原肽的直接接触区域,且CDR3很大程度上决定了TCR的多样性。TCR-BERT利用未标记的TCR CDR3序列来学习TCR序列的一般表示,从而使下游任务能够预测TCR的抗原特异性[46]。
4.5 抗体大语言模型近年来,抗体大语言模型在免疫学和生物医学研究中逐渐崭露头角。这些模型采用类似于蛋白质语言模型的思路,可以预测抗体的结构、功能、互作、亲和力等关键属性。AntiBERTa[56]学习抗体的“语言”,可完成跟踪抗体的B细胞来源、量化免疫原性和预测结合位点等任务。ParaAntiProt[57]是一种深度学习辅助的结合表位预测方法,利用预训练的蛋白质和抗体语言模型,提取了高效的嵌入信息用于结合表位预测,该方法仅依赖氨基酸序列且与抗原无关。由于抗体的基因重排、互补决定区多样性等原因,抗体结构预测是蛋白质结构预测领域中的一个重大难点。IgFold[58]是一个在5.58亿自然抗体序列上预训练的语言模型,可以直接预测抗体结构的原子坐标,其预测准确度能与AlphaFold模型相当,但速度更快。AlphaFold3在抗体结构预测方面相比其前身取得了显著的进展,尤其在预测对抗原结合的特异性和亲和力极为重要的重链可变区3 (complementarity determining region H3, CDR H3)方面,AlphaFold3展现了更高的准确性,成功将CDR H3的预测均方根偏差从2.74 Å降至 1.34 Å[40] 。抗体大语言模型使抗体的设计、筛选和优化更加高效,能够为新药靶点的发现提供支持,并为精准医学、疫苗开发以及抗体药物的优化奠定基础。
5 单细胞多组学大语言模型:整合药靶发掘多维数据大语言模型可以分别从基因组学、转录组学、蛋白质组学等维度进行分析,为药物靶点的筛选提出见解。与此同时,单细胞多组学大语言模型通过多维度信息的融合,进一步拓宽了药物靶点发现的视野,挖掘以往无法触及的潜在靶点。在系统医学时代,多组学技术在加速药物发现中发挥着重要的作用[59],多组学分析通过整合基因组学、转录组学、蛋白质组学和代谢组学等不同层面的生物信息,对组学数据进行比较和分析,揭示与疾病相关的通路和关键调节因子,从而筛选出潜在的药物靶点。这种分析方法能够提供更全面的疾病发生发展机制分析,并指导药物设计和优化,以提高药效和减少副作用。
近年来,大语言模型在多组学分析中的应用展现出了巨大的潜力与优势。scGPT[60]利用单细胞多组学数据结合遗传调控的多种视角,在单细胞水平上捕捉了基因与基因之间的相互作用,提供了额外的可解释性。scMVP[61]模型专门为整合单细胞RNA-seq和ATAC-seq数据设计,可在同一细胞中分析基因表达和染色质可及性。DeepMAPS[62]从scMultiomics数据(包括SCRNA-seq、SCATAC-seq和CITE-seq)中进行生物网络推断和数据整合,该模型以基因和细胞为节点构建图,并学习区域和全局特征以建立细胞和基因之间的关系。单细胞多组学数据的研究通过在单细胞水平上整合不同组学技术信息,为解决数据多变性、稀缺性和细胞异质性等难题提供了解决方案[46]。
6 总结和展望药物研发是一个漫长且昂贵的过程,药物靶点发现是研发的核心,涉及识别在疾病中起关键作用的生物分子或途径。然而,由于其极高的难度和复杂性,已确定的成功药物靶点极为有限。实验方法、多组学方法和计算方法等技术的进步和突破推动了药物靶点发现策略的发展,但基于实验及多组学的方法多是资源密集型的,且实验结果严重受到生物样本质量的限制。人工智能和大语言模型正在逐步重塑药物研发的全过程。这些模型基于Transformer架构,通过处理大量文本数据学习语言模式,理解和生成人类语言。本文对用于药物靶点发现的大语言模型进行了总结(表 1),自然语言模型可以进行全面文献综述和专利分析,BioBERT等专用模型通过理解自然语言和解释复杂科学概念,提高了生物医学自然语言处理任务的准确性和效率。此外,大语言模型在基因组学、转录组学、蛋白质组学以及单细胞多组学中也展现出了巨大的潜力。基因组学大语言模型深化了对基因功能、调控和相互作用的理解,提升了对致病变异和基因表达的预测能力。Geneformer等转录组学大语言模型预测基因网络动力学,为药物靶点发现提供丰富的生物学信息。蛋白质组学语言模型的应用在结构和功能预测、药物靶点筛选与设计等方面展现潜力,不断提高研究效率并降低成本。单细胞多组学大语言模型整合不同组学技术信息,揭示疾病相关通路和关键调节因子。这些模型的应用加深了我们对生物学的认识,加速靶点发现和药物研发进程,也为精准医疗和个性化治疗提供了新的可能性。
| 表 1 药物靶点发现中的大语言模型 |
尽管人工智能大语言模型在驱动药物靶点发现中取得了不少进展,但这些技术在实际应用中也面临着诸多问题和挑战。首先,人工智能算法在预测中的可解释性仍有待提高,这对于预测靶点获得科学界和医学界的信任和接受至关重要。目前这些大语言模型在药物靶点发现中的应用集中在通过提高对表达调控网络中关键因子的序列、功能、结构的理解间接预测药物靶点,对药物靶点需具备的特异性、可达性及安全性等其他性质考虑较少,可能会制约预测药物靶点的实际应用。其次,数据偏差对模型训练构成了重大障碍,在有偏差的数据集上训练的大语言模型可能会延续甚至加剧其预测中现有的偏差[18]。解决这个问题需要多样化的训练数据,使得模型在处理背景不同的任务时具有更好的普适性。另外值得注意的是,大语言模型的训练需要大量的数据支持,在数据收集及模型构建的过程中,道德考虑、数据隐私和监管框架也是待解决的关键问题[3]。总之,人工智能大语言模型在药物研发中的应用将继续扩大,提供新的分析方法和靶点发现途径,加速药物研发进程。随着技术的不断进步,大语言模型将使药物靶点发现和新药研发更加高效、经济,并推动行业持续创新,为医药领域带来深刻的变革。
| [1] |
Hinkson IV, Madej B, Stahlberg EA. Accelerating therapeutics for opportunities in medicine: a paradigm shift in drug discovery. Front Pharmacol, 2020, 11: 770. DOI:10.3389/fphar.2020.00770 |
| [2] |
Zhou Y, Zhang YT, Lian XC, et al. Therapeutic target database update 2022: facilitating drug discovery with enriched comparative data of targeted agents. Nucleic Acids Res, 2022, 50: D1398-D407. DOI:10.1093/nar/gkab953 |
| [3] |
Pun FW, Ozerov IV, Zhavoronkov A. AI-powered therapeutic target discovery. Trends Pharmacol Sci, 2023, 44: 561-72. DOI:10.1016/j.tips.2023.06.010 |
| [4] |
Gangwal A, Ansari A, Ahmad I, et al. Generative artificial intelligence in drug discovery: basic framework, recent advances, challenges, and opportunities. Front Pharmacol, 2024, 15: 1331062. DOI:10.3389/fphar.2024.1331062 |
| [5] |
Ren F, Aliper A, Chen J, et al. A small-molecule TNIK inhibitor targets fibrosis in preclinical and clinical models. Nat Biotechnol, 2024, 43: 63-75. |
| [6] |
Pun FW, Liu BHM, Long X, et al. Identification of therapeutic targets for amyotrophic lateral sclerosis using pandaomics - an AI-enabled biological target discovery platform. Front Aging Neurosci, 2022, 14: 914017. DOI:10.3389/fnagi.2022.914017 |
| [7] |
Vaswani A, Shazeer N, Parmar N, et al. Attention Is All You Need[C] //31st Annual Conference on Neural Information Processing Systems (NIPS 2017). Long Beach, CA, USA: Neural Information Processing Systems, 2017
|
| [8] |
Zheng YZ, Koh HY, Yang M. Large language models in drug discovery and development: from disease mechanisms to clinical trials. arXiv, 2024. DOI:10.48550/arXiv.2409.04481 |
| [9] |
Lin ZM, Akin H, Rao RS, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [10] |
AI4Science MR, Quantum MA. The impact of large language models on scientific discovery: a preliminary study using gpt-4. arXiv, 2023. |
| [11] |
Devlin J, Chang MW, Lee K, et al. BERT: Pre-training of deep bidirectional transformers for language understanding. arXiv, 2018. DOI:10.48550/arXiv.1810.04805 |
| [12] |
Savage N. Drug discovery companies are customizing ChatGPT: here's how. Nat Biotechnol, 2023, 41: 585-6. DOI:10.1038/s41587-023-01788-7 |
| [13] |
Zhou HJ, Liu FL, Gu BY, et al. A survey of large language models in medicine: progress, application, and challenge. arXiv, 2023. DOI:10.48550/arXiv.2311.05112 |
| [14] |
Lee J, Yoon W, Kim S, et al. BioBERT: a pre-trained biomedical language representation model for biomedical text mining. Bioinformatics, 2020, 36: 1234-40. DOI:10.1093/bioinformatics/btz682 |
| [15] |
Gu Y, Tinn R, Cheng H, et al. Domain-specific language model pretraining for biomedical natural language processing. ACM Trans Comput Healthcare, 2021, 3: 1-23. |
| [16] |
Luo R, Sun L, Xia Y, et al. BioGPT: generative pre-trained transformer for biomedical text generation and mining. Brief Bioinform, 2022, 23: bbac409. DOI:10.1093/bib/bbac409 |
| [17] |
Park G, Yoon BJ, Luo X, et al. Automated extraction of molecular interactions and pathway knowledge using large language model, Galactica: opportunities and Challenges. [C]//The 22nd Workshop on Biomedical Natural Language Processing and BioNLP Shared Tasks, 2023: 255–64
|
| [18] |
Sarumi OA, Heider D. Large language models and their applications in bioinformatics. Comput Struct Biotechnol J, 2024, 23: 3498-505. DOI:10.1016/j.csbj.2024.09.031 |
| [19] |
Li RF, Li LX, Xu YG, et al. Machine learning meets omics: applications and perspectives. Brief Bioinform, 2022, 23: bbab460. DOI:10.1093/bib/bbab460 |
| [20] |
Ji YR, Zhou ZH, Liu H, et al. DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome. Bioinformatics, 2021, 37: 2112-20. DOI:10.1093/bioinformatics/btab083 |
| [21] |
Yang M, Huang LC, Huang HP, et al. Integrating convolution and self-attention improves language model of human genome for interpreting non-coding regions at base-resolution. Nucleic Acids Res, 2022, 50: e81. DOI:10.1093/nar/gkac326 |
| [22] |
Nguyen E, Poli M, Durrant MG, et al. Sequence modeling and design from molecular to genome scale with Evo. Science, 2024, 386: eado9336. DOI:10.1126/science.ado9336 |
| [23] |
Joachimiak MP, Caufield JH, Harris NL, et al. Gene set summarization using large language models. arXiv, 2023. DOI:10.48550/arXiv.2305.13338 |
| [24] |
Avsec Ž, Agarwal V, Visentin D, et al. Effective gene expression prediction from sequence by integrating long-range interactions. Nat Methods, 2021, 18: 1196-203. DOI:10.1038/s41592-021-01252-x |
| [25] |
Tsukiyama S, Hasan MM, Deng HW, et al. BERT6mA: prediction of DNA N6-methyladenine site using deep learning-based approaches. Brief Bioinform, 2022, 23: bbac053. DOI:10.1093/bib/bbac053 |
| [26] |
Yu YY, He WJ, Jin JR, et al. iDNA-ABT: advanced deep learning model for detecting DNA methylation with adaptive features and transductive information maximization. Bioinformatics, 2021, 37: 4603-10. DOI:10.1093/bioinformatics/btab677 |
| [27] |
Zeng WH, Gautam A, Huson DH. MuLan-methyl-multiple transformer-based language models for accurate DNA methylation prediction. Gigascience, 2022, 12: giad054. DOI:10.1093/gigascience/giad054 |
| [28] |
Akiyama M, Sakakibara Y. Informative RNA base embedding for RNA structural alignment and clustering by deep representation learning. NAR Genom Bioinform, 2022, 4: lqac012. DOI:10.1093/nargab/lqac012 |
| [29] |
Shen T, Hu ZH, Sun SQ, et al. Accurate RNA 3D structure prediction using a language model-based deep learning approach. Nat Methods, 2024, 21: 2287-98. DOI:10.1038/s41592-024-02487-0 |
| [30] |
Theodoris CV, Xiao L, Chopra A, et al. Transfer learning enables predictions in network biology. Nature, 2023, 618: 616-24. DOI:10.1038/s41586-023-06139-9 |
| [31] |
Yang XD, Liu GL, Feng GH, et al. GeneCompass: deciphering universal gene regulatory mechanisms with a knowledge-informed cross-species foundation model. Cell Res, 2024, 34: 830-45. DOI:10.1038/s41422-024-01034-y |
| [32] |
Wong CK, Choo A, Cheng ECC. Lomics: generation of pathways and gene sets using large language models for transcriptomic analysis. arXiv, 2024. DOI:10.48550/arXiv.2407.09089 |
| [33] |
Yang F, Wang WC, Wang F, et al. scBERT as a large-scale pretrained deep language model for cell type annotation of single-cell RNA-seq data. Nat Mach Intell, 2022, 4: 852-66. DOI:10.1038/s42256-022-00534-z |
| [34] |
Hao MS, Gong J, Zeng X, et al. Large-scale foundation model on single-cell transcriptomics. Nat Methods, 2024, 21: 1481-91. DOI:10.1038/s41592-024-02305-7 |
| [35] |
Chen K, Zhou Y, Ding ML, et al. Self-supervised learning on millions of primary RNA sequences from 72 vertebrates improves sequence-based RNA splicing prediction. Brief Bioinform, 2024, 25: bbae163. DOI:10.1093/bib/bbae163 |
| [36] |
Feng HQ, Wang SC, Wang Y, et al. LncCat: an ORF attention model to identify LncRNA based on ensemble learning strategy and fused sequence information. Comput Struct Biotechnol J, 2023, 21: 1433-47. DOI:10.1016/j.csbj.2023.02.012 |
| [37] |
Zhang L, Qin XY, Liu M, et al. BERT-m7G: a transformer architecture based on BERT and stacking ensemble to identify RNA N7-methylguanosine sites from sequence information. Comput Math Methods Med, 2021, 2021: 7764764. |
| [38] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-9. DOI:10.1038/s41586-021-03819-2 |
| [39] |
Baek M, DiMaio F, Anishchenko I, et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science, 2021, 373: 871-6. DOI:10.1126/science.abj8754 |
| [40] |
Abramson J, Adler J, Dunger J, et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature, 2024, 630: 493-500. DOI:10.1038/s41586-024-07487-w |
| [41] |
Yao MH, Miller GW, Vardarajan BN, et al. Deciphering proteins in Alzheimer's disease: a new Mendelian randomization method integrated with AlphaFold3 for 3D structure prediction. Cell Genom, 2024, 4: 100700. DOI:10.1016/j.xgen.2024.100700 |
| [42] |
Chowdhury R, Bouatta N, Biswas S, et al. Single-sequence protein structure prediction using a language model and deep learning. Nat Biotechnol, 2022, 40: 1617-23. DOI:10.1038/s41587-022-01432-w |
| [43] |
Bertoline LMF, Lima AN, Krieger JE, et al. Before and after AlphaFold2: an overview of protein structure prediction. Front Bioinform, 2023, 3: 1120370. DOI:10.3389/fbinf.2023.1120370 |
| [44] |
Nijkamp E, Ruffolo JA, Weinstein EN, et al. ProGen2: exploring the boundaries of protein language models. Cell Syst, 2023, 14: 968-78. DOI:10.1016/j.cels.2023.10.002 |
| [45] |
Ferruz N, Schmidt S, Höcker B. ProtGPT2 is a deep unsupervised language model for protein design. Nat Commun, 2022, 13: 4348. DOI:10.1038/s41467-022-32007-7 |
| [46] |
Liu JJ, Yang MY, Yu YK. Large language models in bioinformatics: applications and perspectives. arXiv, 2024. DOI:10.48550/arXiv.2401.04155 |
| [47] |
Brandes N, Ofer D, Peleg Y, et al. ProteinBERT: a universal deep-learning model of protein sequence and function. Bioinformatics, 2022, 38: 2102-10. DOI:10.1093/bioinformatics/btac020 |
| [48] |
Xu MH, Yuan XY, Miret S, et al. Protst: multi-modality learning of protein sequences and biomedical texts. arXiv, 2023. DOI:10.48550/arXiv.2301.12040 |
| [49] |
Chen JX, Gu ZH, Xu YJ, et al. QuoteTarget: a sequence-based transformer protein language model to identify potentially druggable protein targets. Protein Sci, 2023, 32: e4555. DOI:10.1002/pro.4555 |
| [50] |
Pang YH, Liu B. DisoFLAG: accurate prediction of protein intrinsic disorder and its functions using graph-based interaction protein language model. BMC Biol, 2024, 22: 3. DOI:10.1186/s12915-023-01803-y |
| [51] |
Li GQ, Liu Y, Hu HX, et al. Evolution of innovative drug R & D in China. Nat Rev Drug Discov, 2022, 21: 553-4. DOI:10.1038/d41573-022-00058-6 |
| [52] |
Li T, Li YP, Zhu XY, et al. Artificial intelligence in cancer immunotherapy: applications in neoantigen recognition, antibody design and immunotherapy response prediction. Semin Cancer Biol, 2023, 91: 50-69. DOI:10.1016/j.semcancer.2023.02.007 |
| [53] |
Wang FX, Wang HY, Wang LZ, et al. MHCRoBERTa: pan-specific peptide-MHC class Ⅰ binding prediction through transfer learning with label-agnostic protein sequences. Brief Bioinform, 2022, 23: bbab595. DOI:10.1093/bib/bbab595 |
| [54] |
Cheng J, Bendjama K, Rittner K, et al. BERTMHC: improved MHC-peptide class Ⅱ interaction prediction with transformer and multiple instance learning. Bioinformatics, 2021, 37: 4172-9. DOI:10.1093/bioinformatics/btab422 |
| [55] |
Wu K, Yost K, Daniel B, et al. TCR-BERT: learning the grammar of T-cell receptors for flexible antigen-binding analyses. bioRxiv, 2021. DOI:10.1101/2021.11.18.469186 |
| [56] |
Leem J, Mitchell LS, Farmery JHR, et al. Deciphering the language of antibodies using self-supervised learning. Patterns (N Y), 2022, 3: 100513. DOI:10.1016/j.patter.2022.100513 |
| [57] |
Kalemati M, Noroozi A, Shahbakhsh A, et al. ParaAntiProt provides paratope prediction using antibody and protein language models. Sci Rep, 2024, 14: 29141. DOI:10.1038/s41598-024-80940-y |
| [58] |
Ruffolo JA, Chu LS, Mahajan SP, et al. Fast, accurate antibody structure prediction from deep learning on massive set of natural antibodies. Nat Commun, 2023, 14: 2389. DOI:10.1038/s41467-023-38063-x |
| [59] |
Wang MY, Zhang Z, Liu JF, et al. Gefitinib and fostamatinib target EGFR and SYK to attenuate silicosis: a multi-omics study with drug exploration. Signal Transduct Target Ther, 2022, 7: 157. DOI:10.1038/s41392-022-00959-3 |
| [60] |
Cui HT, Wang C, Maan H, et al. scGPT: toward building a foundation model for single-cell multi-omics using generative AI. Nat Methods, 2024, 21: 1470-80. DOI:10.1038/s41592-024-02201-0 |
| [61] |
Li GY, Fu SL, Wang SG, et al. A deep generative model for multi-view profiling of single-cell RNA-seq and ATAC-seq data. Genome Biol, 2022, 23: 20. DOI:10.1186/s13059-021-02595-6 |
| [62] |
Ma AJ, Wang XY, Li JX, et al. Single-cell biological network inference using a heterogeneous graph transformer. Nat Commun, 2023, 14: 964. DOI:10.1038/s41467-023-36559-0 |
 2025, Vol. 37
2025, Vol. 37 

 应天雷,复旦大学特聘教授、博导,教育部长江学者特聘教授,上海合成免疫工程技术研究中心主任,治疗性疫苗国家工程实验室执行主任,医学分子病毒学教育部/卫生健康委重点实验室副主任,上海市肺部炎症与损伤重点实验室副主任,复旦大学交叉研究发展中心主任,中国生物工程学会抗体工程分委会副主任委员。长期从事抗体工程药物研究,近年来在Cell、Cell Host Microbe等国际知名期刊发表170余篇SCI论文,被引8 000余次,申请30余项专利,完成10余项重大成果转化,推进多项新药临床试验,获美国联邦技术转让奖、盖茨基金会大挑战青年科学家奖、教育部科学研究优秀成果奖一等奖、上海市科技进步奖一等奖、转化医学创新奖、上海市优秀学术带头人、上海青年科技英才、上海市青年五四奖章等
应天雷,复旦大学特聘教授、博导,教育部长江学者特聘教授,上海合成免疫工程技术研究中心主任,治疗性疫苗国家工程实验室执行主任,医学分子病毒学教育部/卫生健康委重点实验室副主任,上海市肺部炎症与损伤重点实验室副主任,复旦大学交叉研究发展中心主任,中国生物工程学会抗体工程分委会副主任委员。长期从事抗体工程药物研究,近年来在Cell、Cell Host Microbe等国际知名期刊发表170余篇SCI论文,被引8 000余次,申请30余项专利,完成10余项重大成果转化,推进多项新药临床试验,获美国联邦技术转让奖、盖茨基金会大挑战青年科学家奖、教育部科学研究优秀成果奖一等奖、上海市科技进步奖一等奖、转化医学创新奖、上海市优秀学术带头人、上海青年科技英才、上海市青年五四奖章等