蛋白质作为生命活动的主要执行者,其序列、结构与功能研究一直是生物学关注的核心。蛋白质大模型特指利用大规模深度学习架构(通常是基于Transformer[1]或其变体),在海量蛋白质序列和(或)结构数据上训练得到的模型。蛋白质大模型代表了深度学习在生物信息学、结构生物学和整个生命科学领域的革命性突破,极大地加速了我们对生命基本组成部分的理解和调控能力。
蛋白质计算模型的发展经历了从基于物理的模拟和同源建模,到采用经典机器学习模型,再到当前以Transformer架构为核心的深度学习大模型的演变历程。这一转变的关键驱动在于基因测序技术爆发式增长带来的海量序列数据(如UniProt[2]数据库已达数十亿级别),以及PDB等结构数据库提供的宝贵三维结构信息(约20万+)。传统方法无法捕捉序列中的长程依赖和复杂折叠规律,而大模型通过自监督学习(如掩码语言建模)从海量数据中蒸馏进化、结构和功能的深层关联,形成了不同层次的蛋白质大模型,使人类首次具备了系统性地设计功能蛋白质的能力。这一领域的里程碑式突破——AlphaFold2[3]的成功,标志着蛋白质结构预测问题在很大程度上得到解决,并将研究焦点进一步推向蛋白质动态构象预测、复杂生物分子组装体建模以及功能导向的蛋白质生成。
这些模型包括蛋白质大语言模型(protein large language model, PLLM)、蛋白质结构预测模型、蛋白质性质与功能预测模型和蛋白质生成模型。就像语言大模型(如GPT系列)学习人类语言的统计规律和语义一样,蛋白质大模型学习氨基酸序列中蕴含的进化、结构和功能的深层模式与规则。给定一个氨基酸序列(一级结构),蛋白质结构预测模型可预测其折叠成的三维结构(三级结构),这是理解蛋白质功能的关键。蛋白质性质与功能预测模型可以利用或超越结构,预测蛋白质的稳定性、溶解度、与其他分子的相互作用(如蛋白质-蛋白质、蛋白质-配体、蛋白质-DNA)、功能位点、突变效应等。而生成式人工智能在生成新型蛋白质方面,通过学习蛋白质的“语法”,可以设计自然界中不存在但具有特定功能和特性的全新蛋白质序列。
1 蛋白质语言模型的核心架构大模型通过在超大规模数据上预训练,学习到通用的、可迁移的蛋白质表示(Embeddings)。这种表示编码丰富的生物物理和进化信息,可以作为下游各种任务(结构预测、功能预测、设计)的强大起点,显著减少对特定任务标注数据的需求(迁移学习)。随着深度学习技术的发展,蛋白质语言模型经历了从基于循环神经网络(RNN)到基于注意力机制的架构演进,在模型规模、预测精度和应用范围等方面不断突破。特别是近年来Transformer架构的引入,使得模型能够更好地捕捉蛋白质序列中的长距离依赖关系,为结构生物学、蛋白质工程和药物设计等领域带来了革命性变化。
蛋白质序列与自然语言的token序列具有天然的相似性:氨基酸可以类比为单词,蛋白质序列可以看作是由这些“单词”组成的“句子”,而蛋白质家族则类似于“语料库”中的不同主题文本。这种结构上的相似性促使研究者将自然语言处理技术引入蛋白质研究领域。语言模型(LM)作为从大规模序列数据库中学习“内容感知”数据表示的强大范式出现[4],被广泛用于自然语言处理(NLP)中的机器翻译、问答,甚至扩展到计算机视觉、分子等领域。由于蛋白质和人类语言之间的相似性,LMs逐渐演化为蛋白质语言模型(pLM)来处理各种蛋白质数据,专门匹配蛋白质序列以学习可用于结构与功能的表示。受NLP方法的启发,蛋白质表示学习成为一个活跃的研究领域,学习用于各种下游任务的表示[5]。然而,特定任务的标记蛋白质可能非常稀缺,因为标记蛋白质功能通常需要耗时且资源密集的实验室验证。为了缓解甚至解决这个问题,蛋白质语言模型通常开发为预训练模型,通过在源任务上预训练模型获得知识,并通过在具有较少标签的新目标任务上微调模型来改善学习。
蛋白质大模型的预训练核心在于通过自监督学习范式,从海量无标注蛋白质序列中蒸馏进化蕴含的通用生物规律。不同于依赖有限标注数据的传统方法,预训练模型利用蛋白质序列自身的统计特性构建学习目标:最典型的是掩码语言建模(Masked Language Modeling, MLM)——随机遮盖序列中部分氨基酸残基,给定周围序列重建被破坏的token,迫使模型根据上下文预测被遮盖单元。这一过程使模型隐式捕获残基间的共进化约束、空间邻近关联及折叠稳定性的物理化学规则。早期的预训练序列编码器包括TAPE Transformer[1]、ProteinBert[6]、ProtTrans[7]和ESM-1b[8],它们通过预测序列中的掩码残基进行训练。模型通过处理数十亿级宏基因组数据(如Meta的ESM模型训练自2.5亿条序列),学习到跨越数百万物种的进化保守性模式,例如跨物种高度保守的活性位点残基组合、跨蛋白家族共享的结构域组装逻辑。这些知识被编码为高维向量表示,形成一种蛋白质生物语义的通用词典。
在预训练策略方面,研究者们也进行了多种创新探索,不仅预测单个被掩码的氨基酸,还考虑多个掩码位置之间的相互依赖关系,这种设计更好地模拟了蛋白质进化过程中的协同突变现象。CPCProt[9]则采用了对比学习策略,通过最大化序列表示与结构特征的交互信息,使模型学习到更具生物学意义的嵌入空间。这些创新不仅提升了模型性能,也丰富了我们对如何从序列数据中提取结构功能信息的理解。
预训练表示的本质是功能结构的数学映射器。当模型处理新序列时,其输出的表示可携带多重信息,比如物理化学属性(如疏水性、电荷分布)、结构倾向性(如二级结构偏好、环区/螺旋区概率)以及功能潜势(如可能的催化残基或蛋白结合界面)。这种表示具有强大的迁移能力:只需微调轻量级下游模块,即可应用于突变致病性预测(如从表示变化推断稳定性损失)、抗原-抗体结合热点识别,甚至跨物种功能注释等任务。例如AlphaFold的Evoformer在预训练阶段学习的共进化模式,使其能从未知蛋白的MSA中提取出决定β折叠层堆积角度的关键残基对相互作用。这种预训练范式将蛋白质序列转化为机器可读的生物语义载体,成为解码结构-功能关系的通用计算基石。ConPLe[10]利用预先训练的蛋白质语言模型(PLex),结合蛋白质锚定的对比共嵌入(Con),实现通过蛋白质语言空间的对比学习来预测药物和目标蛋白质之间的相互作用。
蛋白质语言模型毕竟不是真正的自然语言模型,具有缺乏自然语言能力和指令理解不足的局限性。ProLLaMA利用预训练的通用LLM (例如LLaMA2)持续学习蛋白质语言,同时保持其自然语言知识,这样持续预训练的模型具备基本的蛋白质序列处理能力,并保留其原有的自然语言理解和生成能力。在此基础上,使用多任务指令数据集对模型进行微调,使其能够理解和执行各种蛋白质语言处理任务,让模型学会根据指令完成蛋白质生成、性质预测等任务[11]。
在深度学习技术应用于蛋白质序列分析的早期阶段,长短时记忆网络(LSTM)及其变体成为主流架构选择。这类模型通过门控机制控制信息流动,能够在一定程度上解决传统RNN存在的梯度消失问题,适合处理蛋白质序列这类具有时序特性的数据。UniRep[12]是这一时期的代表性工作,它创新性地使用乘法LSTM (mLSTM)架构,将任意长度的蛋白质序列压缩为固定长度的向量表示。这种方法的最大优势在于完全基于序列信息,无需任何结构或进化数据即可生成具有泛化能力的蛋白质表示,为后续研究提供了重要启示。
NetSurfP-2.0[13]采用了CNN与双向LSTM (BiLSTM)的混合架构。这种设计结合了CNN在局部特征提取方面的优势和BiLSTM在序列建模方面的能力,能够同时预测蛋白质的多种结构特征,包括二级结构、溶剂可及性等。特别值得注意的是,该模型的输入不仅包含原始序列,还整合了多序列比对(MSA)信息,通过结合进化信息提升了预测精度。SPIDER3-Single[14]则展示了另一种思路,它仅基于单序列输入,通过LSTM-BRNN架构就能有效预测二级结构,摆脱了对MSA信息的依赖,为处理缺乏同源序列的蛋白质提供了可行方案。
蛋白质大语言模型学习到的序列表示天然蕴含了功能信息,使其能够直接或通过微调用于蛋白质功能预测。例如,ESM系列模型的嵌入已被成功用于预测酶的EC编号、基因本体(GO)术语注释以及蛋白质-蛋白质相互作用; DeepFRI[15]等模型进一步结合蛋白质结构和序列信息,使用图卷积网络对蛋白质结构进行建模,实现了更精确的功能注释; 对于突变效应的评估,ESM-1v模型展示了在仅凭序列的情况下,通过零样本预测(zero-shot prediction)即可达到与基于多序列比对的传统方法相媲美的性能,能够识别出导致疾病或影响功能的错义突变。这些功能预测能力将蛋白质大模型的应用从结构层面延伸至功能层面,为系统性的功能注释和致病性突变解读提供了强大工具。表 1对几种主流蛋白质语言模型进行了总结。
| 表 1 几种主流蛋白质语言模型的总结 |
ProSE[4]模型代表了LSTM-based架构的进一步创新,它在传统语言模型训练目标的基础上,引入了残基接触预测和结构相似性预测等结构监督任务。这种多任务学习框架使模型能够更好地捕捉蛋白质的结构语义信息,生成的嵌入表示包含了更丰富的结构特征。然而,尽管这些基于LSTM的模型取得了一定成功,其固有的序列计算特性导致处理长蛋白质序列时效率低下,且难以建模复杂的全局依赖关系,这些局限性最终促使研究者转向更具潜力的Transformer架构。
Transformer架构凭借其独特的自注意力机制,彻底改变了蛋白质语言模型的发展轨迹,已成为当前主流范式。与LSTM不同,Transformer能够直接建模序列中任意两个氨基酸之间的相互作用,不受距离限制,这对理解蛋白质的三维结构和功能至关重要。在蛋白质结构中,空间上相距很远的氨基酸可能通过折叠发生关键相互作用。Transformer架构的自注意力机制天然擅长捕捉这种长程依赖关系,这是传统CNN或RNN难以企及的。ESM-1b[8]是这一方向的里程碑式工作,它采用深层Transformer架构,通过掩码语言模型(MLM)预训练任务,使模型学会从上下文预测被掩码的氨基酸。这一过程使模型隐式地学习到了残基间的共进化信息,生成的嵌入表示在多种下游任务中表现出色。Evoformer[18]作为AlphaFold2的核心组件,展示了Transformer架构在蛋白质结构预测中的惊人潜力。
2 蛋白质结构预测大模型:单体与复合物,静态结构与动态结构蛋白质结构预测领域近年来取得了革命性进展,这很大程度上归功于专用蛋白质语言模型的创新。AlphaFold2 (AF2)[3]和RosettaFold[18]的成功标志着这一领域的重要转折,激发了应用pLMs解决尚未解决的挑战性问题,包括使用pLMs进行无进化信息的蛋白质结构预测、预测蛋白质复合物结构[19]、发现蛋白质折叠背后的机制等。
2.1 进化信息(MSA-多序列比对)与结构信息的关联蛋白质结构大模型的核心技术和流程通常包括:
2.1.1 输入表示:序列输入氨基酸序列被转化为数值向量(Embeddings)。除了基本的氨基酸类型Embedding,通常还会加入:
A:位置编码/Embedding。这表示氨基酸在序列中的位置。
B:进化信息(MSA-多序列比对)。这是最关键的输入之一。模型通常输入一个代表目标序列及其进化相关序列(同源序列)的MSA。MSA提供了强大的进化约束信号,暗示了哪些位置是保守的(对功能或结构至关重要),哪些位置可以共同进化(暗示空间接近或相互作用)。MSA信息通常通过构建“Pair Repression”(表示每对氨基酸序列的共进化关系)和“MSA Repression”(表示整个比对信息)输入模型。
C:模板信息(可选)。如果已知结构相似的蛋白质(模板),其结构信息可以作为额外的输入线索。
2.1.2 核心模型架构A:基于Transformer的变体。这是主流架构,但进行了重要领域适应性改造。Evoformer (AlphaFold2核心):DeepMind在AlphaFold2中提出的革命性架构。它不是单一Transformer,而是由两个主要模块组成。MSA Stack:处理MSA表示,在行(序列)和列(比对位置)两个维度上应用改进的注意力机制,提取序列内和序列间的进化信息。Pair Stack:处理Pair Representation (氨基酸对表示)。它使用轴向注意力(沿行和列分别操作,降低计算复杂度)和外积等操作,不断更新表示每对氨基酸之间相互作用可能性的矩阵。MSA Stack和Pair Stack之间有密集的信息交换,让共进化信息和成对相互作用信息深度融合。改进的注意力机制:引入三角不等式感知的注意力、门控机制等,更好地建模蛋白质结构的几何约束(如距离、角度)。
B:几何表示与约束。模型内部或输出端会显式地预测和利用几何信息。距离/距离分布:预测每对氨基酸残基之间的距离(或距离的概率分布)。二面角:预测主链的二面角(Phi, Psi)。框架:预测每个残基的局部坐标系。
C:迭代精炼。模型通常是迭代的(如AlphaFold2的“结构模块”),将初步预测的结构信息反馈回网络,进行多轮精炼,逐步优化预测结果。
2.1.3 训练目标A: 监督学习。在已知结构的蛋白质数据集(如PDB)上训练。损失函数通常结合多个目标。结构损失:预测结构与真实结构之间的差异。常用Frame Aligned Point Error衡量局部结构准确性,Distance Matrices Error衡量全局折叠准确性。辅助损失:预测接触图、二面角、溶剂可及表面积等的准确性。
B: 自监督学习。利用海量无标签序列数据(如UniRef)进行预训练。常见任务包括以下三种。掩码语言建模:随机掩盖序列中的部分氨基酸,让模型预测被掩盖的部分(类似BERT)。对比学习:学习区分相似序列(同源)和不相似序列。进化相关预测:预测序列间的进化距离或是否同源。
2.1.4 输出A: 三维原子坐标。AF3直接输出蛋白质主链和侧链原子的3D坐标。
B: 置信度分数。对预测结构的局部(每个残基)和全局可靠性进行评分(如AlphaFold2的pLDDT和pTMscore)。
C: 中间表示。模型学习到的MSA Embeddings和Pair Embeddings本身也是强大的输出,可用于其他下游任务。
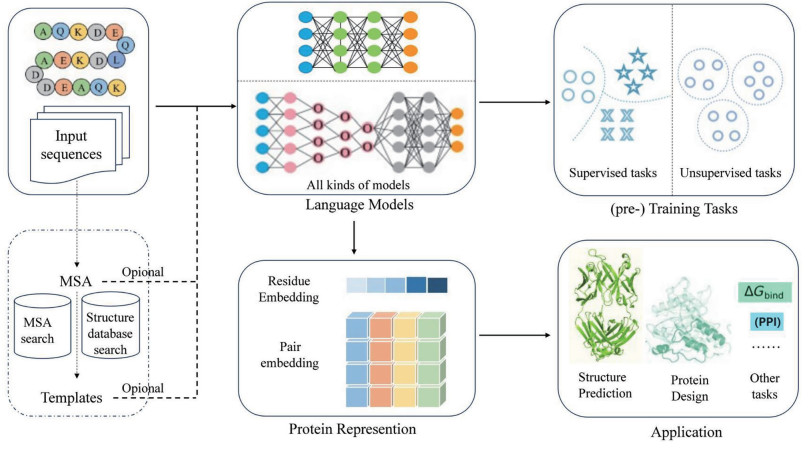
基于语言模型的蛋白质表示学习与应用流程总结见图 1。
|
先输入蛋白质序列(可加MSA/模板等辅助信息),经各类语言模型提取单氨基酸、氨基酸配对的特征,再通过有/无监督任务训练模型,最终用这些特征完成蛋白质结构预测、设计及结合自由能等功能任务。 图 1 基于语言模型的蛋白质表示学习与应用流程 |
AF2的优异表现得力于两个技术的应用:(1)多序列比对(MSA)对空间特征结构的关联; (2)使用Transformer处理多序列比对(MSA)与结构信息,融合进化与空间特征,构建残基间关联。它创新性地设计了轴向注意力机制,分别处理MSA表示中的行(序列)方向和列(位点)方向信息,通过交替更新这两种表示捕获不同层次的进化约束,将MSA信息和残基对表示深度融合。特别值得注意的是,Evoformer创新的结构模块(IPA)引入了三角不等式约束,确保预测的距离矩阵满足几何一致性,这种将物理约束融入深度学习框架的思路对提升预测精度起到了关键作用。
进化标度建模(Evolutionary Scale Model, ESM)系列是目前最具代表性的蛋白质语言模型家族,NetSurfP-3.0[20]利用ESM-1b替代传统MSA流程,将结构特征预测的运行时间缩短了两个数量级以上,同时保持与之前方法相当的精度。这类工具的出现使得大规模蛋白质组的结构注释成为可能,为系统生物学研究提供了重要支持。从ESM-1b (6.69亿参数)到ESM-2 (150亿参数)又产生了能力的巨大提升。ESM-2[21]代表了另一条结构预测技术路线,它仅依靠单序列信息,通过大规模预训练就能获得高质量的结构预测能力。尽管ESM-2已蕴含蛋白质的进化信息,但其预测接触时依赖的是局部序列上下文的共进化信息,而非完整的蛋白质折叠(fold)信息[22]。这一特性挑战了“必须依赖大量同源序列才能实现准确结构预测”的传统认知,为缺乏同源序列的“孤儿蛋白”结构预测提供了新思路。OmegaFold进一步推进了这一方向,它采用门控注意力单元(GAU)替代标准Transformer中的注意力机制,配合几何变换保持空间一致性,在计算效率和预测精度之间取得了良好平衡。但是就精度而言,对人源蛋白酶的系统结构预测表明AlphaFold2比ESMFold更为精确[23]。
2.2 从单体到复合物从单链蛋白质结构预测进一步到蛋白质复合物的结构预测并非顺理成章,基于AF2算法,AlphaFold-Multimer[19]做了部分调整以满足复合物结合界面结构的特殊需要。近来发现,对于天然无序蛋白形成的复合物,AlphaFold-Multimer常常也能预测出正确的复合物结构[24]。为了改善计算机内存和算力需求,AlphaFold-Multimer对蛋白质进行裁剪,这些裁剪区域是最多可达384个残基的连续的残基块,裁剪区域包含多个链,力求扩大链覆盖度、截断片段多样性,从而能更好地预测结合界面的结构。AlphaFold-Multimer添加了额外的位置编码来表示给定的一对氨基酸是否对应于不同的链,以及它们是否属于不同的同源链或异源链。AlphaFold模型用predicted TM-score (pTM)估计内在模型精度。AlphaFold-Multimer采取类似方案,但是更专注于界面预测的准确性。因此建立了不同链残基之间相互作用的评分系统——Interface pTM (ipTM)。
随着蛋白质链数增加,AlphaFold等方法的精度显著下降,且受GPU内存限制难以直接处理。针对这一问题,研究者提出了基于子组件预测和蒙特卡洛树搜索(MCTS)组装的创新策略[25]。该方法首先用AlphaFold-Multimer预测二聚体和三聚体子结构,然后以mpDockQ (结合界面plDDT和接触数的复合指标)为指导,通过MCTS探索最优组装路径。在175个10~30链的测试复合物中,成功组装91个,其中30个达到高精度(TM-score≥0.8)。分析表明,子组件质量、复合物对称性和有效序列数是影响组装成功的关键因素。与传统方法相比,这种分层组装策略具有明显优势。一方面,它突破了全原子建模的计算限制,使大型复合物预测成为可能; 另一方面,MCTS的启发式搜索大大减少了构象空间探索的复杂度。特别值得注意的是,该方法对于对称复合物的预测效果显著优于不对称复合物。
2.3 扩散模型是蛋白质大模型的又一个里程碑AlphaFold3 (AF3)[26]的发表表明蛋白质单链和复合物静态结构预测达到新的历史性高度。AF3核心除了改进Evoformer模块,其强大功能源于其新一代的架构和训练。AF3使用生成式扩散(diffusion)网络进行预测,扩散过程从加噪的原子坐标开始,最终直接生成准确的分子结构。AF2只是预测蛋白质的主链原子结构,然后用分子力学的方式引入和优化侧链。采用生成式扩散模型,AF3直接预测蛋白质单链和复合物静态结构,同时包括蛋白质和核酸、小分子、离子的复合物。在蛋白质-配体相互作用方面的预测精度远高于当前最先进的分子对接工具,在蛋白质-核酸相互作用方面的预测精度远高于专门针对核酸的预测工具,在抗体-抗原相互作用方面的预测精度也显著高于AlphaFold-Multimer v2。
蛋白质生成式扩散模型的另一个成功的范例是RFdiffusion[27]系列模型,它将RoseTTAFold结构预测模型微调为去噪生成器,可以在无条件或条件约束下生成蛋白质结构和支架,并与下游用于序列生成的ProteinMPNN[28]、LigandMPNN[29]一起,构成了适用于各种蛋白质设计应用的完整工具箱。与AF3类似,RoseTTAFold All Atom (RFAA)超越了多肽链限制,涵盖了整个生物分子系统的复杂性和多样性,包括蛋白质、核酸、小分子、金属离子和各种共价修饰。RFAA允许基于输入序列和化学结构对复杂的生物分子组件进行建模。通过对去噪任务进行微调来细化RFAA创建的RFdiffusion All Atom (RFdiffusionAA)[30]能够产生专门容纳感兴趣的小分子的de novo蛋白质结构。将RFdiffusion用于抗体设计的RFantibody在抗体设计上也得到冷冻电镜结果的验证。设计的抗体-抗原复合物的CDR与实验相差无几(RMSD值分别为:CDRH1=0.4 Å,CDRH2=0.3 Å,CDRH3=0.7 Å; CDRL1=0.2 Å,CDRL2=1.1 Å,CDRL3=0.2 Å)[31]。
扩散模型在蛋白质大模型中得到广泛应用。GeoFlow-V2是一个基于原子的扩展模型,同时具备对蛋白质、核酸、小分子进行结构预测和蛋白质从头生成的功能[32]。GeoFlow-V2同时也考虑实验约束和前期知识的输入作为辅助条件。Chroma[33]引入了一种尊重聚合物构象统计的扩散过程,通过亚二次方缩放将高位分布降低为简单分布,用低温采样来提高采样骨架的结构准确,并纳入对称性、片段约束、结构语义甚至自然语言提示等条件,能够实现更加新颖的结构和更大尺寸蛋白质复合物的计算设计。TopoDiff[34]利用扩散模型的生成能力和变分自编码器(VAE)对特征空间的表征和压缩能力,能够更好地全局拓扑特征,提高骨架生成的覆盖性,特别是对β拓扑结构的采样,设计出了主要为β二级结构的新折叠。
蛋白质在真实细胞环境中并非静态结构,而是通过构象变化、动态组装和变构效应执行功能,如多亚基复合物的精确组装、信号转导中的构象转换,以及与核酸/配体/膜环境的瞬时互作。现有模型主要基于静态晶体结构数据训练,难以捕捉这些涉及毫秒级时间尺度和能量景观变化的动态过程。扩散模型的应用也使蛋白质结构预测从单一的静态结构预测进入到预测蛋白质的动态构象分布。这一方向首先的尝试是利用分子动力学(MD)模拟产生蛋白质的动态构象,然后利用深度学习产生的数据集来预测动态构象分布。微软亚洲研究院Distributional Graphormer利用已有的GPCR模拟数据集和补充的分子动力学模拟结果训练的模型,能够预测一些蛋白质的动态构象[35]; 微软的另一个团队开发了BioEmu-1,本质上也是基于分子动力学模拟产生蛋白质的动态构象作为训练集,做到了更为精确的效果[36]。Deepconfomer采用了不同的路径,为了避免分子动力学中分子力场精度的限制,只利用已有的实验结构作为训练集,该模型学习了实验结构中隐含的能量景观和动态关联,利用扩散模型预测的蛋白质动态结构能够复现分子动力学模拟的氨基酸平均波动(RMSF)和单点突变引起的大幅度构象转变[37]。
尽管蛋白质大模型共享相似的底层架构(如Transformer),但它们在设计理念、输入要求、资源消耗和适用场景上存在显著差异,这些差异决定了其具体应用。为提供更清晰的视角,表 2系统比较了代表性模型的关键特征。从上表可以看出,模型选择存在明确的权衡。AlphaFold2在精度上无可匹敌,但以计算资源和时间为代价; ESMFold[38]和OmegaFold[39]提供了快速但精度稍逊的替代方案,尤其适用于缺乏同源序列的场景。在生成任务中,RFdiffusion系列在结构创新性上领先,而ProGen2[17]则在序列的自然度和属性控制上表现出色。对于复杂的生物分子相互作用,AlphaFold3是目前最全面的工具,但其可用性目前受到限制。理解这些模型的比较优势,对于研究者根据具体任务(是追求精度还是速度,是进行预测还是设计)选择合适的工具至关重要。
| 表 2 主流蛋白质大模型特征比较 |
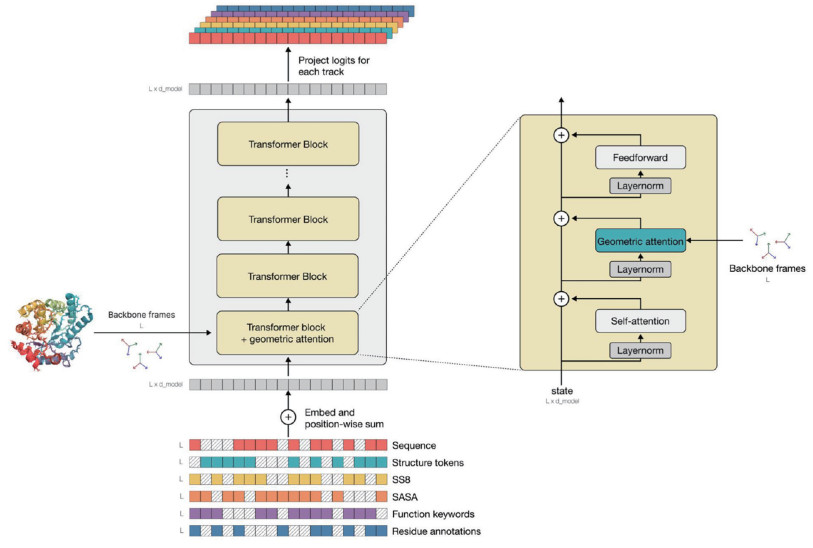
ESM系列最新模型ESM3作为多模态生成模型,统一编码序列、结构、功能令牌,通过提示组合实现可编程设计,能够推理蛋白质序列、结构和功能。ESM3的训练过程囊括了地球自然环境的多样性——数十亿种蛋白质,其规模比上一代ESM大大扩展,数据量提高了60倍,训练计算量提高了25倍,并且具有原生多模态的生成模型。ESM3随规模增加逐渐涌现能力,其中一个重要的能力就是原子级协调,使蛋白质设计任务达到原子级精度。ESM3利用了具有SE(3)不变性的几何注意力机制,通过变换来实现局部参考框架和全局框架的交互,将局部和全局框架加入到常规注意力计算中,形成的几何注意力机制使ESM3模型能够高效处理蛋白质的三维结构信息。如图 2所示,第一个transformer模块中的几何注意力层允许模型直接使用用户提供的原子坐标,大幅提升了ESM3对原子坐标的处理能力。应用实例如:用ESM3生成与天然荧光蛋白序列差异达58%的新型荧光蛋白esmGFP,荧光强度与天然蛋白相当; 将丝氨酸蛋白酶活性位点移植至全新折叠架构,序列长度压缩33%仍保持催化功能[40]。
|
ESM3能够综合推理蛋白质的序列、结构和功能,该模型将三者分别表示为输入输出端的token轨道。在训练过程中,系统对各轨道随机采样掩码,并在输出端预测被掩蔽的token位置。 图 2 ESM3架构详解 |
除序列信息之外,在蛋白质预训练模型中也开始融合结构信息,融合了结构信息的蛋白质多模态预训练模型取得了较大进展[41]。SaProt利用Foldseek将蛋白质进行编码,生成了一维的3D结构序列(使用了Foldseek的结构词表,每种3D token代表不同的局部结构),这样的结构序列与氨基酸序列是等长的。因此,Su等[42]使用了一种简单而有效的结构嵌入方式:利用结构词表和氨基酸词表计算笛卡尔积(即两两组合),形成新的结构感知词表。这样对于蛋白质的每个位点,其氨基酸类型和对应的局部结构都能组合成新词表中的某个元素,从而让模型同时考虑到蛋白质的序列与结构信息。随后,利用Bert架构进行掩码语言建模预训练,得到结构感知的融合序列与结构蛋白质语言模型SaProt。直接把结构的细节信息,如主链Psi和Phi选择角度、侧链角度以及alphafold2 pLDDT分数等信息,汇入序列信息微调ESM-2,也可以得到与SaProt相当或更好的模型[43]。
清华大学团队开发的ESM-AA是首个融合氨基酸与原子信息处理的蛋白质预训练语言模型[44]。模型通过整合多尺度信息,展现出稳健且卓越的性能。ESM-AA的多尺度预训练目标包括掩码语言建模和成对距离恢复(PDR)。掩码语言建模通过遮盖氨基酸和原子,要求模型根据周围的上下文进行预测,这一训练任务可以在氨基酸和原子两个尺度上进行。而PDR则要求模型准确预测不同原子之间的欧几里得距离,以训练模型理解原子级的结构信息。
3 蛋白质设计与生成大模型由于蛋白质的序列空间非常庞大,从头设计通常被拆解为骨架生成和序列搜索两个步骤。在骨架生成步骤中,一般需要产生的骨架二级结构规范、折叠良好,以提高稳定性和可折叠性,这样一个可设计的骨架能够先验地限制序列采样空间。TopoBuilder[45]将蛋白质骨架的拓扑参数化为理想二级结构元件的分层排列,构建连接环,并在Rosetta能量函数的指导下优化拓扑元件的排列组装,从而使蛋白质骨架类似天然的结构,提高可设计性。
3.1 流匹配模型的兴起去噪扩散概率模型和流匹配模型是当前应用于蛋白质骨架生成的两个相似的深度学习框架,它们通过学习天然蛋白质结构的分布,来生成相似的结构输出,也有一些研究试图综合两者的特点[46]。最近的一些模型比如OriginFlow,表明流匹配生成的蛋白质成功率更高,在无约束蛋白质单体的生成、功能基序支架设计、结构约束的蛋白生成、指定靶点的结合蛋白设计等方面表现优异[47],在17个后验功能位点设计任务中,OriginFlow成功完成16个(> 94%),成功率领先RFdiffusion、EvoDiff等当前主流模型,RMSD均值< 1 Å,预测结构pLDDT > 85。Proteina是另一个新型、大规模的基于流的蛋白质骨架生成器。它利用层次化的折叠类别标签(hierarchical fold class labels)进行条件控制,并依赖于一个可扩展的Transformer架构,其训练数据扩展到了数百万个合成蛋白质结构。为了更好地适应蛋白质骨架生成,训练和采样包括了针对蛋白质骨架的LoRA微调策略、新的引导方法(如适用于蛋白质骨架的无分类器引导(classifier-free guidance)和自动引导(autoguidance),以及新的调整训练目标。Proteina的亮点在于能生成长度前所未有(高达800个残基)的蛋白质,利用特定折叠合成技术在生成结构中成功实现了β折叠结构的可控增强[48]。
Jing、Berger和Jaakkola把AlphaFold和ESMFold与流匹配模型结合用来生成蛋白质构象的系棕[49]。P2Dflow利用分子动力学模拟的构象训练流匹配模型用来预测蛋白质构象的系棕分布[50]也取得了良好的结果,为了区分具有不同能量的构象集,模型引入了“近似能量”这一新维度。这一维度通过将分子动力学模拟的结果投影到由回转半径(RG)和均方根偏差(RMSD)定义的二维平面上,计算高斯核密度来获得。这样的设计使得模型在生成时能够更好地避免非存在的中间状态。
3.2 蛋白质序列的生成和蛋白质结构-序列的共生成蛋白质序列的生成可以分为基于特定蛋白质特征的序列生成、基于蛋白质骨架的序列生成和无限制的蛋白质序列生成。设计能够自主折叠成给定骨架的氨基酸序列,也称为蛋白质的逆折叠问题。早期基于物理的解法一般将其视为一个优化问题,具体方法是基于残基分布的统计规律和最小化能量函数来设计,如ABACUS[51]和RosettaDesign[52]。然而,人工设计的能量函数难以考虑复杂的高阶非线性耦合,深度学习作为一个万能函数,可以不依赖于现行近似。
深度学习的训练策略一般是对残基进行掩码,然后让模型根据蛋白质的全局或局部结构进行推断恢复。早期的尝试一般基于编码器-解码器的架构,并使用不同的模型架构来表征蛋白质,例如ProteinMPNN[28]使用的消息传递神经网络MPNN,和ABACUS-R[53]使用的Transformer。AlphaFold系列工具的问世为人们提供了建模序列-结构关系更加精细化和模块化的方法,反转AlphaFold2网络架构用于学习单点和成对残基表示,将蛋白质的三维结构转化为一维序列也被提出[54]; CarbonDesign[55]在此基础上,在序列循环搜索中整合ESM2序列嵌入,将进化和结构约束融合起来; RSO[56]直接通过基于AlphaFold2网络的梯度下降来更新输入序列,使其接近骨架结构,这种方法也被称为“幻觉”。
ProGen是一个通用的蛋白质序列大语言模型,用了2.81亿去冗余的蛋白质序列训练,训练时采用了两类控制标签:(1)关键词标签; (2)分类标签。关键词标签遵循UniProtKB受控层级化关键词词表(其中许多关键词源自基因本体(GO)术语)的定义,控制关键词标签涵盖了1 100个术语,包括细胞组分、生物过程和分子功能三大范畴的术语。分类标签则包含来自NCBI分类体系的100 000个术语,涵盖了八级标准分类阶元,这样ProGen可以按照标签生成特定蛋白质特征序列[57]。若采用更大的Profluent Protein Atlas v1数据集(包含34亿个全长蛋白质和1.1万亿个氨基酸标记),升级的ProGen3能够生成更为多样化且功能真实的蛋白质,准确率比小模型高出近两倍,显示出更广泛的生物学潜力[58]。进一步,研究人员通过使用来自五个溶菌酶的55 948条序列对模型进行微调并生成了100万个合成序列。依据氨基酸语义语法的自然程度,选择100个在实验中进行表达量与活性的测试。通过色谱峰和条带可视化测定发现,72%的蛋白质表达良好,即使与自然蛋白差异不断增大,人工蛋白也能表达良好,并且与100个具有代表性的天然蛋白表达质量相当。在活性测试中,73%的蛋白质序列表现出了与鸡蛋清溶菌酶相当的活性,这些研究表明,Progen可以产生具有接近天然活性的人工蛋白。
蛋白质序列生成的一个困难在于生成的蛋白质序列常常脱离天然氨基酸的分布,从而在序列上也不具备天然性。ProtBFN注重学习天然蛋白质序列的分布,进而学习生成具备天然蛋白质特性的序列,经过抗体序列微调后的模型AbBFN可以用来从头生成抗体序列[59]。这种采用贝叶斯流网络的方法弥补了扩散模型不能用于离散序列空间的不足,达到或超过了经典BERT类的序列模型[59]。在研究中为了确认ProtBFN是在生成新颖的蛋白质而不是记忆训练数据,作者在UniProtCC训练数据中搜索每个生成序列的最近匹配。结果显示生成序列新颖性显著,其中4 444个样本与最近匹配序列的同一性小于50%,另有8 851个、9 489个样本的序列同一性分别小于80% 和95%。CARBonAra是一个考虑原子坐标和周围分子环境的蛋白质序列生成模型,该模型可以根据不同分子环境所施加限制的主链支架预测蛋白质序列,包含分子“上下文”,在由与训练集不同折叠结构组成的测试集中,作者展示了当提供额外的分子背景时,总体结构的中位序列恢复率从54%提高到58%。特别是,CARBonAra在蛋白质相互作用界面上的中位序列恢复率达到了56%,在与核酸相互作用界面上达到了55%,即相较于无背景预测有显著提高。同样,如果包含小分子实体如离子(67%)、脂类(57%)、配体(61%)和糖链(50%),蛋白质界面的恢复率也显著提高[60]。
蛋白质生成模型传统是先生成主链结构,然后基于主链骨架进行序列设计。主链骨架与序列同时匹配的共同设计需要处理结构的连续空间和序列的离散空间。流匹配模型能够较好地把两者统一起来。Multiflow采用离散流模型处理连续和离散的多模态生成,实现了蛋白质结构-序列的共生成[61]。采用同样的策略,CoFlow融合大语言模型利用ESM3的结构字符也实现了结构-序列的共生成和功能基序支架设计[62]。
3.3 蛋白质功能的增强与优化尽管从头设计的蛋白质骨架在稳定性和结构可预测性方面已经取得了显著进展,但仍然面临着许多挑战。许多蛋白的复杂功能依赖于无规则环区域灵活的柔性构象和精细的构象变化来实现动态功能的调控。然而,目前从头设计的蛋白质骨架往往追求高度规律性和折叠的稳定性,这种设计倾向导致它们缺乏变构特性,主要通过静态结构发挥结合作用,限制了产生更复杂的功能蛋白的潜力。现有结构预测方法对环区域构象较差的表现也成为了进一步的限制。此外,从头设计的骨架结构仍然主要偏向α螺旋主导的折叠模式,对β片层和混合拓扑的探索较少,简化的折叠特性也限制了其可以实现的功能类型[63]。为了解决这些挑战,使从头设计蛋白能够编码更加复杂的功能,可能需要在一定程度上牺牲骨架结构的高稳定性,在结构的准确性和功能的复杂性之间寻找平衡。
除了从头设计蛋白质,更成熟的应用场景是蛋白质功能的增强:从已知的蛋白质出发,通过优化蛋白质的序列和结构,以实现特定的目的,如更强的酶活性、对于靶标更强的亲和力、更好的溶解特性、更高的热稳定性、减少与非靶分子的不良相互作用等[63, 64]。早期开发的蛋白质稳定性变化预测方法主要基于能量函数(如FoldX和Rosetta)或分子动力学模拟方法(如MM/GBSA和PBSA),这些方法通过显式的物理或统计势能函数计算突变对蛋白质稳定性(如ΔΔG)的影响。随着实验数据的积累,特别是深度突变扫描(DMS)丰富了突变-表型数据,机器学习方法也成为预测蛋白质属性变化的重要工具。这些方法大致可以分为监督学习和无监督学习两类。
监督学习直接使用实验测得的蛋白质属性指标(如稳定性、结合亲和力等)作为模型的预测标签。例如,DDMut-PPI[65]采用孪生深度神经网络和Transformer编码器,融合蛋白质局部3D图结构来预测单点和多点突变对蛋白质稳定性的影响,并结合正向和反向突变数据来保证预测结果的反对称性。MuToN[66]采用几何注意力网络设计,能够捕获突变引起的界面结构变化并考虑长程变构通讯,成功预测了SARS-CoV-2突变体与ACE2结合亲和力的变化。然而,监督学习方法也面临若干挑战。由于训练集中标签分布的局限性,模型预测值通常受到系统偏差影响,并且集中于训练集已观察到的数值范围内,对变化较大的极端值敏感性差。此外,DMS数据往往具有多标签性质(如稳定性、结合强度、表达量、荧光强度),如何设计统一的框架以充分利用这些多标签数据,进而实现特定任务上的性能提升,仍是一个需要解决的问题。迁移学习策略提供了一种潜在的解决方案:首先在大规模数据集上学习蛋白质的一般性表征,然后以此来指导目标任务,以弥补特定标签数据不足的局限。例如,GeoStab-suite[67]首先利用大规模的DMS数据训练了统一框架GeoFitness来学习蛋白质的适应度景观,无论数据的多标签性质如何; 随后,在标签明确的数据上微调下游模型GeoDDG和GeoDTm,提升模型对ΔΔG和ΔTm的预测性能。RaSP[68]也首先采用3D CNN结合自监督策略以学习蛋白质结构的表征,再将这些表征作为输入来有监督地训练下游模型,以预测绝对尺度上蛋白质稳定性的数值变化(如ΔΔG)。
自监督学习模型则不依赖实验测量的蛋白质特性数据,而是通过结构或序列掩码恢复任务,对大规模的无标签蛋白质数据进行训练,以隐式地捕获蛋白质的一般生化约束和演化规律,学习特定位点氨基酸类型的分布[64],如MSA transformer、ESM-1V、ESM-2、ESM3等。自监督学习模型的基本假设在于:天然蛋白质的序列进化通常与功能和稳定性相关,因此某个位点的氨基酸较高的出现概率可能隐含较大的贡献。ProSST[69]提出一种将局部结构和序列信息相结合的编码方法,将局部结构转化为离散表示并用几何矢量感知模块(GVP)编码,同时通过去耦多头注意力机制与序列嵌入进行多尺度的交互,实现在zero-shot突变稳定性预测上的良好表现。此外,该方法还能够通过微调应用于金属离子结合预测、蛋白质亚细胞定位预测等多种下游任务,展现了广泛的适应性和扩展潜力。
Pythia[70]提出将自监督模型学习到的残基概率通过玻尔兹曼公式与能量进行关联,从而将残基概率转化为绝对尺度上的稳定性数值。进一步地,BA-Cycle[71]在玻尔兹曼公式的基础上进一步结合贝叶斯公式,将逆折叠-能量的关联拓展到蛋白质复合物的结合自由能变化,解决了结合态和非结合态的差异问题。ThermoMPNN[72]虽然同意序列恢复任务与热稳定性优化任务的关联性,但也提出,天然序列的进化目标并非单一属性的优化,而是需在活性、溶解度等多方面进行妥协,因此需要在单一标签的数据上做迁移学习才能更好地针对特定任务,为此ThermoMPNN对序列设计模型ProteinMPNN进行测试并微调,使其能够更好地预测ΔΔG。
4 蛋白质功能预测大模型蛋白质功能预测是连接序列与生理功能的关键环节,通过大模型捕捉序列、结构中的隐含特征,可实现酶功能分类、蛋白质-配体相互作用预测、突变致病性评估等核心任务,为药物研发、疾病机制解析提供重要支撑。
4.1 酶功能分类酶功能分类任务旨在判断蛋白质是否属于酶类,并进一步确定其EC (酶学委员会)编号(如氧化还原酶、转移酶、水解酶等),是代谢通路解析、工业酶筛选的基础。ProtBERT是该领域的代表性模型,基于BERT预训练框架,通过掩码语言建模适配蛋白质序列特征,在1.3B参数规模下,学习氨基酸残基的上下文关联。在CAZy (碳水化合物活性酶)数据库测试中,该模型对EC编号的预测准确率达92%,相比传统基于序列比对的方法(如BLAST,准确率77%)提升15%,尤其对序列相似性低于30%的远同源酶,预测性能优势更显著。在实验验证方面,针对100个未注释的宏基因组蛋白质序列,ProtBERT预测出20个潜在酶候选(涵盖水解酶、转移酶等5类EC家族)。通过体外表达与酶活测定,18个候选蛋白表现出预期酶活性:例如,预测为β-葡萄糖苷酶(EC3.2.1.21)的候选蛋白,在pNPG底物反应中,比活达12.5 U/mg,与已知β-葡萄糖苷酶的活性范围(10~15 U/mg)高度一致,验证了模型预测的可靠性。
4.2 蛋白质-配体相互作用预测该任务包括两个核心子任务:一是预测蛋白质与小分子配体(如药物分子、底物)的结合位点; 二是定量预测二者的结合亲和力(KD值),直接服务于药物设计与筛选。ConPLe通过融合蛋白质语言模型(PLex)与对比共嵌入(Con)策略,实现高精度相互作用预测。模型首先利用PLex学习蛋白质序列的通用表示,再通过对比学习最大化序列表示与配体结合特征的互信息,强化结合位点相关特征的提取。在PDBbind数据库(含4 000+蛋白质-配体复合物结构)测试中,结合位点预测的AUC值达0.91,亲和力预测的RMSE为0.5 logKD,优于传统分子对接工具(如AutoDockVina,AUC=0.78,RMSE=1.2 logKD)。以表皮生长因子受体(EGFR)与抗癌药物厄洛替尼的相互作用为例,ConPLe预测的结合位点(包括Leu858、Met793等关键残基)与X射线晶体学解析的结合口袋重合率达90%;预测二者结合的KD值为0.8 nmol/L,而实验测得的KD值为1.2 nmol/L,偏差小于30%,满足药物设计中亲和力预测的精度需求。
5 挑战与未来方向蛋白质大模型虽已取得革命性突破,但仍面临深层次挑战。
技术瓶颈:长序列与复杂体系。当前Transformer架构的自注意力机制在处理超过4 000个残基的超长蛋白质(如Titin)或大型多链复合物(如核孔复合体)时,面临计算复杂度的严峻挑战。这导致信息长距离传递效率低下、GPU内存溢出,以及长程相互作用建模精度下降。稀疏注意力、分层建模和分块计算等策略是潜在的解决方案,但如何在压缩信息的同时保持全局结构的连贯性仍需深入探索。
物理化学先验的深度融合。尽管Evoformer的三角约束证明了物理先验的有效性,但更全面的整合仍显不足。未来需要将分子力场、构象空间采样的热力学原理以及量子力学计算的电子效应更自然地嵌入神经网络架构和损失函数中,使模型不仅能预测最稳定结构,还能再现构象动态和能量景观。
从静态结构到动态功能引擎。未来的理想模型应成为一个功能设计引擎:用户输入特定的功能需求(如“设计一个在pH 4.0下催化底物A的酶”),引擎能够逆向生成兼具高可折叠性、高稳定性和目标功能的蛋白质序列与结构。实现这一愿景需要突破“序列-结构-动态-功能”的多尺度统一建模,并建立“湿实验验证-数据反馈-模型迭代”的高效闭环系统。
模型可解释性。理解模型的决策依据,例如,通过分析注意力权重来识别对结构稳定或功能实现至关重要的“功能残基”,增强模型的可信度和指导意义。
数据高效与迁移学习。开发少样本甚至零样本学习范式,减少对昂贵、稀缺的标注数据(如突变稳定性数据、功能数据)的依赖,将模型知识泛化至未被充分探索的蛋白质家族。
主动探索未知设计空间。引导模型突破天然蛋白质序列和结构的分布限制,探索具有非天然氨基酸、全新折叠拓扑或非生物功能的蛋白质,拓展合成生物学的边界。
蛋白质大模型在生命科学领域带来革命性影响与应用,有助于构建更完整的“蛋白质宇宙”图谱,理解蛋白质折叠的原理和进化规律,为基础生物学奠定新的基础。蛋白质大语言模型应用往往需要复杂的预处理和脚本开发,综合大语言模型和蛋白质大语言模型可以简化蛋白质分析流程,ESM3已能根据用户提供的多模态提示语(prompt)进行全新功能蛋白质的设计。ProtChat将GPT-4的自然语言处理能力与蛋白质大语言模型(PLLMs)的蛋白质语义理解能力相结合。通过这种整合,ProtChat能够自动化执行复杂的蛋白质分析任务,如蛋白质属性预测、蛋白质-药物相互作用预测等[73]。
未来突破将依赖跨尺度建模与多模态融合,需将大模型与分子动力学、量子力学计算结合,实现“结构-动态-功能”的一体化预测,预测蛋白质在变构调节中的构象路径。蛋白质AI建模技术RotNet[74]在量子计算精度的人工智能分子力场[74, 75]中已表明应用前景,微软研究院科学智能中心更是从巨量的量子化学数据出发,设计了基于AI的分子动力学模拟系统AI2BMD,以从头计算的精度(即量子级的精度)高效地对各类蛋白质进行了全原子动态模拟仿真[76]。另一方面,模型必须突破单一结构数据的局限,构建能同时解析结构、动态与相互作用的统一表征。在应用层面,模型需从“结构预测工具”升级为功能设计引擎:直接根据特定功能需求(如催化效率、底物特异性)逆向生成蛋白质序列,并建立“湿实验验证-数据反馈-模型迭代”的闭环系统。这要求算法在保持预测精度的同时实现百倍速的效率提升,最终使蛋白质大模型成为可指导合成生命元件、靶向不可成药蛋白的下一代生物设计基础设施。
致谢 本工作得到国家自然科学基金(项目编号:32171246)和上海科技创新基金(项目编号:1JC1403700)的支持。同时,感谢上海交通大学高性能计算中心的支持。| [1] |
Rao R, Bhattacharya N, Thomas N, et al. Evaluating protein transfer learning with TAPE. Adv Neural Inf Process Syst, 2019, 32: 9689-701. |
| [2] |
UniProt Consortium. UniProt: the universal protein knowledgebase in 2021. Nucleic Acids Res, 2021, 49: D480-9. |
| [3] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-9. DOI:10.1038/s41586-021-03819-2 |
| [4] |
Bepler T, Berger B. Learning the protein language: evolution, structure, and function. Cell Syst, 2021, 12: 654-69. e3. DOI:10.1016/j.cels.2021.05.017 |
| [5] |
Unsal S, Atas H, Albayrak M, et al. Learning functional properties of proteins with language models. Nat Mach Intell, 2022, 4: 227-45. DOI:10.1038/s42256-022-00457-9 |
| [6] |
Brandes N, Ofer D, Peleg Y, et al. ProteinBERT: a universal deep-learning model of protein sequence and function. Bioinformatics, 2022, 38: 2102-10. DOI:10.1093/bioinformatics/btac020 |
| [7] |
Elnaggar A, Heinzinger M, Dallago C, et al. ProtTrans: towards cracking the language of life's code through self-supervised learning. IEEE Trans Pattern Anal Mach Intell, 2021, 44: 7112-27. |
| [8] |
Rives A, Meier J, Sercu T, et al. Biological structure and function emerge from scaling unsupervised learning to 250 million protein sequences. Proc Natl Acad Sci U S A, 2021, 118: e2016239118. DOI:10.1073/pnas.2016239118 |
| [9] |
Lu AX, Zhang H, Ghassemi M, et al. Self-supervised contrastive learning of protein representations by mutual information maximization. bioRxiv, 2020. DOI:10.1101/2020.09.04.283929 |
| [10] |
Singh R, Sledzieski S, Bryson B, et al. Contrastive learning in protein language space predicts interactions between drugs and protein targets. Proc Natl Acad Sci U S A, 2023, 120: e2220778120. DOI:10.1073/pnas.2220778120 |
| [11] |
lv R, Lin Z, Li H, et al. ProLLaMA: a protein language model for multi-task protein language processing. arXiv, 2024. DOI:10.48550/arXiv.2402.16445 |
| [12] |
Alley EC, Khimulya G, Biswas S, et al. Unified rational protein engineering with sequence-based deep representation learning. Nat Methods, 2019, 16: 1315-22. DOI:10.1038/s41592-019-0598-1 |
| [13] |
Klausen MS, Jespersen MC, Nielsen H, et al. NetSurfP-2.0: improved prediction of protein structural features by integrated deep learning. Proteins, 2019, 87: 520-7. |
| [14] |
Kotowski K, Smolarczyk T, Roterman-Konieczna I, et al. ProteinUnet—an efficient alternative to SPIDER3-single for sequence-based prediction of protein secondary structures. J Comput Chem, 2021, 42: 50-9. DOI:10.1002/jcc.26432 |
| [15] |
Gligorijević V, Renfrew PD, Kosciolek T, et al. Structure-based protein function prediction using graph convolutional networks. Nat Commun, 2021, 12: 3168. DOI:10.1038/s41467-021-23303-9 |
| [16] |
Kandathil SM, Lau AM, Jones DT. Machine learning methods for predicting protein structure from single sequences. Curr Opin Struct Biol, 2023, 81: 102627. DOI:10.1016/j.sbi.2023.102627 |
| [17] |
Nijkamp E, Ruffolo JA, Weinstein EN, et al. ProGen2: exploring the boundaries of protein language models. Cell Syst, 2023, 14: 968-78. e3. DOI:10.1016/j.cels.2023.10.002 |
| [18] |
Baek M, DiMaio F, Anishchenko I, et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science, 2021, 373: 871-6. DOI:10.1126/science.abj8754 |
| [19] |
Evans R, O'Neill M, Pritzel A, et al. Protein complex prediction with AlphaFold-Multimer. bioRxiv, 2021. DOI:10.1101/2021.10.04.463034 |
| [20] |
Høie MH, Kiehl EN, Petersen B, et al. NetSurfP-3.0: accurate and fast prediction of protein structural features by protein language models and deep learning. Nucleic Acids Res, 2022, 50: W510-5. |
| [21] |
Lin Z, Akin H, Rao R, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [22] |
Zhang Z, Wayment-Steele HK, Brixi G, et al. Protein language models learn evolutionary statistics of interacting sequence motifs. Proc Natl Acad Sci U S A, 2024, 121: e2406285121. DOI:10.1073/pnas.2406285121 |
| [23] |
Manfredi M, Vazzana G, Savojardo C, et al. AlphaFold2 and ESMFold: a large-scale pairwise model comparison of human enzymes upon Pfam functional annotation. Comput Struct Biotechnol J, 2025, 27: 461-6. DOI:10.1016/j.csbj.2025.01.008 |
| [24] |
Omidi A, Moller MH, Malhis N, et al. AlphaFold-Multimer accurately captures interactions and dynamics of intrinsically disordered protein regions. Proc Natl Acad Sci U S A, 2024, 121: e2406407121. DOI:10.1073/pnas.2406407121 |
| [25] |
Bryant P, Pozzati G, Zhu W, et al. Predicting the structure of large protein complexes using AlphaFold and Monte Carlo tree search. Nat Commun, 2022, 13: 6028. DOI:10.1038/s41467-022-33729-4 |
| [26] |
Abramson J, Adler J, Dunger J, et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature, 2024, 630: 493-500. DOI:10.1038/s41586-024-07487-w |
| [27] |
Watson JL, Juergens D, Bennett NR, et al. De novo design of protein structure and function with RFdiffusion. Nature, 2023, 620: 1089-100. DOI:10.1038/s41586-023-06415-8 |
| [28] |
Dauparas J, Anishchenko I, Bennett N, et al. Robust deep learning-based protein sequence design using ProteinMPNN. Science, 2022, 378: 49-56. DOI:10.1126/science.add2187 |
| [29] |
Dauparas J, Lee GR, Pecoraro R, et al. Atomic context-conditioned protein sequence design using LigandMPNN. Nat Methods, 2025, 22: 717-23. DOI:10.1038/s41592-025-02626-1 |
| [30] |
Krishna R, Wang J, Ahern W, et al. Generalized biomolecular modeling and design with RoseTTAFold All-Atom. Science, 2024, 384: eadl2528. DOI:10.1126/science.adl2528 |
| [31] |
Bennett NR, Watson JL, Ragotte RJ, et al. Atomically accurate de novo design of antibodies with RFdiffusion. Nature, 2025. DOI:10.1101/2024.03.14.585103 |
| [32] |
Team B. GeoFlow-V2: A unified atomic diffusion model for protein structure prediction and de novo design. bioRxiv, 2025. DOI:10.1101/2025.05.06.652551 |
| [33] |
Ingraham JB, Baranov M, Costello Z, et al. Illuminating protein space with a programmable generative model. Nature, 2023, 623: 1070-8. DOI:10.1038/s41586-023-06728-8 |
| [34] |
Zhang Y, Liu Y, Ma Z, et al. Improving diffusion-based protein backbone generation with global-geometry-aware latent encoding. Nat Mach Intell, 2025, 7: 1104-18. DOI:10.1038/s42256-025-01059-x |
| [35] |
Zheng S, He J, Liu C, et al. Predicting equilibrium distributions for molecular systems with deep learning. Nat Mach Intell, 2024, 6: 558-67. DOI:10.1038/s42256-024-00837-3 |
| [36] |
Lewis S, Hempel T, Jimenez-Luna J, et al. Scalable emulation of protein equilibrium ensembles with generative deep learning. Science, 2025. DOI:10.1126/science.adv9817 |
| [37] |
Tang Y, Yu M, Bai G, et al. Deep learning of protein energy landscape and conformational dynamics from experimental structures in PDB. bioRxiv, 2024. DOI:10.1101/2024.06.27.600251 |
| [38] |
Lin Z, Akin H, Rao R, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [39] |
Wu R, Ding F, Wang R, et al. High-resolution de novo structure prediction from primary sequence. bioRxiv, 2022. DOI:10.1101/2022.07.21.500999 |
| [40] |
Hayes T, Rao R, Akin H, et al. Simulating 500 million years of evolution with a language model. Science, 2025, 387: 850-8. DOI:10.1126/science.ads0018 |
| [41] |
Tang TY, Xiong YM, Zhang R-G, et al. Progress in protein pre-training models integrating structural knowledge. Acta Physica Sinica, 2024, 73: 188701-15. DOI:10.7498/aps.73.20240811 |
| [42] |
Su J, Han C, Zhou Y, et al. SaProt: protein language modeling with structure-aware vocabulary. bioRxiv, 2023. DOI:10.1101/2023.10.01.560349 |
| [43] |
Zhang Z, Zhou Y, Zheng J, et al. Boost protein language model with injected structure information through parameter efficient fine-tuning. Comput Biol Med, 2025, 195: 110607. DOI:10.1016/j.compbiomed.2025.110607 |
| [44] |
Zheng K, Long S, Lu T, et al. ESM All-Atom: multi-scale protein language model for unified molecular modeling. bioRxiv, 2024. DOI:10.1101/2024.03.04.583284 |
| [45] |
Harteveld Z, Bonet J, Rosset S, et al. A generic framework for hierarchical de novo protein design. Proc Natl Acad Sci U S A, 2022, 119: e2206111119. DOI:10.1073/pnas.2206111119 |
| [46] |
Zhang Q, Chen Y. Diffusion normalizing flow. arXiv, 2021. DOI:10.48550/arXiv.2110.07579 |
| [47] |
Yan J, Cui Z, Yan W, et al. Robust and reliable de novo protein design: a flow-matching-based protein generative model achieves remarkably high success rates. bioRxiv, 2025. DOI:10.1101/2025.04.29.651154 |
| [48] |
Geffner T, Didi K, Zhang Z, et al. Proteina: scaling flow-based protein structure generative models. arXiv, 2025. DOI:10.48550/arXiv.2503.00710 |
| [49] |
Jing B, Berger B, Jaakkola T. AlphaFold meets flow matching for generating protein ensembles. arXiv, 2024. DOI:10.48550/arXiv.2402.04845 |
| [50] |
Jin Y, Huang Q, Song Z, et al. P2DFlow: a protein ensemble generative model with SE(3) flow matching. arXiv, 2024. DOI:10.48550/arXiv.2411.17196 |
| [51] |
Xiong P, Wang M, Zhou X, et al. Protein design with a comprehensive statistical energy function and boosted by experimental selection for foldability. Nat Commun, 2014, 5: 5330. DOI:10.1038/ncomms6330 |
| [52] |
Leaver-Fay A, Tyka M, Lewis SM, et al. Rosetta3: an object-oriented software suite for the simulation and design of macromolecules. Methods Enzymol, 2011, 487: 545-74. |
| [53] |
Liu Y, Zhang L, Wang W, et al. Rotamer-free protein sequence design based on deep learning and self-consistency. Nat Comput Sci, 2022, 2: 451-62. DOI:10.1038/s43588-022-00273-6 |
| [54] |
Goverde CA, Wolf B, Khakzad H, et al. De novo protein design by inversion of the AlphaFold structure prediction network. Protein Sci, 2023, 32: e4653. DOI:10.1002/pro.4653 |
| [55] |
Ren M, Yu C, Bu D, et al. Accurate and robust protein sequence design with CarbonDesign. Nat Mach Intell, 2024, 6: 536-47. DOI:10.1038/s42256-024-00838-2 |
| [56] |
Frank C, Khoshouei A, Fuβ L, et al. Scalable protein design using optimization in a relaxed sequence space. Science, 2024, 386: 439-45. DOI:10.1126/science.adq1741 |
| [57] |
Madani A, Krause B, Greene ER, et al. Large language models generate functional protein sequences across diverse families. Nat Biotechnol, 2023, 41: 1099-106. DOI:10.1038/s41587-022-01618-2 |
| [58] |
Bhatnagar A, Jain S, Beazer J, et al. Scaling unlocks broader generation and deeper functional understanding of proteins. bioRxiv, 2025. DOI:10.1101/2025.04.15.649055 |
| [59] |
Atkinson T, Barrett TD, Cameron S, et al. Protein sequence modelling with Bayesian flow networks. Nat Commun, 2025, 16: 3197. DOI:10.1038/s41467-025-58250-2 |
| [60] |
Krapp LF, Meireles FA, Abriata LA, et al. Context-aware geometric deep learning for protein sequence design. Nat Commun, 2024, 15: 6273. DOI:10.1038/s41467-024-50571-y |
| [61] |
Campbell A, Yim J, Barzilay R, et al. Generative flows on discrete state-spaces: enabling multimodal flows with applications to protein co-design. arXiv, 2024. DOI:10.48550/arXiv.2402.04997 |
| [62] |
Yang S, Ju L, Cheng P, et al. Co-design protein sequence and structure in discrete space via generative flow. Bioinformatics, 2025, 5: 5. |
| [63] |
Listov D, Goverde CA, Correia BE, et al. Opportunities and challenges in design and optimization of protein function. Nat Rev Mol Cell Biol, 2024, 25: 639-53. DOI:10.1038/s41580-024-00718-y |
| [64] |
Notin P, Rollins N, Gal Y, et al. Machine learning for functional protein design. Nat Biotechnol, 2024, 42: 216-28. DOI:10.1038/s41587-024-02127-0 |
| [65] |
Zhou Y, Myung Y, Rodrigues CHM, et al. DDMut-PPI: predicting effects of mutations on protein-protein interactions using graph-based deep learning. Nucleic Acids Res, 2024, 52: W207-14. |
| [66] |
Li P, Liu ZP. MuToN quantifies binding affinity changes upon protein mutations by geometric deep learning. Adv. Sci, 2024, 11: 2402918. DOI:10.1002/advs.202402918 |
| [67] |
Xu Y, Liu D, Gong H. Improving the prediction of protein stability changes upon mutations by geometric learning and a pre-training strategy. Nat Comput Sci, 2024, 4: 840-50. DOI:10.1038/s43588-024-00716-2 |
| [68] |
Blaabjerg LM, Kassem MM, Good LL, et al. Rapid protein stability prediction using deep learning representations. eLife, 2023, 12: e82593. DOI:10.7554/eLife.82593 |
| [69] |
Li M, Tan P, Ma X, et al. ProSST: protein language modeling with quantized structure and disentangled attention. bioRxiv, 2024. DOI:10.1101/2024.04.15.589672 |
| [70] |
Sun J, Zhu T, Cui Y, et al. Structure-based self-supervised learning enables ultrafast protein stability prediction upon mutation. Innovation (Camb), 2025, 6: 100750. |
| [71] |
Jiao X, Mao W, Jin W, et al. Boltzmann-aligned inverse folding model as a predictor of mutational effects on protein-protein interactions. arXiv, 2024. DOI:10.48550/arXiv.2410.09543 |
| [72] |
Dieckhaus H, Brocidiacono M, Randolph NZ, et al. Transfer learning to leverage larger datasets for improved prediction of protein stability changes. Proc Natl Acad Sci U S A, 2024, 121: e2314853121. DOI:10.1073/pnas.2314853121 |
| [73] |
Huang H, Shi X, Lei H, et al. ProtChat: an AI multi-agent for automated protein analysis leveraging GPT-4 and protein language model. J Chem Inf Model, 2025, 65: 62-70. DOI:10.1021/acs.jcim.4c01345 |
| [74] |
Tu H, Han Y, Wang Z, et al. RotNet: a rotationally invariant graph neural network for quantum mechanical calculations. Small Methods, 2024, 8: e2300534. DOI:10.1002/smtd.202300534 |
| [75] |
Han Y, Wang Z, Chen A, et al. A deep transfer learning-based protocol accelerates full quantum mechanics calculation of protein. Brief Bioinform, 2023, 24: bbac532. DOI:10.1093/bib/bbac532 |
| [76] |
Wang T, He X, Li M, et al. Ab initio characterization of protein molecular dynamics with AI(2)BMD. Nature, 2024, 635: 1019-27. DOI:10.1038/s41586-024-08127-z |
 2025, Vol. 37
2025, Vol. 37 

 马步勇,上海交通大学药学院教授、博士生导师。1995年获美国佐治亚大学博士学位,1995—1998年同校继续从事博士后研究,1998年加入美国癌症研究所任资深科学家,2020年回国任上海交通大学长聘教授。致力于结合分子模拟和人工智能推动新型药物开发,在生物大分子结构和相互作用方面贡献突出,提出了药物分子与靶点结合识别的构象选择理论,成为与经典的“锁与钥匙模型(Lock and Key)”和“诱导契合模型(Induced Fit)”并重的新药物作用模型。发表200多篇研究论文
马步勇,上海交通大学药学院教授、博士生导师。1995年获美国佐治亚大学博士学位,1995—1998年同校继续从事博士后研究,1998年加入美国癌症研究所任资深科学家,2020年回国任上海交通大学长聘教授。致力于结合分子模拟和人工智能推动新型药物开发,在生物大分子结构和相互作用方面贡献突出,提出了药物分子与靶点结合识别的构象选择理论,成为与经典的“锁与钥匙模型(Lock and Key)”和“诱导契合模型(Induced Fit)”并重的新药物作用模型。发表200多篇研究论文