(2 上海交通大学生命科学与技术学院, 上海 200240)
(2 School of Life Sciences and Biotechnology, Shanghai Jiaotong University, Shanghai 200240, China)
目前,人工智能技术和数据驱动的研究方法被广泛应用于研究蛋白质序列、结构和功能的关系,其里程碑式的蛋白质结构预测模型AlphaFold[1],解决了长期困扰科学界的蛋白质折叠问题,能够以高精度预测蛋白质的三维结构,极大地加速了生物医学研究,其主要开发者Demis Hassabis和John Jumper荣膺2024年诺贝尔化学奖[2]。AlphaFold作为蛋白质领域的代表性AI模型,揭示了蛋白质序列与结构的关系,更进一步地推动了对蛋白质序列与功能之间关系的研究。在此背景下,蛋白质语言模型(protein language models, 简称PLMs)作为一种能够连接蛋白质序列与功能的强大AI工具,受到了学术界和工业界的广泛关注,并得到了飞速发展。PLMs的出现为理解蛋白质序列如何决定其生物功能,以及如何根据所需功能挖掘或设计相应序列等蛋白质工程的关键研究问题,提供了全新的工具。
PLMs借鉴了自然语言处理领域大模型的成功经验,将蛋白质序列视为一种“生命语言”,通过海量的序列数据进行自监督学习,从而捕捉到蛋白质序列中复杂的模式和语义信息,生成通用高质量的表征。在预训练阶段,PLMs在大量的不带有功能标签的蛋白质序列数据上通过语言建模任务进行自监督任务学习,以捕捉语言的深层结构和语义信息。具有代表性的两种语言建模是掩码语言建模[3](masked language modeling, MLM)和自回归语言建模[4](又称因果语言模型,causal language modeling, CLM)。MLM随机遮盖蛋白质序列中的部分氨基酸,要求模型根据未遮盖部分的氨基酸序列来预测被遮盖的氨基酸,若模型预测错误,则施加惩罚,从而迫使模型学习蛋白质序列内部氨基酸之间的上下文依赖关系。CLM则要求模型根据序列中已出现的氨基酸来预测下一个氨基酸,这种单向的预测机制使得模型能够学习到序列的生成模式和长距离依赖关系,适用于全新蛋白质序列生成或序列补全等任务。通过预训练,PLMs能够深入理解蛋白质“语言”的语法规则,这些预训练模型可以直接应用于各类生物学任务,或者针对特定的下游任务(如蛋白质功能注释、突变预测或家族蛋白生成)进行微调。在微调阶段,研究人员通常会使用少量带有标注数据的数据集进行迁移学习和有监督训练,从而调整模型参数,使PLMs更好地适应特定的任务需求。相较于传统的机器学习模型,PLMs的优势在于其无需手工构造蛋白质序列的特征。相反,PLMs在预训练阶段充分利用了丰富的无监督蛋白质序列数据,不仅能够生成更为通用的表征,还能降低下游任务对标注数据量的需求。此外,与自然语言模型类似,随着模型规模的扩大(例如从百万级参数量提升至亿级参数量)和训练数据量的增加,PLMs的语言建模性能也呈现出可预测的提升,符合“扩展定律”(scaling laws)[5-8]。
本文围绕着蛋白质语言模型的分类、应用和发展展开,从语言建模目标和实际应用场景两个维度对蛋白质语言模型进行分类,并讨论了蛋白质语言模型的最新进展、面临的挑战和未来展望(图 1)。
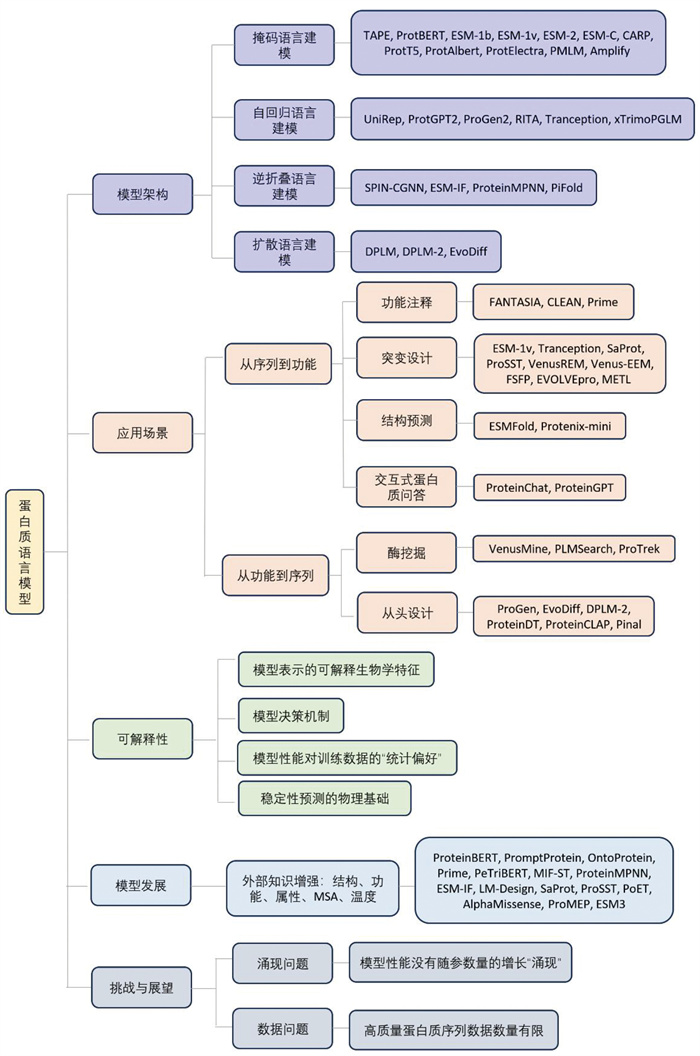
|
图 1 蛋白质语言模型综述框架图 |
蛋白质语言模型的性能与其训练数据的规模和多样性息息相关。正如自然语言模型需要海量的文本语料库,PLMs的强大能力同样根植于对亿万级别蛋白质序列数据的学习。然而,近年来随着模型规模的迅速扩张,高质量训练数据的稀缺已成为限制其性能进一步提升的“数据墙”(data wall)[9]。本节将概述PLMs训练数据的主要来源、发展趋势以及为突破“数据墙”而构建的大规模蛋白质数据集。
最初,PLMs的训练主要依赖于公开的蛋白质序列数据库,其中最核心的是UniProt知识库及其经过聚类去冗余后形成的UniRef系列数据集[10]。例如,被广泛使用的UniRef50和UniRef90是众多开创性PLMs (如ESM系列)的训练基础。这些数据库整合了来自全球学术实验的成果,为模型提供了宝贵的初始数据。然而,这些传统数据库相对于宏基因组数据库数据量仍显不足。
为了突破这一瓶颈,研究人员开始构建规模空前、专门用于AI模型训练的蛋白质数据集。如图 2所示,这些数据集通过整合多个上游数据库(如UniProt、MGnify宏基因组数据库[11]、JGI IMG[12]、PDB结构数据库[13]等),并应用先进的生物信息学流程进行挖掘和处理,极大地扩展了可用于训练的序列空间。这些为训练PLMs而生的大规模数据集合并了来自不同源头的数据,其规模通常达到了数十亿级别。例如,Dayhoff Atlas[14]包含33.4亿条序列,Profluent Protein Atlas v1[15]包含34亿条序列,OpenMeta- Genomic[16]包含31亿条序列,为ESM-3模型构建的ESM-3 dataset[17]拥有27.8亿条序列,BFD[1]数据集包含23亿条序列,UniMeta200B[8]则整合了20亿条序列。即便是主要面向结构预测的Colab-FoldDB[18],其序列量也达到了7亿。这些超大规模数据集为训练更大、更强的PLMs提供了可能。
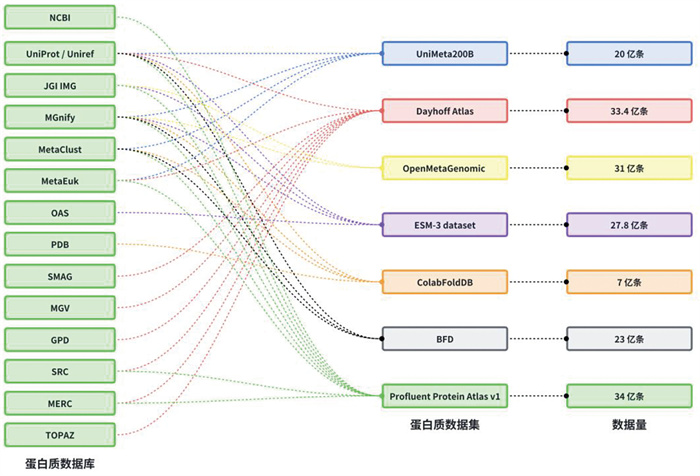
|
图 2 主流蛋白质数据集的来源与规模 |
从概率的视角来看,PLMs是一个用于描述蛋白质序列分布的模型。PLMs学习自然界中蛋白质序列的潜在概率分布p(X),其中X=(x1, x2, ..., xL)是由氨基酸组成的序列。通过在大量数据上进行训练,模型能够捕捉到决定序列有效性的规律,包括进化所偏好的氨基酸组合、结构基序和功能模式等。该分布具备两个功能:(1)判别:评估一个给定序列的似然度,即判断其是否像一个真实的蛋白质;(2)生成:从这个分布中采样,生成全新的蛋白质序列。此外,为了高效地建模蛋白质序列的概率分布,PLMs在学习过程中还将离散的氨基酸转换为信息密集的连续向量表示,即嵌入向量(embeddings)。与传统的独热编码(one-hot encoding)等简单的查表式映射不同,PLMs生成的嵌入向量是上下文感知的,即同一个氨基酸会因其在序列中所处的上下文环境不同而获得不同的向量表示。蛋白质序列中所有氨基酸编码向量构成了蛋白质的表征。这些表征在一个稠密的高维空间中,编码了关于蛋白质结构倾向、功能角色和进化关系等宝贵的生物学信息,因此可以直接作为强大的特征输入,服务于各类下游预测任务,例如结构预测、功能注释等。
不同的模型架构通过各异的数学范式来实现上述的判别与生成目标。下文将分别探讨四种主流架构:以学习深度上下文表示为目标的掩码语言建模,以直接序列生成为目标的自回归模型,以及在它们基础上进一步发展而来的、用于条件生成的逆折叠模型与扩散模型。
3.1 掩码语言建模掩码语言建模(MLM)是一种通过在输入序列中随机遮蔽一部分氨基酸,训练模型来预测还原这些被掩盖的原始氨基酸,从而学习氨基酸的表征和蛋白质序列的表征。为了准确预测被掩盖的位置,模型必须深刻理解该位置两侧的上下文信息。这种双向的上下文依赖性,使得MLM能够学习上下文感知的氨基酸嵌入表示。表 1列举了目前常见的蛋白质掩码语言模型。
| 表 1 基于掩码语言建模的PLMs |
在技术实现上,与自然语言类似,MLM通常采用基于Transformer编码器的架构,如BERT及其在蛋白质领域的变体,例如Meta团队开发的ESM系列模型、西湖大学开发的SaProt模型、上海交通大学团队开发的ProSST模型等。其训练过程遵循以下步骤:首先,对输入的蛋白质序列X进行随机掩盖,产生一个被加噪的序列
| $ \mathrm{L}_{M L M}=-\sum\limits_{m \in M} \log p\left(x_m \mid \widetilde{X}\right) $ | (1) |
其中,LMLM表示掩码语言建模的损失函数;M表示被掩码的氨基酸位置集合;
自回归语言建模是另一种主流的序列概率建模范式,其将序列的联合概率分布p(X) 分解为序列中每个氨基酸的条件概率的乘积。依据概率论的链式法则,一个长度为L的序列X的概率表示如公式2所示:
| $ p(X)=p\left(x_1, x_2, \ldots, x_L\right)=\prod\limits_{i=1}^L p\left(x_i \mid x_{<i}\right) $ | (2) |
其中,x< i表示在位置i之前的所有氨基酸子序列(x1, x2, ..., xi-1)。在位置i出现某个氨基酸的概率,取决于它之前的所有氨基酸。这种从左到右(或从右到左)的单向依赖性被称作“自回归过程”或者因果语言建模。表 2列举了目前常见的蛋白质掩码语言模型。
| 表 2 基于自回归语言建模的PLMs |
在架构上,自回归模型通常采用Transformer的解码器(Decoder)部分。在训练阶段,模型的目标是最大化整个训练序列的对数似然,即最小化负对数似然损失,如公式3所示:
| $ \mathrm{L}_{A R}=-\sum\limits_{i=1}^L \log p\left(x_i \mid x_{<i}\right) $ | (3) |
其中,LAR为自回归损失函数,即整个序列负对数似然的累加。为了保证严格的单向依赖性,解码器中的自注意力机制会使用因果掩码,确保在计算位置i的表示时,只能关注到i及之前的位置,而无法看到未来的信息。自回归语言模型凭借其连续生成的特性,天然适用于蛋白质序列的生成任务。一旦模型训练完成,就可以通过迭代采样的方式生成新序列:从一个起始符开始,模型首先预测第一个氨基酸的概率分布并从中采样;然后将采样到的氨基酸作为下一个时间步的输入,继续预测第二个氨基酸的分布,如此循环往复,直到生成一个终止符或达到预设长度。此外,在生成的过程中可以通过Prompt、微调等手段使其生成具有特定功能的蛋白质。ProtGPT2模型和ProGen系列模型是自回归蛋白质语言模型的代表,它们能够生成功能多样的全新蛋白质序列。ProGen模型通过在溶菌酶家族蛋白上微调,能够生成全新的溶菌酶序列。基因组语言模型同样可以生成蛋白质,例如Evo模型[39, 40],通过在基因编辑酶家族上微调,能够生成全新的基因编辑酶序列。
3.3 逆折叠语言建模逆折叠语言建模是一种有条件的蛋白质生成的语言模型。给定一个期望的蛋白质三维骨架结构S (通常由Ca、N、C、O等骨干原子坐标定义),推断并生成一个能够稳定折叠成该特定构象的氨基酸序列X。此任务常被直观地描述为结构预测的逆问题。从概率视角来看,逆折叠模型的目标为学习条件概率分布P(X | S)。在实际应用中,此概率分布既可以作为生成器,通过从该分布中采样,可实现全新的、定制化的蛋白质序列设计;其二,作为评分函数,可计算一个给定序列与目标结构之间的适配似然度,为蛋白质工程中的序列优化提供先验。表 3列举了目前常见的逆折叠语言模型。
| 表 3 用于逆折叠的PLMs |
在架构实现上,逆折叠模型通常遵循编码器-解码器(encoder-decoder)的设计。编码器的功能是将输入的原子坐标这一原始三维数据,转化为一个能够被后续模块有效利用的高维向量表示。由于蛋白质结构具有图状拓扑特性和空间几何关系,通常采用图神经网络或基于等变性的网络作为编码器。以ESM-IF模型为例,其GNN编码器将残基视作图节点,根据空间邻近关系构建边,并将残基间的相对距离和方向编码为边特征。通过在图上执行迭代的消息传递,每个节点的嵌入向量得以捕捉其局部三维结构信息。
解码器部分则通常采用自回归的方式,基于编码器输出的结构嵌入来逐个生成氨基酸。在生成第i个氨基酸xi时,解码器不仅会接收其先前已生成的氨基酸序列x< i作为输入,还会接收编码器提供的结构嵌入向量HS。因此,其条件概率可以表示为p(xi|x< i, S)。整个序列的生成概率是这些条件概率的连乘积。在训练阶段,模型的目标同样是最大化整个训练序列的对数似然,即最小化负对数似然损失,如公式4所示:
| $ \mathrm{L}_{A R}=-\sum\limits_{i=1}^L \log p\left(x_i \mid x_{<i}, S\right) $ | (4) |
其中,S代表给定的蛋白质三维骨架结构条件;
然而,这种依赖于结构的建模方式存在一定的局限性。首先,高质量的PDB结构数据仅约20万个,远比序列数据稀缺,限制了模型学习蛋白质空间完整多样性的能力。其次,对于功能并非由单一静态结构介导的蛋白质,如包含本质无序区的蛋白,基于静态结构的设计方法本身就是不适用的。
3.4 离散扩散语言建模离散扩散模型(discrete diffusion model)是近年来新兴的生成模型,其通过学习逆转一个逐步向数据中注入噪声的“前向过程”来实现高质量的样本生成。在应用于蛋白质等离散序列数据时,这一框架表现出天然的灵活性。具体而言,前向过程通过逐步添加随机噪声的方式扰动一个真实的蛋白质序列X0。与应用于图像的连续高斯噪声不同,离散扩散根据一个预设的状态转移矩阵(如均匀突变或基于BLOSUM62的突变矩阵)来改变氨基酸的类型,直到序列Xt变得与随机噪声无法区分。相应地,一个参数化的神经网络,通常是Transformer,被训练来学习“反向过程”,即从一个被加噪的序列Xt预测出加噪前一步状态Xt-1。生成过程则从一个完全随机或被掩码的序列XT开始,通过迭代地应用这个学习到的去噪网络,逐步提纯序列,最终生成一个全新蛋白质序列X0。
表 4列举了目前场景的离散扩散蛋白质语言模型。EvoDiff模型是一个完全基于序列的扩散模型框架,在训练时仅利用序列数据。其证明了离散扩散语言模型在学习蛋白质序列方面的可行性,并且说明模型能生成多样化、结构合理且覆盖自然功能空间的蛋白质,甚至在不依赖任何结构信息的情况下,仅通过序列条件就能完成对功能基序的骨架构建。DPLM-2模型将离散扩散框架扩展为一种多模态基础模型,能够同时生成蛋白质的序列与结构。其能够生成结构的关键在于其引入了一个结构标记器,将连续的三维坐标转换为离散的符号。通过对拼接后的序列-结构符号进行联合去噪学习,DPLM-2能够同时生成高度兼容的氨基酸序列及其对应的三维结构。
| 表 4 基于离散扩散生成的蛋白质语言模型 |
综上所述,四种语言建模架构在建模目标、条件信息利用和应用场景上存在显著差异。掩码语言建模通过双向上下文信息学习高质量的氨基酸表征,适用于功能注释、突变效应预测等下游判别任务,但其非生成式的本质使其不直接支持序列生成。自回归语言建模基于单向因果依赖逐个生成氨基酸,这种从左到右的生成机制使其天然适合从头设计和序列补全等生成任务。逆折叠语言建模以三维骨架结构为强约束条件,实现从结构到序列的映射,主要服务于结构导向的蛋白质设计,但受限于高质量结构数据的稀缺。离散扩散语言建模通过学习逆转噪声注入过程来实现序列乃至结构的生成,在生成质量和多样性上具有优势,尤其适合多模态蛋白质生成任务,但其迭代去噪的推理过程通常比自回归生成更为耗时。在训练数据需求上,掩码和自回归模型仅需序列数据即可训练,数据获取相对容易;逆折叠模型则依赖于结构-序列配对数据,数据规模受到一定限制。
4 应用场景蛋白质语言模型的应用围绕着蛋白质序列与结构三者之间相互关系开展。这些应用可以被归纳为两个对偶方向:从已知的序列推断其功能,或者反过来,从期望的功能出发,设计出满足要求的序列。表 5总结了PLMs的主要应用场景及其代表性模型。
| 表 5 蛋白质语言模型的应用场景 |
根据蛋白质的序列预测蛋白质的功能是PLMs应用最为广泛的任务。其中两个典型的案例是功能注释和突变设计。
4.1.1 功能注释功能注释指针对序列数据库中仅包含测序信息的蛋白质。传统的功能注释方法主要依赖于序列比对来对蛋白质进行同源搜索,但当一个蛋白质与其已知功能的同源物序列相似性较低时,这些方法的精度会受到影响。
PLMs能够为任意的蛋白质序列生成表征,这些通用的表征蕴含蛋白质的功能信息。利用PLMs将蛋白质序列编码为信息丰富的嵌入向量作为下游机器学习分类器的输入特征,结合具体的功能注释数据集和机器学习或者深度学习算法,即可通过微调获得功能注释的模型。在实际的应用中,研究人员只需收集一个带有功能标签(如GO标签、EC号等)的小型数据集,训练一个简单的分类器(如逻辑回归或多层感知机),就能实现对海量未知序列的高效功能预测。例如,FANTASIA这样的工具就是基于这一原理,实现了对整个蛋白质组的高通量功能注释。此外,还可以通过收集蛋白质的物理化学性质数据集,通过微调来获得蛋白质Tm预测模型[53]、最优催化温度预测模型[54]、酸碱稳定性预测模型[55]等。在此类任务中,更精巧的学习策略也能带来性能提升。例如,CLEAN模型[56]便是一个代表。它并没有对整个蛋白质语言模型进行微调,而是采用了一种高效的迁移学习方法:首先,使用预训练好的ESM-1b模型(冻结其参数)来提取蛋白质序列的嵌入向量;然后,通过对比学习来训练一个映射模块,将该序列向量与对应的酶功能(EC号)的文本描述在同一个表示空间中进行对齐。通过这种方式,CLEAN学会了精准地将蛋白质序列与其功能描述关联起来,在酶功能注释任务上表现出优异的性能。
4.1.2 突变设计突变设计指预测单个或多个氨基酸突变对蛋白质功能(如稳定性、活性、结合亲和力)的影响。PLMs能够从多场景出发来预测蛋白质的突变功能,包括单点突变预测和数据驱动的高点位突变预测。
4.1.2.1 单点突变预测凭借在预训练中学习到的蛋白质序列概率分布建模能力,PLMs能够对突变进行似然度排序。其基本思想是,自然进化倾向于保留功能正常的蛋白质,因此天然序列的似然度应该较高[57]。一个导致功能丧失的突变会产生一个“不自然”的序列,其似然度也相应较低。因此,通过计算野生型序列和突变型序列的似然度对数比,就可以直接评估突变的效应。这种预测事先无需目标蛋白的突变功能数据,仅依靠蛋白质的序列信息,因此被称作零样本突变效应预测[25]。基于这一原理,多款PLMs被开发出来用于预测蛋白质突变效应,包括ESM-1v模型[25]、Tranception模型[38]、SaProt模型[58]、ProSST模型[59]、VenusREM模型[60]、Venus-EEM模型[61]等。此外,还有专门用于小样本预测的模型FSFP[62],其能够在仅利用几十个数据的情况下显著提升蛋白质语言模型单点位突变预测的阳性率。哈佛大学医学院和牛津大学研究人员建立了ProteinGym高通量基准测试平台[63],上海交通大学建立了Venus-MutHub低通量基准测试平台[64],它们能够从多个维度衡量蛋白质语言模型的零样本突变预测效果。其中,VenusMutHub的系统性评估进一步揭示了不同模型的能力边界与适用场景。研究发现,模型的性能表现出强烈的任务依赖性:例如,基于结构信息的模型在稳定性预测中占优,而依赖进化信息的模型则在活性预测上表现更佳。同时,该基准测试也暴露了当前所有PLMs的共同短板,即在处理多点突变时难以捕捉上位效应(epistatic effects),以及在预测选择性等更复杂功能时表现普遍不佳,这为未来模型的发展指明了需要攻克的方向。
4.1.2.2 数据驱动的高点位突变预测一般来说,蛋白质工程改造蛋白质达到理想的性能指标依赖于高点突变。PLMs能够从单点位或高点位突变中学习突变序列到功能的映射,并且能够以多轮迭代的方式提升模型预测的精度,最终完成大幅度的模型提高。上海交通大学提出了PRIME蛋白质语言模型[53],其不仅能够以零样本的方式预测蛋白质单点位突变的性能,还能够通过多轮迭代的方式提升模型的预测精度,其预测在湿实验中得到了验证,超过30%的推荐突变体在热稳定性、催化活性或结合亲和力等方面显著提升。EVOLVEpro模型[65]则利用主动学习框架,能够仅利用少量实验数据指导蛋白质进化。EVOLVEpro模型将PLMs提取的突变序列的嵌入向量与机器学习模型结合,通过几轮“设计-构建-测试-学习”的迭代,能够从极少量的实验数据点中学习序列与功能的关系。与依赖进化信息的模型不同,METL框架[66]为解决数据稀缺问题提供了新思路。它创新地在生物物理模拟数据(而非进化序列)上进行预训练,通过学习预测数百万突变体的物理属性(如能量、稳定性),为模型注入“物理先验知识”。这使得METL在微调时仅需极少量实验数据,就能在小样本和跨任务中表现出色。实验验证,仅用64个GFP数据点,METL就成功设计出多个具有活性的高阶突变体,证明了融合物理学知识能有效提升模型在蛋白质工程中的泛化能力。
4.1.3 结构预测尽管AlphaFold2在结构预测上取得了巨大成功,但其严重依赖于多序列比对(MSA)的输入,对于缺乏同源序列的孤儿蛋白(orphan protein)效果不佳。蛋白质语言模型为此提供了新的解决思路。由于PLMs在预训练阶段已经学习了蕴含在海量序列中的进化和结构规律,它们有潜力直接从单条序列预测三维结构。代表性工作是Meta AI开发的ESMFold[26]。该模型在大型语言模型ESM-2的基础上,加入了一个结构预测模块(folding head),实现了端到端、无需MSA的结构预测。虽然其总体精度略低于AlphaFold2,但在没有同源序列的情况下,ESMFold的表现尤为出色。更重要的是,通过完全绕过传统模型中最为耗时的MSA搜索环节,ESMFold的推理速度得到了极大提升。这种显著的效率优势使其在需要对数百万乃至数十亿序列进行预测的高通量场景中,如宏基因组结构注释(metagenomic structure annotation),具有无可替代的独特价值,为快速、大规模的结构预测以及孤儿蛋白的研究提供了强大的工具。
随着AlphaFold3[67]等新一代扩散模型的出现,结构预测的精度再创新高,但其巨大的计算开销也为大规模应用带来了挑战。针对这一问题,近期提出的Protenix-Mini模型[68]探索了构建轻量化、高效率结构预测器的新途径。在其Protenix-Mini-ESM变体中,研究者用预训练好的PLM(ESM2-3B)的嵌入向量完全替代了耗时的MSA搜索与处理模块,并通过一种混合训练策略,让模型学会同时从MSA或PLM嵌入中提取信息。在推理时,可以完全绕过MSA,实现快速预测。更重要的是,Protenix-Mini还通过精简模型架构(如减少Transformer层数)和优化采样算法(将数百步的扩散采样降至仅需两步),在仅牺牲1%~5%精度的前提下,大幅降低了计算成本。这一系列工作表明PLMs不仅是解决孤儿蛋白预测问题的关键,更是实现“高精度”与“高效率”平衡、推动结构预测技术走向更广泛实际应用的重要引擎。
4.1.4 交互式蛋白质问答近期,研究者们开始将蛋白质语言模型与通用大语言模型(LLMs)相结合,构建了交互式的蛋白质问答系统,进一步提升了从序列/结构中获取功能信息的能力。这类模型,如ProteinChat[69]和ProteinGPT[70],允许用户以自然语言对话的方式对蛋白质进行提问。用户可以上传一个蛋白质的序列或三维结构,然后直接询问“这个蛋白的催化位点在哪里?”或“它与哪些药物可能相互作用?”。模型内部通过PLMs将蛋白质信息编码为向量,再将其与用户的问题一同输入到一个大型语言模型中进行理解和回答。这种交互式范式极大地降低了生物信息分析的门槛,为研究人员提供了一个直观探索蛋白质功能的新途径。
4.2 从功能到序列从功能到序列是从序列到功能的对偶任务,即根据期望的功能,推出对应的氨基酸序列。以下介绍两种从功能到序列的研究方法:酶挖掘和从头设计。
4.2.1 酶挖掘酶挖掘的目标是从天然或宏基因组序列数据库中,发现具有特定催化活性的新酶序列。PLMs为两个蛋白质序列计算出的嵌入向量的相似度在一定程度上能够反映两个蛋白质的功能相似性。例如,上海交通大学洪亮教授团队开发的VenusMine模型[71]将PLMs与三维结构分析相结合。首先利用PLMs提取的高维特征对海量序列进行聚类和初步筛选,然后评估与已知具备PET水解功能的酶结构相似的候选蛋白。这种优先考虑结构相似性而非序列同源性的策略,成功地从数千万个候选蛋白中发现了多种全新的、催化效率和热稳定性均优于已知酶的PET水解酶,证明了蛋白质语言模型在酶挖掘方面的巨大潜力。此外,PLMs同样可以通过微调直接估计两条蛋白质的结构相似性。PLMSearch[72]是一种仅仅依赖序列输入的快速同源搜索方法,其利用预训练语言模型生成的深度表征来训练一个能够预测蛋白质间结构相似度(TM-score)的模型,使得PLMSearch能够捕捉到传统序列比对方法BLAST无法识别的远缘同源关系,在保持极高搜索速度的同时,其灵敏度甚至超越了基于三维结构的搜索方法,尤其擅长发现序列差异大但结构和功能相似的新酶。
在此基础上,近期由西湖大学原发杰教授团队开发的ProTrek模型[73]在多模态蛋白质搜索方面取得了进一步的突破。ProTrek是一个创新的三模态语言模型,它通过对比学习将蛋白质的序列、三维结构以及由自然语言描述的功能信息统一到一个联合嵌入空间中。这种设计使得ProTrek不仅能进行传统的序列或结构比对,更开创性地实现了基于文本描述的蛋白质搜索。研究者可以直接用自然语言(例如“能够从DNA中移除尿嘧啶的糖基化酶”)来查询庞大的蛋白质数据库,精准地找到具备特定功能的候选酶,甚至能够发现那些序列和结构上无显著同源性,但功能上趋同进化的蛋白质。
4.2.2 从头设计从头设计(de novo design)旨在创造出自然界中不存在的、具有全新或优化功能的蛋白质序列。自回归蛋白质语言模型是实现从头设计的重要工具。ProGen模型[37]通过在包含功能标签的2.8亿条序列上进行训练,学会了根据指定的标签生成相应的序列。其实验验证工作表明ProGen生成的多种全新溶菌酶的序列与任何已知天然蛋白的同一性低至31.4%,但在体外实验中表现出与天然溶菌酶相当的催化活性,表明了PLMs具备了生成新蛋白的能力。扩散蛋白质模型同样能够完成从头设计任务,例如EvoDiff[50]等模型能够在无结构信息的条件下,仅从序列出发生成多样化且结构合理的蛋白质。而多模态扩散模型如DPLM-2[49]则能同时生成序列和结构,确保了设计出的序列能够折叠成稳定的三维形态。
一些最新的研究前沿甚至开始探索从自然语言描述直接设计蛋白质。例如,ProteinDT模型[74]利用蛋白质的文本描述来指导蛋白质设计。首先,通过对比学习模型ProteinCLAP对齐文本和蛋白质的表示;其次,由一个促进器从文本生成蛋白质表示;最后,一个解码器根据此表示生成蛋白质序列。实验证明,ProteinDT在文本引导的蛋白质生成、零样本蛋白质编辑和属性预测等多项任务中表现出色,验证了融合文本信息在蛋白质设计中的有效性。Pinal模型[75]则提出了一种两阶段方法:首先将用户的自然语言指令(如“设计一个能结合X靶点的蛋白”)翻译成一个抽象的蛋白质结构表示,然后再利用逆折叠模型生成符合该结构和语言描述的序列。这种方法通过在更小的结构空间中进行搜索,有效地约束了广阔的序列空间,比直接进行端到端的文本到序列生成更为高效和可靠。
5 可解释性:从统计规律到物理原理尽管蛋白质语言模型在各项任务中取得了显著成功,但其如同“黑箱”般的内部工作机制限制了我们对其决策过程的深入理解与信任。因此,可解释性研究旨在揭示模型“知其然”背后的“所以然”。本节将遵循“表征特征发现-决策机制解析-物理基础验证”的递进逻辑,深入探讨PLMs是仅仅记忆了序列中的统计规律,还是在某种程度上学习到了驱动蛋白质功能的物理化学原理,从而在AI模型与生物物理第一性原理之间建立桥梁。
5.1 表征特征发现:模型嵌入向量中的生物学信息可解释性研究的首要任务是打开“黑箱”,理解其高维表示中编码了何种生物学信息。研究发现,直接分析单个神经元难以与特定生物学概念对应。因此,研究者致力于将复杂的内部表示分解为人类可理解的特征。近期,以稀疏自编码器(SAE)为代表的字典学习方法取得了显著进展。例如,InterPLM框架[76]在ESM-2模型上应用SAE,成功将其内部表征分解为数千个与生物学概念(如结合位点、结构基序)高度对齐的、更稀疏且更可解释的“特征”。这类研究证实,PLMs的内部表示确实捕捉到了丰富的、有意义的生物学概念,这为后续的决策机制分析奠定了基础。
5.2 决策机制解析:统计记忆还是物理模拟?在确认模型学到了生物学特征之后,下一个关键问题是:PLMs在进行预测时,究竟是像物理引擎一样进行模拟,还是更像一个高效的“查询系统”,仅依赖于对进化统计规律的记忆与调用?
多项研究证据倾向于后者。一项针对ESM-2的研究[77]发现,模型主要依赖局部序列基序进行接触预测,并且其内部的共进化信号与传统统计模型高度相似,这表明其行为更接近于查找统计规律。对模型“偏好”的进一步研究也支持了这一假说。Gordon等[78]发现,PLMs在零样本突变预测任务上的表现,与其对野生型序列的“偏好程度”(通过似然度量化)密切相关,而这种偏好主要源于训练集中同源序列的存在与相似度。这些证据共同表明,当前PLMs的能力主要源于其对海量数据中进化统计规律的高效记忆和调用,而非对物理折叠过程的模拟。
5.3 物理基础验证:连接统计规律与物理原理然而,将PLMs的能力完全归结于统计记忆可能忽略了其学习到的规律背后所蕴含的物理意义。Frellsen等[79]的工作为模型的统计预测能力与物理现实之间建立了坚实的桥梁。他们从热力学第一性原理出发,严格推导了逆折叠模型计算的似然度对数比与蛋白质稳定性自由能变(ΔΔG)之间的数学关系。该研究阐明,当前普遍使用的零样本预测方法,在理论上是物理模型的一个有效近似,这解释了其预测结果为何与实验有很高的相关性。这项工作巧妙地证明了PLMs学习到的进化统计规律,在特定任务(如稳定性预测)上可以成为复杂生物物理原理的有效代理(proxy)。它不仅为AI模型的黑箱决策提供了物理解释,也为基于物理理解改进模型(如通过引入结构系综)指明了方向。
6 模型的发展仅基于序列的语言模型没有考虑蛋白质赖以发挥功能的三维结构。为了让模型学习到更符合生物规律的知识,研究者尝试向蛋白质语言模型中引入多模态信息,其中最关键的就是结构信息,可以使用蛋白质的二级结构、三级结构或者是序列化的结构来增强蛋白质语言模型的性能。同样地,功能信息、MSA信息等也可以被用于增强蛋白质语言模型。此外,自然语言作为一种人类使用的对蛋白质功能的理解符号,也可以被用来增强蛋白质语言模型的性能,尤其是在引入蛋白质的功能约束,例如ESM-3模型[17]就采用了此方案。表 6列举了目前通过引入多模态信息增强的蛋白质语言模型。多模态信息的融合通常采用以下几种策略:(1)输入层融合:将结构或功能信息的嵌入向量直接与序列嵌入拼接或求和,或者通过扩增词表来表示。例如,SaProt模型[60]利用Foldseek工具将蛋白质三维结构离散化为结构字母表,与氨基酸序列拼接形成“结构感知序列”作为模型输入;ProSST模型[61]通过VQ-VAE将局部结构量化为离散符号,与序列符号交替编码。(2)中间层交互:利用交叉注意力机制或提示学习在模型的Transformer层中引入外部模态信息。例如,LM-Design模型[87]通过交叉注意力机制使语言模型在生成序列时动态关注结构编码器提供的空间信息;PromptProtein模型[83]将结构和属性信息编码为软提示向量,插入到Transformer中间层引导模型学习;ESM-IF模型[45]和ProteinMPNN模型[48]均采用编码器-解码器架构,在解码器的每一层通过交叉注意力接收结构编码器的嵌入表示。(3)输出层对齐:通过对比学习等目标函数,在共享的潜在空间中对齐序列与结构/功能的表征。例如,OntoProtein模型[84]利用对比学习将蛋白质序列表征与基因本体知识图谱嵌入对齐,使表征隐式编码功能语义;ProTrek模型[75]通过三模态对比学习,将序列、结构和自然语言功能描述统一到同一嵌入空间,实现跨模态检索。
| 表 6 引入外部知识增强的蛋白质语言模型 |
尽管蛋白质语言模型已经取得了较大的发展,但其仍面临着技术挑战,本节对涌现问题和数据问题对蛋白质语言模型的挑战做了分析,并且展望了蛋白质语言模型的发展。
7.1 涌现问题在自然语言处理领域,扩展定律[5]与涌现现象[89]是驱动大型语言模型参数规模持续增长的重要原因。扩展定律揭示了模型性能与模型参数量、训练数据量及计算资源之间存在着可预测的幂律关系。而“涌现”则指当模型规模突破某一临界点后,会表现出小型模型所不具备的、解决复杂任务的全新能力。这两种现象表明模型越大,能力越强,推动了学界和业界对更大规模模型的探索。
然而,蛋白质语言模型的扩展定律与涌现现象并没有明显的证据。已有研究,如Cheng等[8]的工作确实初步证实了PLMs中存在扩展定律:随着模型参数和计算量的增加,预训练任务的损失函数值显著下降。尽管如此,预训练损失的降低并未稳定地转化为下游任务性能的提升。研究表明,PLMs的性能与参数量之间并非简单的正相关关系。例如,Chen等[7]在评估xTrimoPGLM时发现,仅不到一半占比的下游任务性能随模型增大而改善,甚至有部分的任务表现出性能随规模增大反而下降的逆缩放现象。同样,Hesslow等[6]在RITA模型的研究中也未观察到酶功能及突变功能预测任务存在“涌现”,其性能增长是渐进式的。Lin等[26]对ESM-2模型的评估也得出了类似的结论,即模型性能随参数增加的提升幅度缓慢。
综上所述,当前研究尚未在蛋白质语言模型中发现明显的涌现现象,因此“模型越大越好”的假设在蛋白质领域尚未得到充分验证。蛋白质语言模型暂未发现涌现现象的原因可能是蛋白质序列与自然语言文本具有本质的差别,与NLP中常出现涌现的离散型、认知型任务不同,蛋白质功能预测多为回归任务,旨在预测连续的生物物理值。其评价指标(如皮尔逊相关系数、RMSE)天然地反映了渐进式的性能提升,这是一个平滑的量变过程,而非“从无到有”的突变,因此难以观测到涌现现象。蛋白质序列缺乏明确的词汇单元,其功能又由三维空间结构决定,导致序列中存在大量复杂的长程依赖关系。加之生物学下游任务的标记数据往往非常稀疏,有限的标记数据量为模型性能设定了“天花板”,当模型规模大到足以完全拟合这些数据时,继续增大参数量将无法带来收益;且实验数据本身可能包含系统性误差和固有噪声,过度强大的模型反而容易过拟合这些噪声,导致泛化能力下降。这些因素共同导致了在数据受限的情况下,单纯扩大模型规模可能难以带来预期收益,甚至中小规模的模型表现可能更优[90]。
7.2 数据问题自然语言和蛋白质语言模型的研究都表明,模型扩展必须伴随着高质量数据的同步增长。虽然近年来测序技术的发展为蛋白质语言模型提供了大量潜在训练数据,但与自然语言动辄数十万亿的数据量相比,蛋白质语言模型所能使用的高质量序列仍有限。更严重的问题在于,不同数据库对蛋白质序列的定义标准尚未统一。例如,RefSeq数据库[91]根据RNA确定蛋白质序列,而其他数据库,例如Ensembl数据库[92],可能依据DNA或实验数据进行识别。在UniProt数据库[22, 28]中,真正经过实验验证的蛋白质序列仅有约百万条[33],其余数亿条多源于宏基因组拼接或同源比对,缺乏足够的功能验证支持。这种数据规模不足与质量欠佳的双重制约,显著阻碍了蛋白质语言模型的发展。
7.3 极端环境数据问题极端环境数据对于提升模型泛化能力至关重要。目前用于训练蛋白质语言模型的数据主要来源于常规环境的生物体系,导致模型在应对极端环境蛋白质时可能会存在偏差。极端环境(高温、高压、强酸、强碱)中的蛋白质序列在氨基酸组成、结构折叠模式方面与常规环境的蛋白质不同,因此构建具备环境多样性的训练数据是提升模型性能一个重要方法。应用极端环境的数据,能够提升模型在工业生产环境下的能力,设计出更符合极端工业生产环境的蛋白质。
在高温环境中,如海底热泉生态系统(温度可达300 ℃以上),嗜热菌体内的蛋白质表现出独特的结构稳定性。研究表明,这类嗜热蛋白通常具有独特的结构性质和氨基酸组成模式,例如通过谷氨酸替代天冬氨酸,增强静电相互作用稳定性[93, 94]。然而,相关序列数据在公共数据库(如UniProt、BFD)中的覆盖率极低,使得蛋白质语言模型难以充分学习热稳定性特征。
在高压生态系统中,如马里亚纳海沟及其深渊带(可达1 100个大气压),深海嗜压生物的蛋白质表现出与常压生物显著不同的序列分布与动力学特征。研究表明,这些蛋白质往往通过减少内部空腔体积、强化范德华相互作用、优化极性残基分布来缓解压缩效应[95-98]。此类数据对模型学习体积依赖性、稳定性与压强敏感残基替换模式具有重要价值,但目前仅占公开序列数据库的极少部分,可能会导致模型在耐高压的场景下的生成与预测性能不足。上海交通大学肖湘教授牵头发起的“MEER计划”[99],依托“奋斗者”号载人潜水器开展深海取样与生态研究,系统采集了沉积物、水体、宏生物与微生物样本,在6 000~11 000米水深区域获得超过2 000份样本,构建多样性丰富深渊微生物数据库[100],对模型学习“高压适应”特征极具补充意义。
在碱性环境中,如青海湖(pH 9~11),嗜碱性生物蛋白质展现出显著的电荷调控特征。例如,研究发现青海湖裸鲤(Gymnocypris przewalskii) 在高盐碱环境中上调与离子稳态、折叠伴侣及抗氧化相关的基因表达,其蛋白质序列普遍减少赖氨酸与精氨酸、增加天冬氨酸与谷氨酸残基,从而调节整体电荷分布[101-103]。这些序列对模型捕捉pH依赖性结构变化极为关键,但因样本稀缺而在预训练语料中可能会导致利用不足。
在酸性环境中,如酸性矿区或工业酸性废水(pH 1~2),嗜酸菌的蛋白质需要抵御质子化与金属离子胁迫。研究发现,这类蛋白质表面富集酸性残基以形成稳定电荷屏障,同时增强疏水核心和金属结合能力,这种序列适应还伴随抗氧化酶的扩增与蛋白质自修复机制[104, 105]。这些环境相关序列反映了多层级的适应模式,但目前尚未系统地纳入模型训练语料中。
从数据多样性角度看,极端环境蛋白质不仅扩展了序列空间的覆盖范围,也揭示了氨基酸替换模式与结构稳定性之间的多样耦合机制。将此类序列系统性纳入蛋白质语言模型的训练,可有效缓解模型在常规环境下的过拟合问题,并提升模型在蛋白质稳定性预测、功能注释与定向设计中的可靠性。未来研究应重点构建覆盖温度、压强、pH与盐度梯度的多源蛋白质数据库,结合环境元数据实现条件化语言建模,以实现对真实生物环境更具泛化性的蛋白质设计。
7.4 核心科学问题的再思考:超越序列统计尽管PLMs在模仿自然进化产物方面表现出色,但要真正实现“按需设计”的工程目标,领域内仍有几个深层次的科学问题亟待突破。这不仅是技术层面的优化,更是对研究范式的重新审视。
首先,如何从“统计模拟”迈向“物理理解”?当前PLMs本质上是高效的统计相关性学习器,它们通过记忆海量进化数据中的共现模式来“猜测”规律,而非通过理解物理首要原理(如热力学稳定性、动力学路径)来进行推理。这种差异导致模型在面对非自然序列(de novo design)时容易产生“幻觉”——生成的蛋白质可能在序列上看似合理,但无法在物理世界中折叠或行使功能。未来的研究需要探索如何将可微的生物物理约束(differentiable biophysics)显式地嵌入到语言模型的损失函数中,使模型不仅学习“像不像天然蛋白”,更能学习“能不能稳定存在”。其次,数据困境的破局之道在于“主动”而非“被动”。盲目追求模型参数和数据量的扩展(scaling laws)在生物学领域可能面临边际效应递减的风险,因为高质量的湿实验标签数据永远无法像互联网文本那样廉价获取。因此,未来的范式应从“被动的大数据喂养”转向“主动的智能探索”。这就要求赋予PLMs“好奇心”——即利用主动学习(active learning)和贝叶斯优化框架,让模型能够识别自身知识的边界,主动筛选那些“最能证伪模型假设”的高价值序列进行湿实验验证。这种“干湿闭环”(dry-wet loop)的迭代模式,将是突破数据瓶颈、实现小样本学习的关键。最后,探索蛋白质空间的“暗物质”。现有的训练数据主要覆盖了自然界中经过亿万年进化筛选的“成功者”,而忽略了无数在进化中被淘汰的“失败者”(负样本),以及尚未被自然界探索的广阔序列空间。这种“幸存者偏差”限制了模型对蛋白质适应性景观(fitness landscape)全貌的理解。未来的创新方向之一是利用物理模拟生成大量的负样本数据,或者系统性地收集突变实验中的失效数据,教会模型“什么是错误的”,这对于提升模型在从头设计任务中的鲁棒性可能比学习“什么是正确的”更为重要。
8 总结蛋白质语言模型是人工智能与生命科学深度交叉的前沿领域,在理解蛋白质“序列-功能”的关系中发挥了重要作用。本综述总结了蛋白质语言模型的模型架构、应用场景和发展趋势,并对其当前面临的挑战与未来发展趋势进行了展望。
在过去数年间,该领域见证了显著的进展。模型架构已从早期的判别式模型演进至功能多样的生成式模型,其中包括旨在根据结构或功能约束生成序列的自回归与逆折叠模型,以及近期涌现的、在生成质量和灵活性上更具优势的扩散模型。这一发展轨迹清晰地反映出该领域核心目标的转变:从最初对现有蛋白质的理解,迈向了对全新功能蛋白的挖掘与设计。
然而蛋白质语言模型仍面临若干瓶颈。首先,数据基础仍需夯实,要求进一步挖掘现有生物数据库,并扩充高质量、多样化的数据资源。其次,多模态信息的融合是提升模型能力的关键,通过有效整合结构、功能及进化信息,有望显著增强模型的表征精度与生成性能。
综上所述,尽管蛋白质语言模型已在多个方向取得令人鼓舞的成果,但其领域的爆炸性突破尚未到来。如何持续提升模型性能、拓展应用场景,并揭示其背后潜在的生物学规律,仍是AI辅助蛋白质工程领域的核心议题。推进这一进程不仅将为合成生物学的发展注入新的动力,更将为生命健康及相关产业的创新与突破提供强有力的计算引擎。
| [1] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-9. DOI:10.1038/s41586-021-03819-2 |
| [2] |
余元玺, 钟博子韬, 洪亮. 人工智能的诺奖时刻: 重塑科学的未来. 物理, 2025, 54: 25-9. |
| [3] |
Devlin J, Chang M, Lee K, et al. Bert: pre-training of deep bidirectional transformers for language understanding. North American chapter of the Association for Computational Linguistics, 2019, 4171-86. |
| [4] |
Radford A, Narasimhan K, Salimans T, et al. Improving language understanding by generative pre-training. OpenAI, 2018, 2018: 1-12. |
| [5] |
Kaplan J, McCandlish S, Henighan T, et al. Scaling laws for neural language models. arXiv, 2020, doi: 10.48550/arXiv.2001.08361
|
| [6] |
Hesslow D, Zanichelli N, Notin P, et al. Rita: a study on scaling up generative protein sequence models. arXiv, 2022, doi: 10.48550/arXiv.2205.05789
|
| [7] |
Chen B, Cheng X, Li P, et al. xTrimoPGLM: unified 100-billion-parameter pretrained transformer for deciphering the language of proteins. Nat Methods, 2025, 22: 1028-39. DOI:10.1038/s41592-025-02636-z |
| [8] |
Cheng X, Chen B, Li P, et al. Training compute-optimal protein language models[C]//Proceeding of the 38th International Conference on Neural Information Processing Systems. New York, USA: Curran Associates Inc, 2024: 69386-418
|
| [9] |
Vince O, Oldach P, Pereno V, et al. Breaking through biology's data wall: expanding the known tree of life by over 10x using a global biodiscovery pipeline. bioRxiv, 2025, doi: 10.1101/2025.06.11.658620
|
| [10] |
UniProt: the universal protein knowledgebase in 2023. Nucleic Acids Res, 2023, 51: D523-D31
|
| [11] |
Mitchell AL, Almeida A, Beracochea M, et al. MGnify: the microbiome analysis resource in 2020. Nucleic Acids Res, 2020, 48: D570-D8. |
| [12] |
Markowitz VM, Korzeniewski F, Palaniappan K, et al. The integrated microbial genomes (IMG) system. Nucleic Acids Res, 2006, 34: D344-D8. DOI:10.1093/nar/gkj024 |
| [13] |
Berman HM, Westbrook J, Feng Z, et al. The protein data bank. Nucleic Acids Res, 2000, 28: 235-42. DOI:10.1093/nar/28.1.235 |
| [14] |
Yang KK, Alamdari S, Lee AJ, et al. The Dayhoff Atlas: scaling sequence diversity for improved protein generation. bioRxiv, 2025, doi: 10.1101/2025.07.21.665991
|
| [15] |
Bhatnagar A, Jain S, Beazer J, et al. Scaling unlocks broader generation and deeper functional understanding of proteins. bioRxiv, 2025, doi: 10.1101/2025.04.15.649055
|
| [16] |
Cornman A, West-Roberts J, Camargo AP, et al. The OMG dataset: an Open MetaGenomic corpus for mixed-modality genomic language modeling. bioRxiv, 2024, doi: 10.1101/2024.08.14.607850
|
| [17] |
Hayes T, Rao R, Akin H, et al. Simulating 500 million years of evolution with a language model. Science, 2025, 387: 850-8. DOI:10.1126/science.ads0018 |
| [18] |
Mirdita M, Schütze K, Moriwaki Y, et al. ColabFold: making protein folding accessible to all. Nat Methods, 2022, 19: 679-82. DOI:10.1038/s41592-022-01488-1 |
| [19] |
Rao R, Bhattacharya N, Thomas N, et al. Evaluating protein transfer learning with TAPE. Adv Neural Inf Process Syst, 2019, 32: 9689-701. |
| [20] |
Bateman A, Coin L, Durbin R, et al. The Pfam protein families database. Nucleic Acids Res, 2004, 32: D138-D41. DOI:10.1093/nar/gkh121 |
| [21] |
Elnaggar A, Heinzinger M, Dallago C, et al. ProtTrans: toward understanding the language of life through self-supervised learning. IEEE Trans Pattern Anal Mach Intell, 2022, 44: 7112-27. DOI:10.1109/TPAMI.2021.3095381 |
| [22] |
Suzek BE, Wang Y, Huang H, et al. UniRef clusters: a comprehensive and scalable alternative for improving sequence similarity searches. Bioinformatics, 2014, 31: 926-32. |
| [23] |
Steinegger M, Mirdita M, Söding J. Protein-level assembly increases protein sequence recovery from metagenomic samples manyfold. Nat Methods, 2019, 16: 603-6. DOI:10.1038/s41592-019-0437-4 |
| [24] |
Rives A, Meier J, Sercu T, et al. Biological structure and function emerge from scaling unsupervised learning to 250 million protein sequences. Proc Natl Acad Sci USA, 2021, 118: e2016239118. DOI:10.1073/pnas.2016239118 |
| [25] |
Meier J, Rao R, Verkuil R, et al. Language models enable zero-shot prediction of the effects of mutations on protein function. Adv Neural Inf Process Syst, 2021, 29287-303. |
| [26] |
Lin Z, Akin H, Rao R, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [27] |
EvolutionaryScale. ESM Cambrian: revealing the mysteries of proteins with unsupervised learning2024[EB/OL]. https://www.evolutionaryscale.ai/blog/esm-cambrian
|
| [28] |
Consortium TU. UniProt: the universal protein knowle-dgebase in 2025. Nucleic Acids Res, 2024, 53: D609-D17. |
| [29] |
Richardson L, Allen B, Baldi G, et al. MGnify: the microbiome sequence data analysis resource in 2023. Nucleic Acids Res, 2022, 51: D753-D9. |
| [30] |
Grigoriev IV, Nordberg H, Shabalov I, et al. The genome portal of the department of energy joint genome institute. Nucleic Acids Res, 2011, 40: D26-D32. |
| [31] |
Yang KK, Fusi N, Lu AX. Convolutions are competitive with transformers for protein sequence pretraining. Cell Syst, 2024, 15: 286-94. DOI:10.1016/j.cels.2024.01.008 |
| [32] |
He L, Jin P, Min Y, et al. SFM-Protein: integrative co-evolutionary pre-training for advanced protein sequence representation. arXiv, 2024, doi: 10.48550/arXiv.2410.24022
|
| [33] |
Fournier Q, Vernon RM, van der Sloot A, et al. Protein language models: is scaling necessary? bioRxiv, 2024, doi: 10.1101/09.23.614603
|
| [34] |
Olsen TH, Boyles F, Deane CM. Observed antibody space: a diverse database of cleaned, annotated, and translated unpaired and paired antibody sequences. Protein Sci, 2022, 31: 141-6. DOI:10.1002/pro.4205 |
| [35] |
Alley EC, Khimulya G, Biswas S, et al. Unified rational protein engineering with sequence-based deep representation learning. Nat Methods, 2019, 16: 1315-22. DOI:10.1038/s41592-019-0598-1 |
| [36] |
Ferruz N, Schmidt S, Höcker B. ProtGPT2 is a deep unsupervised language model for protein design. Nat Commun, 2022, 13: 4348. DOI:10.1038/s41467-022-32007-7 |
| [37] |
Madani A, Krause B, Greene ER, et al. Large language models generate functional protein sequences across diverse families. Nat Biotechnol, 2023, 41: 1099-106. DOI:10.1038/s41587-022-01618-2 |
| [38] |
Notin P, Dias M, Frazer J, et al. Tranception: protein fitness prediction with autoregressive transformers and inference-time retrieval. PMLR, 2022, 162: 16990-7017. |
| [39] |
Nguyen E, Poli M, Durrant MG, et al. Sequence modeling and design from molecular to genome scale with Evo. Science, 2024, 386: eado9336. DOI:10.1126/science.ado9336 |
| [40] |
Brixi G, Durrant MG, Ku J, et al. Genome modeling and design across all domains of life with Evo2. bioRxiv, 2025, doi: 10.1101/02.18.638918v1
|
| [41] |
Zhang X, Yin H, Ling F, et al. SPIN-CGNN: improved fixed backbone protein design with contact map-based graph construction and contact graph neural network. PLoS Comput Biol, 2023, 19: e1011330. DOI:10.1371/journal.pcbi.1011330 |
| [42] |
Sillitoe I, Bordin N, Dawson N, et al. CATH: increased structural coverage of functional space. Nucleic Acids Res, 2021, 49: D266-D73. DOI:10.1093/nar/gkaa1079 |
| [43] |
Hsu C, Verkuil R, Liu J, et al. Learning inverse folding from millions of predicted structures. International Conference on Machine Learning, 2022, 8946-70. |
| [44] |
Barrio Hernandez I, Yeo J, Jänes J, et al. Clustering predicted structures at the scale of the known protein universe. Nature, 2023, 622: 637-45. DOI:10.1038/s41586-023-06510-w |
| [45] |
Varadi M, Anyango S, Deshpande M, et al. AlphaFold protein structure database: massively expanding the structural coverage of protein-sequence space with high-accuracy models. Nucleic Acids Res, 2022, 50: D439-D44. DOI:10.1093/nar/gkab1061 |
| [46] |
Dauparas J, Anishchenko I, Bennett N, et al. Robust deep learning-based protein sequence design using ProteinMPNN. Science, 2022, 378: 49-56. DOI:10.1126/science.add2187 |
| [47] |
Gao Z, Tan C, Li SZ, et al. PiFold: toward effective and efficient protein inverse folding. arXiv, 2022, doi: 10.48550/arXiv.2209.12643
|
| [48] |
Wang X, Zheng Z, Ye F, et al. Diffusion language models are versatile protein learners. arXiv, 2024, doi: 10.48550/arXiv.2402.18567
|
| [49] |
Wang X, Zheng Z, Xue D, et al. DPLM-2: a multimodal diffusion protein language model. arXiv, 2024, doi: 10.48550/arXiv.2410.13782
|
| [50] |
Alamdari S, Thakkar N, Van Den Berg R, et al. Protein generation with evolutionary diffusion: sequence is all you need. bioRxiv, 2023, doi: 10.1101/2023.09.11.556673
|
| [51] |
Ahdritz G, Bouatta N, Floristean C, et al. OpenFold: retraining alphaFold2 yields new insights into its learning mechanisms and capacity for generalization. Nat Methods, 2024, 21: 1514-24. DOI:10.1038/s41592-024-02272-z |
| [52] |
Lin Z, Hao L, Lv L, et al. Taxdiff: taxonomic-guided diffusion model for protein sequence generation. Sci China Inf Sci, 2025, 68: 149101. DOI:10.1007/s11432-024-4296-6 |
| [53] |
Jiang F, Li M, Dong J, et al. A general temperature-guided language model to design proteins of enhanced stability and activity. Sci Adv, 2024, 10: eadr2641. DOI:10.1126/sciadv.adr2641 |
| [54] |
Li M, Zhang L, Wang Z, et al. Learning temperature-aware representations from millions of annotated protein sequences. Neurips 2024 Workshop Foundation Models for Science: Progress, Opportunities, and Challenges. |
| [55] |
Zhang L, Luo K, Zhou Z, et al. A deep retrieval-enhanced meta-learning framework for enzyme optimum pH prediction. J Chem Inf Modeling, 2025, 65: 3761-70. DOI:10.1021/acs.jcim.4c02291 |
| [56] |
Yu T, Cui H, Li JC, et al. Enzyme function prediction using contrastive learning. Science, 2023, 379: 1358-63. DOI:10.1126/science.adf2465 |
| [57] |
Riesselman AJ, Ingraham JB, Marks DS. Deep generative models of genetic variation capture the effects of mutations. Nat Methods, 2018, 15: 816-22. DOI:10.1038/s41592-018-0138-4 |
| [58] |
Su J, Han C, Zhou Y, et al. SaProt: protein language modeling with structure-aware vocabulary. biorxiv, 2024, doi: 10.1101/2023.10.01.560349
|
| [59] |
Li M YT, Ma X, Zhong B, et al. ProSST: protein language modeling with quantized structure and disentangled attention. Adv Neural Inf Process Syst, 2024, 35700-26. |
| [60] |
Tan Y, Wang R, Wu B, et al. From high-throughput evaluation to wet-lab studies: advancing mutation effect prediction with a retrieval-enhanced model. Bioinformatics, 2025, 41: i401-i9. DOI:10.1093/bioinformatics/btaf189 |
| [61] |
Yu Y, Jiang F, Zhong B, et al. Entropy-driven zero-shot deep learning model selection for viral proteins. Phys Rev Res, 2025, 7: 013229. DOI:10.1103/PhysRevResearch.7.013229 |
| [62] |
Zhou Z, Zhang L, Yu Y, et al. Enhancing efficiency of protein language models with minimal wet-lab data through few-shot learning. Nat Commun, 2024, 15: 5566. DOI:10.1038/s41467-024-49798-6 |
| [63] |
Notin P, Kollasch A, Ritter D, et al. ProteinGym: large-scale benchmarks for protein fitness prediction and design. Adv Neural Inf Process Syst, 2023, 64331-79. |
| [64] |
Zhang L, Pang H, Zhang C, et al. VenusMutHub: a systematic evaluation of protein mutation effect predictors on small-scale experimental data. Acta Pharm Sin B, 2025, 15: 2454-67. DOI:10.1016/j.apsb.2025.03.028 |
| [65] |
Jiang K, Yan Z, Di Bernardo M, et al. Rapid in silico directed evolution by a protein language model with EVOLVEpro. Science, 2025, 387: eadr6006. DOI:10.1126/science.adr6006 |
| [66] |
Gelman S, Johnson B, Freschlin C, et al. Biophysics-based protein language models for protein engineering. Nat Methods, 2025, 22: 1868-79. DOI:10.1038/s41592-025-02776-2 |
| [67] |
Abramson J, Adler J, Dunger J, et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature, 2024, 630: 493-500. DOI:10.1038/s41586-024-07487-w |
| [68] |
Gong C, Chen X, Zhang Y, et al. Protenix-mini: efficient structure predictor via compact architecture, few-step diffusion and switchable PLM. arXiv, 2025, doi: 10.48550/arXiv.2507.11839
|
| [69] |
Guo H, Huo M, Zhang R, et al. Proteinchat: towards achieving ChatGPT-like functionalities on protein 3D structures. Authorea, 2023. DOI:10.36227/techrxiv.23120606 |
| [70] |
Xiao Y, Sun E, Jin Y, et al. Proteingpt: multimodal llm for protein property prediction and structure understanding. arXiv, 2024, doi: 10.48550/arXiv.2408.11363
|
| [71] |
Wu B, Zhong B, Zheng L, et al. Harnessing protein language model for structure-based discovery of highly efficient and robust PET hydrolases. Nat Commun, 2025, 16: 6211. DOI:10.1038/s41467-025-61599-z |
| [72] |
Liu W, Wang Z, You R, et al. PLMSearch: protein language model powers accurate and fast sequence search for remote homology. Nat Commun, 2024, 15: 2775. DOI:10.1038/s41467-024-46808-5 |
| [73] |
Su J, He Y, You S, et al. A trimodal protein language model enables advanced protein searches. Nat Biotechnol, 2025. DOI:10.1038/s41587-025-02836-0 |
| [74] |
Liu S, Li Y, Li Z, et al. A text-guided protein design framework. Nat Mach Intell, 2025, 7: 580-91. DOI:10.1038/s42256-025-01011-z |
| [75] |
Dai F, You S, Wang C, et al. Toward de novo protein design from natural language. bioRxiv, 2024, doi: 10.1101/2024.08.01.606258
|
| [76] |
Simon E, Zou J. Interplm: discovering interpretable features in protein language models via sparse autoencoders. Nat Methods, 2025, 22: 2107-17. DOI:10.1038/s41592-025-02836-7 |
| [77] |
Zhang Z, Wayment-Steele HK, Brixi G, et al. Protein language models learn evolutionary statistics of interacting sequence motifs. Proc Natl Acad Sci USA, 2024, 121: e2406285121. DOI:10.1073/pnas.2406285121 |
| [78] |
Gordon C, Lu AX, Abbeel P. Protein language model fitness is a matter of preference. bioRxiv, 2024, doi: 10.1101/2024.10.03.616542
|
| [79] |
Frellsen J, Kassem MM, Bengtsen T, et al. Zero-shot protein stability prediction by inverse folding models: a free energy interpretation. arXiv, 2025, doi: 10.48550/arXiv.2506.05596
|
| [80] |
Brandes N, Ofer D, Peleg Y, et al. ProteinBERT: a universal deep-learning model of protein sequence and function. Bioinformatics, 2022, 38: 2102-10. DOI:10.1093/bioinformatics/btac020 |
| [81] |
Wang Z, Zhang Q, Hu S, et al. Multi-level protein structure pre-training via prompt learning. International Conference on Learning Representations, 2023, 1-10. |
| [82] |
Zhang N, Bi Z, Liang X, et al. OntoProtein: protein pretraining with gene ontology embedding. International Conference on Learning Representations, 2022, 1-10. |
| [83] |
Dumortier B, Liutkus A, Carré C, et al. PeTriBERT: augmenting BERT with tridimensional encoding for inverse protein folding and design. bioRxiv, 2022, doi: 10.1101/2022.08.10.503344
|
| [84] |
Yang KK, Zanichelli N, Yeh H. Masked inverse folding with sequence transfer for protein representation learning. Protein Eng Des Select, 2022, 36: gzad015. |
| [85] |
Zhang Z, Lu J, Chenthamarakshan V, et al. Structure-informed protein language model. arXiv, 2024, doi: 10.48550/arXiv.2402.05856
|
| [86] |
Cheng J, Novati G, Pan J, et al. Accurate proteome-wide missense variant effect prediction with AlphaMissense. Science, 2023, 381: eadg7492. DOI:10.1126/science.adg7492 |
| [87] |
Truong TF, J r, Bepler T. PoET: a generative model of protein families as sequences-of-sequences. Adv Neural Inf Process Syst, 2023, 77379-415. |
| [88] |
Cheng P, Mao C, Tang J, et al. Zero-shot prediction of mutation effects with multimodal deep representation learning guides protein engineering. Cell Res, 2024, 34: 630-47. DOI:10.1038/s41422-024-00989-2 |
| [89] |
Wei J, Tay Y, Bommasani R. Emergent abilities of large language models. Transact Mach Learning Res, 2022, 1: 1-30. |
| [90] |
Vieira LC, Handojo ML, Wilke CO. Scaling down for efficiency: medium-sized protein language models perform well at transfer learning on realistic datasets. bioRxiv, 2025, doi: 10.1101/2024.11.22.624936
|
| [91] |
Goldfarb T, Kodali Vamsi K, Pujar S, et al. NCBI RefSeq: reference sequence standards through 25 years of curation and annotation. Nucleic Acids Res, 2024, 53: D243-D57. |
| [92] |
Dyer SC, Austine-Orimoloye O, Azov AG, et al. Ensembl 2025. Nucleic Acids Res, 2024, 53: D948-D57. |
| [93] |
Fontanillas E, Galzitskaya OV, Lecompte O, et al. Proteome evolution of deep-sea hydrothermal vent alvinellid polychaetes supports the ancestry of thermophily and subsequent adaptation to cold in some lineages. Genome Biol Evol, 2017, 9: 279-96. |
| [94] |
Minic Z, Thongbam PD. The biological deep sea hydrothermal vent as a model to study carbon dioxide capturing enzymes. Mar Drugs, 2011, 9: 719-38. DOI:10.3390/md9050719 |
| [95] |
Wang K, Shen Y, Yang Y, et al. Morphology and genome of a snailfish from the Mariana Trench provide insights into deep-sea adaptation. Nat Ecol Evol, 2019, 3: 823-33. DOI:10.1038/s41559-019-0864-8 |
| [96] |
Caliò A, Dubois C, Fontanay S, et al. Unravelling the adaptation mechanisms to high pressure in proteins. Int J Mol Sci, 2022, 23: 8469. DOI:10.3390/ijms23158469 |
| [97] |
Xiao X, Zhang Y, Wang F. Hydrostatic pressure is the universal key driver of microbial evolution in the deep ocean and beyond. Environ Microbiol Rep, 2021, 13: 68-72. DOI:10.1111/1758-2229.12915 |
| [98] |
Xu H, Fang C, Xu W, et al. Evolution and genetic adaptation of fishes to the deep sea. Cell, 2025, 188: 1393-408. e13. DOI:10.1016/j.cell.2025.01.002 |
| [99] |
Xiao X, Wang J, Ding K. MEER: extraordinary flourishing ecosystem in the deepest ocean. Cell, 2025, 188: 1175-7. DOI:10.1016/j.cell.2024.12.037 |
| [100] |
Xiao X, Zhao W, Song Z, et al. Microbial ecosystems and ecological driving forces in the deepest ocean sediments. Cell, 2025, 188: 1363-77. e9. DOI:10.1016/j.cell.2024.12.036 |
| [101] |
Wei F, Liang J, Tian W, et al. Transcriptomic and proteomic analyses provide insights into the adaptive responses to the combined impact of salinity and alkalinity in Gymnocypris przewalskii. Bioresour Bioprocess, 2022, 9: 104. DOI:10.1186/s40643-022-00589-1 |
| [102] |
Zhang R, Ludwig A, Zhang C, et al. Local adaptation of Gymnocypris przewalskii (Cyprinidae) on the Tibetan Plateau. Sci Rep, 2015, 5: 9780. DOI:10.1038/srep09780 |
| [103] |
Tong C, Li M. Genomic signature of accelerated evolution in a saline-alkaline lake-dwelling Schizothoracine fish. Int J Biol Macromol, 2020, 149: 341-7. DOI:10.1016/j.ijbiomac.2020.01.207 |
| [104] |
Méndez-García C, Peláez AI, Mesa V, et al. Microbial diversity and metabolic networks in acid mine drainage habitats. Front Microbiol, 2015, 6: 475. |
| [105] |
She Z, Wang J, He C, et al. New insights into microbial interactions with dissolved organic matter in acid mine drainage with the integration of microbial community and chemical composition analysis. ACS ES&T Water, 2022, 2: 278-87. |
 2025, Vol. 37
2025, Vol. 37 

 洪亮,上海交通大学自然科学研究院/物理与天文学院/药学院/张江高等研究院特聘教授,上海交通大学张江高研院人工智能生物医药中心主任。以计算、人工智能和实验相结合的方式进行分子生物物理和蛋白质设计研究。2016年入选国家高层次人才青年专家,2021年入选教育部长江学者。在Nature、Proceedings of the National Academy of Sciences of the United States of America、Nature Physics等期刊上发表SCI论文70余篇。参与并主导开发了多个创新算法来提升功能蛋白和小分子药物的研发效率
洪亮,上海交通大学自然科学研究院/物理与天文学院/药学院/张江高等研究院特聘教授,上海交通大学张江高研院人工智能生物医药中心主任。以计算、人工智能和实验相结合的方式进行分子生物物理和蛋白质设计研究。2016年入选国家高层次人才青年专家,2021年入选教育部长江学者。在Nature、Proceedings of the National Academy of Sciences of the United States of America、Nature Physics等期刊上发表SCI论文70余篇。参与并主导开发了多个创新算法来提升功能蛋白和小分子药物的研发效率