(2 上海临床研究中心, 上海 201210)
(2 Shanghai Clinical Research and Trial Center, Shanghai 201210, China)
在现代医疗体系中,循证医学已成为指导临床决策的金标准,要求医疗实践建立在最佳可获得的科学证据基础上。然而,随着医学研究的爆发式增长,医疗专业人员正面临前所未有的信息过载挑战。全球医学文献数量巨大,临床医生难以跟上学科发展速度[1]。与此同时,传统文献检索方法存在诸多局限性,比如:知识更新存在延迟,大约平均需要17年才能将研究发现转化为临床实践;医学知识呈现高度碎片化状态,分散在各种专业期刊、临床指南和病历系统中;证据质量参差不齐,医生难以在有限时间内有效评估和整合[2]。
在复杂患者案例面前,尤其是多病共存、罕见病或个体化治疗决策场景下,传统计算机辅助决策系统往往基于简单规则或统计模型,缺乏对医学知识丰富语义关系的深度理解,无法满足临床决策的复杂性和动态需求[3, 4]。如何有效整合、表示和利用海量医学知识以支持循证医学决策,成为医疗信息学领域亟待解决的问题。
1.2 技术机遇知识图谱作为一种新兴的知识表示与推理技术,近年来在生物医学领域的应用呈现爆发式增长。生物医学知识图谱通过图结构将医学概念(如疾病、药物、基因等)作为节点,将它们之间的关系(如治疗、禁忌、因果等)作为边,构建成一个复杂的语义网络,为医学知识的系统化表示与推理提供了框架[5]。目前,已有多个大型生物医学知识资源被广泛应用,如统一医学语言系统(Unified Medical Language System, UMLS)整合了约200万个医学概念和500多万个医学词汇;语义医学数据库(Semantic MEDLINE Database, SemMedDB)从PubMed文献中提取了超过5 760万个谓词关系[6];SNOMED CT提供了超过35万个医学概念的标准化术语体系[7]。图 1展示了SNOMED CT术语模型的逻辑结构,该模型通过分层组织医学概念,支持多源异构医学信息的语义统一。该结构为临床术语的标准化、互操作提供了基础,有助于支撑后续的本体构建和知识图谱结构化设计[8]。
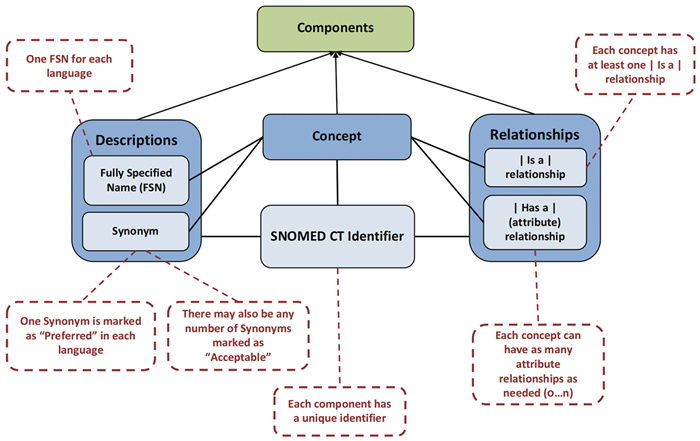
|
图 1 SNOMED CT的逻辑模型图解[8] |
表 1列举了SNOMED CT中定义的19个顶级概念,包括临床发现、操作、标本等。这些顶层语义类别为构建结构化医学知识图谱提供了重要的语义基础与术语支持。
| 表 1 SNOMED CT的19个顶级概念[8] |
COVID-19大流行期间,知识图谱技术展现出加速证据合成的独特优势。例如,研究人员基于知识图谱构建了COVID-19知识整合平台,在短时间内整合了数十万篇相关文献和临床数据,有效支持了药物重定位、疫苗研发和临床诊疗决策[9]。该案例充分展示了知识图谱在整合异构医学数据、加速证据合成和辅助临床决策方面的巨大潜力,为循证医学实践提供了新的技术途径。
1.3 研究进展与综述价值本综述旨在系统梳理知识图谱与循证医学的交叉研究领域。我们在PubMed、IEEE Xplore等数据库中检索了相关文献,并对其进行分析。根据“证据-知识-决策”转化框架,揭示了从原始医学文献到结构化知识,再到可操作临床决策的系统化路径[10]。
目前,相关的医学知识图谱构建与应用系统已被开发出来。例如,通过整合大量患者电子健康记录和临床指南,构建了包含数百万三元组的医学知识网络,并在多家三甲医院进行临床试点,在多病种共患管理和罕见病诊断方面取得了积极成果[1, 4]。本综述在系统总结领域技术发展的同时,也分析了现存挑战并提出可能的解决路径。
2 基础理论与技术路径 2.1 基础概念体系 2.1.1 循证医学决策的PICO框架循证医学(Evidence-based medicine, EBM)强调将最佳研究证据、临床专业知识和患者价值观有机整合,以指导临床决策[11]。PICO框架如图 2所示,是循证医学决策的黄金标准,由四个要素组成:患者群体(Population)、干预措施(Intervention)、对照措施(Comparison)和结局指标(Outcome)。PICO框架规范了临床问题的形成过程,为系统化文献检索和证据评价奠定了基础[11]。
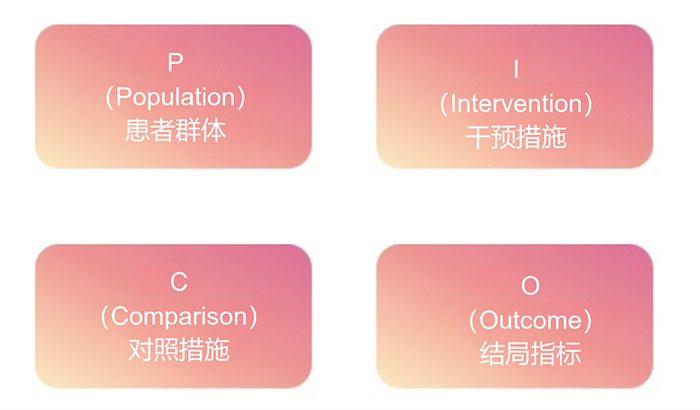
|
图 2 PICO框架 |
然而,PICO框架在应用层面也面临挑战,比如:医学文献中PICO元素往往隐含在文本中而非显式标注;临床决策中涉及的多维因素难以完全纳入框架;临床研究与实际患者情况存在差异,限制了证据的外部有效度[12]。而知识图谱技术可以通过对PICO元素的自动化识别、语义关联分析和情境感知推理,为循证医学决策提供更精准、全面的支持。
2.1.2 生物医学知识图谱的构成要素生物医学知识图谱是一种特殊的语义网络结构,通过节点和边表示医学领域的实体及其关系[13]。节点类型主要包括疾病节点、症状节点、药物节点、基因/蛋白质节点、实验室检查节点、解剖结构节点、证据节点和患者特征节点[4, 14]。
这些节点通过丰富的关系类型相互连接,主要包括治疗关系(如“药物A-治疗-疾病B”)、禁忌关系、因果关系、关联关系、分类关系、时序关系、解剖关系和证据支持关系[7]。这种节点-关系结构使生物医学知识图谱不仅能表示医学事实,还能捕捉复杂的医学知识语义和推理路径,为循证医学决策提供丰富的知识基础。图 3所示的BioCypher是一个面向生物医学的知识图谱构建框架,通过ETL (抽取(Extract)、转换(Transform)、加载(Load))过程接入异构数据源并利用本体进行概念映射,实现了灵活的数据融合和图谱生成。该框架支持从异构源数据中提取结构化信息,并基于本体进行实体标准化与语义映射,最终构建统一的图谱结构,用于支持下游的知识整合与推理任务[15]。
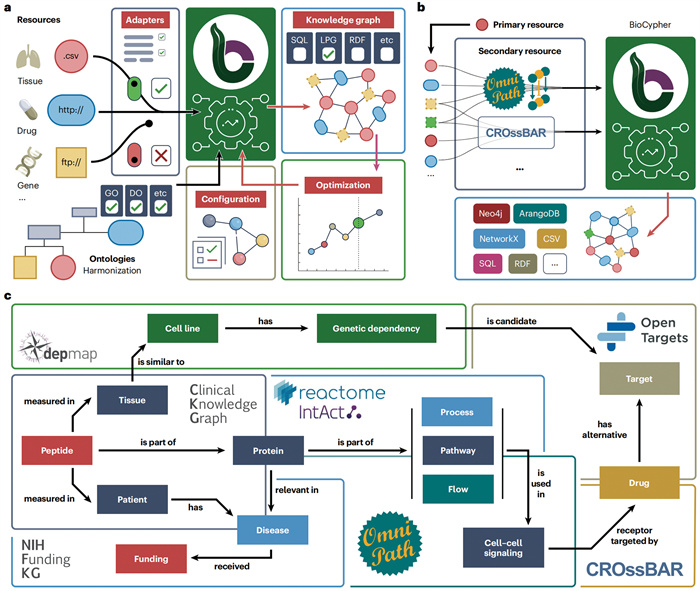
|
图 3 BioCypher框架[15] |
不同于传统关系数据库,知识图谱的灵活结构允许动态添加新的节点类型和关系类型,使其能够适应医学知识的不断演变和扩展。这种灵活性对于捕捉医学领域快速变化的新发现至关重要。
生物医学知识图谱的构建在实践中通常包括以下关键步骤。(1)数据源选择与整合:确定包括电子健康记录(Electronic health record, EHR)、公共数据库(如DrugBank、DisGeNET、PubMed)、临床指南和注册临床试验等多源数据;(2)数据清洗与标准化:对异构数据进行去噪和规范化处理,例如通过UMLS映射不同术语的同义关系,实现语义一致性;(3)实体识别与关系抽取:利用自然语言处理工具(如MetaMap、SciSpacy)从文本中识别疾病、药物、基因等实体及其语义关系;(4)本体对齐与三元组生成:将抽取结果与标准本体(如SNOMED CT、Gene Ontology)对齐,生成规范化三元组h, r, t (头实体Head entity),关系(Relation),目标实体(Target));(5)图数据库构建与存储:采用Neo4j、Amazon Neptune等图数据库存储知识三元组,实现高效图查询;(6)知识验证与更新:引入专家评审与自动化规则校验,对三元组置信度分级,并支持增量更新机制[6, 13, 14]。
2.2 关键技术路径 2.2.1 知识获取技术生物医学知识图谱的构建依赖于从多源异构数据中获取医学知识,主要包括结构化数据整合和非结构化文本挖掘两大技术路径[16]。结构化数据来源包括临床试验数据库(如ClinicalTrials.gov)、公共生物医学数据库(如DrugBank、DisGeNET等)和电子健康记录中的结构化数据[17]。
结构化数据整合面临的主要挑战是处理数据源之间的异质性。有研究者开发了基于本体映射的数据集成方法,如通过UMLS、SNOMED CT等标准本体作为中介,实现异构数据源之间的语义互通[7, 10]。
在非结构化文本挖掘方面,自然语言处理(Natural language processing, NLP)技术是从临床指南、医学文献和病历记录等文本数据中提取知识的核心工具。NLP技术链通常包括命名实体识别、关系抽取、共指消解和时序信息提取。
针对PICO框架的自动文本处理技术,能从医学文献中识别患者特征、干预措施、对比组和结果指标,支持循证医学决策自动化[11]。
通过基于多层次注意力机制的医学文本知识提取框架,引入医学本体知识,可以有效提高实体关系抽取的准确率。例如,Sousa等[18]提出的K-RET系统通过整合多源本体知识提高了关系抽取的准确性和覆盖面。
2.2.2 知识表示方法知识表示是将获取的医学知识转化为计算机可处理的形式,是生物医学知识图谱发挥决策支持作用的关键环节,而知识图谱作为知识表示的一种体现方式,将概念与关系以节点-边的形式进行直观体现,实现了异构医学知识的统一表示和语义互联。
与传统基于符号的规则或纯向量嵌入不同,知识图谱兼具可解释性和灵活性,可支持跨来源数据的语义整合和复杂关系推理,从而补充了其他表示方法的不足[14]。本体构建作为知识图谱的语义基础,通过形式化定义领域内的核心概念、关系和约束,为知识图谱提供统一的语义框架。这一方法在医疗、生物等领域已得到广泛应用,如SNOMED CT、Gene Ontology等,本体被广泛应用于知识图谱的基础构建中[7]。
图 4展示了SNOMED CT中一个医学概念的详细描述格式,包括该概念的唯一标识符、全称、术语类型以及与其他概念之间的关系信息。这种结构化的描述方式为构建具有语义清晰性的医学本体奠定了坚实基础。
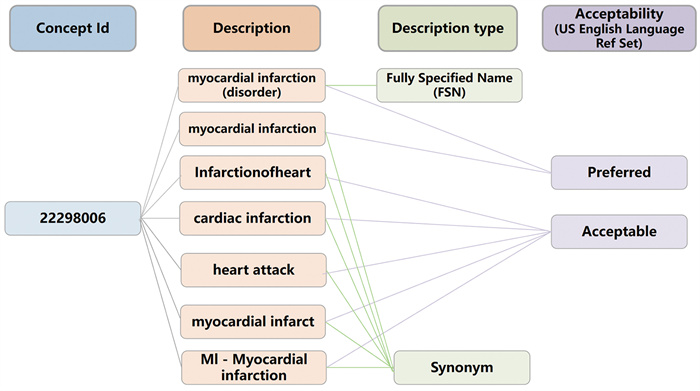
|
图 4 SNOMED CT单个概念的描述示例[19] |
在生物医学领域,KG-BERT (Knowledge Graph, KG)等模型将预训练语言模型与知识图谱相结合,实现了医学概念的上下文感知表示[20]。通过在临床文档和医学知识图谱上联合训练模型,能够在药物-疾病关系预测任务上取得显著提升,因此也可以看出通过引入知识图谱可以有效提升传统医学预测的准确率。
此外,多模态知识表示也日益受到重视,通过整合文本、图像和分子结构等多源信息,为我们全面理解复杂医学概念提供了更好的思路。这些多模态的知识表示方法也为生物医学知识图谱提供了强大的语义表达能力。
2.2.3 决策支持应用基于生物医学知识图谱的决策支持应用正在为循证医学变革提供实践。患者个性化治疗推荐系统是典型的应用,通过整合患者特征、疾病机制和治疗证据,能为医生提供定制化治疗方案。例如,IBM Watson for Oncology (https://www.cancertaipei.tw/watson癌症治疗辅助系统/?lang=zh-hans)基于超过1 500万页医学资料构建的知识图谱,为癌症患者提供个性化治疗建议,考虑患者的基因突变、共病情况和用药史等因素。
图 5为“证据-知识-决策”转化框架,从原始医学文献出发,经过信息抽取、知识表示、图谱构建与推理等步骤,最终支持循证医学中的临床决策任务。该流程体现了知识图谱在医学决策支持中的端到端作用路径。
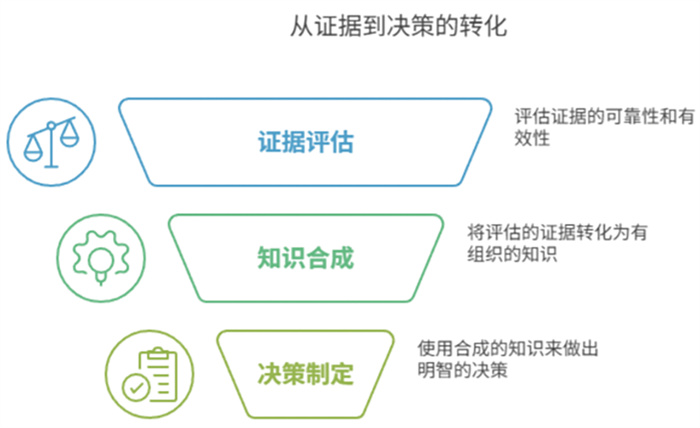
|
图 5 “证据-知识-决策”转化框架图 |
临床路径优化是另一重要应用,通过分析大量患者治疗轨迹与预后关系,发现最佳诊疗路径并提供实时指导。可以基于知识图谱进行临床路径系统的开发,通过整合患者诊疗数据和医学指南,优化患者的诊疗路径并提高治疗依从性。
将知识图谱与大型语言模型(LLMs)相结合,可提高决策支持系统的准确性[21]。例如,2024年的研究提出了知识超图增强的LLM生成模型,特别针对循证医学场景,通过重要性驱动的证据优先级算法支持复杂临床查询[22]。
3 关键应用场景分析 3.1 药物重定位与精准治疗药物重定位是知识图谱在循证医学中最具影响力的应用场景之一,具有显著缩短药物开发周期和降低成本的潜力[23]。Rephetio项目构建了包含4.7万个节点的11类关系,以及225万条关系的生物医学知识图谱Hetionet,整合了来自29个公共数据源的信息,该项目采用基于元路径的方法预测药物-疾病关系,并通过逻辑回归将多种路径证据整合为统一的重定位分数,预测了20多万种化合物-疾病配对的治疗可能性[24]。
Rephetio成功预测了多个高置信度的药物重定位机会,如预测抗癫痫药物托吡酯对炎症性肠病的治疗效果,随后在动物模型中得到证实。在COVID-19大流行期间,多个研究团队基于知识图谱快速识别了潜在的抗SARS-CoV-2药物,如巴瑞替尼,该药随后在临床试验中显示出积极结果并获得紧急使用授权[8, 25]。
因此,我们发现通过整合基因表达谱、蛋白质相互作用网络和临床试验数据得到的层次化知识图谱框架,可以用于自身免疫性疾病的药物重定位。该框架在结节病、儿童肾脏癌症等疾病的回溯性验证中,成功将大部分已知有效药物排在候选列表的前列,显著优于传统的单一数据源方法。该框架的一个创新点在于引入了“治疗证据强度”层次结构,将不同类型的证据(从分子机制到临床验证)整合到统一的评分体系中[26]。
3.2 罕见病诊断辅助罕见病诊断是医学领域的重大挑战,罕见病影响全球约6%~10%的人口,并且患者普遍面临诊断延迟(25%需5~30年确诊)、误诊(40%)、不当治疗及信息沟通不畅问题[27]。而生物医学知识图谱通过整合分散的罕见病知识和复杂表型-基因型关联,正在革新罕见病的诊断流程。
FindZebra系统是知识图谱辅助罕见病诊断的典型案例,基于包含罕见病和相关表型的知识图谱,通过深度学习模型将患者症状映射到标准化人类表型本体(Human Phenotype Ontology, HPO)术语,然后计算与知识库中疾病表型的相似度生成诊断建议。在56个测试案例中,FindZebra将正确诊断排在前10位的准确率达到62.5%,显著高于通用搜索引擎[28]。图 6为FindZebra官网的界面示意图(https://www.findzebra.com/search?q=sinus),该系统基于知识图谱和人类表型本体(HPO)构建,通过输入患者症状自动匹配可能的罕见疾病,为临床提供辅助诊断建议,体现了知识图谱在实际诊断工具中的应用价值。
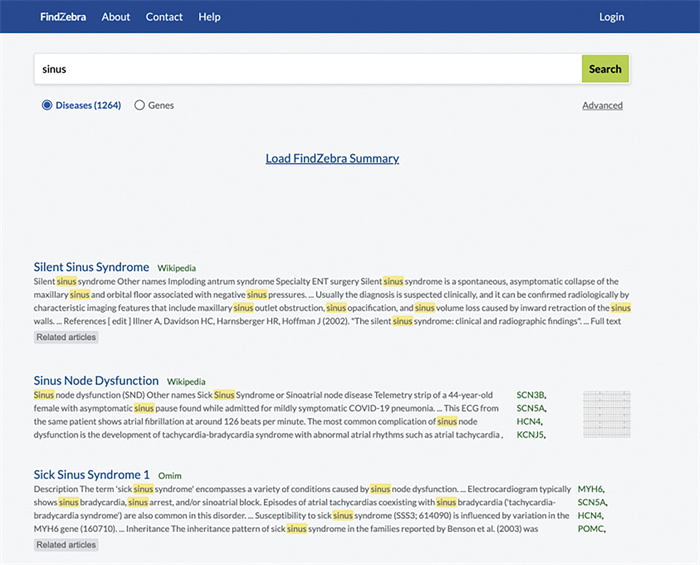
|
图 6 FindZebra系统 |
除此之外,RareNet系统整合了患者表型特征、基因变异和代谢通路信息,构建了多层次的患者-疾病相似性计算框架。该系统利用图注意力网络学习不同表型特征的重要性权重,并结合基因-表型关联网络进行推理,在复杂罕见病测试集上,将正确诊断排在前列的准确率较传统表型匹配方法有显著提高,并在临床试点中辅助诊断了多例困难病例。
图 7为RareNet官网的示意图(http://www.rarenet.eu/symposium-2016),该系统整合了表型、基因变异和通路信息,结合图神经网络与注意力机制,实现对复杂罕见病的辅助诊断推理,进一步证明了知识图谱与深度学习技术融合的前景。
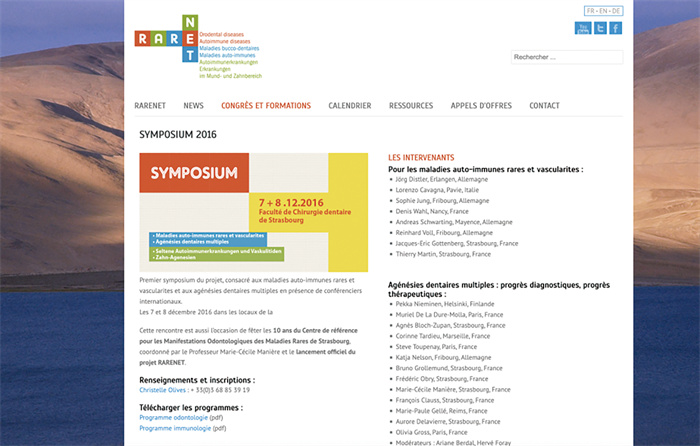
|
图 7 RareNet系统 |
多病种共患(multimorbidity)已成为现代医疗体系的重大挑战,超过60%的老年患者同时患有多种慢性疾病[4]。传统循证医学框架面临严重局限,因为临床指南通常遵循单病种方法,缺乏对疾病间复杂相互作用的考量[11, 29]。
知识图谱通过其独特的网络结构,为多病种共患管理提供了创新性解决方案。通过构建疾病关联网络,将疾病作为节点,通过多种关系类型(如共同分子机制、药物相互作用、时序模式)连接相关疾病[1]。另外我们发现,用药管理是多病种共患的核心挑战,当患者服用5种以上药物,就会导致药物相互作用风险显著增加[17]。
基于知识图谱的决策支持系统,可以通过整合患者的电子健康记录和临床指南,构建三元组的医学知识网络,为患者提供个性化用药建议和风险预警,基于知识图谱推理和患者相似度计算推荐治疗方案。通过知识图谱技术的加持,能够让临床辅助决策系统大大减少多病种患者的药物相互作用不良事件,并提高医嘱符合率,改善患者的临床结局。
4 现存挑战与解决策略 4.1 证据质量评估难题证据质量评估是循证医学决策的核心环节,在知识图谱语境下面临独特挑战[11]。知识图谱中的关系往往源自多种异质数据源,具有不同的可靠性水平和知识粒度[6]。
我们发现,虽然循证医学领域已建立成熟的证据分级系统(如Grading of Recommendations Assessment, Development and Evaluation, GRADE),但这些框架难以直接应用于图结构中的三元组关系[11],并且知识图谱中的关系也缺乏标准化的质量标识。比如,同样表示为“药物A-治疗-疾病B”的关系,可能源自随机对照试验的高强度证据,也可能来自病例报告的低级别证据。
因此,可以根据证据来源类型、置信度评分和跨数据源一致性,为三元组关系分配动态质量评分,建立起一种基于证据级联的知识图谱质量评估框架。这种框架在对大规模医学三元组的评估中,能够自动识别出一定比例的矛盾关系和低可信度关系,为知识图谱的质量控制提供了有效工具。
未来研究方向可以包括开发针对知识图谱的标准化证据评级体系,构建大规模专家标注的证据质量数据集,以及探索将形式化论证理论与知识图谱技术相结合,更好地表示和推理复杂的证据关系。
4.2 动态更新难题生物医学知识以前所未有的速度扩展,因此需要持续更新以反映最新的研究发现[1]。传统知识库构建采用周期性更新模式,难以满足实时决策支持的需求[30]。现有生物医学知识图谱大多采用静态构建方法,存在知识及时性不足、更新过程资源密集、知识演变追踪困难等多重局限。
为解决这些挑战,可以通过增量更新的方法来解决这一难题,该方法仅处理新增或变更的信息,避免重建整个知识图谱[31]。这种基于主动学习的知识图谱增量更新框架可以通过评估文献的信息价值和新颖性得分,优先处理可能带来显著知识更新的内容。该框架在实验中可以仅需处理较少比例的新增医学文献即可捕获大部分重要知识更新,显著提高了计算效率和知识时效性。
4.3 临床可解释性瓶颈临床可解释性是生物医学知识图谱应用于循证医学决策支持的关键挑战[32]。虽然知识图谱本质上具有较好的可解释性基础,但在实际临床应用中仍面临多重瓶颈。首先,推理复杂度与可解释性存在根本矛盾。现代生物医学知识图谱通常包含数千万甚至数亿三元组关系,基于此进行的推理可能涉及数十到数百步骤的路径分析或复杂的概率计算[14]。因此,医学专业人员在有限时间内难以完全理解过于复杂的推理链。其次,知识图谱嵌入模型的“黑盒”特性加剧了可解释性挑战。这类模型将实体和关系映射到低维向量空间,通过向量运算实现高效推理,但向量空间中的操作难以映射回人类可理解的医学概念[20]。
4.4 医疗数据共享障碍国内的医疗健康数据的共享还面临多重障碍,研究者在共享数据时往往要克服繁琐的行政审批和数据库访问权限限制,而且对于共享后可能导致的隐私泄露十分担忧。这些担忧因缺乏明确的法律和伦理规定来规范医疗数据的研究使用而更加严重。另外,2021‒2022年中国医院信息化调查指出,三级医院在医疗数据共享方面面临一个关键挑战,即数据科学家的人才较为匮乏,这也导致了大量的数据难以被很好地利用,大约在600多家医院中,有75.15%的医院认为数据科学家的短缺是主要障碍[33]。
5 跨学科融合与发展前景 5.1 因果推理与联邦学习的整合生物医学知识图谱与循证医学决策支持的发展正展现出跨学科融合的趋势,特别是在因果推理与知识图谱的结合以及联邦学习解决数据隐私问题两个关键方向[34]。因果推理长期作为循证医学的基石,强调识别干预措施与预期结果之间的因果关系,而非简单相关性[11]。传统知识图谱虽能表示实体间关系,但难以区分相关与因果,限制了其在临床决策中的应用深度。将因果推理框架与知识图谱有机结合可形成因果知识图谱(Causal Knowledge Graph, CKG)[35]。在CKG中,边不再是简单的关联,而是代表有向干预效应,支持反事实推理(如果使用药物A而非药物B,患者结局会如何变化?)[36]。在技术实现上,结构因果模型与图神经网络的融合创造了新型推理架构,能同时利用领域知识与观测数据[35]。
联邦学习也正逐渐成为解决医疗数据隐私与安全挑战的关键技术。医疗数据的隐私敏感性和监管限制使传统的数据集中方法难以实施。联邦学习通过“模型到数据”而非“数据到模型”的范式转变,允许算法在不同机构的本地数据上训练,只共享模型参数而非原始数据[37, 38]。
5.2 评价指标体系与临床整合评价生物医学知识图谱辅助循证医学决策的效果,需要平衡技术指标与临床实用性,构建多维度的评价指标体系。传统知识图谱评价主要关注技术精确度,如通过链接预测任务评估图嵌入质量,这些指标在临床环境中意义有限。在循证医学决策支持背景下,评价指标体系应兼顾技术性能(知识覆盖率、知识准确性等)、临床相关性(决策相关性、证据质量等)和实施影响(用户满意度、工作流集成度等)三个维度[39]。相关的多中心临床评估研究提出了一种综合评价框架,通过平衡技术性能指标与临床影响指标来评估知识图谱决策支持系统的实际价值。在临床试点中,单纯依靠技术准确率指标可能导致临床价值评估的偏差。与临床工作流的无缝整合、对医生决策习惯的尊重以及系统响应时间,往往对系统实际采纳更为重要。
5.3 未来发展趋势生物医学知识图谱辅助循证医学决策的未来发展将主要集中在以下几个方向。首先,实时证据整合系统将彻底革新医学知识更新模式,通过自动化文献理解、证据质量评估和知识图谱动态更新。这类系统正从周期性批量更新向连续学习模式转变,能够自主监控医学文献发布平台,即时提取关键发现并评估其对现有知识体系的影响。其次,人机协同决策界面将发展为交互式决策支持环境,通过自适应解释机制、多模态交互和情境感知推荐,创造医生与AI系统的最佳协作模式[32]。这种设计理念将AI视为“智能助手”而非“自动决策者”,保留医生的专业自主权,同时提供认知增强支持。第三,知识图谱与大型语言模型的深度融合将创造新一代循证医学决策支持系统[21, 22]。通过将结构化知识与自然语言理解能力相结合,这些系统能够更自然地与医生交互,理解复杂的临床查询,并提供可追溯、可验证的循证支持。最后,基于知识图谱的“活指南”(living guidelines)概念将推动临床指南的动态更新与个体化应用[40]。研究人员正在探索构建包含多层次证据、动态更新机制和个性化调整能力的知识框架,实现临床指南的持续优化。目前,这种动态知识驱动的指南系统能够提高医疗决策与科学证据的一致性,特别是在快速发展的领域和多疾病共存的复杂患者中表现较好。
未来生物医学领域的研究趋势之一是如何构建更大规模并开放共享的知识图谱资源。目前已有框架(如KG-Hub、ClinicalKG、BioCypher、KaBOB等)实现了跨数据库的知识图谱构建与交换[41]。例如,Nature Biotechnology报道的临床知识图谱平台已经集成了实验数据、公共数据库和文献中的信息,节点近2 000万,关系超过2.2亿条,可用于增强组学数据分析并辅助临床决策[42]。这些进展表明,未来通过开放共享知识图谱,将为循证医学提供更丰富的结构化知识和决策支持。
6 总结生物医学知识图谱正在推动循证医学决策从“基于文献”向“基于知识”的范式转型,这一转变具有深远意义[4, 11]。知识图谱通过将分散的医学证据转化为结构化、可计算的知识网络,实现了医学知识的系统化表示与智能化应用。在这一新范式下,证据不再是静态的文献集合,而是动态演化的知识网络,能够自动整合最新研究发现,捕捉概念间的复杂关系,并支持多层次的临床推理[14]。
尽管生物医学知识图谱在辅助循证医学决策方面展现出巨大潜力,但仍面临多重挑战。证据质量评估需要开发针对知识图谱的标准化评级体系;动态更新机制需要更高效的增量学习方法;临床可解释性需要更贴近医生认知模式的解释框架。已有研究工作针对这些挑战提出了一系列解决方案,例如证据级联的质量评估框架、基于主动学习的知识增量更新方法和多级临床可解释性框架。这些方法在临床实践中被证明有助于提升知识图谱辅助决策系统的实用性和可接受度[29, 31, 32, 39]。
生物医学知识图谱是循证医学发展的新方向,通过建立从数据到知识,再到智能决策的完整链条,为提高医疗质量、优化资源配置和改善患者预后提供了强大工具[14, 32]。未来的发展将依赖于技术创新、临床验证和跨学科合作的协同推进,共同构建更智能、更精准、更人性化的医疗决策支持体系。
| [1] |
Rotmensch M, Halpern Y, Tlimat A, et al. Learning a health knowledge graph from electronic medical records. Sci Rep, 2017, 7: 5994. DOI:10.1038/s41598-017-05778-z |
| [2] |
Morris ZS, Wooding S, Grant J. The answer is 17 years, what is the question: understanding time lags in translational research. J R Soc Med, 2011, 104: 510-20. DOI:10.1258/jrsm.2011.110180 |
| [3] |
Callahan TJ, Tripodi IJ, Pielke-Lombardo H, et al. Knowledge-based biomedical data science. Ann Rev Biomed Data Sci, 2020, 3: 23-41. DOI:10.1146/annurev-biodatasci-010820-091627 |
| [4] |
Barnett K, Mercer S W, Norbury M, et al. Epidemiology of multimorbidity and implications for health care, research, and medical education: a cross-sectional study. Lancet, 2012, 380: 37-43. DOI:10.1016/S0140-6736(12)60240-2 |
| [5] |
Qi Z, Wang H, Zhang H. A dual-store structure for knowledge graphs. IEEE Trans Knowl Data Eng, 2021, 35: 1104-18. |
| [6] |
Kilicoglu H, Shin D, Fiszman M, et al. SemMedDB: a PubMed-scale repository of biomedical semantic predications. Bioinformatics, 2012, 28: 3158-60. DOI:10.1093/bioinformatics/bts591 |
| [7] |
Donnelly K. SNOMED-CT: the advanced terminology and coding system for eHealth. Stud Health Technol Inform, 2006, 121: 279. |
| [8] |
Bhattacharyya SB. Introduction to SNOMED CT: Singapore: Springer, 2015.
|
| [9] |
Domingo-Fernández D, Baksi S, Schultz B, et al. COVID-19 Knowledge Graph: a computable, multi-modal, cause-and-effect knowledge model of COVID-19 pathophysiology. Bioinformatics, 2021, 37: 1332-4. DOI:10.1093/bioinformatics/btaa834 |
| [10] |
Bodenreider O. The unified medical language system (UMLS): integrating biomedical terminology. Nucleic Acids Res, 2004, 32: D267-70. DOI:10.1093/nar/gkh061 |
| [11] |
Sackett DL, Rosenberg WMC, Gray JAM, et al. Evidence based medicine: what it is and what it isn't. BMJ, 1996, 312: 71-2. |
| [12] |
Jin D, Szolovits P. PICO element detection in medical text via long short-term memory neural networks[C]//. Melbourne, Australia: Proceedings of the BioNLP 2018 Workshop, 2018: 67-75
|
| [13] |
Nigel Shadbolt N, Berners-Lee T, Hall W. The semantic web revisited. IEEE Intell Syst, 2006, 21: 96-101. |
| [14] |
Nicholson DN, Greene CS. Constructing knowledge graphs and their biomedical applications. Comput Struct Biotechnol J, 2020, 18: 1414-28. DOI:10.1016/j.csbj.2020.05.017 |
| [15] |
Lobentanzer S, Aloy P, Baumbach J, et al. Democratizing knowledge representation with BioCypher. Nat Biotechnol, 2023, 41: 1056-9. DOI:10.1038/s41587-023-01848-y |
| [16] |
Lee D, Shin H. Disease causality extraction based on lexical semantics and document-clause frequency from biomedical literature. BMC Med Inform Decis Mak, 2017, 17: 53. DOI:10.1186/s12911-017-0448-y |
| [17] |
Shen Y, Yuan K, Dai J, et al. KGDDS: a system for drug-drug similarity measure in therapeutic substitution based on knowledge graph curation. J Med Syst, 2019, 43: 92. DOI:10.1007/s10916-019-1182-z |
| [18] |
Sousa F, Couto FM. K-RET: knowledgeable biomedical relation extraction system. Bioinformatics, 2023, 39: btad174. DOI:10.1093/bioinformatics/btad174 |
| [19] |
OMAHA联盟. 统一的力量: 临床医学术语标准化的展望[EB/OL]. (2016-11-28)[2025-05-15]. https:/www.imit.org.cn/data/upload/portal/20200811/imit白皮书第二期:统一的力量:临床医学术语标准化的展望.pdf
|
| [20] |
Jia N, Yao C. ShallowBKGC: a BERT-enhanced shallow neural network model for knowledge graph completion. PeerJ Comput Sci, 2024, 10: e2058. DOI:10.7717/peerj-cs.2058 |
| [21] |
Liu H, Wang S, Zhu Y, et al. Knowledge graph-enhanced large language models via path selection. arXiv, 2024, https://doi.org/10.48550/arXiv.2406.13862
|
| [22] |
Pan S, Luo L, Wang Y, et al. Unifying large language models and knowledge graphs: a roadmap. IEEE Trans Knowl Data Eng, 2024, 36: 3580-99. DOI:10.1109/TKDE.2024.3352100 |
| [23] |
Pushpakom S, Iorio F, Eyers PA, et al. Drug repurposing: progress, challenges and recommendations. Nat Rev Drug Discov, 2019, 18: 41-58. DOI:10.1038/nrd.2018.168 |
| [24] |
Himmelstein DS, Lizee A, Hessler C, et al. Systematic integration of biomedical knowledge prioritizes drugs for repurposing. elife, 2017, 6: e26726. DOI:10.7554/eLife.26726 |
| [25] |
Richardson P, Griffin I, Tucker C, et al. Baricitinib as potential treatment for 2019-nCoV acute respiratory disease. Lancet, 2020, 395: e30-1. DOI:10.1016/S0140-6736(20)30304-4 |
| [26] |
Sosa DN, Derry A, Guo M, et al. A literature-based knowledge graph embedding method for identifying drug repurposing opportunities in rare diseases. Pac Symp Biocomp, 2020, 25: 463-74. |
| [27] |
Knight AW, Senior TP. The common problem of rare disease in general practice. Med J Aust, 2006, 185: 82-3. DOI:10.5694/j.1326-5377.2006.tb00477.x |
| [28] |
Dragusin R, Petcu P, Lioma C, et al. FindZebra: a search engine for rare diseases. Int J Med Inform, 2013, 82: 528-38. DOI:10.1016/j.ijmedinf.2013.01.005 |
| [29] |
Hughes LD, McMurdo MET, Guthrie B. Guidelines for people not for diseases: the challenges of applying UK clinical guidelines to people with multimorbidity. Age Ageing, 2013, 42: 62-9. DOI:10.1093/ageing/afs100 |
| [30] |
Gao M, Lu J, Chen F. Medical knowledge graph completion based on word embeddings. Information, 2022, 13: 205. DOI:10.3390/info13040205 |
| [31] |
Pareja A, Domeniconi G, Chen J, et al. Evolvegcn: evolving graph convolutional networks for dynamic graphs. Proc AAAI Confer Artif Intell, 2020, 34: 5363-70. |
| [32] |
Holzinger A, Langs G, Denk H, et al. Causability and explainability of artificial intelligence in medicine. Wiley Interdiscip Rev Data Min Knowl Discov, 2019, 9: e1312. DOI:10.1002/widm.1312 |
| [33] |
Li X, Cong Y. Exploring barriers and ethical challenges to medical data sharing: perspectives from Chinese researchers. BMC Med Ethics, 2024, 25: 132. DOI:10.1186/s12910-024-01135-8 |
| [34] |
Wang X, Li Q, Yu D, et al. Causal disentanglement for semantic-aware intent learning in recommendation. IEEE Trans Knowl Data Eng, 2022, 35: 9836-49. |
| [35] |
Huang H. Causal relationship over knowledge graphs[C]//Atlanta, GA: Proceedings of the 31st Acm International Conference On Information & Knowledge Management, 2022: 5116-9
|
| [36] |
Peng C, Xia F, Naseriparsa M, et al. Knowledge graphs: opportunities and challenges. Artif Intell Rev, 2023, 56: 13071-102. DOI:10.1007/s10462-023-10465-9 |
| [37] |
Rieke N, Hancox J, Li W, et al. The future of digital health with federated learning. NPJ Digit Med, 2020, 3: 119. DOI:10.1038/s41746-020-00323-1 |
| [38] |
Li Q, Wen Z, Wu Z, et al. A survey on federated learning systems: vision, hype and reality for data privacy and protection. IEEE Trans Knowl Data Eng, 2021, 35: 3347-66. |
| [39] |
Amann J, Blasimme A, Vayena E, et al. Explainability for artificial intelligence in healthcare: a multidisciplinary perspective. BMC Med Inform Decis Mak, 2020, 20: 310. DOI:10.1186/s12911-020-01332-6 |
| [40] |
Marshall IJ, Nye B, Kuiper J, et al. Trialstreamer: a living, automatically updated database of clinical trial reports. J Am Med Inform Assoc, 2020, 27: 1903-12. DOI:10.1093/jamia/ocaa163 |
| [41] |
Callahan TJ, Tripodi IJ, Stefanski AL, et al. An open source knowledge graph ecosystem for the life sciences. Sci Data, 2024, 11: 363. DOI:10.1038/s41597-024-03171-w |
| [42] |
Santos A, Colaço AR, Nielsen AB, et al. A knowledge graph to interpret clinical proteomics data. Nat Biotechnol, 2022, 40: 692-702. DOI:10.1038/s41587-021-01145-6 |
 2025, Vol. 37
2025, Vol. 37 

 范海巍,正高级工程师,上海临床研究中心信息化总监。自2000年起,先后工作于中国科学院上海生命科学信息中心、上海市科委研发公共服务平台管理中心、国家蛋白质设施、中国科学院条财局重大设施处。上海市计算机协会高性能计算专委会委员、中国研究型医院学会临床数据与样本资源库专业委员会委员、上海市智能建筑建设协会青年专家委员会委员、《健康医疗数据要素安全共享技术要求》起草者、2023年浦东新区明珠工程师。长期从事科研领域信息化工作,充分掌握该领域的前沿技术及国内外发展态势,兼具科研项目管理及工程组织实践能力。对生命科学、医疗健康、药物设计与筛选等高性能计算领域具有较好的熟知深度;对数据中心、智慧园区、云计算、科研大数据、信息化基础设施平台、信息化运营服务等领域具有较为丰富的规划实施与运行管理经验。牵头负责项目有上海科技大学智能化信息系统基础设施(上海市发展与改革委)、国家蛋白质设施数据与计算分析系统(国家发展与改革委)、中国科学院重大科技基础设施共享服务平台(中国科学院级)、硬X射线自由电子激光装置项目束线站高性能数据管理系统(国家发展与改革委、上海市发展与改革委)、上海市临床研究中心智慧信息系统建设(上海市发展与改革委),及主要负责SNP单核苷酸多态性检索数据库系统、HLPP人类肝脏蛋白质组学数据库、ArrayLIMS生物芯片在线数据存储及分析系统、EPGD真核旁系同源数据库系统、基于影像电子病历的远程诊断系统、长三角大型仪器数据汇交系统、长三角大型仪器技术转移系统、长三角文献全文传递系统、长三角资源条件保障系统、专业行业情报及美国政府公报检索系统、上海市科技基础条件资源数据中心、万户工程用户库系统、研发平台一站式跨库检索、大型仪器网二期改造实验基地评估系统、河北省科学仪器共享服务联盟平台等多个专业科研领域与跨域联动项目。2018年曾协调协助上海科技大学超算队伍,获两次世界超算大赛第二、第四名
范海巍,正高级工程师,上海临床研究中心信息化总监。自2000年起,先后工作于中国科学院上海生命科学信息中心、上海市科委研发公共服务平台管理中心、国家蛋白质设施、中国科学院条财局重大设施处。上海市计算机协会高性能计算专委会委员、中国研究型医院学会临床数据与样本资源库专业委员会委员、上海市智能建筑建设协会青年专家委员会委员、《健康医疗数据要素安全共享技术要求》起草者、2023年浦东新区明珠工程师。长期从事科研领域信息化工作,充分掌握该领域的前沿技术及国内外发展态势,兼具科研项目管理及工程组织实践能力。对生命科学、医疗健康、药物设计与筛选等高性能计算领域具有较好的熟知深度;对数据中心、智慧园区、云计算、科研大数据、信息化基础设施平台、信息化运营服务等领域具有较为丰富的规划实施与运行管理经验。牵头负责项目有上海科技大学智能化信息系统基础设施(上海市发展与改革委)、国家蛋白质设施数据与计算分析系统(国家发展与改革委)、中国科学院重大科技基础设施共享服务平台(中国科学院级)、硬X射线自由电子激光装置项目束线站高性能数据管理系统(国家发展与改革委、上海市发展与改革委)、上海市临床研究中心智慧信息系统建设(上海市发展与改革委),及主要负责SNP单核苷酸多态性检索数据库系统、HLPP人类肝脏蛋白质组学数据库、ArrayLIMS生物芯片在线数据存储及分析系统、EPGD真核旁系同源数据库系统、基于影像电子病历的远程诊断系统、长三角大型仪器数据汇交系统、长三角大型仪器技术转移系统、长三角文献全文传递系统、长三角资源条件保障系统、专业行业情报及美国政府公报检索系统、上海市科技基础条件资源数据中心、万户工程用户库系统、研发平台一站式跨库检索、大型仪器网二期改造实验基地评估系统、河北省科学仪器共享服务联盟平台等多个专业科研领域与跨域联动项目。2018年曾协调协助上海科技大学超算队伍,获两次世界超算大赛第二、第四名