(2 中国科学院上海营养与健康研究所, 上海 200032)
(2 Shanghai Institute of Nutrition and Health, Chinese Academy of Sciences, Shanghai 200032, China)
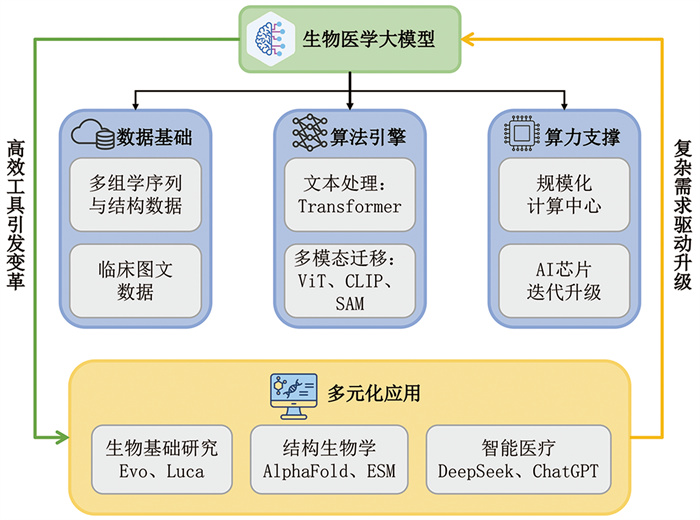
生物医学大模型作为面向生物医学领域设计、基于超大规模预训练策略与多模态学习技术构建的人工智能模型,是人工智能与生命科学交叉融合的重要成果。一方面,生命科学领域长期积累的多模态生物医学数据,与信息时代所积累的海量通用文本数据,成为驱动人工智能模型系统演进的关键生产要素。另一方面,智能时代的算法迭代与算力基础设施的提升,促进了生物医学数据的深度挖掘与知识发现。这一协同演进体系以数据为基础、以算法为引擎、以算力为支撑,并通过多元化应用场景,实现了双向赋能:大模型技术为生物医学领域基础研究与应用提供了高效工具,而复杂多样的生物医学问题又反向驱动大模型架构的迭代升级。大模型技术与生物医学领域的深度融合,正以前所未有的速度推动两种学科的范式转型与跨越式发展(图 1)。
|
图 1 生物医学大模型协同演进体系 |
测序技术的发展产生了海量多组学数据,与全球医学信息化时代积累的临床数据共同组成了人工智能赋能“大健康”产业发展的数据生产要素[1],是生物医学大模型训练的基础。2003年,人类基因组计划(HGP)的完成[2]不仅标志着基因组时代的到来,也是基因组数据标准化采集以及公共数据库系统性建设的开始。2010年之后,以Illumina、华大BGI平台为代表的新一代测序技术(NGS)的发展将单碱基测序成本降至HGP时期的十万分之一[3],使得各类公共数据库规模进入指数级增长阶段。截至2025年5月,美国国立生物技术信息中心参考序列库(NCBI Reference Sequence Database, RefSeq)[4]收录了超过16万个物种的4亿多个蛋白质与超过7 000万条转录本的序列信息,基因组分类数据库(Genome Taxonomy Database, GTDB)[5]收集了超过71万细菌、1.7万古细菌的基因组信息,蛋白质数据银行(Protein Data Bank, PDB)[6]已发布超过23.8万个结构数据,并仍以每年7%左右的增长率持续扩增规模。同时,临床信息化平台以及医学相关研究已积累亿级规模的临床数据,涵盖医学影像、电子病历、电子健康记录、生物医学文献等非结构化图像及文本信息,为多模态生物医学大模型的训练提供训练数据。如常用于医疗大模型训练与医学能力评估的MIMIC-Ⅲ数据集[7]收录了超过6万个患者的临床医疗记录,成立于2001年的PubMed Central[8]收录了超过1 100万份生物医学领域文献,为生物医学大模型的训练提供了领域专用语料资源。多组学数据反映了生物系统不同层面的遗传与表型信息,与临床数据组成了跨尺度、多模态的复杂生物医学数据体系,为揭示疾病发生机制、药物发现以及精准医疗提供了丰富的素材,使得当前时代比历史上任何时期都更接近全面解析生物系统功能与疾病发生发展机制的目标。
1.2 算法迭代在数据大幅增长的背景下,深度学习算法的持续演进为跨尺度多模态生物医学数据分析及生物医学大模型构建提供了强力引擎。深度学习算法在过去近十年间飞速发展,其中2017年推出的Transformer架构[9]打破了传统循环神经网络(RNN)和卷积神经网络(CNN)在长序列数据处理方面的局限性,通过注意力机制(self-attention)理解词元(Token)之间的关系,在长序列建模、多头注意力机制与上下文捕捉能力等方面具备显著优势,成为深度学习技术发展的重要里程碑。2018年,OpenAI发布了第一代生成式预训练Transformer (GPT-1)[10];同年,Google提出了基于Transformer的开源预训练模型BERT[11],开启了AI开源开放的新阶段。此后,Hugging Face开源Transformers库整合了GPT、BERT等多种预训练模型,极大地降低了自然语言处理的研究门槛。基于GPT与BERT的大规模预训练语言模型,在自然语言理解与生成任务中取得了显著成果,并因其能高效处理大规模文本序列并表征复杂上下文依赖关系,而逐步扩展至生物信息学领域,诞生了DNABERT[12]等模型。2020—2023年间,Vision Transformer (ViT)[13]、对比语言-图像预训练(Contrastive Language-Image Pre-training, CLIP)[14]、Segment Anything Model (SAM)[15]等标志性工作相继出现,为生物医学多模态大模型的构建提供了可迁移、可扩展的技术支持。
1.3 算力提升智能时代,算力成为驱动生物医学大模型持续演进的核心支撑。特别是在模型参数规模、精度和多模态能力不断扩展的背景下,高效、可扩展的计算系统对模型训练和推理的支持作用愈发关键。算力提升主要体现在两方面:大规模计算中心的集群化建设与AI专用计算芯片的持续迭代升级。首先,超大规模计算中心的建设使得训练千亿级别参数模型成为可能。从GPT-1到GPT-3,模型参数量从1.17亿增加到1 750亿,后者单次训练所需算力高达3 640 PFLOP/s-day[16, 17]。以谷歌为例,为支撑千亿甚至万亿级别参数模型的训练,构建了由4 096个TPU v4芯片组成的超级计算平台,单芯片性能较v3提升约2.1倍,整体规模扩大4倍,使得平台整体算力提高了近10倍,BF16算力峰值达到1.126 ExaFLOPs,支撑了Med-PaLM等千亿级别参数模型的训练[18]。其次,AI专用芯片的迭代为模型训练提供更强的单卡算力支持。以英伟达为例,其2022年发布的H100 Tensor Core GPU基于Hopper架构,在FP8精度的峰值算力较上一代的A100提升约6倍[19],使得ESM-2 (650M)[20]模型在相同数量的H100上训练速度较A100提升1倍。
1.4 多元化应用生物医学大模型的多元化应用正在引发传统研究范式的改变,主要包括三类场景应用:其一,驱动基础生命科学研究,如Evo系列模型能理解并按需设计基因组序列[21, 22],Luca系列模型[23, 24]能综合学习遗传和蛋白质组语言;其二,革新蛋白质结构解析与设计方法,如AlphaFold系列模型改变了蛋白质结构预测主要依赖物理模型的现状[25-28],ESM[20, 29, 30]系列模型能够根据提示信息设计蛋白质;其三,临床诊疗智能化革新,DeepSeek[31]、ChatGPT[16, 32]、通义千问[33]等为代表的大语言模型能整合电子健康记录、医学影像等异构信息,辅助制定个性化治疗方案,促进智能诊疗模式升级。
2 创新应用引发范式变革生物医学大模型结合了海量多模态数据、创新算法架构和超大规模算力,最终通过多元化的场景应用正在引发一场前所未有的范式变革,推动生物医学研究从传统实验驱动向“AI+”智能驱动模式转变。以Evo、AlphaFold、DeepSeek等为代表的大模型,已在序列分析、结构预测、临床辅助决策等领域取得突破性进展,极大提升了工作效率,改变了传统研究或问题解决的方式。随着大模型与生物医学研究进一步深度融合,我们有望见证更高效的靶点发现、药物研发以及个性化治疗方案,彻底重塑生命科学研究范式。
2.1 序列大模型基因组等高通量组学驱动的序列大模型已经能从海量序列中学习生物遗传序列的底层规律。Evo系列模型是迄今为止参数规模最大的生物大模型,训练数据主要来自RefSeq、GTDB在内的多个基因组数据库。Evo通过在覆盖整个物种进化树的海量基因组数据上进行训练,实现了对现有DNA的理解以及从头编写DNA序列的能力,打破了传统生物设计的局限,实现了从序列到功能的直接映射,开创了生物工程序列设计的新范式,为合成生物学研究提供了全新的方法和生物设计工具。2024年2月发布的Evo模型基于StripedHyena架构,在270万个原核生物和噬菌体基因组上进行预训练,实现了从分子到基因组尺度上精准的生物序列预测和生成任务[21]。2025年2月发布的Evo2模型采用了创新的Striped Hyena 2架构,其训练数据规模扩展至12.8万个基因组的9.3万亿个DNA碱基,涵盖人类、其他动植物以及真核生物,上下文长度可以达到100万个碱基对,其序列预测和生成的能力优于上一代模型[22]。Evo模型能够不依赖相似序列生成CRISPR-Cas系统等复杂分子化合物。Arc研究团队通过微调Evo,实现了CRISPR-Cas分子复合物和转座系统的生成式设计,并成功验证其功能活性[21]。在乳腺癌相关研究中,Evo2能够以90%的准确率预测BRCA1基因突变的有害性,指导癌症的精准治疗[22]。
Luca系列模型则进一步拓展了序列大模型的能力边界。2024年7月份首次发布的LucaOne模型基于来自16.9万个物种的核酸与蛋白质序列,采用面向DNA、RNA、蛋白质的统一架构进行自监督与半监督学习训练[23],实现了对分子生物学“中心法则”的高效学习与泛化理解,帮助研究人员更加深入地理解生物世界的底层逻辑。在此基础上,2025年6月发布的LucaVirus模型作为LucaOne在病毒学方向的衍生模型,专门用于病毒基因组与蛋白质序列的理解,在多种病毒学任务中表现出色[23],为病毒学基础研究及疫情防控提供了强大的工具。未来随着更丰富模态、更多数据的加入以及模型的持续升级,Evo、Luca等生物大模型将更深入揭示生物系统的结构、功能与调控规律。
2.2 结构大模型结构预测与蛋白质设计模型极大地加速了结构生物学研究。AlphaFold系列模型是用于蛋白质与DNA、RNA等生物分子三维结构预测的领域专用模型。该系列模型主要基于PDB数据训练,并在模型迭代过程中逐渐加入BFD、UniRef90、UniProt、RNAcentral、PubChem等数据库,最终实现了对生物分子结构的高精度预测,将蛋白质结构的获取方式从原来主要依赖冷冻电镜等实验方法转变为大模型预测的新范式。2018年,DeepMind团队首次将深度学习引入蛋白质结构预测领域并发布首个AlphaFold模型,展示了深度学习在蛋白质结构预测方面的潜力。2020年发布的AlphaFold2首次实现无同源模板条件下原子级精度蛋白单体结构预测。借助AlphaFold2,人类蛋白质组的结构覆盖率从原来的17%扩展至98.5% [34]。2024年发布的Alpha- Fold3基于全新Pairformer模块与扩散生成架构,摆脱对多序列比对数据的依赖,能够对包含蛋白质、核酸、小分子、离子和修饰残基在内的复合物进行联合结构预测,其在多种生物分子相互作用预测精度上超越专门工具。截至2024年,成立仅三年的AlphaFold蛋白质结构数据库(AlphaFold DB)共累计收录超过2.14亿个预测蛋白质结构[35],其数据已被整合到PDB、UniProt、Ensemble等主要数据资源中,对结构生物学的发展产生了重大影响。2024年诺贝尔化学奖的一半奖项被授予AlphaFold核心开发者John Jumper与Demis Hassabis,以表彰他们在蛋白质结构预测方面的成就[36],标志着AlphaFold系列模型在结构生物学乃至更多相关研究领域产生了革命性影响。
基于大型语言模型的ESM系列模型在蛋白质结构预测方法取得了显著进步,其训练数据主要来自UniRef50、PDB、AlphaFold DB等数据库。Meta团队于2021年推出了具有650 M参数的蛋白质语言模型ESM-1b[29],并在2023年推出了蛋白质语言模型ESM2和蛋白质结构预测模型ESMFold[20],实现了基于蛋白质序列的端到端结构预测。与依赖多重序列比对和模板结构的AlphaFold不同,ESMFold支持基于单条输入序列从头设计蛋白质,如宏基因组蛋白质结构预测场景,且计算效率更高。最新的ESM宏基因组图谱包含了超过7亿个预测蛋白质结构[20]。2025年发布的ESM3支持多模态信息输入,能根据功能关键字等提示信息推理蛋白质序列、结构与功能[30]。Meta团队利用ESM3通过关键残基序列、功能关键词等提示信息,迭代生成了与现有绿色荧光蛋白进化距离超过5亿年的绿色荧光蛋白esmGFP,证明了其在蛋白质设计领域的巨大潜力。未来随着模型规模和数据量的增加,ESM3有望生成更加复杂和全新的蛋白质,革新蛋白质工程领域的研究进程。
在当前,结构大模型凭借其精准的分子结构预测与设计能力,已在抗体开发、免疫治疗等产业应用领域展现出了革命性应用潜力。在抗体设计领域,多模态生成模型Chai-2[37]能够在零样本条件下以16%的命中率设计功能性抗体,较传统计算方法提升超100倍,成功将抗体研发周期压缩至2周内,标志着抗体研发从经验驱动走向计算主导设计的重大跃迁。在免疫治疗领域,Householder等[38]、Johansen等[39]以及David Baker/刘炳旭团队[40]通过RFdiffusion、ProteinMPNN、AlphaFold等生成式人工智能工具设计出具有高亲和力与高特异性的pMHC结合蛋白,能更精准地针对肿瘤抗原进行靶向治疗,为个性化精准免疫治疗提供了可行路径。
2.3 智能医疗大语言模型大语言模型凭借其强大的自然语言理解与生成能力,正逐步改变医疗诊断、临床决策与医疗知识应用模式。ChatGPT是由OpenAI于2022年推出的生成式人工智能聊天机器人,能通过多模态输入理解人类语言并生成上下文相关的互动响应。在临床医疗领域,ChatGPT展现出广泛的应用潜力:Ayers等[41]对比了ChatGPT和医生针对患者在线提问的回答,发现ChatGPT的回答质量和同理心评分均高于医生;基于GPT-4的模型已通过包括美国医师执照考试(USMLE)在内的多个专业考试,在医学问答以及病历总结等任务中展现出接近专家的水平[42];ChatGPT可以作为教学工具,帮助医学生理解复杂的医学概念,并通过模拟患者互动来提高他们的沟通技巧[43]。
国产大模型体系在医疗智能化应用方面取得积极进展,其中DeepSeek系列模型表现尤为突出。DeepSeek的出现打破了大模型领域由少数科技公司主导的局面,通过工程创新大幅降低了大模型训练和应用的门槛,促进了尖端AI技术的民主化,并通过开源推动了海量大模型应用,尤其是需要本地化部署的医疗应用。2025年1月20日,深度求索发布了其第一代开源推理模型DeepSeek-R1-Zero和DeepSeek-R1,因其低成本、高性能和开源优势而备受关注[31]。DeepSeek的开放权重框架允许用户进行定制与微调,加速了其在医疗领域的普及与应用。Sandmann等[44]基于125例多病种病例基准测试表明,DeepSeek-R1在诊断和治疗建议任务中的表现至少与现有的专有大语言模型相当;Tordjman等[45]发现,DeepSeek-R1在多模态任务中同样展现稳定性能,在临床推理能力上尤其是需要复杂推理的场景中表现突出,显著优于ChatGPT-o1和Llama 3.1-405B。这些结果表明,DeepSeek的诊断推理能力已达临床可用水平,具有作为开源医疗AI基座的潜力,有望通过持续微调发展成为安全可控的临床决策支持系统。目前,DeepSeek已在国内超过300多家医院中完成部署,参与临床诊断与决策支持、科学研究、医院管理等多项任务[46, 47]。
此外,国内生物医学大模型研究正逐步形成从基础研究到临床转化的系统性格局,在结构预测、单细胞建模、医学影像与临床决策等方向均取得重要进展。在基础研究方面,深圳湾实验室开发的RhoFold+模型[48]采用端到端深度学习策略,直接从RNA序列预测三维结构,解决了RNA结构灵活性带来的预测挑战;阿里与中国科学技术大学联合发布的GENERator模型[49]专注于基因区域的训练,在序列分类、设计与生成等任务中表现优异;中山大学与华为合作研发的CellFM模型[50]基于超1亿人类单细胞数据和8亿参数的学习框架,在细胞注释、扰动预测和基因功能预测等任务上显著优于现有模型;中国科学院自动化研究所开发的磐石·科学基础大模型[51]及其衍生的X-Cell数字细胞大模型致力于实现从基因序列到细胞表型的整体建模;崖州湾国家实验室联合团队发布的丰登·基因科学家智能体[52],通过构建“基因-性状-环境”三维知识图谱和科研推理链数据库,成功挖掘出数十个未报道的功能基因;中国科学院深圳先进技术研究院的SYMPLEX模型[53]融合领域大语言模型与合成生物学专家知识,从文献中自动挖掘功能基因元件,并在mRNA疫苗加帽酶设计中获得催化效率提升2倍以上的成果。
在临床转化应用方面,阿里巴巴推出的Lingshu[54]多模态医疗模型在多项医疗视觉问答和报告生成任务中达到先进水平,尤其在multimodal QA任务上超越多个开源模型;腾讯联合高校开发的M³FM模型[55]支持多语种、多领域的零样本临床诊断与疾病报告生成,展现了跨语言和跨中心的泛化能力;上海东方医院与中国科学院软件所联合开发的Med-Go模型(基于Qwen2-72B微调)[56]在200亿条高质量医学数据上训练,深度融合临床指南与真实世界病历数据,在评测中夺冠,目前已集成至医院HIS系统,部署于浦东新区15家社区卫生服务中心及多个省市级医院,用于辅助诊断、病历质控和罕见病分析场景,显著提升基层医疗机构的诊疗效率与质量一致性。同时,中医药大模型在产学研协同推动下快速发展,截至2025年累计发布30余款模型,融合古籍文献与现代医学知识,构建智能辨证系统,促进中医药服务的标准化与普及化,推动中医药从经验医学向数据驱动转型。总体来看,国内生物医学大模型的系统性进展正逐步实现从实验室研究到临床应用的转化,通过深度融合行业知识、优化数据治理和强化多模态推理能力,为构建自主可控、符合本土需求的智能临床决策支持体系奠定了重要基础。
表 1对2020—2025年生物医学领域代表性大模型进行了总结。
| 表 1 2020—2025年生物医学领域代表性大模型 |
随着序列大模型、结构大模型以及医疗大语言模型的快速发展,生物医学研究正逐步迈向跨模态、跨尺度的整合阶段。相比单一模态的学习任务,多组学与多模态融合不仅能够捕捉生命系统的不同层次信息,更有助于揭示分子-细胞-组织-个体之间的动态关联,因此成为引领下一轮范式变革的关键方向。
在数据层面,多模态基础模型预训练依赖大规模、多样化的多组学数据集,包括批量测序、单细胞分析、空间转录组学、染色质可及性和蛋白质组学等。当前人类生物分子图谱计划(HuBMAP)[68]、人类细胞图谱(HCA)[69]、国际人类表观基因组联盟(IHEC)[70]等国际性项目积累了海量多组学数据,为模型预训练奠定了坚实基础。同时,10x Multiome、ASAP-seq等新兴测序技术能够在单细胞或同一样本层面获得跨模态配对信息,为多模态数据整合提供了关键锚点,尤其有助于贯通DNA-RNA-蛋白质这一中心法则链条。
在算法层面,首先需要实现多模态数据统一的词元化(Tokenization),即为不同组学与类型的原始数据设计通用最小分析单元与编码方案,并将其映射到共享的嵌入空间[71]。其次,基于Transformer架构的多级混合注意力机制,能够同时捕捉局部模态内关联(如基因-基因、蛋白质-蛋白质)与全局跨模态依赖(如RNA-蛋白质、顺式元件-转录因子),从而生成兼具局部细节与全局语义的多尺度表征。AlphaFold3[28]、scGPT[64]等模型的成功验证了该架构在复杂生物分子相互作用建模中的潜力。
在学习范式上,多模态基础模型通常采用自监督学习策略,通过模态内掩码重构、跨模态对比学习与跨模态预测等任务,引导模型自动捕捉潜在的生物学规律。此外,结合生物领域本体知识(如Gene Ontology[72]、Reactome[73])或专业语料(如PubMed文献)进行检索增强与知识约束,也有助于提升模型的可解释性与生物学一致性。总体而言,多组学与多模态基础模型的快速发展正在推动生命科学研究从单一模态向跨模态、跨尺度、跨层级方向转变,使模型能够更全面地捕捉生命系统的复杂规律,为疾病机制研究、药物靶点发现与精准干预提供更加坚实的技术基础。
3 变革中的技术挑战目前生物医学大模型在多元应用场景中取得了显著进展,并正在推动生物医学研究范式转型,但相较于通用大模型,其在数据、算法与应用方面会面临更严峻的挑战。
3.1 数据孤岛、异构标准与标注鸿沟生物医学大模型依赖大规模、多中心、高质量的数据进行预训练,而医疗数据具备高度敏感性、异构性和跨机构孤岛化特征,现有开源医学数据集数量和规模远低于通用人工智能领域。
当前医学术语标准(如国际疾病分类ICD、医学系统命名法SNOMED、逻辑观测标识符名称和代码LOINC)、医学影像格式(如医学数字成像和通信标准DICOM及其变体)以及组学注释体系存在版本差异,严重制约了多中心生物医学大模型的协同训练与共享应用。同时,医学数据标注严重依赖具备专业医学背景的临床医生,导致标注过程成本高昂、工作量庞大且标注一致性存在差异,导致高质量监督数据稀缺,进一步限制了大模型的监督微调、指令微调及迁移学习能力的充分发挥。比如Mohanty等[74]提到,面部皮肤病数据因涉及高度敏感的个人信息,导致医疗机构间形成数据孤岛,致使针对红斑痤疮等疾病的计算机辅助诊断算法开发难以获取足够规模的训练数据。针对上述问题,近期提出的BiomedCoOp提示词学习框架[75],通过高效利用提示词来进行少样本学习,引导BiomedCLIP模型针对稀缺样本进行高效训练,显著提升了生物医学图像分类任务中的准确性与泛化性能,为缓解标注鸿沟与数据稀缺困境提供了切实可行的解决思路。
3.2 跨尺度多模态耦合建模困难生命系统天然具备跨尺度层级结构,从基因、蛋白质、细胞、组织、器官到表型和行为,涉及不同时间尺度与空间分辨率。生物医学大模型需同时处理纳米尺度组学、微米尺度影像、宏观临床文本与时间序列长期监测等多模态数据,存在典型的多模态-多尺度耦合问题。
主流Transformer及其衍生结构虽擅长统一编码,但在动态分辨率建模与跨尺度因果路径解析方面存在短板,限制了对复杂疾病进展过程、组学-影像-临床表型联动机制的有效表征。Warner等[76]提到,生物医学领域正在经历由多模态数据驱动的大模型变革,这些数据如何整合到统一的向量空间中,如何转换为机器学习算法能够处理的向量形式,如何维持不同数据模态之间的语态结构关系都是需要面对的挑战。针对该问题,谷歌发布的Med-Gemini通过构建多模态大语言模型框架,率先实现CT/MRI等3D影像数据与X射线等2D图像协同处理,并支持端到端CT报告生成[77],为复杂多模态建模体提供了有效的技术参考。
3.3 模型训练与应用的风险责任生物医学AI系统的伦理风险高于通用领域,涉及患者生命安全、数据主权、代际隐私与医疗责任归属等敏感问题,未经明确知情同意使用患者健康数据亦可能违反现行数据权益保护法规。即便经过匿名化处理,凭借少量的时空数据点,仍可能实现患者身份的再识别,导致严重的患者隐私泄露。Ong等[78]指出,在生物医学大模型训练过程中,若直接使用可识别患者身份的数据而缺乏有效的数据保护机制,模型存在潜在的敏感信息记忆与泄露风险。
在当前生物医学人工智能领域,常用的数据隐私技术如联邦学习与合成数据生成,有效解决了医疗数据孤岛与隐私泄露风险之间的矛盾,实现了在保护患者隐私的前提下跨机构协同建模。这些技术显著提升了模型训练的合规性与数据安全性,推动了AI在医疗场景中的落地应用。如联邦学习允许多个机构在不共享原始数据的情况下协同训练模型,通过在本地设备或服务器上训练模型,然后将更新后的模型参数发送到中央服务器进行聚合,从而能够有效地保护本地医疗数据的隐私性,因此适合用于生物医学AI模型训练。Stripelis等[79]提出了一种名为MetisFL的可扩展、安全且私密的联邦学习架构,使得生物医学机构不用共享患者敏感数据(如脑MRI影像),仅加密传输模型参数就能联合训练AI模型,还能够依靠加密抵御外部攻击,依靠梯度噪声抵御内部攻击。另外在医学AI训练,尤其是在医学影像领域中,合成数据是一种常用的数据生成方法,通过生成与真实数据相似的数据集作为模型的训练集,将图像特征和患者数据进行了有效隔离,无需访问敏感信息。Dorjsembe等[80]提出了首个用于3D语义脑部MRI合成的扩散模型Med-DDPM,该模型能够生成稳定、多样、高保真度的脑部MRI图像,大幅减少模型训练对于真实数据的依赖。
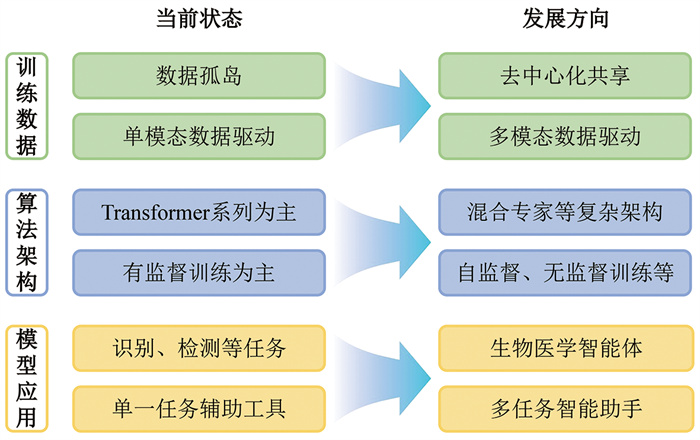
4 生物医学大模型发展前瞻生物医学大模型的未来发展需要应对复杂的数据问题,在技术方面将深度融合通用智能体的核心能力——多模态理解、自主进化与人机协同;未来5到10年,生物医学大模型有望持续迭代,实现从“辅助工具”到“智能协作者”的变革发展(图 2)。
|
图 2 生物医学大模型发展对比 |
数据壁垒和标注稀缺问题将随着联邦学习、隐私计算与合成数据技术的成熟得到缓解。未来的生物医学大模型训练可能依托于去中心化的“医疗数据联盟”,通过区块链技术实现跨机构数据共享与权益分配,同时确保患者隐私,比如允许模型在加密数据上训练而无需原始数据迁移。此外,自监督学习与主动学习的结合将减少对人工标注的依赖:模型可通过分析未标注的电子病历、医学影像和科研文献,自动挖掘潜在关联并生成高质量伪标签。合成生物学与器官芯片技术的进步还将提供仿真生物数据,弥补真实数据在罕见病或长周期研究中的不足。
4.2 技术融合与架构创新:迈向通用生物医学智能未来的生物医学大模型将突破当前单一任务或模态的局限,向多模态耦合、跨尺度推理的通用生物医学智能体演进。一方面,模型架构将更注重生物系统的层级特性,通过引入混合专家(mixture of experts, MoE)架构、扩散模型与物理建模的混合框架,实现对基因调控网络、蛋白质互作动态等跨尺度生物过程的统一表征。例如,结合AlphaFold3的分子扩散生成能力与Evo2的基因组设计能力,未来模型或能直接模拟“基因突变-蛋白质构象变化-细胞功能异常”的全链条机制,为复杂机制研究提供端到端解决方案。另一方面,多模态融合技术的进步将推动文本、影像、组学与实时传感器数据的深度整合。类似Med-Gemini[77]的框架可能进一步扩展至单细胞测序、穿戴设备数据甚至手术机器人实时反馈,形成“感知-分析-决策-执行”的闭环医疗系统。
4.3 长期愿景:生物医学的“AlphaFold时刻”在基础研究领域,生物医学大模型将加速“干湿结合”的实验范式。例如,通过预测蛋白质-药物结合位点并自动生成实验方案,模型可指导机器人实验室如Strateos[81]或Emerald[82]完成高通量筛选,将传统数月的研究压缩至数天。在药物研发中,大模型有望覆盖从靶点发现到临床试验设计的全流程,生成具有特定药理性质的分子结构,如辉瑞已利用AI设计COVID-19药物Paxlovid的候选分子从而大幅降低研发成本。
在临床层面,生物医学大模型将推动个性化医疗的普及。通过整合基因组、代谢组与生活方式数据,模型可为患者生成动态健康风险图谱,并实时优化治疗方案。例如,未来的肿瘤诊疗可能由大模型根据患者突变谱自动匹配靶向药物组合,并同步调整放疗计划。此外,结合增强现实(AR)与手术机器人,模型还能在术中提供实时解剖导航与风险预警。
类似AlphaFold引发结构生物学革命,未来模型可能通过模拟细胞代谢、免疫响应等复杂系统,发现人类尚未认知的生物规律。更进一步,大模型与自动化实验平台如AI-driven lab深度融合,科学发现可能进入“自我驱动”时代——模型生成假设→机器人验证→反馈优化模型,形成“AI-实验”闭环。
5 结论生物医学大模型作为人工智能与生物医学深度融合的产物,正以极快的速度融入各类生物医学应用场景,引发智能化范式变革,其未来发展不仅限于简单的技术迭代,更有机会促成生物医学认知方法论的重构。随着生物医学大模型的持续发展,其有望成为“生物医学领域的通用智能体”,实现从“辅助工具”到“智能协作者”的转型,并在基础研究、工业运用、临床诊疗等领域引发更加深刻的变革,而生物医学的边界也将被重新定义。
| [1] |
张国庆, 赵国屏, 李亦学. 生物医学大数据生产要素价值的实现: 从数据元素起步. 生命科学, 2023, 35: 1545-52. |
| [2] |
Collins FS, Morgan M, Patrinos A. The human genome project: lessons from large-scale biology. Science, 2003, 300: 286-90. DOI:10.1126/science.1084564 |
| [3] |
Goodwin S, McPherson JD, McCombie WR. Coming of age: ten years of next-generation sequencing technologies. Nat Rev Genet, 2016, 17: 333-51. |
| [4] |
Goldfarb T, Kodali VK, Pujar S, et al. NCBI RefSeq: reference sequence standards through 25 years of curation and annotation. Nucleic Acids Res, 2024, 53: D243-57. |
| [5] |
Parks DH, Chuvochina M, Rinke C, et al. GTDB: an ongoing census of bacterial and archaeal diversity through a phylogenetically consistent, rank normalized and complete genome-based taxonomy. Nucleic Acids Res, 2022, 50: D785-94. DOI:10.1093/nar/gkab776 |
| [6] |
Burley SK, Bhatt R, Bhikadiya C, et al. Updated resources for exploring experimentally-determined PDB structures and computed structure models at the RCSB Protein Data Bank. Nucleic Acids Res, 2025, 53: D564-74. DOI:10.1093/nar/gkae1091 |
| [7] |
Johnson AEW, Pollard TJ, Shen L, et al. MIMIC-Ⅲ, a freely accessible critical care database. Sci Data, 2016, 3: 1-9. |
| [8] |
Roberts RJ. PubMed Central: the GenBank of the published literature. Proc Natl Acad Sci U S A, 2001, 98: 381-2. DOI:10.1073/pnas.98.2.381 |
| [9] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need[C]. Proceedings of the 31st International Conference on Neural Information Processing Systems, California, 2017: 5998-6008
|
| [10] |
Radford A, Narasimhan K, Salimans T, et al. Improving language understanding by generative pre-training[EB/OL]. 2018. https://www.mikecaptain.com/resources/pdf/GPT-1.pdf
|
| [11] |
Devlin J, Chang MW, Lee K, et al. Bert: pre-training of deep bidirectional transformers for language understanding[C]. Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies, Minneapolis, 2019: 4171-86
|
| [12] |
Ji Y, Zhou Z, Liu H, et al. DNABERT: pre-trained bidirectional encoder representations from transformers model for DNA-language in genome. Bioinformatics, 2021, 37: 2112-20. DOI:10.1093/bioinformatics/btab083 |
| [13] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16x16 words: transformers for image recognition at scale. arXiv, 2021, https://doi.org/10.48550/arXiv.2010.11929
|
| [14] |
Radford A, Kim JW, Hallacy C, et al. Learning transferable visual models from natural language supervision[C]. Proceedings of the 38 th International Conference on Machine Learning, PMLR, 2021: 8748-63
|
| [15] |
Kirillov A, Mintun E, Ravi N, et al. Segment aything[C]. Proceedings of the IEEE/CVF international conference on computer vision, Paris, 2023: 4015-26
|
| [16] |
Brown T, Mann B, Ryder N, et al. Language models are few-shot learners. Adv Neural Inf Process Syst, 2020, 33: 1877-901. |
| [17] |
Kaplan J, McCandlish S, Henighan T, et al. Scaling laws for neural language models. arXiv, 2020, https://arxiv.org/pdf/2001.08361/1000
|
| [18] |
Jouppi N, Kurian G, Li S, et al. Tpu v4:an optically reconfigurable supercomputer for machine learning with hardware support for embeddings. Proc Annu Int Symp Comput Archit, 2023, 82: 1-14. |
| [19] |
Choquette J. Nvidia hopper H100 GPU scaling performance. IEEE Micro, 2023, 43: 9-17. |
| [20] |
Lin ZM, Akin H, Rao RS, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023, 379: 1123-30. DOI:10.1126/science.ade2574 |
| [21] |
Nguyen E, Poli M, Durrant MG, et al. Sequence modeling and design from molecular to genome scale with Evo. Science, 2024, 386: eado9336. DOI:10.1126/science.ado9336 |
| [22] |
Brixi G, Durrant MG, Ku J, et al. Genome modeling and design across all domains of life with Evo 2. bioRxiv, 2025, https://doi.org/10.1101/2025.02.18.638918
|
| [23] |
He Y, Fang P, Shan Y, et al. Generalized biological foundation model with unified nucleic acid and protein language. Nat Mach Intell, 2025, 7: 942-53. DOI:10.1038/s42256-025-01044-4 |
| [24] |
Pan YF, He Y, Liu YQ, et al. Predicting the evolutionary and functional landscapes of viruses with a unified nucleotide-protein language model: LucaVirus. bioRxiv, 2025, https://doi.org/10.1101/2025.06.14.659722
|
| [25] |
Rajpurkar P, Chen E, Banerjee O, et al. AI in health and medicine. Nat Med, 2022, 28: 31-8. DOI:10.1038/s41591-021-01614-0 |
| [26] |
Senior AW, Evans R, Jumper J, et al. Improved protein structure prediction using potentials from deep learning. Nature, 2020, 577: 706-10. DOI:10.1038/s41586-019-1923-7 |
| [27] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-9. DOI:10.1038/s41586-021-03819-2 |
| [28] |
Abramson J, Adler J, Dunger J, et al. Accurate structure prediction of biomolecular interactions with AlphaFold3. Nature, 2024, 630: 493-500. DOI:10.1038/s41586-024-07487-w |
| [29] |
Rives A, Meier J, Sercu T, et al. Biological structure and function emerge from scaling unsupervised learning to 250 million protein sequences. Proc Natl Acad Sci U S A, 2021, 118: e2016239118. DOI:10.1073/pnas.2016239118 |
| [30] |
Hayes T, Rao R, Akin H, et al. Simulating 500 million years of evolution with a language model. Science, 2025, 387: 850-8. DOI:10.1126/science.ads0018 |
| [31] |
Xiong L, Wang H, Chen X, et al. DeepSeek: paradigm shifts and technical evolution in large AI models. IEEE/CAA J Autom Sin, 2025, 12: 841-58. DOI:10.1109/JAS.2025.125495 |
| [32] |
Achiam J, Adler S, Agarwal S, et al. GPT-4 technical report. arXiv, 2023, https://doi.org/10.48550/arXiv.2303.08774
|
| [33] |
Bai J, Bai S, Chu Y, et al. Qwen technical report. arXiv, 2023, https://doi.org/10.48550/arXiv.2309.16609
|
| [34] |
Tunyasuvunakool K, Adler J, Wu Z, et al. Highly accurate protein structure prediction for the human proteome. Nature, 2021, 596: 590-6. DOI:10.1038/s41586-021-03828-1 |
| [35] |
Varadi M, Bertoni D, Magana P, et al. AlphaFold protein structure database in 2024:providing structure coverage for over 214 million protein sequences. Nucleic Acids Res, 2024, 52: D368-75. DOI:10.1093/nar/gkad1011 |
| [36] |
Abriata LA. The Nobel Prize in Chemistry: past, present, and future of AI in biology. Commun Biol, 2024, 7: 1409. DOI:10.1038/s42003-024-07113-5 |
| [37] |
Team CD, Boitreaud J, Dent J, et al. Zero-shot antibody design in a 24-well plate. bioRxiv, 2025, https://doi.org/10.1101/2025.07.05.663018
|
| [38] |
Householder KD, Xiang X, Jude KM, et al. De novo design and structure of a peptide-centric TCR mimic binding module. Science, 2025, 389: 375-9. DOI:10.1126/science.adv3813 |
| [39] |
Johansen KH, Wolff DS, Scapolo B, et al. De novo-designed pMHC binders facilitate T cell-mediated cytotoxicity toward cancer cells. Science, 2025, 389: 380-5. DOI:10.1126/science.adv0422 |
| [40] |
Liu B, Greenwood NF, Bonzanini JE, et al. Design of high-specificity binders for peptide-MHC-I complexes. Science, 2025, 389: 386-91. DOI:10.1126/science.adv0185 |
| [41] |
Ayers JW, Poliak A, Dredze M, et al. Comparing physician and artificial intelligence chatbot responses to patient questions posted to a public social media forum. JAMA Intern Med, 2023, 183: 589-96. DOI:10.1001/jamainternmed.2023.1838 |
| [42] |
Nori H, King N, McKinney SM, et al. Capabilities of GPT-4 on medical challenge problems. arXiv, 2023, https://doi.org/10.48550/arXiv.2303.13375
|
| [43] |
Khan RA, Jawaid M, Khan AR, et al. ChatGPT-Reshaping medical education and clinical management. Pak J Med Sci, 2023, 39: 605-7. |
| [44] |
Sandmann S, Hegselmann S, Fujarski M, et al. Benchmark evaluation of DeepSeek large language models in clinical decision-making. Nat Med, 2025, 31: 2546-9. DOI:10.1038/s41591-025-03727-2 |
| [45] |
Tordjman M, Liu Z, Yuce M, et al. Comparative benchmarking of the DeepSeek large language model on medical tasks and clinical reasoning. Nat Med, 2025, 31: 2550-5. DOI:10.1038/s41591-025-03726-3 |
| [46] |
Zeng D, Qin Y, Sheng B, et al. DeepSeek's "Low-Cost" adoption across China's hospital systems: too fast, too soon?. JAMA, 2025, 333: 1866-9. DOI:10.1001/jama.2025.6571 |
| [47] |
Chen J, Miao C. DeepSeek deployed in 90 Chinese tertiary hospitals: how artificial intelligence is transforming clinical practice. J Med Syst, 2025, 49: 1-3. DOI:10.1007/s10916-024-02133-4 |
| [48] |
Shen T, Hu Z, Sun S, et al. Accurate RNA 3D structure prediction using a language model-based deep learning approach. Nat Methods, 2024, 21: 2287-98. DOI:10.1038/s41592-024-02487-0 |
| [49] |
Wu W, Li Q, Li M, et al. GENERator: a long-context generative genomic foundation model. arXiv, 2025, https://doi.org/10.48550/arXiv.2502.07272
|
| [50] |
Zeng Y, Xie J, Shangguan N, et al. CellFM: a large-scale foundation model pre-trained on transcriptomics of 100 million human cells. Nat Commun, 2025, 16: 4679. DOI:10.1038/s41467-025-59926-5 |
| [51] |
ScienceOne. AI for Science research intelligence platform[EB/OL]. 2025. https://www.scienceos.ai/
|
| [52] |
Yang F, Kong H, Ying J, et al. SeedLLM·Rice: a large language model integrated with rice biological knowledge graph. Mol Plant, 2025, 18: 1118-29. DOI:10.1016/j.molp.2025.05.013 |
| [53] |
Wang T, Qin BR, Li S, et al. Discovery of diverse and high-quality mRNA capping enzymes through a language model-enabled platform. Sci Adv, 2025, 11: eadt0402. DOI:10.1126/sciadv.adt0402 |
| [54] |
Xu W, Chan HP, Li L, et al. Lingshu: a generalist foundation model for unified multimodal medical understanding and reasoning. arXiv, 2025, https://doi.org/10.48550/arXiv.2506.07044
|
| [55] |
Liu F, Li Z, Yin Q, et al. A multimodal multidomain multilingual medical foundation model for zero shot clinical diagnosis. NPJ Digit Med, 2025, 8: 86. DOI:10.1038/s41746-024-01339-7 |
| [56] |
Zhang H, An B. MedGo: a Chinese medical large language model. arXiv, 2024, https://doi.org/10.48550/arXiv.2410.20428
|
| [57] |
Shin H-C, Zhang Y, Bakhturina E, et al. BioMegatron: larger biomedical domain language model. arXiv, 2020, https://doi.org/10.48550/arXiv.2010.06060
|
| [58] |
Baek M, DiMaio F, Anishchenko I, et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science, 2021, 373: 871-6. DOI:10.1126/science.abj8754 |
| [59] |
Luo RQ, Sun LA, Xia YC, et al. BioGPT: generative pre-trained transformer for biomedical text generation and mining. Brief Bioinform, 2022, 23: bbac409. DOI:10.1093/bib/bbac409 |
| [60] |
Dauparas J, Anishchenko I, Bennett N, et al. Robust deep learning-based protein sequence design using ProteinMPNN. Science, 2022, 378: 49-56. DOI:10.1126/science.add2187 |
| [61] |
Yang X, Chen A, PourNejatian N, et al. Gatortron: a large clinical language model to unlock patient information from unstructured electronic health records. arXiv, 2022, https://doi.org/10.48550/arXiv.2203.03540
|
| [62] |
Singhal K, Azizi S, Tu T, et al. Large language models encode clinical knowledge. arXiv, 2022, https://doi.org/10.48550/arXiv.2212.13138
|
| [63] |
Zhu WW, Wang X. Shennong-tcm: a traditional Chinese medicine large language model[EB/OL]. (2023-06-26). https://github.com/michael-wzhu/ShenNong-TCM-LLM
|
| [64] |
Cui H, Wang C, Maan H, et al. scGPT: toward building a foundation model for single-cell multi-omics using generative AI. Nat Methods, 2024, 21: 1470-80. DOI:10.1038/s41592-024-02201-0 |
| [65] |
Luo Y, Zhang J, Fan S, et al. Biomedgpt: an open multimodal large language model for biomedicine. IEEE J Biomed Health Inform, 2024, 1-12. |
| [66] |
Singhal K, Tu T, Gottweis J, et al. Toward expert-level medical question answering with large language models. Nat Med, 2025, 31: 943-50. DOI:10.1038/s41591-024-03423-7 |
| [67] |
Tu T, Schaekermann M, Palepu A, et al. Towards conversational diagnostic artificial intelligence. Nature, 2025, 642: 442-50. DOI:10.1038/s41586-025-08866-7 |
| [68] |
Jain S, Pei L, Spraggins JM, et al. Advances and prospects for the human BioMolecular atlas Program (HuBMAP). Nat Cell Biol, 2023, 25: 1089-100. DOI:10.1038/s41556-023-01194-w |
| [69] |
Regev A, Teichmann S, Rozenblatt-Rosen O, et al. The human cell atlas white paper. arXiv, 2018, https://doi.org/10.48550/arXiv.1810.05192
|
| [70] |
Bujold D, de Lima Morais DA, Gauthier C, et al. The international human epigenome consortium data portal. Cell Syst, 2016, 3: 496-9.e2. DOI:10.1016/j.cels.2016.10.019 |
| [71] |
Team C. Chameleon: mixed-modal early-fusion foundation models. arXiv, 2024, https://doi.org/10.48550/arXiv.2405.09818
|
| [72] |
Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the unification of biology. Nat Genet, 2000, 25: 25-9. DOI:10.1038/75556 |
| [73] |
Fabregat A, Jupe S, Matthews L, et al. The reactome pathway knowledgebase. Nucleic Acids Res, 2018, 46: D649-55. DOI:10.1093/nar/gkx1132 |
| [74] |
Mohanty A, Sutherland A, Bezbradica M, et al. High-fidelity synthetic face generation for rosacea skin condition from limited data. Electronics, 2024, 13: 395. DOI:10.3390/electronics13020395 |
| [75] |
Koleilat T, Asgariandehkordi H, Rivaz H, et al. Biomedcoop: learning to prompt for biomedical vision-language models[C]. Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition, Nashville, 2025: 14766-76
|
| [76] |
Warner E, Lee J, Hsu W, et al. Multimodal machine learning in image-based and clinical biomedicine: survey and prospects. Int J Comput Vis, 2024, 132: 3753-69. DOI:10.1007/s11263-024-02032-8 |
| [77] |
Saab K, Tu T, Weng WH, et al. Capabilities of gemini models in medicine. arXiv, 2024, https://doi.org/10.48550/arXiv.2404.18416
|
| [78] |
Ong JCL, Chang SYH, William W, et al. Ethical and regulatory challenges of large language models in medicine. Lancet Digit Health, 2024, 6: e428-32. DOI:10.1016/S2589-7500(24)00061-X |
| [79] |
Stripelis D, Gupta U, Saleem H, et al. A federated learning architecture for secure and private neuroimaging analysis. Patterns, 2024, 5: 101031. DOI:10.1016/j.patter.2024.101031 |
| [80] |
Dorjsembe Z, Pao HK, Odonchimed S, et al. Conditional diffusion models for semantic 3D brain MRI synthesis. IEEE J Biomed Health Inform, 2024, 28: 4084-93. DOI:10.1109/JBHI.2024.3385504 |
| [81] |
Kamuntavičius G, Prat A, Paquet T, et al. Accelerated hit identification with target evaluation, deep learning and automated labs: prospective validation in IRAK1. J Cheminform, 2024, 16: 127. DOI:10.1186/s13321-024-00914-0 |
| [82] |
Qian H, Andresen D. Emerald: enhance scientific workflow performance with computation offloading to the cloud[C]. Proceedings of the IEEE/ACIS International Conference on Computer and Information, Las Vegas, 2015: 443-8
|
 2025, Vol. 37
2025, Vol. 37 

 林新华,上海交通大学网络信息中心副主任、博士生导师、CCF杰出会员,长期负责“交我算”校级计算平台。博士毕业于东京工业大学,研究方向为高性能计算。全球计算联盟GCC高性能计算产发委(HPC DG)主任、上海市计算机学会高性能计算专委会(上海高专委)主任、CCF高专委员常委。担任国际期刊FGCS及国内期刊《计算机科学》编委,担任IEEE Cluster 2024 (CCF-B)联合主席。承担并参与20多项各类科研项目,包括国家重点研发计划专题、国家自然科学基金等。发表国内外期刊及会议论文30多篇,包括Cell (共同一作)、Cancer Cell (共同通讯),获IEEE/ACM CCGrid24最佳论文奖(唯一通讯)。作为副主编编写“十四五”国家重点出版物图书一套,获2022年国家教学成果奖二等奖(排第2)
林新华,上海交通大学网络信息中心副主任、博士生导师、CCF杰出会员,长期负责“交我算”校级计算平台。博士毕业于东京工业大学,研究方向为高性能计算。全球计算联盟GCC高性能计算产发委(HPC DG)主任、上海市计算机学会高性能计算专委会(上海高专委)主任、CCF高专委员常委。担任国际期刊FGCS及国内期刊《计算机科学》编委,担任IEEE Cluster 2024 (CCF-B)联合主席。承担并参与20多项各类科研项目,包括国家重点研发计划专题、国家自然科学基金等。发表国内外期刊及会议论文30多篇,包括Cell (共同一作)、Cancer Cell (共同通讯),获IEEE/ACM CCGrid24最佳论文奖(唯一通讯)。作为副主编编写“十四五”国家重点出版物图书一套,获2022年国家教学成果奖二等奖(排第2)