(2 中国科学院大学, 北京 100049)
(2 University of Chinese Academy of Sciences, Beijing 100049, China)
近年来,基因编辑技术在精准性、高效性和操作简便性方面取得显著进展,推动其在基础研究与临床转化中的广泛应用。然而,随着技术的不断深化,其在临床应用中也面临着诸多安全与伦理挑战。本文在回顾基因编辑技术发展历程及医学应用态势的基础上,重点分析其在医学领域应用中的安全风险识别与评估方法,剖析存在的伦理问题,并提出综合治理的总体思路,旨在为技术的安全可控和规范化应用提供支撑。
1 基因编辑技术发展及医学应用现状 1.1 基因编辑技术研究持续迭代升级基因编辑技术的发展经历了从宽泛到精准、低效到高效、复杂到简便的演进。这一过程不仅体现了技术本身的突破,更反映了多学科交叉融合对生物医学领域的深远影响。
基因编辑工具不断扩展与优化,技术更加高效与精准。新型编辑系统不断涌现并持续改进,工具体系进一步拓展,由基于双链断裂(double-strand break, DSB)的编辑系统(如ZFN、TALEN、CRISPR)到改进的碱基编辑(base editing, BE)、先导编辑(prime editing, PE)、表观遗传编辑(epigenetic editing, EE)等方向拓展,推动实现基因组的高效、精准、多位点与大片段编辑[1]。在提高编辑效率方面,线粒体A-to-G碱基编辑器eTd-mtABEs[2]将编辑效率提升至87%,比初代BE工具高出145倍;胞嘧啶碱基编辑器haA3A-CBE[3]可兼顾高精准度和极低脱靶活性;Td-CGBE与Td-CBEs[4]、QBEmax[5]的研发提高了BE编辑产物的纯度,减少了脱靶效应等潜在副作用。同时,PE7[6]融合了小RNA结合外切核酸酶保护因子La与PE蛋白,能够大幅提高PE效率;eePASSIGE[7]将PE技术和新型重组酶相结合,可在哺乳动物细胞中高效定点整合大片段基因,编辑效率比类似方法高出16倍。在大片段DNA插入方面,双先导编辑(twin prime editing, twinPE)实现在人类细胞中插入长达40 kb的基因[8]、EvoCAST[9]实现了特定位点的精准长链DNA片段(超过10 kb)的插入,以及PCE[10]系统实现了千碱基(kb)到兆碱基(Mb)级别DNA的高效、精准、无痕编辑,提升了真核生物基因组的操纵尺度和能力。
为提升基因编辑的靶向性、安全性与应用潜力,递送载体、脱靶检测工具的研发不断取得突破。从递送载体来看,慢病毒、腺病毒因递送效率高而被广泛使用,但存在免疫原性风险及载体容量限制等问题。近年来,脂质纳米颗粒、仿生纳米颗粒、细胞外囊泡等非病毒递送载体在靶向性、特异性方面持续改进[11],取得多项成果。如脂质纳米颗粒载体LNP-168[12]无需结合抗体即可实现骨髓的高效靶向,Cas9-EDV[13]兼具细胞靶向特异性及基因编辑功能,类病毒颗粒载体RIDE[14]、ENVLPE[15]具有递送效率高、脱靶风险低的特点等。从脱靶检测工具来看,新一代脱靶检测技术不断涌现,灵敏度和特异性持续提升,为基因编辑的安全性评估提供保障,主要包括体外预测(如Digenom-seq、DIG-seq、CIRCLE-seq、PEAC-seq[16])与体内检测方法(GUIDE-tag、DISCOVER-seq、VIVO)[17]。同时,高通量测序分析、多组学整合、空间组学技术进一步提升了脱靶位点检测灵敏度与分辨率,增强了基因编辑安全评估的深度和可靠性。
此外,人工智能(artificial intelligence, AI)尤其是生成式人工智能的深度应用,正驱动重塑基因编辑工具的设计、效率预测与治疗方案开发,加速推动基因编辑迈向更高维度的创新。例如,完全由生成式AI从头设计的基因编辑工具OpenCRISPR-1,实现了人类DNA的高效精准编辑,且OpenCRISPR-1已开源,可用于科学研究或基因编辑疗法开发[18]。基于RFdiffusion和AlphaFold3设计、筛选的MLH1小型结合蛋白(MLH1-SB),可与当前的各种先导编辑器兼容适配,显著提高了PE的编辑效率[19]。同时,自动化实验平台与AI模型的结合,使大规模文库筛选和精准修饰成为可能,推动基因编辑向系统化、规模化应用演进。
1.2 基因编辑技术医学应用不断深入基因编辑技术在医学领域的应用已从早期的基础研究逐步迈向临床转化阶段。截至2025年8月,ClinicalTrials.gov数据库中登记的CRISPR基因编辑相关临床试验超过60项,主要集中在肿瘤、血液系统疾病和眼科疾病等领域。整体来看,基因编辑的医学应用呈现精准化、个体化与多样化的特点,展现出应用领域持续拓宽、治疗模式不断多元化、临床应用转化有序推进的发展态势。
基因编辑技术的临床应用领域持续扩展,由单基因疾病向多基因复杂疾病延伸。基因编辑疗法最初主要用于治疗单基因遗传性疾病,如β-地中海贫血症、镰状细胞病等。随着技术的不断进步与成熟,基因编辑疗法逐步向更多的疾病领域扩展,包括肿瘤、代谢性疾病、心血管疾病等(表 1)[20]。在单基因遗传性疾病治疗方面,全球首个基因编辑疗法于2023年获批上市,用于治疗镰状细胞病(sickle-cell disease, SCD)和输血依赖性地中海贫血(transfusion-dependent thalassemia, TDT)。在肿瘤治疗方面,针对乳腺癌、食管癌、非小细胞肺癌、肝细胞癌等疾病的临床试验取得积极进展,通过基因编辑技术改造的T细胞疗法、NK细胞疗法已显示出显著疗效。同时,基因编辑在代谢性疾病治疗中展现出潜力,尤其在高胆固醇血症、糖尿病和糖原贮积病等方面已进入临床探索,例如,全球首例由CRISPR-Cas基因编辑技术获得的供体胰岛β细胞,在未使用免疫抑制剂的情况下,能够在1型糖尿病患者体内成功存活且持续发挥生理功能达12周,为1型糖尿病治疗开辟了新途径[21]。
| 表 1 基因编辑技术在人类疾病中的临床应用(例举) |
此外,BE、PE等新型编辑工具在临床中展现出良好效果,进一步拓宽了基因编辑技术的临床应用前景。目前,全球基因编辑疗法相关的临床试验以CRISPR疗法为主,基于BE或PE的相关疗法正在陆续推进中。截至2025年6月,全球已开展20项基于BE或PE的临床试验,主要适应证为β-地中海贫血、慢性肉芽肿病、T细胞白血病、家族性高胆固醇血症等,且多为临床Ⅰ、Ⅱ期试验[22]。其中,9项试验已发布初步结果,显示出积极的临床试验结果。如全球首例定制化BE疗法成功治疗新生儿CPS1酶缺乏症[23]、PM359疗法在慢性肉芽肿病患者的临床试验中获得积极结果[24]。尽管这些技术仍处于临床初期,但在安全性、精准性和治疗潜力上已展现出优势,标志着基因治疗进入了一个更具个性化和系统化的新时代。
2 基因编辑技术医学应用的安全风险 2.1 基因编辑技术应用的安全风险识别随着基因编辑疗法的快速发展与应用场景的不断拓展,其引发的安全问题持续受到关注。基因编辑技术本身的固有局限,叠加技术应用衍生的潜在健康风险,共同构成了制约其临床应用的核心瓶颈[25]。
2.1.1 技术自身风险:脱靶效应、嵌合现象、免疫反应基因编辑技术的临床应用与转化受限于其技术自身存在的安全风险,主要集中在由编辑准确率低带来的脱靶效应,编辑效率不足引发的嵌合现象,以及递送系统固有风险所导致的免疫反应三个方面,这些风险在不同层面对技术的安全性和有效性提出了挑战。
一是编辑准确率低带来的脱靶效应。脱靶效应是基因编辑领域最受关注的技术安全风险,主要指编辑工具非预期作用于靶位点以外的基因组区域,从而引发非预期的遗传改变。CRISPR-Cas系统受gRNA的序列特异性、PAM序列依赖性、Cas蛋白的结构特性等因素的影响,会导致错误切割非目标位点、基因组结构突变,诱发染色体大片段缺失、倒位或易位等问题[26]。虽然BE、PE等技术通过避免形成DSB降低了结构变异风险,但仍存在独特的脱靶问题。例如,根据作用对象不同,BE系统会导致DNA和RNA脱靶,包括单碱基编辑BE3存在全基因组范围内的脱靶、胞嘧啶碱基编辑(cytosine base editor, CBE)产生了全基因组的脱靶单核苷酸变异、腺嘌呤碱基编辑(adenine base editor, ABE)会引起大量随机的RNA脱靶编辑等[27]。这些风险使得“精准”编辑仍存在挑战。
二是编辑效率低导致的嵌合现象。基因编辑过程往往难以实现对所有细胞的同步修改,结果导致部分细胞成功获得目标修饰,而另一部分细胞仍保留原始基因状态,从而形成基因型嵌合。例如,在人类胚胎MYBPC3基因编辑实验中,近30%的胚胎出现了非预期的DNA片段插入或缺失,未修复的突变细胞可能引发心肌功能异常[28]。嵌合现象会直接影响治疗的有效性,还可能在发育过程中产生复杂的生理效应,增加对治疗反应的不可预测性。
三是编辑组件及递送系统的固有风险引发的免疫反应。在基因编辑工具中,基因编辑组件(如Cas9蛋白或sgRNA)可能会引发机体免疫反应,如约78%的健康人血液中检测到针对SaCas9的IgG抗体,58%有针对SpCas9的抗体,且细胞免疫与体液免疫呈现高度一致性[29]。同时,基因编辑工具的体内应用依赖递送系统将编辑组件递送至靶细胞,而递送载体的选择直接影响技术安全性。目前大多数的基因疗法利用病毒载体进行基因递送,而病毒载体递送系统可能引起机体炎症、诱发免疫反应,会造成外源基因随机整合破坏基因组,这也制约了技术的临床应用。
2.1.2 健康安全风险:从个体表型到跨代际安全影响基因编辑技术层面的缺陷最终会通过细胞功能异常、组织损伤或遗传传递等路径,逐步转化为长期健康风险。不仅如此,基因编辑可能在个体及遗传层面引发多重健康隐患,且部分风险的潜伏期长达数年,甚至存在跨代传递的可能,给安全评估带来巨大挑战。
从个体层面来看,健康安全风险主要集中在免疫应答异常、炎症反应与器官渐进性损伤等方面。编辑后的细胞可能表达异常蛋白,引发毒性或过敏反应,在基因治疗中可能导致器官损伤、炎症风暴或患癌风险增加。例如,研究发现CRISPR-Cas9/AAV6介导的对造血干细胞的基因编辑会带来衰老和炎症等不良后果,从而影响治疗结果与安全性[30];CRISPR-Cas9基因编辑可以通过逆转录转座过程引起DNA大重排,从而可能会引发癌症[31]。同时,目前全球已出现多例基因编辑治疗死亡案例,如大剂量AAV基因治疗的杜氏肌营养不良症患者主要因治疗中出现先天性免疫反应导致的急性呼吸窘迫综合征而死亡[32];以及因出现不良安全事件而暂停临床试验的情况,如Editas于2022年宣布暂停全球首个体内基因编辑疗法EDIT-101的临床研究、Verve Therapeutics于2024年暂停体内碱基编辑疗法VERVE-10的杂合子家族性高胆固醇血症临床Ib期试验Heart-1研究的招募等。
此外,基因编辑在遗传与跨代层面的风险同样不容忽视。脱靶变异、大结构重排、表观遗传改变及与环境的复杂交互,都可能在后代中以潜伏、延迟的方式显现。基于小鼠[33]等动物模型的相关实验已证实基因编辑产生的安全风险可通过生殖遗传至子代,这一问题已成为基因编辑安全评估中不可忽视的关键议题。
2.2 基因编辑技术应用的安全风险评估基因编辑技术的安全风险是一个随着技术发展与认知深入而不断动态演进的过程。为构建一个科学、审慎且可操作的基因编辑风险评估体系,需要综合考虑“固有风险、研究阶段、应用场景”三大维度,以“风险-获益”平衡为核心逻辑,在“治愈疾病的巨大潜力”与“未知的、潜在的严重风险”之间找到平衡点,从而为监管决策和临床转化提供清晰指引。
技术固有风险是安全风险评估的“基础阈值”,是最直接的风险来源,主要涉及脱靶效应、免疫反应、嵌合现象及基因组稳定性等问题。这些风险取决于基因编辑工具本身的特性,尽管预测算法和检测技术不断改进,但脱靶事件仍难以完全避免,因此在风险分级体系中必须将其置于核心位置。同时,在评估中需重点聚焦基因编辑工具本身的风险可控性与潜在危害,通过可量化的指标来构建风险基线,并关注检测与预测工具的敏感度和精确性,以促进尽早识别潜在的非预期编辑事件,从而实现在技术固有风险得到充分量化和控制的前提下,保证其安全性。
技术研究阶段影响风险评估的“证据支撑力度”,需按研究进展分阶段设定评估重点。在基础研究阶段,主要风险表现为模型外推性不足以及潜在分子机制的不确定性,重点在于识别和验证潜在不良效应;进入临床前阶段,需评估在动物模型和体外系统中的疗效与安全性是否具备可重复性与可推广性,特别是长期脱靶效应和意外插入突变的稳定性问题;而在临床试验阶段,患者个体差异、免疫反应强度、治疗持久性及长期安全性成为风险评估的主要内容。即便在获批与应用后,仍需依赖长期随访与药物警戒体系监测可能的罕见或延迟性风险。评估时的关键在于建立一套可动态更新的体系,使风险分级能够随着研究进展不断修正,并通过数据积累形成完整的安全证据链,从而支持临床与监管决策。
技术应用场景决定了安全风险的“影响范围”,涉及体细胞与生殖系细胞的编辑等。在体细胞基因编辑中,相关治疗需在严格监管下推进临床研究,风险主要集中于基因治疗个体本身,其风险评估逻辑是传统医疗风险-获益分析框架,核心判断在于临床收益是否显著超过其潜在的副作用风险,可通过剂量控制、监测机制和再治疗策略实现一定程度的风险控制;但在涉及生殖细胞或早期胚胎的研究中,风险不仅表现为个体层面的不良后果,还具有不可逆的、代际传递的特性,对基因组稳定性造成长期影响,因此对安全性的要求更高。从伦理规范与监管层面看,《人类基因组编辑研究伦理指引》[34]已明确规定,目前不允许进行任何生殖系基因组编辑的临床研究。只有在对获益与风险以及其他可供选择的方案进行充分理解和权衡,安全性和有效性问题得以解决,经严格审慎的评估并在严格监管下,才可考虑开展临床研究。
3 基因编辑技术医学应用的伦理问题 3.1 基因编辑技术在医学领域的应用可能引发系列伦理问题基因编辑技术的应用在推动疾病治疗和健康改善方面展现出巨大潜能,同时在基础与临床研究、社会和生态方面的潜在伦理问题也日益引起关注。
3.1.1 基础和临床伦理层面,面临隐私保护与应用边界界定的挑战基因编辑技术在基础与临床研究方面的潜在伦理问题,已成为社会各界关注的重点议题,核心问题集中在知情同意与隐私保护、治疗与增强的界限等方面。
人类基因编辑研究需要大量的人类组织、细胞或基因样本,若缺乏严格规范约束或操作不当,易引发信息泄露、知情同意不充分等问题。其中,基因信息能够揭示致病或缺陷基因与个体疾病表型间的内在关系,具有高度敏感性,其采集、分析及应用阶段都有可能存在隐私安全问题,包括采集环节“形式同意而非实质知情”的被动参与问题,分析环节因数据加密不足或权限管理疏漏导致的信息暴露,应用环节因研究目的变更或数据共享失控引发的隐私风险[35]。例如,美国基因检测公司23andMe的数据库在2023年遭遇黑客入侵,导致近700万用户的DNA数据、家族信息等个人信息泄露。这些数据还被用于非法售卖,严重侵犯了用户隐私,引发了公众对基因数据安全与生物信息隐私保护的广泛讨论[36]。
同时,基因编辑技术在医学领域的应用模糊了“治疗”与“优化”的边界,对现有伦理框架造成挑战。传统医学以疾病治疗与健康恢复为核心目标,而基因编辑技术具备改造人类基因组的能力,使得“疾病治疗”与“功能增强”之间的界限日益模糊。例如,通过基因编辑预防或治疗遗传性疾病属于治疗范畴,但若用于提升智力、体能等非疾病相关领域,则可能走向“优生学”的伦理禁区。这种边界的模糊不仅挑战了医学伦理的基本原则,还可能重塑社会对“正常”与“异常”的定义,从而影响个体身份认同与价值观,乃至导致“基因歧视”。因此,明确基因编辑的应用边界,合理界定其在医学实践中的适用范围,是守护伦理底线与维系社会稳定的关键[37]。
此外,2025年8月全球首例基因编辑猪肺成功移植到人体[38],这标志着异种器官移植迈出重要一步,也引发了多重伦理争议。一是动物的过度“工具化”,挑战了对动物主体性及生命尊严的认知。二是物种界限的“模糊化”,引发关于人类身份与生物伦理边界的反思。三是异种病毒传播风险,猪内源性逆转录病毒(PERVs)的潜在激活和跨物种感染,可能引发全球性的生物安全危机,其长期安全性与伦理可接受度仍需进一步论证与规范。
3.1.2 社会和生态伦理层面,对社会公平与基因生态平衡造成冲击基因编辑技术不仅在个体层面引发伦理争议,也对社会结构和生态系统带来挑战,其应用可能重塑社会公平格局并影响原有生态伦理秩序。在社会公平方面,基因编辑的研发成本高、实施流程复杂,导致相关疗法价格昂贵。例如,全球首个获批的基因编辑疗法Casgevy定价在220万美元,使得绝大多数患者无法承担,可能加剧个人、家庭,甚至全球范围内的医疗资源分配不均[39]。同时,基因编辑的核心技术专利高度集中在欧美企业当中,可能进一步加深“技术鸿沟”,从而影响基因编辑技术医学应用的普及[40]。
此外,基因编辑技术的出现打破了自然进化的规律,其编辑产物的环境释放可能对生物多样性和生态系统稳定性造成不可预测的风险。在当前技术水平下,不能确保经过编辑的基因片段在代际遗传过程中完全保持稳定,其可能产生不可预见的突变或新的基因缺陷。这不仅增加了人类遗传性疾病的潜在发生风险,也可能对人类基因池的长期安全构成潜在威胁。人类作为生态系统中的成员,应该对整个生态系统的健康与稳定负责任,不能以牺牲生态平衡为代价来谋求自身利益[41]。
3.2 基因编辑研发及应用全流程的伦理关注重点各有侧重基因编辑研发及应用的全流程体系贯穿从实验室研究到临床应用的完整链条,涵盖基础研究、临床前研究、临床试验、审批与上市以及上市后监测等不同阶段。其涉及的伦理关注重点,包括技术层面的有效性与规范性,以及实践层面的受试者权益保障等。同时,基因编辑伦理研究、审查与监管贯穿基因编辑研究的全流程。只有建立全面的伦理审查机制和监管体系,才能确保实验安全、保护受试者权益,还能有效防止潜在的社会、生态及法律风险,从而实现科学进步与伦理责任的同步平衡(图 1)。
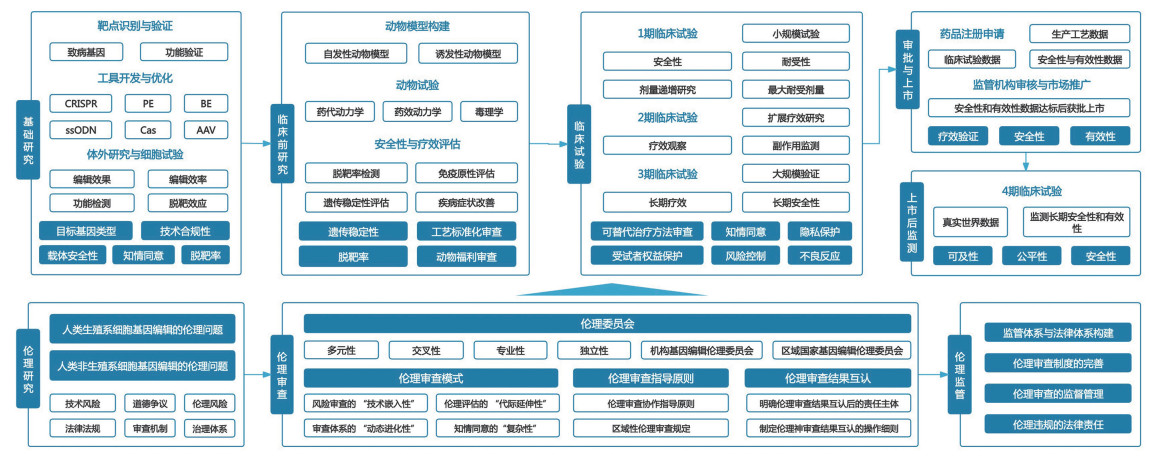
|
图 1 从实验室到临床的基因编辑研究全流程的伦理审查体系 |
基因编辑基础研究阶段涉及靶点识别与验证、工具开发与优化、体外研究及细胞实验等各个环节,其伦理审查重点各有侧重,但总体聚焦于技术规范性与整体安全性。在这个阶段,伦理审查的核心目标是确保研究设计符合科学伦理规范,同时最大程度降低潜在风险,为后续临床前研究奠定安全基础。
具体来看,目标基因的选择与验证是关键环节,常见编辑对象包括致病基因或与特定疾病相关的基因,须确认其是否符合伦理标准。同时,需要严格评估技术的规范性与载体的安全性。技术规范性方面,确保所使用技术符合国际规范,禁止使用未经验证的高风险工具;载体安全性方面,应重点评估病毒载体(如腺相关病毒、慢病毒)及非病毒载体(如脂质纳米颗粒)的生物安全性,优先选择低免疫原性且瞬时表达的递送方式。此外,还需在体外培养的细胞中进行整体安全性预评估,通过实验评估编辑效率、效果及脱靶情况,以确保基因编辑方法在细胞水平上精准、有效,并具有可控性。
3.2.2 临床前研究阶段,重视动物福利与工艺标准化在临床前研究阶段,动物模型构建与试验、安全性与疗效评估是核心环节。该阶段旨在保证科学研究目标实现的同时,最大程度减少动物痛苦与潜在风险,确定标准化的工艺流程,为后续临床应用提供可靠的科学与伦理依据。
动物伦理审查重点在于判断是否遵循3R原则(Replacement、Reduction、Refinement,即替代、减少、优化),并审视动物模型构建的必要性。在药代动力学和药效动力学研究中应兼顾科学目的与动物福利,避免进行不必要的实验操作,从而保障动物在研究过程中的基本权益。同时,工艺标准化是确保研究可追溯和可靠的关键环节。所有操作需符合标准化操作手册(standard operating procedure, SOP)要求,实验记录和数据必须完整可追溯,为伦理审查和后续临床转化提供依据。
安全性与疗效评估在临床前研究阶段也十分重要,需要对脱靶率、免疫原性及遗传稳定性等指标进行审查。若基因编辑可能引发严重免疫反应或遗传异常,即便存在潜在疗效,也需重新评估伦理可行性。疾病症状改善与动物健康之间的平衡是关键,是确保临床前研究在科学有效性与伦理合规性之间实现合理权衡的关键。
3.2.3 临床试验阶段,聚焦风险控制与受试者权益保护在临床试验阶段,受试者的权益与安全是核心关切点。该阶段聚焦不同临床阶段的特定风险,既包括研究目的的合法性与科学性,也涵盖受试者在参与过程中的权利保护[42]。
在此过程中,可替代治疗方法的评估是重要环节。体细胞基因编辑临床研究通常旨在治疗或预防疾病,其合法性与合理性需建立在基础研究和临床前证据基础上,包括必要的动物实验和体外实验,以充分验证其安全性。在此基础上,还需与其他可替代治疗方法,如小分子疗法、生物制剂治疗、其他基因治疗等方法进行综合比较,从安全性、有效性、可及性和卫生经济学等多个维度展开系统评估,充分论证体细胞基因组编辑临床研究的科学性和合理性。
与此同时,受试者权益保护是基因编辑伦理审查的关键。具体而言,知情同意需清晰透明、避免诱导,并完整披露潜在风险;未成年人和孕妇等弱势群体应得到重点关注,需通过强化知情同意程序,确保其对技术不确定性形成充分认知;治疗机会的公平性必须得到保障;隐私保护方案则需严格合规,以防敏感信息泄露。通过这些多维度的审查与约束,才能在保证科学探索的同时实现对受试者的全面保护[43]。
3.2.4 上市及上市后监测阶段,侧重伦理合规性与社会影响评估在基因编辑产品进入上市与上市后监测阶段,伦理关注的重点逐渐从临床试验层面的风险控制,转向对产品的安全性、有效性以及社会的公平性、可及性的考量。这个阶段的核心在于确保基因编辑成果不仅满足医学安全与疗效的基本要求,同时能够在社会层面实现合理分配与公平使用,从而防范技术滥用并规避伦理失衡。
在安全性与伦理合规性方面,监管机构需要全面审查临床试验数据的完整性与真实性,验证生产工艺的稳定性与可控性,通过全链条审查确保产品的潜在风险可控,避免存在安全隐患的产品流入市场。同时,公平性与可及性审查是该阶段伦理评估的另一核心,需要对上市后产品的价格合理性与分配公平性进行系统评估,防止因价格过高或资源配置不均而加剧社会不公,确保创新成果能够真正服务于公共健康需求,而非扩大健康差距。
4 思考与建议基因编辑技术在医学领域的应用正处于从实验室向临床转化的关键时期,面对安全与伦理的多重挑战,亟需建立与技术发展相匹配的、前瞻性的评估与治理体系,既要厘清“风险点”与“放行点”,明确“应该做什么”与“应该如何做”,更需形成“标准可依、风险可控、监管全程”的协同机制,以促进基因编辑技术的健康发展。具体而言,可从三方面推进体系建设:
一是构建基于技术迭代的动态风险评估框架。当前基因编辑工具迭代迅速,从CRISPR-Cas到碱基编辑、先导编辑,其脱靶效应、编辑效率和安全特征不断演变。监管制度需与技术发展保持同步优化,构建动态风险知识库,持续纳入新型编辑工具的安全性数据;同时结合人工智能与新型脱靶检测技术,实现风险识别的精准化和前瞻化。建议设立国家级的基因编辑安全监测平台,统一收集、分析和共享不良反应事件,形成“评估-监测-反馈-优化”的闭环管理。
二是完善贯穿研发全周期的伦理安全审查体系。基因编辑技术的伦理安全风险随着研发进程而变化,需要实施分阶段管理。临床前阶段应重点完善长期毒理学评价和生殖系转移风险评估;临床阶段强化实时监测与长期随访机制,重点关注受试者权益保护,并动态追踪脱靶情况;上市后阶段应建立基于真实世界数据的安全预警体系。
三是实施基于应用场景的分级分类精准管控。根据应用对象和风险等级实施差异化监管,体细胞基因编辑疗法应严格遵循风险-获益平衡原则,在严格控制质量与安全的基础上推进应用;而涉及不可逆的遗传改变及代际影响的生殖系统编辑,应继续维持当前最高安全标准限制。建议依据编辑类型、递送方式、靶向组织等关键参数制定风险分级标准,为不同应用场景提供明确的操作指引和监管要求。
| [1] |
Tao J, Bauer DE, Chiarle R. Assessing and advancing the safety of CRISPR-Cas tools: from DNA to RNA editing. Nat Commun, 2023, 14: 212. |
| [2] |
Chen L, Hong M, Luan C, et al. Efficient mitochondrial A-to-G base editors for the generation of mitochondrial disease models. Nat Biotechnol, 2025. DOI:10.1038/s41587-025-02685-x |
| [3] |
Yang L, Huo Y, Wang M, et al. Engineering APOBEC3A deaminase for highly accurate and efficient base editing. Nat Chem Biol, 2024, 20: 1176-87. DOI:10.1038/s41589-024-01595-4 |
| [4] |
Chen L, Zhu B, Ru G, et al. Re-engineering the adenine deaminase TadA-8e for efficient and specific CRISPR-based cytosine base editing. Nat Biotechnol, 2023, 41: 663-72. DOI:10.1038/s41587-022-01532-7 |
| [5] |
Hu J, Guo M, Gao Q, et al. QBEmax is a sequence-permuted and internally protected base editor. Nat Biotechnol, 2025. DOI:10.1038/s41587-025-02641-9 |
| [6] |
Yan J, Oyler-Castrillo P, Ravisankar P, et al. Improving prime editing with an endogenous small RNA-binding protein. Nature, 2024, 628: 639-47. DOI:10.1038/s41586-024-07259-6 |
| [7] |
Pandey S, Gao XD, Krasnow NA, et al. Efficient site-specific integration of large genes in mammalian cells via continuously evolved recombinases and prime editing. Nat Biomed Eng, 2025, 9: 22-39. |
| [8] |
Anzalone AV, Gao XD, Podracky CJ, et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nat Biotechnol, 2022, 40: 731-40. DOI:10.1038/s41587-021-01133-w |
| [9] |
Witte IP, Lampe GD, Eitzinger S, et al. Programmable gene insertion in human cells with a laboratory-evolved CRISPR-associated transposase. Science, 2025, 388: eadt5199. DOI:10.1126/science.adt5199 |
| [10] |
Sun C, Li H, Liu Y, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales. Cell, 2025, 188: 4693-710. e15. DOI:10.1016/j.cell.2025.07.011 |
| [11] |
Madigan V, Zhang F, Dahlman JE. Drug delivery systems for CRISPR-based genome editors. Nat Rev Drug Discov, 2023, 22: 875-94. DOI:10.1038/s41573-023-00762-x |
| [12] |
Xu S, Liang D, Wang Q, et al. In vivo genome editing of human haematopoietic stem cells for treatment of blood disorders using mRNA delivery. Nat Biomed Eng, 2025. DOI:10.1038/s41551-025-01480-y |
| [13] |
Hamilton JR, Chen E, Perez BS, et al. In vivo human T cell engineering with enveloped delivery vehicles. Nat Biotechnol, 2024, 42: 1684-92. DOI:10.1038/s41587-023-02085-z |
| [14] |
Ling S, Zhang X, Dai Y, et al. Customizable virus-like particles deliver CRISPR-Cas9 ribonucleoprotein for effective ocular neovascular and Huntington's disease gene therapy. Nat Nanotechnol, 2025, 20: 543-53. DOI:10.1038/s41565-024-01851-7 |
| [15] |
Geilenkeuser J, Armbrust N, Steinmaßl E, et al. Engineered nucleocytosolic vehicles for loading of programmable editors. Cell, 2025, 188: 2637-55. e31. DOI:10.1016/j.cell.2025.03.015 |
| [16] |
Yu Z, Lu Z, Li J, et al. PEAC-seq adopts prime editor to detect CRISPR off-target and DNA translocation. Nat Commun, 2022, 13: 7545. DOI:10.1038/s41467-022-35086-8 |
| [17] |
Pena-Gutierrez I, Olalla-Sastre B, Rio P, et al. Beyond precision: evaluation of off-target clustered regularly interspaced short palindromic repeats/Cas9–mediated genome editing. Cytotherapy, 2025, 27: 279-86. DOI:10.1016/j.jcyt.2024.10.010 |
| [18] |
Ruffolo JA, Nayfach S, Gallagher J, et al. Design of highly functional genome editors by modelling CRISPR–Cas sequences. Nature, 2025, 645: 518-25. DOI:10.1038/s41586-025-09298-z |
| [19] |
Park JC, Uhm H, Kim YW, et al. AI-generated MLH1 small binder improves prime editing efficiency. Cell, 2025, 188: 5831-46. e21. DOI:10.1016/j.cell.2025.07.010 |
| [20] |
Liu D, Cao D, Han R. Recent advances in therapeutic gene-editing technologies. Mol Ther, 2025, 33: 2619-44. DOI:10.1016/j.ymthe.2025.03.026 |
| [21] |
Carlsson PO, Hu X, Scholz H, et al. Survival of transplanted allogeneic β cells with no immunosuppression. N Engl J Med, 2025, 393: 887-94. DOI:10.1056/NEJMoa2503822 |
| [22] |
DeFrancesco L. Precision gene editing medicine makes history, and it's just getting started. Nat Biotechnol, 2025, 43: 1019-22. DOI:10.1038/s41587-025-02741-6 |
| [23] |
Musunuru K, Grandinette SA, Wang X, et al. Patient-specific in vivo gene editing to treat a rare genetic disease. N Engl J Med, 2025, 392: 2235-43. DOI:10.1056/NEJMoa2504747 |
| [24] |
Prime Medicine. Prime Medicine Announces Breakthrough Clinical Data Showing Rapid Restoration of DHR Positivity After Single Infusion of PM359, an Investigational Prime Editor for Chronic Granulomatous Disease[EB/OL]. (2025-05-19)[2025-08-26]. https://investors.primemedicine.com/node/8356/pdf
|
| [25] |
Zhang X, Ma D, Liu F. CRISPR technology and its emerging applications. Genomics Proteomics Bioinformatics, 2025, 23: qzaf034. DOI:10.1093/gpbjnl/qzaf034 |
| [26] |
Manghwar H, Li B, Ding X, et al. CRISPR/Cas systems in genome editing: methodologies and tools for sgRNA design, off-target evaluation, and strategies to mitigate off-target effects. Adv Sci, 2020, 7: 1902312. DOI:10.1002/advs.201902312 |
| [27] |
Jeong YK, Lee SH, Hwang GH, et al. Adenine base editor engineering reduces editing of bystander cytosines. Nat Biotechnol, 2021, 39: 1426-33. DOI:10.1038/s41587-021-00943-2 |
| [28] |
Ma H, Marti-Gutierrez N, Park SW, et al. Correction of a pathogenic gene mutation in human embryos. Nature, 2017, 548: 413-9. DOI:10.1038/nature23305 |
| [29] |
Raghavan R, Friedrich MJ, King I, et al. Rational engineering of minimally immunogenic nucleases for gene therapy. Nat Commun, 2025, 16: 105. DOI:10.1038/s41467-024-55522-1 |
| [30] |
Conti A, Giannetti K, Midena F, et al. Senescence and inflammation are unintended adverse consequences of CRISPR-Cas9/AAV6-mediated gene editing in hematopoietic stem cells. Cell Rep Med, 2025, 6: 102157. |
| [31] |
Tao J, Wang Q, Mendez-Dorantes C, et al. Frequency and mechanisms of LINE-1 retrotransposon insertions at CRISPR/Cas9 sites. Nat Commun, 2022, 13: 3685. DOI:10.1038/s41467-022-31322-3 |
| [32] |
Lek A, Wong B, Keeler A, et al. Death after high-dose rAAV9 gene therapy in a patient with Duchenne's muscular dystrophy. N Engl J Med, 2023, 389: 1203-10. DOI:10.1056/NEJMoa2307798 |
| [33] |
Anderson KR, Haeussler M, Watanabe C, et al. CRISPR off-target analysis in genetically engineered rats and mice. Nat Methods, 2018, 15: 512-4. DOI:10.1038/s41592-018-0011-5 |
| [34] |
中华人民共和国科学技术部. 《人类基因组编辑研究伦理指引》发布[EB/OL]. (2024-07-08)[2025-08-26]. https://www.most.gov.cn/kjbgz/202407/t20240708_191311.html
|
| [35] |
杨涵轲, 蔡红. 人体细胞基因编辑技术伦理问题的法律规制探讨——以英国为例[C]//《法治文化》集刊2024年第1卷——科技伦理的法治表达研究文集, 2024: 110-21
|
| [36] |
安全内参. 美国知名基因测试公司被黑, 或泄露30万华人血缘数据[EB/OL]. (2023-12-06)[2025-10-10]. https://www.secrss.com/articles/61465
|
| [37] |
刘怀远, 陈浩凯. 国内关于基因编辑技术的伦理问题研究述评. 长沙理工大学学报(社会科学版), 2021, 36: 63-70. |
| [38] |
He J, Shi J, Yang C, et al. Pig-to-human lung xenotrans-plantation into a brain-dead recipient. Nat Med, 2025, 31: 2288-93. |
| [39] |
Reuters. Vertex/CRISPR price sickle cell disease gene therapy at. 2 mln[EB/OL]. (2023-12-09)[2025-08-27]. https://www.reuters.com/business/healthcare-phar-maceuticals/vertexcrispr-price-sickle-cell-disease-gene-therapy-22-mln-2023-12-08/
|
| [40] |
Brinegar K, Yetisen AK, Choi S, et al. The commer-cialization of genome-editing technologies. Crit Rev Biotechnol, 2017, 37: 924-32. DOI:10.1080/07388551.2016.1271768 |
| [41] |
刘庭有, 陈晓英. 基因编辑技术的伦理问题研究. 辽宁工业大学学报(社会科学版), 2023, 25: 6-8. |
| [42] |
Singh AN. Ethical controversies and challenges in human genome editing. Int Med J, 2021, 28: 134-7. |
| [43] |
Lei R, Qiu R. Ethical and regulatory issues in human gene editing: Chinese perspective. Biotechnol Appl Biochem, 2020, 67: 880-91. DOI:10.1002/bab.2032 |
 2025, Vol. 37
2025, Vol. 37 

 熊燕,中国科学院上海营养与健康研究所生命科学信息中心研究馆员,长期从事生命科学与生物技术领域的战略研究。主持和参与国家重点研发计划“合成生物学”重点专项“合成生物学生物安全研究”、“生物安全关键技术研发”重点专项“突发生物危害事件评估决策及应急处置集成优化”、国家自然科学基金委-中国科学院学部联合资助项目“合成生物学发展战略研究”等研究课题,为国家和区域科技发展及政策管理提供决策参考
熊燕,中国科学院上海营养与健康研究所生命科学信息中心研究馆员,长期从事生命科学与生物技术领域的战略研究。主持和参与国家重点研发计划“合成生物学”重点专项“合成生物学生物安全研究”、“生物安全关键技术研发”重点专项“突发生物危害事件评估决策及应急处置集成优化”、国家自然科学基金委-中国科学院学部联合资助项目“合成生物学发展战略研究”等研究课题,为国家和区域科技发展及政策管理提供决策参考