(2 上海科技大学,上海 201210)
(2 ShanghaiTech University, Shanghai 201210, China)
国际上首批蛋白质三维结构于20世纪60年代被成功测定,美国和欧洲的部分科学家基于“共享结构数据”的想法在各类正式或非正式会议上开始讨论建立数据库来存储这些结构数据的可能性。1971年夏天,在冷泉港实验室举办的以“三维尺度上的蛋白质结构与功能”为主题的年度会议上,Helen M. Berman (现国际蛋白质结构数据库美国站点名誉主任)和来自包括布鲁克海文国家实验室(Brookhaven National Laboratory, BNL)的杰出晶体学家Walter Hamilton在内的多名科学家提出了设立蛋白质结构数据库(Protein Data Bank, PDB)的构想,同时提出了与英国剑桥晶体学数据中心负责人Olga Kennard共同建设的计划[1]。当年10月,蛋白质结构数据库正式宣布成立[2],成立之初仅收录了7个分子的晶体结构。
随着结构生物学领域对数据开放共享需求的日益迫切,经过多次讨论和倡议并最终在耶鲁大学教授Fred Richards推动下,国际晶体学会(International Union of Crystallography, IUCr)于1989年制定了研究成果在学术期刊发表前必须将数据提交至PDB的政策。这一政策在此后得到了包括《自然》《科学》和《美国国家科学院院刊》等在内的多个学术刊物的积极响应,促使数据库中分子结构数量迎来飞跃式增长。
蛋白质结构数据库涵盖了来源于X-射线晶体学(X-ray crystallography, X-Ray)、核磁共振波谱学(nuclear magnetic resonance spectroscopy, NMR)和电子显微学(electron microscopy, EM)等实验方法测定的生物大分子三维结构数据,以蛋白质的三维结构为主,此外还包括核酸、糖类、蛋白质与核酸复合物的三维结构等[3],已成为生命科学领域最具全球影响力的战略数据资源之一。
1998年,PDB的管理权移交至由罗格斯大学、圣地亚哥超级计算机中心和美国国家标准技术研究所组成的美国结构生物信息蛋白质数据库联合实验室(Research Collaboratory for Structural Bioinformatics, RCSB PDB)[4],目前其主要负责南美洲、北美洲及大洋洲地区的数据审校;欧洲生物信息研究所(European Bioinformatics Institute, EBI)的大分子结构数据库成为PDB数据的投递分中心PDB Europe (PDBe)[5],目前主要负责欧洲和非洲地区的数据审校;2000年,日本大阪大学建立了PDB新的数据投递中心PDB Japan (PDBj)[6],目前主要负责亚洲及中东地区的数据审校。三者于2003年联合组建国际蛋白质结构数据库(Worldwide Protein Data Bank, wwPDB)[7]。随后,生物磁共振数据库(Biological Magnetic Resonance Bank, BMRB)[3]和电子显微学数据库(Electron Microscopy Data Bank, EMDB)[8]于2006年和2021年相继加入国际蛋白质结构数据库,共同承担数据库的运行和全球免费开放。
国际蛋白质结构数据库致力于通过建立符合生物物理学和生物化学科学研究规范的政策、流程和技术标准,确保数据管理体系的标准化、冗余性、安全性、高可用性以及灾备保全,其严格遵循“FAIR”[9] (即可查找Findable、可访问Accessible、可互操作Interoperable、可重用Reusable)和“FACT”[10] (即FAIR和准确Accurate、保密Confidential及透明Transparent)的数据管理原则。
蛋白质结构数据库的愿景是通过分子三维结构信息的高水平存储归档、注释审校、存档管理、共享访问、数据分析、培训和教育等服务,实现数据的开放和可持续的访问[11],整合来自外部可信资源的分子功能和进化信息,不断拓展基础生物学、生物医学、能源科学和生物技术的边界,持续发展壮大涵盖基础生物学和生物化学、生物医学和药物发现、能源科学、生物工程、生物技术和计算机科学等领域的用户社群。
数据库中所有结构均按照标准统一的流程在各个站点使用OneDep系统[12],经过严格的人工数据注释审校后每周由各个数据分发站点进行数据分发。目前年平均经过注释审校和发布的数据量超过15 000条,且持续保持高速增长趋势(图 1)。截至2025年5月,国际蛋白质结构数据库中累积存储超过23万条结构数据,年数据下载量超过30亿次,每年服务全球100余万数据使用者,并且被超过400个生物数据库链接引用,规模庞大,辐射甚众。其严格的数据审核准入标准及产生每条数据所需的大量实验工作,使得其具有不可替代性、权威性、可靠性和永久性等关键特征。
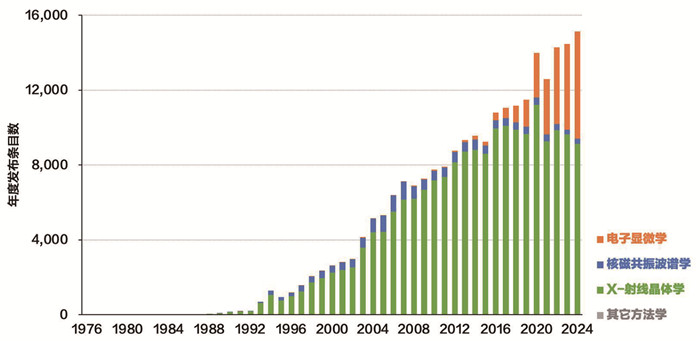
|
图 1 蛋白质结构数据库年度发布数据量趋势与研究方法组成 |
作为面向全球免费开放的生物大分子三维结构数据库,蛋白质结构数据库已经成为基础生命科学、生物医学、生物工程、生物能源、药物发现、精准医疗、现代农业等众多生命科学相关前沿科学研究及产业的核心数据资源[10, 11],同时也是利用人工智能等前沿科学方法进行蛋白质结构预测以及蛋白质设计的核心数据基础。以生命健康与药物研发为例,美国食品药品监督管理局(Food and Drug Administration, FDA) 2010‒2016年间批准了210种新药(其中94%为已知靶标、6%为未知靶标),其中88%的新药靶标在蛋白质结构数据库中都能获取详尽的结构信息,与这些靶标相关的结构数据多达5 914条目[15]。另据不完全统计,2015‒2022年由美国FDA批准上市的322个新药共向蛋白质结构数据库中投递了多达57 564个相关分子结构,涵盖了直接靶标蛋白,药物装载、转运以及代谢相关的蛋白质及药物与靶标的复合体结构。
在过去20年中,基于结构的药物设计(structure-based drug discovery, SBDD)已成为新药研发的关键技术路径之一[16]。蛋白质结构数据库中的生物大分子及小分子复合物三维结构通过精准揭示药物与靶标分子的结合机制,强有力地推动了生物医药产业的革命性发展,助力研发人员不断探索分子结构与药物功效之间的关系[17]。这些三维结构信息也为开发新的创新小分子候选化合物并通过动物及临床试验将其转化为潜在药物时的安全性和有效性平衡提供了极具价值的参考[18]。
此外,单克隆抗体设计因具有靶向性强和毒副作用低等特点成为现代生物药的重要代表,临床上广泛用于治疗恶性肿瘤、自身免疫性疾病以及器官移植排斥反应等。蛋白质结构数据库中的生物大分子间复合物,尤其是抗原-抗体复合物三维结构信息为其提供了重要的数据支撑[19]。
此外,值得一提的是,人工智能(Artificial Intelligence, AI)在生命科学领域的标志性应用AlphaFold[17, 18],即是利用了经过蛋白质结构数据库严格注释审校的高质量蛋白结构数据,对神经网络进行大规模训练,从而获得对蛋白质三维结构预测的突破性成功。当前,AlphaFold预测的蛋白质结构被广泛应用于包括生命科学研究和合成生物学探索等在内的多个领域,充分彰显了高质量科学数据对颠覆性技术创新的基石作用。
2 中国蛋白质结构数据库:为国际科学数据治理体系贡献中国力量半个世纪前,我国老一辈结构生物学家在胰岛素结构研究中取得了世界瞩目的成果,奠定了我国结构生物学的基础。经过半个多世纪的薪火相传,在“国家重点基础研究发展计划(973计划)”“国家重点研发计划”等科技专项,以及中国科学院上海高等研究院国家蛋白质科学研究(上海)设施(以下简称“蛋白质设施”)在内的国家重大科研基础设施的支持下,我国结构生物学发展始终处于国际领先水平。根据近年的统计数据,由我国科学家测定和阐释的生物大分子结构在全球成果中占比不断提高,2024年度结构数据投递数量跃居全球第二,占比超过24% (图 2)。
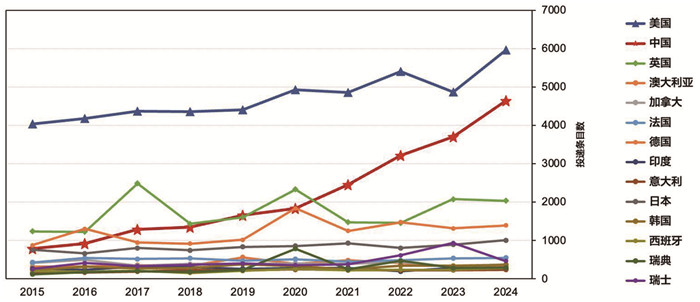
|
图 2 蛋白质结构数据库年度投递数据统计 |
科技基础设施是突破科学前沿、解决国家重大战略需求的“国之重器”。蛋白质设施作为全球首个生命科学集成性大设施,为响应国家“十四五”期间部署的“强化国家战略科技力量”的重要内容,着眼于基础科学和相关产业发展的长远利益,经过长期筹备和多方努力,在国际蛋白质结构数据库成立二十周年之际,与上海科技大学iHuman研究所、免疫化学研究所联合建设中国蛋白质结构数据库(Protein Data Bank China, PDB China),在国内建设便于结构生物学数据汇集、存储和分发的软硬件基础条件和工作机制,特别是组建结构数据质量审校和管理的专门团队,以期其成为我国蛋白质科学研究和相关生物医药产业在数据与智能时代的策源地。
中国蛋白质结构数据库已经建成与wwPDB一致且兼容的标准数据核心单元,包括数据收集系统(Data In)、数据存储核心(Data Core)、数据分发系统(Data Out)等基本单元。自主研发的数据引擎已经进入上线测试阶段,实现了数据全流程管理(提交、审校、归档、发布、检索、下载),初步形成了具有中国特色的软件平台体系;同时,也构建了包括简、繁体中文在内的多语言界面,搭建本地核心数据库,实现综合信息检索,对数据投递、分析、下载、知识分享等模块进行细分个性化建设;构建了数据分析与统计、三维在线交互、内容管理等功能板块。在软件及网络安全和访问负载方面也审慎设计,保障性能与功能的稳定。
中国蛋白质结构数据库积极参与国际合作,于2022年成为国际蛋白质结构数据库成员[22]。作为国际蛋白质结构数据库继美国、欧洲和日本之后第四个国家和地区成员,中国蛋白质结构数据库不仅拥有全球蛋白质结构的完整数据存档,同时具备了高水平的蛋白质结构注释审校能力。2022年10月,由中国蛋白质结构数据库注释审校的首批数据条目向wwPDB全球各站点同步(图 3)。至2024年年末,中国蛋白质结构数据库注释审校的数据条目即已覆盖我国本土投递数据总量的95%。目前,我国本土的蛋白质结构研究结果已经基本实现完全自主审校,并保持与国际数据库互联共享。截至2025年5月,中国团队已累计注释审校数据条目超过六千个(图 4)。

|
图 3 中国蛋白质结构数据库注释审校的首个数据条目 |
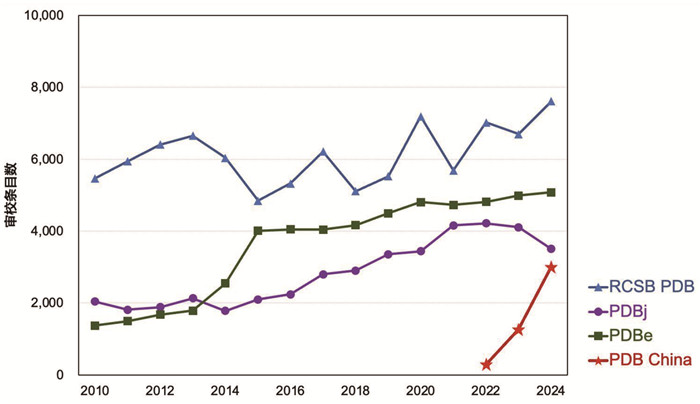
|
图 4 国际蛋白质结构数据库各成员数据注释审校统计 |
中国蛋白质结构数据库的建设,一方面为人工智能时代我国的生物医药产业和诸如蛋白质设计等前沿方向提供核心数据资源保障;另一方面,也是更为重要的,这是我国彰显大国责任、参与国际科学数据治理、加强我国生命科学国际合作的重要举措。
3 面向未来:着力完善国家数据战略基础设施面向未来,瞄准“打造服务于科技创新体系的战略数据中心”这一核心目标,蛋白质设施与上海科技大学紧密协同,对中国蛋白质结构数据库的持续建设和进一步发展进行了前瞻规划。这里就部分要点方向做简要介绍。
3.1 多学科交叉的智能数据管理体系蛋白质结构数据库建设本身是集生命科学、物理、化学、数据科学、计算科学等多学科的综合性工程,研发要素高度密集,其长期稳定发展需要有多学科背景、第次年龄梯队的人才队伍。如何构建跨学科的综合性团队,与多领域生产要素相结合,将生物大分子结构数据库发展成为多学科多领域新质生产力的数据基石,在人工智能等新技术的驱动下,发挥更大的创新价值,释放人工智能驱动创新潜能,是重要的课题。
为支持蛋白质结构数据库的进一步稳定发展,蛋白质设施在开放运行十周年之际,设立“蛋白质结构数据系统”,团队成员学科背景涵盖X-射线晶体学、核磁共振波谱学和电子显微学在内的结构生物学解析技术,同时还整合了信息与计算机科学相关专业背景,形成多学科协同的攻坚力量,有力保障了数据注释审校质量及数据分发系统建设。未来,蛋白质设施与上海科技大学将继续通过持续招募和培养,形成包括数据审校工程师、数据工程师、系统开发工程师、应用工程师、教育与社会服务、合作研究等在内的跨学科交叉战略科技人才梯队,通过与生物、化学、物理、计算、创意等跨学科交叉科学团队的紧密协同攻关,持续发展和开发智能化、综合性的蛋白质科学数据注释审校与分发系统,并积极参与国际蛋白质数据库数据注释审校规范及技术创新,为国际科学数据治理体系贡献中国力量。
3.2 数据的海洋:原始数据集的募集与发掘人工智能等数据科学分析新技术和新范式的发展,为生物结构数据的充分理解带来了新的机遇。比如,在电子显微学原始数据中,仍有大量未被充分挖掘的生物大分子动态信息,蕴含着巨大的潜在科学研究及产业应用价值。
为应对数据科学对更为原始状态的科学数据集日益增长的需求,国际上同类机构建立的电子显微学(如欧洲电子显微学原始数据库EMPIAR[23])、X-射线晶体学(如SBGrid[24]建立的Data Bank项目和日本蛋白质结构数据库的Xtal Raw Data archive项目)等原始数据归档库均在加紧建设,并与蛋白质结构数据库形成标识关联,以便用户能够对原始数据进行溯源和再利用。
依托蛋白质设施的蛋白质结构解析、蛋白质相互作用分析等相关技术系统作为原始数据产生地,可以便捷地打造多种方法学原始数据汇集枢纽,实现从原始数据到三维结构的全链条可溯源性管理,形成源头创新的战略资源储备。
3.3 数据的川流:数据共享与分发网络蛋白质结构数据库的初心即在于充分、高效、便捷地分发和利用科学数据。随着我国科学研究水平及生命医药产业发展持续提升,特别是生物医药等产业的蓬勃发展,在幅员辽阔的国土上有效地开展数据服务,需要大规模分布式数据存储和强健有力的数据网络支持。
目前蛋白质设施利用原有设计的数据库与计算分析系统对蛋白质结构数据库进行业务支撑,但着眼于未来,如何发展面向蛋白质结构数据的压缩算法、面向长距离传输的数据传输协议等技术创新,如何布局与大型科学设施毗邻的在地数据存储中心和分布式区域级数据镜像节点,并与国家生物信息中心等已有或在建中的生命科学数据体系高效协同,依然是迫切的课题(图 5)。
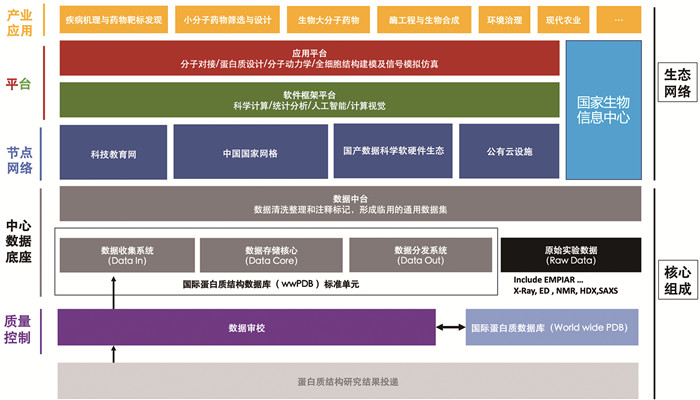
|
图 5 中国蛋白质结构数据库总体设计 |
生物大分子作为地球生命系统的基石分子,三维结构是其独特理化性质和生物学功能的表观,对其充分理解和利用,不仅将为疾病诊疗、新药研发、酶工业及合成生物学等生命科学相关领域赋能,同时也将在环境治理、现代农业、新型生物材料等众多终端产业表现出独特的价值。蛋白质三维结构作为一种高价值科学数据,是典型的“新质生产力要素”,与人工智能等数据科学前沿技术相结合,甚至能够突破传统生命科学边界,在尚未被充分认识的更为广泛的领域发挥影响,成为战略性新兴产业增长的引擎(图 5)。
人工智能相关概念和技术自20世纪50年代以来,已经发展成为一门具有极大商业应用价值的研究学科。特别是过去10年间,随着算力的提升、基础理论和模型的发展以及可用数据集的可用性增强,人工智能技术迎来蓬勃发展并产生了诸多卓越成就,深刻影响了社会发展的方方面面,获得各国工业界、学术界的高度重视,已经成为新一轮科技和产业革命的驱动力。人工智能的突破性发展高度依赖高质量数据的供给,对数据的充分利用,使其在材料科学、物理学、生命科学等学科与细分领域获得了广泛应用,如利用增强学习算法控制聚变反应堆的磁场变化、设计筛选创新药物、基于序列预测蛋白质结构等。其中,最为引人注目的是,AlphaFold等蛋白质结构预测技术利用蛋白质结构数据库中经严格质控的23万条结构数据,实现了颠覆性的性能突破。作为人工智能在结构生物学最重要的应用,AlphaFold相关论文在发布后两年间暴涨了10倍[25]。随后,在蛋白质结构预测能力的支持下,又极大地提高了全新功能蛋白的从头设计能力,为生物医药、合成生物学等领域带来了大量全新的生物活性分子,使生物大分子的研究从结构解析和理解迈向结构设计和创造,催生了生物分子“智造”时代的研究新范式。
当前,以大语言模型为代表的知识融汇技术成为世界各国新一轮竞争热点。蛋白质科学实验测定的结构数据与结构化科研文献语料相结合,经过适当的清洗、整理和专业注释标记,不意外地将再一次成为人工智能系统的高质量数据源,并能够有力地支撑相关人工智能应用的开发、训练和持续进化,使其成为实验数据与人工智能平台及其上层应用体系之间的重要承载桥梁。未来,利用这些经过注释审校的结构化数据,可以为分子对接、分子动力学模拟、蛋白质结构预测以及生物大分子间相互作用预测,提供可直接用于训练的数据集,进而为小分子药物筛选与设计和生物大分子药物研发提供帮助,有效降低药物筛选、动物实验及临床试验的成本。在中国蛋白质结构数据库中,亦将拓展小分子配体质量评估与验证标准和技术方法等相关能力。此外,在蛋白质结构数据库中,也涵盖了细菌、古菌、动物、植物等从原核到真核诸多物种的蛋白质及其小分子复合体结构信息,这些结构信息也会为生物合成、环境微生物治理、绿色生物农药开发、畜牧业病虫害生物防治等方面提供强有力的支撑。
蛋白质科学与人工智能技术的结合俨然已经成为人类社会新一轮技术革命的重要组成部分。蛋白质结构数据库是人类合作探索生命奥秘的典范代表,系统严格的注释审校流程和广泛全面的数据收录,使其成为生命科学领域影响力最深远的数据库。其数据内容是生命的微观分子组成信息,蕴含着巨大的科学价值、社会经济价值和国民健康价值,在人工智能时代,必将成为支撑生命科学及相关产业应用的关键战略资源。
| [1] |
Berman HM, Gierasch LM. How the Protein Data Bank changed biology: an introduction to the JBC reviews thematic series, part 1. J Biol Chem, 2021, 296: 100608. |
| [2] |
Crystallography: Protein Data Bank. Nat New Biol, 1971, 233: 223
|
| [3] |
Berman H, Henrick K, Nakamura H, et al. The worldwide Protein Data Bank (wwPDB): ensuring a single, uniform archive of PDB data. Nucleic Acids Res, 2007, 35: D301-3. |
| [4] |
Berman HM, Westbrook J, Feng Z, et al. The Protein Data Bank. Nucleic Acids Res, 2000, 28: 235-42. |
| [5] |
Velankar S, van Ginkel G, Alhroub Y, et al. PDBe: improved accessibility of macromolecular structure data from PDB and EMDB. Nucleic Acids Res, 2016, 44: D385-95. |
| [6] |
Kinjo AR, Suzuki H, Yamashita R, et al. Protein Data Bank Japan (PDBj): maintaining a structural data archive and resource description framework format. Nucleic Acids Res, 2012, 40: D453-60. |
| [7] |
Berman H, Henrick K, Nakamura H. Announcing the worldwide Protein Data Bank. Nat Struct Biol, 2003, 10: 980. |
| [8] |
Lawson CL, Patwardhan A, Baker ML, et al. EMDataBank unified data resource for 3DEM. Nucleic Acids Res, 2016, 44: D396-403. |
| [9] |
Wilkinson MD, Dumontier M, Aalbersberg IJJ, et al. The FAIR Guiding Principles for scientific data management and stewardship. Sci Data, 2016, 3: 160018. |
| [10] |
Van Der Aalst WMP, Bichler M, Heinzl A. Responsible data science. Bus Informn Syst Eng, 2017, 59: 311-3. |
| [11] |
Rose Y, Duarte JM, Lowe R, et al. RCSB Protein Data Bank: architectural advances towards integrated searching and efficient access to macromolecular structure data from the PDB archive. J Mol Biol, 2021, 433: 166704. |
| [12] |
Young JY, Westbrook JD, Feng Z, et al. OneDep: unified wwPDB system for deposition, biocuration, and validation of macromolecular structures in the PDB archive. Structure, 2017, 25: 536-45. |
| [13] |
Burley SK, Berman HM, Bhikadiya C, et al. RCSB Protein Data Bank: biological macromolecular structures enabling research and education in fundamental biology, biomedicine, biotechnology and energy. Nucleic Acids Res, 2019, 47: D464-74. |
| [14] |
Goodsell DS, Zardecki C, Di Costanzo L, et al. RCSB Protein Data Bank: enabling biomedical research and drug discovery. Protein Sci, 2020, 29: 52-65. |
| [15] |
Westbrook JD, Burley SK. How structural biologists and the Protein Data Bank contributed to recent FDA new drug approvals. Structure, 2019, 27: 211-7. |
| [16] |
Miller EB, Hwang H, Shelley M, et al. Enabling structure-based drug discovery utilizing predicted models. Cell, 2024, 187: 521-5. |
| [17] |
Blundell TL. Protein crystallography and drug discovery: recollections of knowledge exchange between academia and industry. IUCrJ, 2017, 4: 308-21. |
| [18] |
Stoll F, Göller AH, Hillisch A. Utility of protein structures in overcoming ADMET-related issues of drug-like compounds. Drug Discov Today, 2011, 16: 530-8. |
| [19] |
Gilliland GL, Luo J, Vafa O, et al. Leveraging SBDD in protein therapeutic development: antibody engineering. Methods Mol Biol, 2012, 841: 321-49. |
| [20] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596: 583-9. |
| [21] |
Abramson J, Adler J, Dunger J, et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature, 2024, 630: 493-500. |
| [22] |
Xu W, Velankar S, Patwardhan A, et al. Announcing the launch of Protein Data Bank China as an associate member of the Worldwide Protein Data Bank partnership. Acta Crystallogr D Struct Biol, 2023, 79: 792-5. |
| [23] |
Iudin A, Korir PK, Salavert-Torres J, et al. EMPIAR: a public archive for raw electron microscopy image data. Nat Methods, 2016, 13: 387-8. |
| [24] |
Morin A, Eisenbraun B, Key J, et al. Collaboration gets the most out of software. eLife, 2013, 2: e01456. |
| [25] |
Callaway E. What's next for AlphaFold and the AI protein-folding revolution. Nature, 2022, 604: 234-8. |
 2025, Vol. 37
2025, Vol. 37 

 王权,上海科技大学生命科学与技术学院助理教授(Tenure-Track)、博士研究生导师、免疫化学研究所研究员、中国蛋白质结构数据库(PDB China)共同负责人。分别于2009年和2014年在南开大学获得计算科学学士学位和生物化学与分子生物学博士学位,毕业后进入中国科学院生物物理研究所工作,历任助理研究员、副研究员、研究员和青年课题组长。2014‒2015年间曾赴英国牛津大学短暂访学,进行电子显微学技术方法的学习和研究。2020年2月加入上海科技大学至今,主要从事微生物病原体的跨尺度结构与功能研究;
王权,上海科技大学生命科学与技术学院助理教授(Tenure-Track)、博士研究生导师、免疫化学研究所研究员、中国蛋白质结构数据库(PDB China)共同负责人。分别于2009年和2014年在南开大学获得计算科学学士学位和生物化学与分子生物学博士学位,毕业后进入中国科学院生物物理研究所工作,历任助理研究员、副研究员、研究员和青年课题组长。2014‒2015年间曾赴英国牛津大学短暂访学,进行电子显微学技术方法的学习和研究。2020年2月加入上海科技大学至今,主要从事微生物病原体的跨尺度结构与功能研究; 许文青,上海科技大学生命科学与技术学院常任正教授、中国蛋白质结构数据库(PDB China)负责人。许文青教授在国外著名高校长期从事结构生物学研究,为美国华盛顿大学常任正教授。2019年回国加盟上海科技大学,并担任国家蛋白质科学研究(上海)设施主任(2019/01—2023/10)。目前他的研究方向是“细胞信号转导的分子机制与蛋白质设计”
许文青,上海科技大学生命科学与技术学院常任正教授、中国蛋白质结构数据库(PDB China)负责人。许文青教授在国外著名高校长期从事结构生物学研究,为美国华盛顿大学常任正教授。2019年回国加盟上海科技大学,并担任国家蛋白质科学研究(上海)设施主任(2019/01—2023/10)。目前他的研究方向是“细胞信号转导的分子机制与蛋白质设计”