(2 中国科学技术大学生命科学学院,合肥 230026)
(3 复旦大学化学系,上海 200433)
(4 中国科学院上海有机化学研究所,生物与化学交叉研究中心,上海 200032)
(5 中国科学院分子细胞科学卓越创新中心,上海 200031)
(6 上海科技大学iHuman研究所,上海 201210)
(7 中国科学院青岛生物能源与过程研究所,青岛 266101)
(2 School of Life Sciences, University of Science and Technology of China, Hefei 230026, China)
(3 Department of Chemistry, Fudan University, Shanghai 200433, China)
(4 Interdisciplinary Research Center on Biology and Chemistry, Shanghai Institute of Organic Chemistry, Chinese Academy of Sciences, Shanghai 200032, China)
(5 Center for Excellence in Molecular Cell Science, Chinese Academy of Sciences, Shanghai 200031, China)
(6 iHuman Research Institute, ShanghaiTech University, Shanghai 201210, China)
(7 Qingdao Institute of Bioenergy and Bioprocess Technology, Chinese Academy of Sciences, Qingdao 266101, China)
蛋白质的动力学(Dynamics)涉及非常复杂的动态结构变化、构象交换,甚至系综构象的热力学平衡等现象,动力学与功能密切相关[1-3]。从分子运动的角度,蛋白质的动力学可以被定义为原子坐标随时间的波动和协同运动,其时间尺度可以跨越飞秒到秒数量级,从小尺度波动到系综的相变过程,对应着蛋白质不同时间尺度的结构与动力学变化。例如,蛋白质上原子的振动发生在fs~ps时间尺度,侧链的甲基轴对称旋转最快发生在ps~ns时间尺度,酶的活性位点氨基酸残基的动力学被证明发生在ps~μm时间尺度,而蛋白质折叠过程中结构域的运动和系综构象交换等现象可能发生在μs~s时间尺度[1-3]。
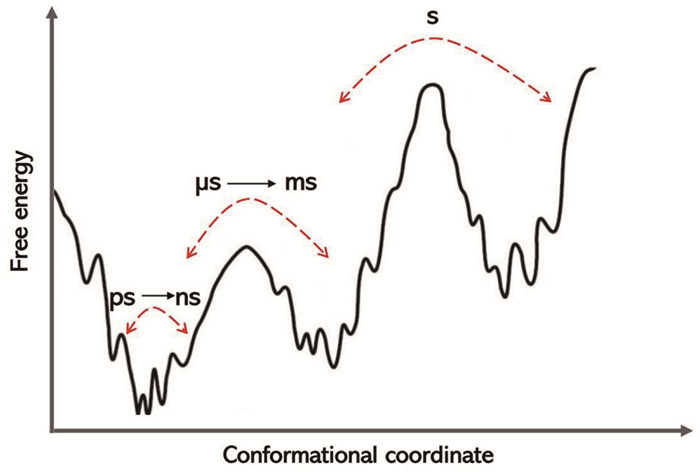
从能量超曲面(Energy Landscapes)模型的角度[4],蛋白质的动力学可以被定义为各种能量状态的结构系综、构象波动及不同状态之间的构象交换现象。关于蛋白质动力学的测量,原则上就是对能量超曲面的动态结构和构象交换现象进行完整的描述(图 1)。理论上,对蛋白质结构与动力学的完整表征需要明确:(1)每个构象状态中每个原子的三维坐标;(2)每个构象状态的布居数;(3)构象状态间的转换速率和路径。核磁共振波谱方法可以同时获得蛋白质的原子分辨率结构、不同状态的布居数、构象交换速率和动态性等不同时间尺度的动力学信息。
|
图中每个状态的布居数(P)由其自由能(G =-KB T log(P))决定,能量最小值对应于布居数最大的稳定状态,而最大值反映的是短暂的过渡状态。能量势垒的高度与状态间随机转换所需的时间尺度成正比。蛋白质内在的结构柔性、分子相互作用和环境因子等影响因素都会导致能量超曲面内的状态再分配和动力学轨迹/轮廓的改变,这些动力学变化都可能伴随着蛋白质功能的调控变化。 图 1 蛋白质的能量超曲面模型(n维空间),在构象坐标系中投影(二维空间)形成的自由能景观示意图 |
原子在不同状态之间进行交换时,原子核周围的化学环境会发生变化,从而引发其核磁信号的改变,这种现象被称作化学交换[1, 5, 6]。化学交换对核磁信号的化学位移、谱峰强度和弛豫速率等各种观察量的改变,为我们提供了研究交换过程中的动力学及热力学性质的机会。为了说明化学交换的机制,本文使用最简单的两态交换模型来描述化学交换过程,我们可以用下式表示核自旋在A、B两种状态之间的交换:
| $ \mathrm{A} \underset{k_2}{\stackrel{k_1}{\rightleftarrows}} \mathrm{~B} $ | (1.1) |
其中k1和k2分别是从A到B和从B到A的转化速率常数。整体的平衡常数定义为:
| $ K_{e q}=\frac{k_1}{k_2} $ | (1.2) |
平衡状态时,两种状态所占的布居数分别为:
| $ \begin{gathered} p_A=\frac{k_2}{k_1+k_2}=\frac{1}{1+K_{e q}}\\ \text {和}\\ p_B=\frac{k_1}{k_1+k_2}=\frac{K_{e q}}{1+K_{e q}} \end{gathered} $ | (1.3) |
为了方便描述不同时间尺度的交换,我们定义表观交换速率Kex和A、B两态间的化学位移差值△ω为:
| $ K_{e x}=k_1+k_2 $ | (1.4) |
| $ {\Delta} \mathtt{ω}=\mathtt{ω}_{{A}}-\mathtt{ω}_{\text{B}} $ | (1.5) |
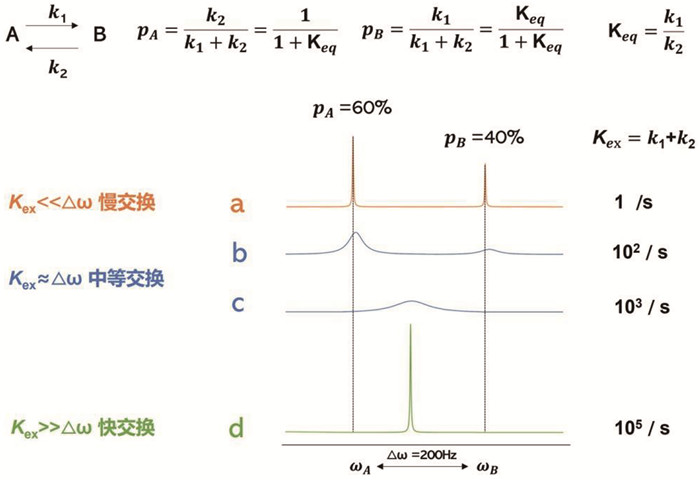
依据Kex与△ω间的大小关系,可以将化学交换分为快交换、慢交换及中等交换三种类型(图 2)。
|
图中a~d展示了A和B两种状态的交换速率从1 Hz到105 Hz时的谱图线型变化。假设A状态和B状态的比例分别占60%和40%,二者化学位移ωA和ωB相差200 Hz。a谱图表示慢交换,b谱图和c谱图表示中等交换,d谱图表示快交换。 图 2 化学交换对核磁共振谱图线型的影响示意图 |
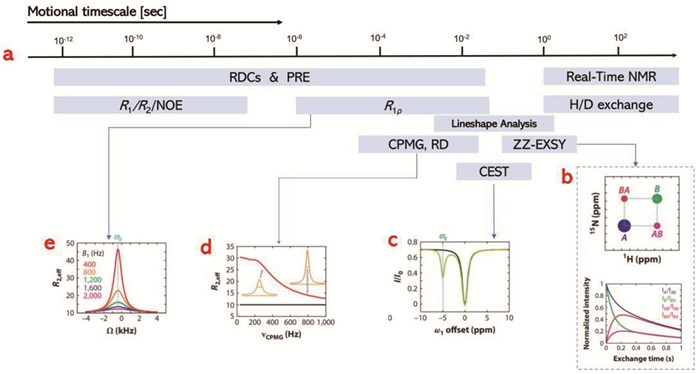
常规的核磁共振动力学方法[7, 8]通过测量1H、13C或1515N核的纵向弛豫速率(R1)、横向弛豫速率(R2)和异核Overhauser (NOE)效应等弛豫相关参数,提供ps~ns时间尺度运动的精细信息(图 3a)。在这些弛豫参数中,异核NOE对ps~ns时间尺度上的快速动态性极为敏感,R2/R1的比值通常反映旋转运动的各向异性,同时还包含着构象交换相关信息。
|
图 3 不同时间尺度的核磁动力学实验方法总结示意图 |
近年来,国际上发展了很多新的核磁共振动力学实验(图 3),用于测量蛋白质的动力学信息及其对蛋白质功能的影响。例如,适合慢交换体系的纵向磁化交换(ZZ-EXSY)实验[9](图 3b)、适合慢到中等交换体系的化学交换饱和转移(CEST)实验[10, 11](图 3c)、适合快交换体系的弛豫色散(CPMG, RD)实验[12, 13](图 3d)和自旋锁定横向弛豫速率(R1ρ)实验(图 3e)[14, 15],这四种实验都可以准确地测量μs~s范围的构象交换信息。此外,残留偶极耦合(RDCs)实验[16, 17]和顺磁弛豫增强(PRE)实验[18]则可以提供更宽时间尺度范围(ps~ms)的动态结构信息。实时核磁(Real-time NMR)实验[19]和氢氘交换(H/D exchange NMR)实验[20, 21] (图 3a)则适合研究时间尺度是秒量级的动力学过程。上述动力学实验方法的具体介绍可以参考其他专门的综述[7, 8, 22],用户可根据研究系统的动态特性来选择最合适的方法。
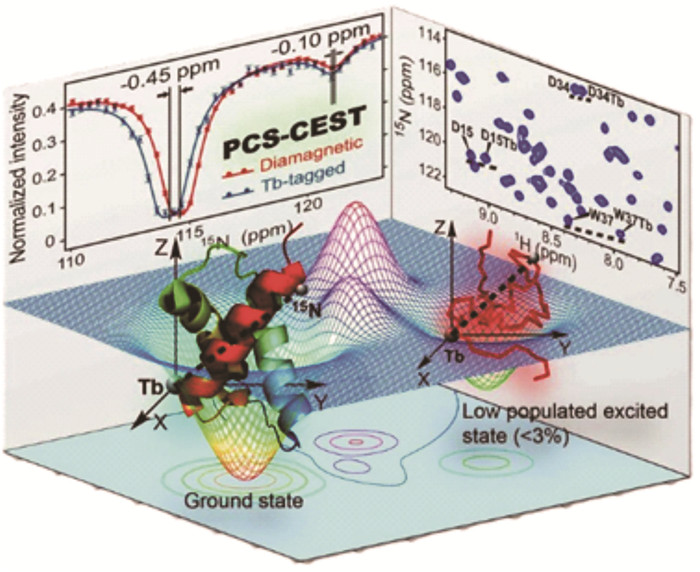
对于存在慢交换动力学特征的蛋白质体系,通过CEST、CPMG (RD)等实验可以获得低丰度激发态的化学位移,但其中缺乏精确的结构信息。通过对蛋白质进行镧系金属顺磁标记可以获取赝接触位移(PCS),其中富含距离、角度结构信息。蛋白质设施的用户——中国科学技术大学阮科课题组将CEST和PCS联用,发展出新的动力学实验方法PCS-CEST,可以测量慢交换蛋白质体系低丰度激发态15N原子的PCS[23](图 4)。该技术通过观测基态信号随照射频率的变化,间接测量出低丰度“不可见”状态的PCS,从而转化成珍贵的距离和角度信息。这对于那些有一种状态信号较差,而另一种状态信号良好的慢交换蛋白质体系尤为有用。
|
图 4 PCS-CEST测量蛋白质激发态15N原子PCS的实验方法示意图[23] |
对于存在中等交换动力学特征的蛋白质与配体相互作用体系,由于谱线加宽非常严重,配体的谱峰甚至可能消失,常规核磁方法很难测量这类情况。阮科课题组通过将PCS与CEST联用的19F-PCS-CEST技术,利用一维CEST实验获取配体19F原子的PCS约束,在分子对接给出的构象池中成功过滤出与实验数据吻合的结合态构象,结果与类似物的晶体复合物结构一致。该方法适用于核磁中等交换、小分子水溶性差等困难情形[24]。同样,通过PCS与RD联用的1H-PCS-RD技术,可以解析传统方法较难研究的中等交换时间尺度的蛋白质与配体复合物结构[25]。
2 蛋白质设施核磁分析系统合作和支撑用户的蛋白质动力学与功能研究成果 2.1 天然无规蛋白质(IDP/IDR)的动力学和相互作用与功能的关系真核生物细胞内存在大量IDP/IDR,其缺乏稳定折叠的三维空间结构,从而挑战了蛋白质生物化学中的经典“结构-功能”关系范式。由于IDP/IDR以快速转换的动态构象系综存在,其潜在的功能优势之一是能以不同的构象与多种配体发生动态相互作用,从而调控生物学功能的动态变化。下面两个研究实例很有代表性。
2.1.1 两个IDP/IDR动态相互作用的结构特征和分子机制IDP/IDR在分子识别过程中表现出极大的多样性。越来越多的IDP/IDR被发现形成“模糊复合物”。模糊性概念广泛,涵盖了IDP/IDR涉及的具有动态和多价特征的蛋白质-蛋白质相互作用,其复合物具有构象异质性、混杂的结合特性和动力学特征。
王文宁课题组通过结合核磁共振方法以及分子动力学模拟,确定了4.1G-CTD与NuMA两个IDP/IDR结合的分子机制[26],其特征为4.1G-CTD中疏水区域(βA和βB基序)之间特定堆积形成分子识别“热点”。NMR实验结果表明,酸性条件导致4.1G-CTD的β-strand二级结构含量减少,加速其骨架动力学并减弱其分子内堆积,表明三级结构属性和稳定的局部结构排列对于二者模糊相互作用中的分子识别至关重要。该工作揭示了IDP分子识别的新机制,表明模糊结合的特异性不仅源于两个IDP上“热点”基序的物理化学互补性,还取决于固有无序的4.1G-CTD紧凑的三维构象特征。
2.1.2 液-液相分离中IDP驱动的瞬态相互作用及其精细调控机制蛋白质的液-液相分离(LLPS)被广泛认为是细胞内构建无膜亚结构、调控信号转导与基因表达等多种关键生命过程的重要机制。这一病理过程与包括肌萎缩侧索硬化症(ALS)在内的多种神经退行性疾病密切相关。
刘聪课题组聚焦于ALS关键致病蛋白FUS的相分离行为研究,发现分子伴侣Hsp27在正常生理条件下以高聚体形式存在,能够有效抑制FUS的LLPS及其向LSPT的转变[27]。通过核磁滴定和PRE实验,研究团队系统揭示了Hsp27通过其天然无序的N端结构特异性结合FUS的低复杂度结构域,抑制其介导的分子间相互作用与相分离。在细胞遭受应激刺激时,Hsp27的N端三个位点会发生磷酸化,导致其高聚体部分解聚并形成磷酸化状态。更为重要的是,磷酸化的Hsp27不仅削弱了其抑制FUS LLPS的能力,还可与FUS发生共相分离,从而以一种保护方式维持FUS处于可逆的液相,有效阻止其进一步转化为致病性淀粉样聚集体。
该研究凸显了核磁共振在解析蛋白质LLPS的分子机制中的独特优势。与传统结构生物学手段(如X射线晶体学或冷冻电镜)相比,核磁方法在测定IDP与其相互作用伙伴之间的高度动态或瞬态的结合界面和机制方面具有明显优势。
2.2 甲基转移酶的构象动态性及功能调控与催化特异性的关系酶“结构与动力学”的关系可以比作“肉体与灵魂”的关系,晶体结构往往仅反映静态稳定构象,难以捕捉酶的柔性区域在溶液中可能存在的动态构象。以下案例巧妙地结合晶体学与液体核磁方法,证明了酶关键残基动力学是其催化特异性的“灵魂”。
2.2.1 构建基于19F-NMR标记的动态结构分析体系,研究辅助蛋白诱导MLL酶活性构象的形成机制组蛋白H3K4位点的甲基化修饰由MLL家族甲基转移酶介导,该家族不同成员在催化能力和产物特异性方面表现出显著差异。MLL家族蛋白本身催化活性较弱,需在辅助蛋白RBBP5-ASH2L的作用下被激活。MLL3SET与MLL1SET在游离状态和与RBBP5-ASH2L复合状态下的结构比较显示,SET-I区域发生显著构象重排。陈勇课题组在SET结构域引入三氟甲基苯丙氨酸(tfmF),并利用19F-NMR对溶液状态下的局部构象进行监测[28]。结果显示,位于SET-I区域的F4827tfmF在游离状态下显示两个峰,提示存在快速交换的多构象状态,而与RBBP5-ASH2L结合后信号转为单峰,表明该区域构象趋于稳定;相比之下,SET-N区域的Y4762tfmF未显示显著变化。这些结果说明SET-I区域的构象动态性受到辅助蛋白的特异性调控。该工作展现了19F-NMR技术在研究柔性区域构象动态变化过程中的独特优势。
2.2.2 保守酪氨酸(Y1和Y2)的动态特性共同决定MLL家族酶的产物特异性尽管MLL家族甲基转移酶在结构上高度保守,不同成员在产物特异性上仍表现出一定的差异。具体表现为它们在催化H3K4从me1至me2以及me2至me3的过程中效率不同。为了揭示其分子基础,陈勇课题组聚焦于活性中心中两个保守酪氨酸残基(Y1和Y2)在不同MLL复合物中的构象状态与动态行为。研究组解析了MLL1复合物与不同甲基化状态的底物(H3K4me0、me1、me2)以及MLL3复合物与H3K4me0结合的晶体结构,结果显示两者活性位点高度相似,赖氨酸K4侧链被一系列保守的芳香族和疏水残基包围。进一步比较发现,MLL1在结合不同甲基化状态的底物时,SET结构域构象基本不变,说明底物甲基化状态并未引发底物结合口袋的构象重排。因此,产物特异性的差异更可能来源于构象动态性的调控,而非静态结构差异。
为探究其动态基础,研究者在两个关键酪氨酸位点(Y1和Y2)引入tfmF,并通过19F-NMR分析其构象变化[29]。结果发现,MLL3中的Y1 (Y4884tfmF)仅显示单一谱峰,表明该位点构象稳定;而MLL1 (Y3942tfmF)和SET1B (Y1898tfmF)则表现为多峰,反映其具有更高的构象灵活性。在Y2位点,SET1B的Y1813tfmF呈现更宽的峰形和更多的构象态,动态性最强,MLL1则相对较弱。这些结果提示,Y1和Y2的构象动态性共同决定不同MLL复合物在多甲基化反应过程中的催化效率和产物特异性。
2.3 单次跨膜蛋白质的胞外区域动态结构、跨膜区多聚结构与受体自抑制及跨膜信号传递的关系受体介导的信号转导在细胞命运中发挥关键作用,其中肿瘤坏死因子受体(TNFR)超家族的跨膜区(TMH)结构长期以来被视为信号传递的“静默参与者”,对膜受体仅起到膜上定位的功能。以下案例充分发挥核磁方法在膜蛋白质结构和动态研究中的优势,打破了传统受体激活必须依赖胞外配体结合的观念。
2.3.1 DR5跨膜螺旋自激活机制,揭示无配体状态下的寡聚网络动态结构基础周界文课题组以死亡受体DR5为模型,采用高分辨率核磁共振方法,系统揭示了其跨膜螺旋TMH在无配体状态下通过自聚集驱动信号激活的全新机制。研究团队首先在类脂质双分子层环境bicelles中重构DR5-TMH,捕捉其在膜环境下的真实构象。通过NOE获取分子间精细接触位点,发现DR5-TMH不仅形成稳定三聚体,还进一步组装为六聚体网状结构。这一高阶寡聚行为由高度保守的GXXXG基序驱动,其中G213和G217之间存在显著的主链-侧链NOE,显示其为二聚体界面核心残基。三聚体界面则依赖T219、A222和I226等位点之间的疏水堆积。通过核磁空间约束信息计算得到的结构模型,不仅描绘了野生型DR5-TMH最小功能单位的空间组装形式,也明确了其各级聚集界面的构象基础[30]。进一步通过细胞切割实验验证该聚集行为的生理功能,发现胞外结构域(ECD)的存在会阻碍TMH聚集,抑制下游信号激活;而一旦去除ECD,TMH即可自发聚集,并促进细胞内信号复合体的组装与激活,展示了跨膜结构本身即具备信号转换的能力,这与前人研究的TNFR家族的其他成员存在明显的差异。
2.3.2 DR5胞外结构域结合配体前的动态结构,揭示细胞因子受体自抑制的动态调控机制在解析受体跨膜区自激活机制后,如何避免受体在静息状态下误激活是理解DR5信号精准调控的核心。周界文课题组针对DR5受体,结合化学位移干扰、PRE和NOE等一系列核磁实验,系统揭示了其ECD在未结合配体时通过特定构象实现跨膜区聚集自抑制的分子机制[31]。研究团队首先制备高纯度的DR5-ECD标记样品,借助特定磷脂,将蛋白单向锚定至含DMPC-DH7PC的双分子层bicelles中,在类膜环境下开展高分辨核磁共振结构测定。核磁谱图显示,DR5-ECD在脂质膜表面发生构象变化,由溶液中可溶单体转变为具有特定聚集性的状态,且其中两个非重叠自缔合界面被清晰解析。随后,利用两种顺磁探针(16-DSA和5-DSA)进行PRE实验,发现DR5-ECD并非直立于膜表面,而是以“匍匐”姿态贴附在脂质层上,导致其与配体TRAIL的结合位点部分被掩埋。这种独特的构象形成了一种功能上非活化的动态的前聚集态,阻止跨膜区自发组装并静默受体活性。研究团队还开发了一种单价纳米抗体,能够靶向干扰DR5的自抑制凝聚界面,进而在无TRAIL存在的情况下诱导其激活。该研究首次从原子层面刻画了细胞因子受体在配体结合前的自抑制构象机制,也为后续靶向ECD的动态聚集状态开发抑制小分子或抗体类激动剂提供了结构基础。
2.4 免疫受体近膜区及其胞内区域的动态相互作用与受体活化调控的关系T细胞作为免疫系统的关键效应细胞,其活化机制尚未完全阐明。它通过其表面抗原受体(TCR)和共刺激、共抑制受体(如CD28和LAG3)等协同识别抗原信号,激活下游信号转导网络,启动获得性免疫应答。如下案例利用核磁共振等方法,揭示了免疫受体结构动态性和免疫应答的关系。
2.4.1 利用核磁共振揭示CD28和TCR的结构动态性调控免疫应答的新机制许琛琦课题组通过核磁共振谱图解析发现CD28的胞内区通过其富含碱性残基(BRS)基序与酸性磷脂发生静电互作,将CD28胞内区完全地屏蔽在膜脂双层中,从而阻止CD28与下游信号分子的自动结合[32]。高分辨率钙成像发现TCR活化后会引发Ca2+在CD28周围的富集,通过核磁共振Ca2+滴定发现其能够通过静电竞争来直接打破CD28与酸性磷脂的静电互作,从而帮助CD28的活化。该发现解释了CD28对TCR依赖的分子机制,提出了TCR-Ca2+-CD28的正反馈环路模型,为理解T细胞的抗原敏感性提供了新思路[33]。此外,为探究T细胞功能多样性和特异性的结构基础,研究团队利用溶剂顺磁弛豫增强(PRE)技术,测定了TCR-CD3复合物中CD3ε胞内区与磷脂结合的构象动态特征,发现部分区域的氨基酸残基PRE效应较低,表明该区域是除BRS基序以外的次级膜结合位点[34]。这些实验证明,膜脂依赖的TCR复合物构象动态特征很可能是TCR传导不同抗原刺激信号的结构基础。该项工作为理解T细胞的抗原特异性提供新的理论模型。
2.4.2 揭示免疫检查点LAG3由泛素化介导的“埋藏-释放”动态调控机制及其临床应用前景免疫检查点LAG3靶向药物于2023年获得美国FDA批准上市,但其配体结合如何触发LAG3活化的分子机制仍不明确。LAG3近膜区含有BRS基序,紧随其后的是关键信号域FSALE以及泛素化位点。为探究LAG3的激活机制,研究团队利用核磁共振检测到LAG3的胞内关键信号域与膜脂互作,被埋藏在细胞膜中,从而隔离信号转导过程;当配体与LAG3受体结合后,泛素化修饰可促使LAG3胞内信号域从细胞膜中释放出来,从而有效激活LAG3的免疫检查点功能[35]。这一泛素化调控的“埋藏-释放”机制为受体活化提供了新范式,并指导团队开发出新型疗效预测标志物,为靶向免疫检查点的精准治疗提供了新策略。
2.5 GPCR膜蛋白质的大幅度动态性与多步骤配体选择性的关系GPCR膜蛋白质是很多药物的作用靶点,GPCR及其与药物的复合物电镜和晶体结构都暗示GPCR在信号传递过程中必然伴随大幅度的结构变化,然而GPCR详细的结构动力学研究难度极大。
上海科技大学的Kurt Wüthrich课题组基于19F-NMR技术研究神经激肽-1受体NK1R与小分子药物阿瑞匹坦(aprepitant)的结合状态,通过二维[19F, 19F]-EXSY等实验发现阿瑞匹坦分子上位于-61.6 ppm和-62.9 ppm的两个三氟甲基存在着慢交换现象,交换速率在6~7 s-1左右。进一步研究显示,阿瑞匹坦连接两个三氟甲基的芳环存在翻转运动,该翻转需要的空间范围至少为6 Å [36]。作者认为其跨膜螺旋可能经历类似“呼吸运动”的大尺度运动,正是这种动态性支持了多步骤的配体选择机制,即GPCR大尺度动态性允许大量不同形状和大小的配体进入并结合,随后通过小尺度的特异性相互作用精筛,最终决定配体的结合效能。该工作展现了液体核磁共振方法的优势和不可或缺性。
2.6 细胞原位环境蛋白质的动力学和相互作用与蛋白质折叠的关系复杂的细胞环境是否影响和如何影响蛋白质动态性和折叠是一个重要,但是悬而未决的问题。如下案例展示了细胞内核磁共振方法的最新技术发展和应用。
2.6.1 构建测量细胞内蛋白质动态性模型方法,研究细胞环境弱相互作用对蛋白质动态性的影响机制姚礼山课题组发展了一种基于蛋白质主链15N横向弛豫速率R2的核磁共振方法,利用细胞内和缓冲液中蛋白质15N R2弛豫速率差异(ΔR2) 来表征细胞内蛋白质主链的动态性。发现蛋白质GB3L环loop1区域具有很强的柔性。细胞内蛋白质构象动态性与缓冲液中获得的结果趋势一致,但是数值大小有显著差异[37]。环loop1的突变体表明,疏水和带正电荷的突变与周围大分子的相互作用变强,会让该区域在细胞内蛋白质环loop1变得更加刚性,即抑制了蛋白质环loop1区域的动态性。该工作证明了细胞内蛋白质与其他生物大分子的弱相互作用会抑制蛋白质环loop的构象动态性,进而可能影响蛋白质的功能,并进一步凸显了核磁共振在细胞内直接研究蛋白质性质和功能的重要性。
2.6.2 发展DMC-CPMG耦合技术,测量细胞内蛋白质的静电相互作用,解析其与细胞内弱相互作用的关联机制研究团队发展了一种基于双突变循环(DMC)的NMR 1H-15N CPMG-RD方法,应用CPMG来测量不同突变体的折叠动力学数据,结合双突变循环方法,实现了对蛋白GB3的8对电荷相互作用的细胞内原位检测[38]。研究结果表明细胞内静电作用在0.8 kcal/mol到+0.5 kcal/mol之间。该方法测量精确度高,静电误差通常小于0.1 kcal/mol。进一步的研究表明,折叠态和解折叠态与细胞环境弱相互作用都可以影响细胞内蛋白质静电相互作用强弱的大小。比起折叠态,细胞环境更易于与解折叠态相互作用,从而削弱蛋白质电荷间的静电相互作用。
2.6.3 测量细胞内蛋白质XH/π相互作用,探究其对细胞内蛋白质构象打开-关闭的影响机制XH/π是一种涉及芳香残基的非共价相互作用,是促进蛋白质折叠的重要作用力,在蛋白质的功能实施过程中起着举足轻重的作用。研究团队采用前期提出的DMC-CPMG耦合技术方法,测量了细胞内蛋白质的N-H/π和C-H/π相互作用[39]。结果表明,与水溶液中的数值相比,细胞内的N-H/π和C-H/π相互作用更弱。XH/π相互作用的改变可能会影响蛋白质的动态性,这一点可以用氢氘(H/D)交换实验来验证。结果显示,细胞内有中等交换的蛋白质氨基酸残基的氢氘交换速度要快于水溶液中的速度,表明细胞内蛋白质局部构象更加容易打开,进而促进氢氘交换。这就证明了细胞内N-H/π和C-H/π相互作用的削弱导致蛋白质动态性的变化。
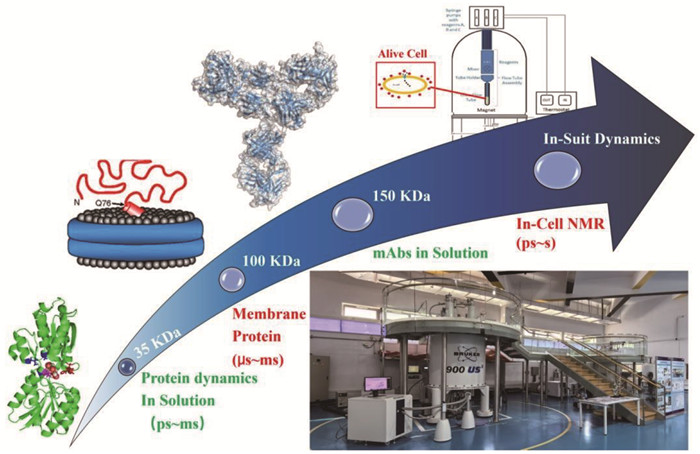
3 总结与展望蛋白质在溶液中通常不会保持一种静态的结构状态,而是在多种构象之间动态切换。蛋白质之间的相互作用和协同作用也表现出高度的动态性,其中协同作用指的是不同分子之间通过相互作用增强彼此功能的现象。在近生理条件下,如何通过实验手段准确测量蛋白质动力学信息,是当前生物物理学和结构生物学领域的重要科学问题。除了分子动力学模拟外,目前可用于准确获取蛋白质结构和动态构象分布的实验手段包括核磁共振、时间分辨X射线晶体学、小角散射、单颗粒冷冻电镜、高分辨质谱和单分子荧光共振能量转移等技术。其中,液体核磁共振技术是唯一能够在近生理条件下同时获取蛋白质动态结构、构象布居数和构象交换速率的技术。它可以在原子分辨率水平上测量跨越皮秒到秒时间尺度的动力学信息(图 5)。
|
图 5 液体核磁共振研究蛋白质结构、动力学与功能关系的技术优势与应用进展示意图 |
未来,结合不同时间尺度的核磁共振动力学实验测定方法与超长(ms级)分子动力学模拟能力,对蛋白质动力学和能量超曲面的深入研究,将有助于对蛋白质结构、动力学、相互作用和功能关系获得更深入的理解。
| [1] |
Kurt W. NMR of Proteins and Nucleic Acids: New York: Wiley Press, 1986: 1-320.
|
| [2] |
Karplus M, Kuriyan J. Molecular dynamics and protein function. Proc Natl Acad Sci U S A, 2005, 102: 6679-85. |
| [3] |
Henzler-Wildman K, Kern D. Dynamic personalities of proteins. Nature, 2007, 450: 964-72. |
| [4] |
Boehr DD, McElheny D, Dyson HJ, et al. The dynamic energy landscape of dihydrofolate reductase catalysis. Science, 2006, 313: 1638-42. |
| [5] |
Perrin CL, Dwyer TJ. Application of two-dimensional NMR to kinetics of chemical exchange. Chem Rev, 1990, 90: 935-67. |
| [6] |
Rule GS, Hitchens TK. Fundamentals of protein NMR spectroscopy: Dordrecht: Springer Science & Business Media, 2006: 1-530.
|
| [7] |
Palmer AG 3rd. NMR characterization of the dynamics of biomacromolecules. Chem Rev, 2004, 104: 3623-40. |
| [8] |
Kleckner IR, Foster MP. An introduction to NMR-based approaches for measuring protein dynamics. Biochim Biophys Acta, 2011, 1814: 942-68. |
| [9] |
Jeener J, Meier BH, Bachmann P, et al. Investigation of exchange processes by two-dimensional NMR spectroscopy. J Chem Phys, 1979, 71: 4546-53. |
| [10] |
Vallurupalli P, Bouvignies G, Kay LE. Studying "invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc, 2012, 134: 8148-61. |
| [11] |
Vallurupalli P, Sekhar A, Yuwen T, et al. Probing conformational dynamics in biomolecules via chemical exchange saturation transfer: a primer. J Biomol NMR, 2017, 67: 243-71. |
| [12] |
Palmer AG 3rd, Kroenke CD, Loria JP. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol, 2001, 339: 204-38. |
| [13] |
Korzhnev DM, Salvatella X, Vendruscolo M, et al. Low-populated folding intermediates of Fyn SH3 characterized by relaxation dispersion NMR. Nature, 2004, 430: 586-90. |
| [14] |
Palmer AG 3rd, Massi F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev, 2006, 106: 1700-19. |
| [15] |
Rangadurai A, Szymaski ES, Kimsey IJ, et al. Characterizing micro-to-millisecond chemical exchange in nucleic acids using off-resonance R(1rho) relaxation dispersion. Prog Nucl Magn Reson Spectrosc, 2019, 112-113: 55-102. |
| [16] |
Lange OF, Lakomek NA, Fares C, et al. Recognition dynamics up to microseconds revealed from an RDC-derived ubiquitin ensemble in solution. Science, 2008, 320: 1471-5. |
| [17] |
Niu X, Bruschweiler-Li L, Davulcu O, et al. Arginine kinase: joint crystallographic and NMR RDC analyses link substrate-associated motions to intrinsic flexibility. J Mol Biol, 2011, 405: 479-96. |
| [18] |
Clore GM, Iwahara J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev, 2009, 109: 4108-39. |
| [19] |
Zeeb M, Balbach J. Protein folding studied by real-time NMR spectroscopy. Methods, 2004, 34: 65-74. |
| [20] |
Bai Y, Englander JJ, Mayne L, et al. Thermodynamic parameters from hydrogen exchange measurements. Methods Enzymol, 1995, 259: 344-56. |
| [21] |
Arrington CB, Robertson AD. Kinetics and thermodynamics of conformational equilibria in native proteins by hydrogen exchange. Methods Enzymol, 2000, 323: 104-24. |
| [22] |
Jin Y, Cui Y, Yuwen T. NMR methods for investigating functionally relevant biomolecular dynamics. Magnetic Resonance Lett, 2025, 200195. |
| [23] |
Ma RS, Li QF, Wang AD, et al. Determination of pseudocontact shifts of low-populated excited states by NMR chemical exchange saturation transfer. Phys Chem Chem Phys, 2016, 18: 13794-8. |
| [24] |
Gao J, Liang E, Ma R, et al. Fluorine pseudocontact shifts used for characterizing the protein-ligand interaction mode in the limit of NMR intermediate exchange. Angew Chem Int Ed Engl, 2017, 56: 12982-6. |
| [25] |
Xu D, Li B, Gao J, et al. Ligand proton pseudocontact shifts determined from paramagnetic relaxation dispersion in the limit of NMR intermediate exchange. J Phys Chem Lett, 2018, 9: 3361-7. |
| [26] |
Wang D, Wu S, Wang D, et al. The importance of the compact disordered state in the fuzzy interactions between intrinsically disordered proteins. Chem Sci, 2022, 13: 2363-77. |
| [27] |
Liu Z, Zhang S, Gu J, et al. Hsp27 chaperones FUS phase separation under the modulation of stress-induced phosphorylation. Nat Struct Mol Biol, 2020, 27: 363-72. |
| [28] |
Li Y, Han J, Zhang Y, et al. Structural basis for activity regulation of MLL family methyltransferases. Nature, 2016, 530: 447-52. |
| [29] |
Li Y, Zhao L, Zhang Y, et al. Structural basis for product specificities of MLL family methyltransferases. Mol Cell, 2022, 82: 3810-25. |
| [30] |
Pan L, Fu TM, Zhao W, et al. Higher-order clustering of the transmembrane anchor of DR5 drives signaling. Cell, 2019, 176: 1477-89. |
| [31] |
Du G, Zhao L, Zheng Y, et al. Autoinhibitory structure of preligand association state implicates a new strategy to attain effective DR5 receptor activation. Cell Res, 2023, 33: 131-46. |
| [32] |
Yang W, Pan W, Chen S, et al. Dynamic regulation of CD28 conformation and signaling by charged lipids and ions. Nat Struct Mol Biol, 2017, 24: 1081-92. |
| [33] |
Li H, Yan C, Guo J, et al. Ionic protein-lipid interactions at the plasma membrane regulate the structure and function of immunoreceptors. Adv Immunol, 2019, 144: 65-85. |
| [34] |
Guo X, Yan C, Li H, et al. Lipid-dependent conformational dynamics underlie the functional versatility of T-cell receptor. Cell Res, 2017, 27: 505-25. |
| [35] |
Jiang Y, Dai A, Huang Y, et al. Ligand-induced ubiquitination unleashes LAG3 immune checkpoint function by hindering membrane sequestration of signaling motifs. Cell, 2025, 188: 2354-71. |
| [36] |
Pan B, Liu D, Yang L, et al. GPCR large-amplitude dynamics by 19F-NMR of aprepitant bound to the neurokinin 1 receptor. Proc Natl Acad Sci U S A, 2022, 119: e2122682119. |
| [37] |
Wang M, Song X, Chen J, et al. Intracellular environment can change protein conformational dynamics in cells through weak interactions. Sci Adv, 2023, 9: eadg9141. |
| [38] |
Song X, Wang M, Chen X, et al. Quantifying protein electrostatic interactions in cells by nuclear magnetic resonance spectroscopy. J Am Chem Soc, 2021, 143: 19606-13. |
| [39] |
Chen X, Zhang X, Chen J, et al. Quantification of CH and NH/pi-stacking interactions in cells using nuclear magnetic resonance spectroscopy. Anal Chem, 2024, 96: 14354-62. |
 2025, Vol. 37
2025, Vol. 37 

 刘志军,正高级工程师、中国生物物理学会生物磁共振分会委员、上海市浦东新区青年联合会第七届委员、中国科学院青年创新促进会会员、九三学社社员。博士毕业于中国科学技术大学,长期从事核磁共振谱仪的技术支撑和应用研究工作。先后参加了我国两个国家级的大科学工程(合肥强磁场设施、上海蛋白质设施)的子系统(核磁共振装置)建设、运行维护和科学研究工作。2012年开始,带领团队建立了包括900兆核磁谱仪在内的一整套先进的、完全开放共享的生物核磁分析系统,积累了一系列先进的生物核磁脉冲技术和实验方法。服务用户的同时,不断地挑战技术极限,帮助用户解决关键技术难题,相关的合作署名文章发表在Molecular Cell、Chemical Science和Nature、Science、Cell等顶尖学术期刊上
刘志军,正高级工程师、中国生物物理学会生物磁共振分会委员、上海市浦东新区青年联合会第七届委员、中国科学院青年创新促进会会员、九三学社社员。博士毕业于中国科学技术大学,长期从事核磁共振谱仪的技术支撑和应用研究工作。先后参加了我国两个国家级的大科学工程(合肥强磁场设施、上海蛋白质设施)的子系统(核磁共振装置)建设、运行维护和科学研究工作。2012年开始,带领团队建立了包括900兆核磁谱仪在内的一整套先进的、完全开放共享的生物核磁分析系统,积累了一系列先进的生物核磁脉冲技术和实验方法。服务用户的同时,不断地挑战技术极限,帮助用户解决关键技术难题,相关的合作署名文章发表在Molecular Cell、Chemical Science和Nature、Science、Cell等顶尖学术期刊上