(2 临港实验室,上海 200031)
(3 上海科技大学物质科学与技术学院,上海 201210)
(4 国科大杭州高等研究院药物与技术学院,杭州 310024)
(5 中国科学院大学,北京 100049)
(2 Lingang Laboratory, Shanghai 200031, China)
(3 School of Physical Science and Technology, ShanghaiTech University, Shanghai 201210, China)
(4 School of Pharmaceutical Science and Technology, Hangzhou Institute for Advanced Study, University of Chinese Academy of Sciences, Hangzhou 310024, China)
(5 University of Chinese Academy of Sciences, Beijing 100049, China)
冠状病毒是一类具有包膜的单链、正义RNA病毒,根据系统分类学可分为α、β、γ和δ四个属,其中β属冠状病毒对人类健康威胁最大[1]。在过去的二十多年时间里,人类社会共经历了三次由冠状病毒暴发引起的重大疫情,分别是2002‒2003年突然大规模暴发的严重急性呼吸综合征(severe acute respiratory syndrome, SARS,简称“非典”)[2]、2012年病死率高达35.3%的中东呼吸综合征(Middle East respiratory syndrome, MERS)[3],以及2019年末席卷全球的新冠病毒肺炎大流行(COVID-19)[4]。COVID-19在全球范围内肆虐,导致感染和死亡人数激增,对公共卫生体系和社会经济造成了前所未有的冲击。为有效应对疫情带来的挑战,全球医药企业和研究机构迅速致力于抗新冠病毒药物的研发[5]。作为冠状病毒复制转录的核心调控元件,3C样蛋白酶(3C-like protease, 3CLpro)因其高度保守性成为抗新冠病毒药物研发的核心靶点之一。
在靶向新冠病毒3CLpro的药物研发过程中,蛋白酶及其与抑制剂复合物的晶体结构解析发挥了关键作用,一如其对人类免疫缺陷病毒(human immunodeficiency virus, HIV)和丙型肝炎病毒(hepatitis C virus, HCV)蛋白酶抑制剂研发的重要贡献[6, 7]。新冠疫情暴发后,研究人员通过解析高分辨率晶体结构,精确揭示了新冠病毒3CLpro蛋白的催化位点及底物结合口袋的三维构象特征,为基于结构的虚拟筛选和理性药物设计提供了重要依据[8]。通过模拟底物与蛋白酶相互作用模式,采用醛基、α-酮酰胺等亲电弹头的首批3CLpro共价拟肽类抑制剂被成功设计与发现[9-11]。随着构效关系研究的不断深入和活性化合物优化策略的迭代,多个候选化合物进入临床研究阶段,其中奈玛特韦(nirmatrelvir)和恩赛特韦(ensitrelvir)已获多国药品监管部门批准上市[12-15]。值得一提的是,中国科学院上海药物研究所联合武汉病毒研究所、先声药业等多个单位,成功研制了我国首个靶向3CLpro的抗新冠病毒药物先诺欣®,成为国内首款获得常规批准的口服抗新冠病毒药物[16]。这些重要研究进展不仅再次验证了病毒蛋白酶抑制剂优异的抗病毒疗效,同时也彰显了蛋白质结构解析及基于结构的药物设计在抗病毒药物研发领域的重要技术优势。本文将系统介绍新冠病毒3CLpro的三维结构、底物识别机制和进化保守性,重点阐述3CLpro抑制剂的最新研发进展及典型案例分析,以期为靶向病毒蛋白酶的广谱抗病毒药物研发提供重要参考。
1 3CL蛋白酶简介 1.1 三维结构与功能3CLpro是冠状病毒基因组编码表达的一种半胱氨酸蛋白酶,在病毒复制过程中,与病毒编码的另外一种半胱氨酸蛋白酶——木瓜样蛋白酶(papain-like protease, PLpro)一起负责对前体多聚蛋白pp1a和pp1ab进行剪切,生成病毒复制所必需的非结构蛋白(non-structural proteins, nsps)。因3CLpro负责绝大部分位点的切割(14个剪切位点中的11个),故而被称为主蛋白酶(main protease, Mpro)[17]。3CLpro在冠状病毒生存周期中扮演重要角色,抑制其水解活性可有效抑制病毒复制,因而成为抗冠状病毒药物开发的核心靶点之一。
3CLpro的结构生物学研究为理解蛋白酶的功能和药物设计提供关键信息。已报道的晶体结构[8, 10]显示,3CLpro通常以二聚体的形式发挥水解活性功能。每个单体包含三个结构域,即分别由6个反平行的β折叠组成的结构域Ⅰ和Ⅱ,以及由5个α螺旋形成的球状结构域Ⅲ。其中,结构域Ⅲ通过一个长环(loop)与结构域Ⅱ相连。此外,N端存在一个独特的指状区(N-finger),在活性二聚体中,N-finger插入到另一单体的底物结合口袋,从而形成稳定而具有水解活性的二聚体(图 1)。3CLpro的底物结合口袋位于结构域Ⅰ和Ⅱ的裂隙中,这个狭长且敞开的口袋又可细分为两个较深(S1和S2)、两个较浅(S1′和S4)以及一个溶剂可及(S3)的亚位点,当然该口袋中还包含对蛋白酶水解活性至关重要且保守的元件——催化二联体(Cys145和His41)和氧阴离子孔穴(Gly143-Cys145)。在催化水解底物时,催化残基His41作为碱夺取残基Cys145巯基(-SH)上的质子使其活化形成亲核性硫醇盐离子(Cys145-S−),随后进攻底物P1-P1′间肽键的羰基碳生成共价硫酯中间体,接着在残基His41以及水分子的协助下肽键发生断裂并再生游离的催化残基Cys145-SH。氧阴离子孔穴则主要用于稳定反应中间体,确保蛋白酶催化反应的顺利进行[18]。值得注意的是,尽管不同冠状病毒3CLpro的序列一致性差异较大,但形成底物结合口袋的关键残基高度保守,尤其是S1和S1′亚位点[19, 20]。如此保守的底物结合口袋特征以及催化机制为靶向3CLpro研发广谱抗冠状病毒药物奠定基石。
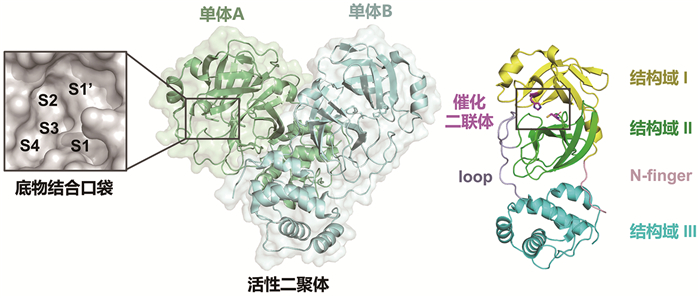
|
图 1 新冠病毒3CLpro的三维结构及其催化位点和底物结合口袋 |
根据Schechter-Berger命名规则,蛋白酶在对底物进行特异性识别与水解切割时,以底物切割位点为界,底物残基从N端到C端依次命名为“……、P2、P1 ↓ P1′、P2′、……”,对应的底物结合亚位点则为“……、S2、S1 ↓ S1′、S2′、……”。由于3CLpro在多聚蛋白上切割11个位点,释放非结构蛋白nsp4~nsp16,因此可以根据这11个切割序列探索其底物特异性,为抑制剂的设计与优化提供重要线索。底物序列分析结果表明,3CLpro对底物P1位谷氨酰胺(Gln)表现出最严格的特异性识别,其次为P2位点,主要为疏水残基亮氨酸(Leu)(图 2)。此外,P1′位也表现出较强的偏好性,多为体积较小的残基,如丝氨酸(Ser)和丙氨酸(Ala)。基于其他6种常见的人类冠状病毒(HCoV-NL63、HCoV-229E、MERS- CoV、SARS-CoV、HCoV-HKU1、HCoV-OC43)的3CLpro底物序列分析也得到类似的结果。简而言之,冠状病毒3CLpro底物特异性程度从高到低排序为P1 > P2 > P1′ > P4 > P3,这一特性提示设计抑制剂时,模拟P1、P2和P1′的保守残基特性可以显著增强抑制剂对蛋白酶的抑制效力和广谱性。
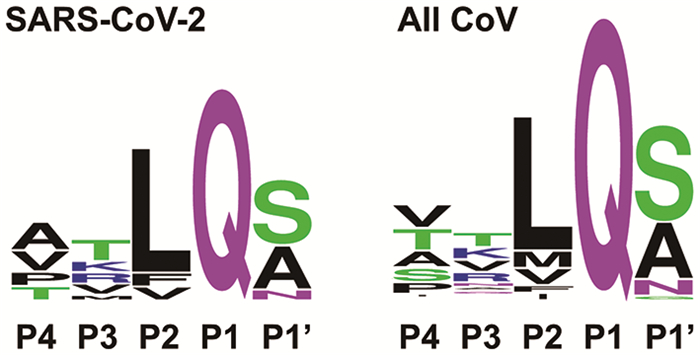
|
图 2 冠状病毒3CLpro底物识别位点的序列保守性 |
病毒进化驱动力源于突变发生速率与群体传播动态的协同作用。虽然遗传变异多呈现随机性,但自然选择通过筛选并保留适应性优势突变塑造进化轨迹。典型例证可见于新冠病毒刺突蛋白的D614G突变,该位点变异通过增强病毒颗粒与宿主细胞受体的结合效率,显著提升了病原体的传播适应性[21, 22]。作为典型的正链RNA病毒科成员,冠状病毒在遗传复制过程中展现出高频突变特性。持续存在的宿主-环境选择压力驱动其发生适应性进化,导致病毒谱系呈现显著表型分化,具体表现为传播优势增强(如ACE2受体结合能力提升)、宿主适应度优化(包括致病性调控)及免疫识别逃逸能力进化等重要生物学特性转变。在此进化框架下,全球监测系统已识别出多个具有显著流行病学影响的新冠病毒变异株(variants of concern, VOC),其持续涌现构成当前冠状病毒防控的核心挑战[21]。据报道,2020年底在英国发现并迅速传播的B.1.1.7毒株为首个VOC (即Alpha变异株)[23, 24]。在随后的几周内,南非和巴西报告了另外两个快速增长的VOC谱系——Beta (B.1.351)[25]和Gamma (P.1)[26]。Delta谱系(B.1.617.2)于2021年5月被确定为VOC,但在此前已在印度传播数月,它迅速取代了之前的VOC,导致全球病例急剧增加[27, 28]。之后,在南非和博茨瓦纳发现的Omicron谱系(BA.1~BA.5)引发了新一轮全球感染浪潮[21, 29]。尽管这些VOCs出现在世界不同地区,但它们共享一组突变(如N501Y、E484K和ΔH69/V70),表明可能存在趋同进化[21]。此外,值得注意的是,这些VOCs的突变大多发生在病毒刺突蛋白(Spike, S)上,相较之下3CLpro的自然突变率较低,目前已鉴定的突变主要有6个,包括G15S (C.37)、T21I (B.1.1.318)、L89F (B.1.2)、K90R (B.1.351)、P132H (B.1.1.529、BA.1、BA.2、BA.4、BA.5、XBB)和L205V (P.2)[30]。这些突变均远离3CLpro的底物结合口袋(图 3),因此对抑制剂活性影响较为微弱[16],这也凸显了3CLpro作为广谱抗冠状病毒药物靶点的优势。
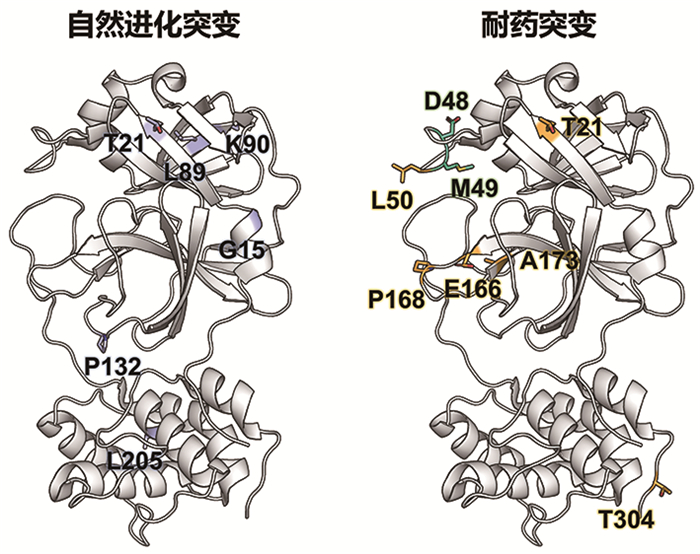
|
图 3 新冠病毒3CLpro的自然进化突变和耐药突变 |
尽管3CLpro在新冠病毒进化过程中表现出高度保守性,但是与HIV和HCV蛋白酶靶向药物应用后产生耐药突变类似,3CLpro抑制剂的广泛应用将引发正向选择压力,诱导产生多位点协同突变,从而影响药物疗效。在连续的细胞传代实验中,研究者发现在最早上市的共价拟肽类药物——奈玛特韦的选择压力下,病毒会通过逐步突变提高耐药性,例如先产生耐药性较弱的T21I、P252L和T304I单突变,随后逐步积累额外突变(如L50F、S144A、E166V和A173V等)以进一步提高耐药性,最终通过多突变协同效应实现对奈玛特韦的耐药[31, 32]。据报道,目前可能对奈玛特韦产生不同程度耐药的突变至少包括E166V、T21I/E166V、L50F/E166V、ΔP168+A173V和A173V/T304I[31-34]。相较于奈玛特韦,另一个获批上市的非共价、非拟肽3CLpro抑制剂——恩赛特韦则表现出不尽相同的耐药突变谱,既有共享耐药突变(如S144A和ΔP168),又有特有耐药突变(如D48Y和M49L)[33, 35],这表明不同结合模式的抑制剂可有效影响病毒的进化方向(图 3)。综上,尽管病毒在自然进化过程中并未产生对现有3CLpro抑制剂药物具有显著抗性的突变体[36],但药物选择压力下的研究结果和临床监测数据提示耐药突变体的出现,这些获得性变异往往通过两种机制实现耐药:其一,通过重构底物结合口袋空间构象来降低抑制剂结合亲和力;其二,通过增强病毒蛋白酶自身的催化活性维持其复制传播能力。此类适应性进化事件凸显冠状病毒耐药性发展的潜在风险,因此仍需开发具有全新结合模式的3CLpro抑制剂,为应对未来可能出现的新致病性和耐药性病毒提供候选分子。
2 靶向3CL蛋白酶的药物研发进展3CLpro作为冠状病毒复制必需且高度保守的靶点,其抑制剂研发自疫情暴发以来一直是全球抗新冠药物开发的焦点,并已取得了突破性进展。2021年美国辉瑞公司开发的奈玛特韦以及2022年日本盐野义公司开发的恩赛特韦相继获批,我国自主研发的首款3CLpro抑制剂先诺特韦(商品名先诺欣®)也于2023年获批上市,标志着国产抗新冠药物研发进入新阶段。此外,还有两款国产靶向3CLpro的药物(来瑞特韦和阿泰特韦)获批上市,同时多个3CLpro抑制剂(如FB2001和olgotrelvir等)处于临床研究阶段。这些突破性进展不仅验证了靶向3CLpro开展药物研发策略的高效性,同时建立了靶点结构解析、活性化合物筛选与优化到临床验证的全链条研发模式,推动抗病毒药物从应急响应模式向中长期战略储备的转变。
2.1 获批上市的3CL蛋白酶抑制剂新冠疫情暴发后,全球产学研界迅速启动针对新冠病毒3CLpro抑制剂研发的集中攻关。迄今,全球共有五款靶向3CLpro的抑制剂获批上市(图 4),本文将根据药物获批上市的时间顺序对这些药物进行系统梳理,重点解析奈玛特韦、恩赛特韦和先诺特韦等代表性品种的分子设计理念与优化路径。
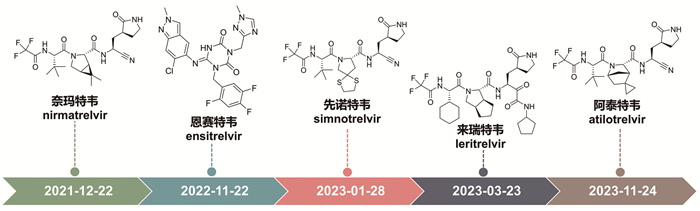
|
图 4 获批上市的新冠病毒3CLpro抑制剂 |
奈玛特韦(nirmatrelvir, PF-07321332)作为辉瑞公司研发的全球首个获FDA紧急使用授权(emergency use authorization, EUA)的SARS-CoV-2 3CLpro抑制剂,是抗新冠复方制剂Paxlovid®的核心活性成分,其成功研发确立了新发传染病应急响应背景下,基于蛋白酶结构的从靶点验证到临床转化的抗病毒药物快速研发范式。该药物分子的开发可追溯至2003年SARS期间,辉瑞公司针对SARS-CoV 3CLpro设计的共价拟肽类抑制剂PF-00835231。然而,SARS的快速消退使得PF-00835231的研发在取得初步进展后便被终止。新冠疫情暴发后,由于SARS-CoV-2和SARS-CoV两种冠状病毒3CLpro的底物结合口袋序列完全一致,研发团队重启了对PF-00835231的结构改造(图 5)。
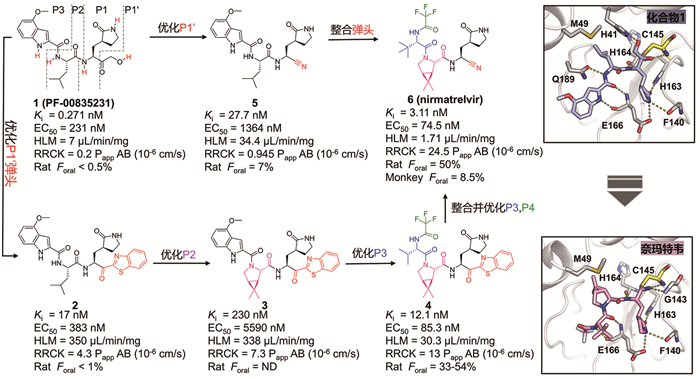
|
图 5 奈玛特韦的设计理念与优化路径 |
初步研究结果表明,PF-00835231对新冠病毒3CLpro表现出极高的亲和力(Ki = 0.271 nM),且具有良好的细胞抗病毒活性(EC50 = 231 nM)和肝微粒体代谢稳定性(HLM = 7 μL/min/mg),但细胞渗透性(Papp = 0.2×10-6 cm/s)和口服生物利用度较差(F < 0.5%)。与新冠病毒3CLpro的复合物晶体结构解析表明,PF-00835231 P1′位弹头的羰基与蛋白酶催化残基Cys145共价结合的同时还形成稳定氢键,而弹头上的羟基则与催化残基His41形成氢键。P1位γ-内酰胺基团通过多重氢键与His163、Glu166侧链以及Phe140主链产生相互作用,P1位主链NH则与His164主链形成氢键,从而将化合物锚定在蛋白酶活性口袋。P2位亮氨酸侧链嵌入S2亚位点,与His41、Met49和Gln189等残基形成疏水相互作用,主链NH则与Gln189侧链羰基形成氢键。P3位吲哚基团通过两根氢键与Glu166主链结合,并与Gln189-Ala191环进行疏水接触,进一步稳定其与蛋白酶的结合[37]。
为改善PF-00835231的口服吸收,同时维持其活性,研发团队对上述参与结合的关键基团进行了系统修饰,并利用计算模拟的方法进行辅助优化[12, 37]。最初,研究人员将P1′位羟甲基酮弹头替换成苯并噻唑酮,以解决渗透性和稳定性方面的挑战。结果显示,设计合成的化合物2相较于PF-00835231,不仅保持了强抑制活性(Ki = 17 nM),还显示出更优的被动渗透性(Papp, 4.3 VS 0.2),这主要归因于减少了一个氢键供体并引入了亲酯基团苯并噻唑酮。然而,该官能团的替换也显著降低了化合物的代谢稳定性(HLM, 350 VS 7 μL/min/mg),其口服生物利用度也并未得到改善。随后,研发人员探究了P3位主链肽键NH的氢键供体是否必要。通过对化合物2 P2位叔亮氨酸与P3主链肽键NH环化后产生的各种衍生物进行计算模拟,最终确定了以偕二甲基双环[3.1.0]脯氨酸为优势基团的化合物3。计算模拟显示,该优势基团与化合物2中的叔亮氨酸及内源性底物中的亮氨酸完全重叠。此外,化合物3的被动渗透性得到改善(Papp, 7.3×10-6 VS 4.3 ×10-6 cm/s),但细胞水平抗病毒活性和稳定性仍较差。进一步将P3位氢键供体吲哚环替换为缬氨酸,同时引入P4位三氟乙酰胺基团,得到化合物4。由于三氟甲基的吸电子效应,该化合物偶极矩降低,亲脂性增强,细胞水平抗病毒活性(EC50 = 85.3 nM)和渗透性(Papp = 13×10-6 cm/s)同步增强,大鼠体内口服生物利用度提升至33%~54%。然而,急性毒理学研究发现该分子具有增加炎症标志物和溶血的风险。回溯性研究发现,将PF-00835231弹头换为氰基(化合物5)会降低细胞活性,但口服吸收有所提高,提示氰基弹头在改善吸收、分布、代谢和排泄(ADME)性质方面的潜力。最终,通过整合氰基弹头与P3/P4位点优势基团,成功获得候选化合物奈玛特韦(6)。
奈玛特韦不仅表现出良好的抗病毒活性,还具备优异的细胞渗透性和药代特性,在大鼠和食蟹猴体内口服生物利用度分别达50%和8.5%。复合物晶体结构解析揭示P1′氰基与Cys145共价结合;P1位γ-内酰胺占据S1亚位点,与Phe140、His163、Glu166形成三根氢键;P2位适配S2亚位点,形成疏水相互作用;P3位叔丁基亮氨酸朝向溶剂区并使P4位三氟乙酰胺选择朝向S4亚位点的构象。然而,后续药代动力学研究发现奈玛特韦易被CYP3A4氧化代谢,导致血药浓度迅速下降。为此,辉瑞公司将其与CYP3A4抑制剂利托那韦联用,开发了全球首款靶向3CLpro的抗新冠病毒口服新药Paxlovid®。临床研究数据显示,与安慰剂相比,Paxlovid®大幅降低患者住院风险(89%)和死亡风险(88%)[38]。尽管利托那韦的联用改善了药效,但其对代谢酶的抑制活性也带来了潜在药物-药物相互作用风险,这也促进了后续非利托那韦联用的3CLpro抑制剂单药的开发。
恩赛特韦(ensitrelvir, S-217622)是日本盐野义公司开发的全球首款,也是目前唯一一款获批上市的非拟肽、非共价3CLpro抑制剂。区别于奈玛特韦的是,它在新冠疫情前并无任何研究基础。该分子仅用两年时间便完成了从早期筛选开始的所有临床前研究和临床试验,并先后通过日本监管机构审评——于2022年11月22日获得日本药品医疗器械管理局的紧急使用授权后,进一步在2024年3月5日取得正式上市批准[39]。恩赛特韦的快速研发是基于结构的药物设计(structure-based drug design, SBDD)策略在广谱抗病毒药物开发领域的一个经典案例,对以结构为指导的计算机辅助药物设计具有非常好的借鉴意义。
恩赛特韦的研发历程大致包含两个关键步骤,即基于分子对接的虚拟筛选和基于结构的化合物优化[13]。首先,研发团队通过对已报道的抑制剂与3CLpro底物口袋的结合模式进行系统分析,发现三种保守的抑制剂-蛋白酶相互作用模式:抑制剂与S1亚位点His163残基形成氢键、与S2亚位点形成疏水作用以及与Glu166残基主链NH形成氢键。随后,利用分子对接对超过十万个分子的内部化合物库进行虚拟筛选,挑选300个打分高且具有上述保守结合特征的化合物。进一步采用质谱分析对这300个化合物进行筛选,得到具有较高结合活性和良好药代性质的苗头化合物7。体外酶活抑制实验结果表明,化合物7抑制新冠病毒3CLpro的IC50值为8.6 μM。药代动力学研究发现,该化合物在人和大鼠肝微粒体(HLM和RLM)中的代谢稳定性分别为97.1%和70.8%,在大鼠体内的血浆清除率为7.3 mL/min/kg,口服生物利用度为111%。化合物7与新冠病毒3CLpro的复合物晶体结构显示该化合物呈现与对接模型相似的结合模式:中间三嗪部分通过与Gly143、Cys145和Glu166主链NH形成的三个氢键,以及通过水分子介导与His41形成的额外氢键,将化合物锚定在底物结合口袋中;甲基酰胺则伸向S1亚位点,羰基氧与His163侧链形成氢键,进一步稳定结合构象;3, 4, 5-三氟苯环则与翻转的His41侧链形成π-π相互作用,进而与S2亚位点进行疏水接触。值得注意的是,复合物晶体结构中化合物7的4-二氟甲氧基-2-甲苯片段伸向S1′亚位点,与Thr26主链形成氢键,而这一独特作用模式在对接构象中并未发现。
基于此,研发团队迅速对化合物7的P1′、P1和P2位官能团进行结构优化(图 6)。为保持与Thr26的独特氢键作用,首先将P1′片段环化成能更好匹配S1′亚位点的6-氯-2-甲基-2H-吲唑基团,所得化合物8不仅保持了良好的代谢稳定性,对3CLpro的抑制活性也提高约90倍。进一步将P1位酰胺环化,同时优化P2,最终得到候选分子恩赛特韦(9),其IC50为13 nM,EC50为370 nM。该分子具有非常卓越的药代属性,代谢稳定性高,血浆清除率低,并且在大鼠、狗和猴体内均具有显著优于拟肽类抑制剂奈玛特韦的口服生物利用度,因此无须与利托那韦联用。复合物晶体结构解析表明,其结合模式与改造前化合物7类似,关键相互作用均得到保持。进一步研究发现,恩赛特韦对新冠病毒Alpha、Beta、Gamma、Delta和Omicron毒株复制具有类似于早期分离株(WT)的抑制活性(EC50为0.29~0.50 μΜ),同时对SARS-CoV、MERS-CoV、HCoV-OC43和HCoV-229E四种人类冠状病毒均有较好抑制活性(EC50为0.07~5.50 μΜ),表明其具有广谱的抗冠状病毒效力。
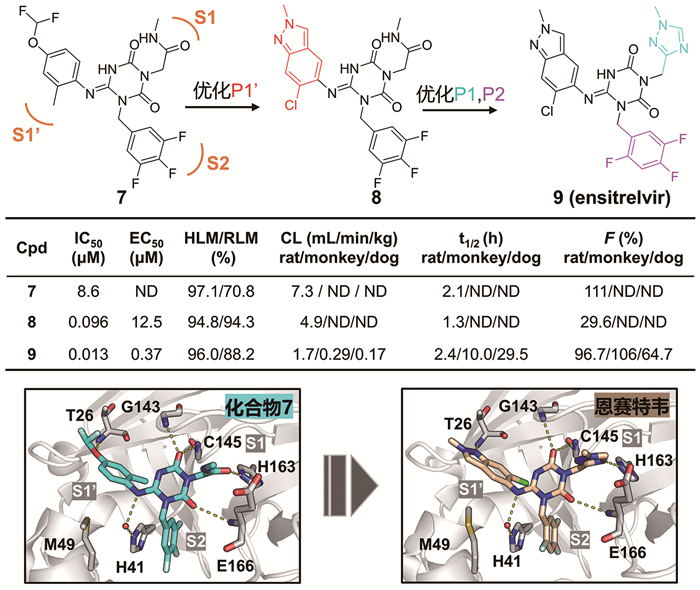
|
图 6 恩赛特韦的设计理念与优化路径 |
先诺特韦(simnotrelvir, SIM0417)是国产首款具有完全自主知识产权的靶向3CLpro口服抗新冠病毒药物,其临床前研发过程同样体现了复合物晶体结构解析以及基于结构的理性设计在活性化合物到候选药物优化过程中的重要性(图 7)[16]。
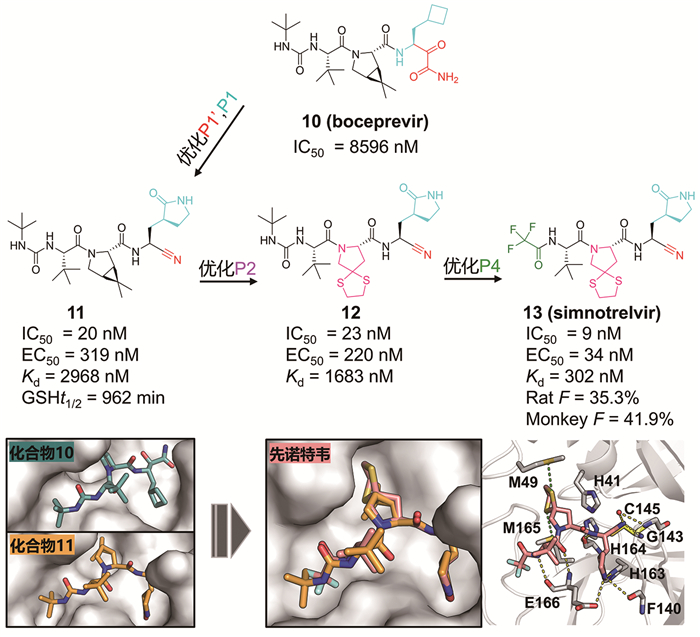
|
图 7 先诺特韦的设计理念与优化路径 |
疫情初期,研发团队发现上市药物博赛匹韦(boceprevir,丙肝病毒蛋白酶抑制剂)对新冠病毒3CLpro具有抑制活性,IC50为8 596 nM。进一步解析其与SARS-CoV-2 3CLpro的复合物晶体结构发现,其P1′位α-酮酰胺弹头与蛋白酶Cys145共价结合,但P1位完全疏水的环丁基仅部分占据由Ser144/His163/Glu166/His172侧链构成的S1亚位点,提示具有良好的改造空间。基于此,研发人员将P1位环丁基替换成广泛应用于3CLpro抑制剂中的γ-内酰胺基团,并选择体积较小、反应性更弱的氰基作为P1′弹头。所得化合物11对3CLpro的抑制活性显著提升429倍,同时具有良好的细胞抗病毒活性(EC50 = 319 nM)。此外,谷胱甘肽(GSH)反应性测试结果表明该弹头具有中等的内在反应性(GSHt1/2 = 962 min),提示其具有较低的脱靶风险。为进一步提高该分子的抗病毒活性,研发人员解析了该化合物与3CLpro的复合物晶体结构,发现该化合物P2位偕二甲基双环[3.1.0]脯氨酸基团未能充分占据S2亚位点,因而选择尺寸更大的疏水基团,如二硫戊烷螺环脯氨酸作为P2位官能团,得到化合物12,其EC50提高至220 nM。P3位叔亮氨酸面向溶剂区,与周围氨基酸无明显相互作用,因此不经修饰直接保留。此外,受奈玛特韦结构启发,将P4位替换成三氟乙酰基,最终得到候选化合物先诺特韦(13)。该化合物对3CLpro的IC50提高至个纳摩尔,细胞水平抗病毒活性也显著提高(EC50 = 34 nM)。复合物晶体结构解析发现,先诺特韦更短、更小的P4三氟乙酰基使其更易通过构象重排占据S4亚位点疏水区,而化合物12中较大的P4叔丁基因空间位阻只能朝向S4亚位点亲水区,这种结构差异显著增强了先诺特韦与蛋白酶的结合焓和结合自由能。此外,研发人员利用等温滴定量热(isothermal titration calorimetry, ITC)实验评估了奈玛特韦和先诺特韦与新冠病毒3CLpro C145G变体的非共价结合自由能,发现两者均由焓驱动结合,但先诺特韦的亲和力更高(Kd, 302 VS 620 nM)。由于两者在结构上的差异仅在P2位(奈玛特韦采用偕二甲基双环[3.1.0]脯氨酸基团,而先诺特韦则为二硫戊烷螺环脯氨酸),因此,先诺特韦亲和力的提高主要得益于其更大的P2基团带来的焓增益。该螺环基团通过结合诱导Met49附近区域发生构象重排,显著扩大了S2亚位点的结合腔体积,并与Met49和Met165建立了独特的硫-硫相互作用,进而与附近残基形成了更多的疏水相互作用,增强了先诺特韦与蛋白酶的结合亲和力。
进一步研究发现,先诺特韦具有良好的安全性,先诺特韦与利托那韦联用表现出了良好的口服药代属性,在大鼠和猴子体内的口服生物利用度分别为35.3%和41.9%。体内抗病毒药效实验显示,该组合能有效抑制转基因小鼠肺组织与脑组织的病毒复制,其病理损伤保护效果显著优于奈玛特韦对照组。Ⅰ期临床结果显示,先诺特韦在健康成人受试者体内表现出良好的安全性、耐受性和药代动力学特征;Ib期临床结果表明在治疗COVID-19时,先诺特韦联用利托那韦具有更好的疗效;Ⅱ/Ⅲ期临床结果显示早期服用先诺特韦联合利托那韦,可显著降低病毒载量,并有效缩短症状改善时间[40-42]。基于此,先诺特韦片/利托那韦片组合包装(商品名:先诺欣®)作为我国首款自主研发的靶向3CL蛋白酶抗新冠病毒新药于2023年附条件获批上市。然而,与奈玛特韦类似,与利托那韦的联用可能引发药物-药物相互作用风险。值得一提的是,先诺特韦不仅对早期毒株(WIVO4、Delta)有效,对后期的VOCs变体、当前流行的Omicron及其变异株(BA.1、JN.1等8种)以及包括SARS-CoV和MERS-CoV在内的其他六种常见人冠状病毒3CLpro均具有强效的抑制活性,对奈玛特韦耐药的3CLpro突变体(Y54A、H172Y、E166V以及T21I/S144A等)也表现出有效抑制,其中包括E166V关键耐药突变体[16, 43]。先诺特韦相较奈玛特韦对E166V突变体的抑制优势,可能是其P2位二硫戊烷螺环结构与蛋白酶突变后结合口袋的空间适应性、硫-硫相互作用及疏水锚定能力共同作用的结果。这一结构差异使先诺特韦能够更有效地应对由E166V突变引起的口袋构象和化学环境变化,而奈玛特韦因缺乏类似的结构特征,在相同突变体中表现出较弱的抑制活性。因此,先诺特韦的广谱抗病毒活性连同提升的耐药遗传屏障,为应对持续进化压力下的新冠病毒变异株防控提供了保障。
来瑞特韦(leritrelvir, RAY1216)是第二款获批上市的国产口服3CLpro抑制剂,由广州医科大学附属第一医院联合众生睿创公司等多家单位共同研发。其研发主要基于上市的HCV蛋白酶抑制剂——特拉匹韦,通过对P1、P2、P3和P4基团的多轮优化,最终得到候选化合物来瑞特韦,其共价弹头为α-酮酰胺,P1~P4位基团分别为γ-内酰胺、环戊基脯氨酸、环己基甘氨酸和三氟乙酰胺。临床前研究[44]表明,来瑞特韦对3CLpro的抑制活性与奈玛特韦相当,但该化合物属于“慢结合、慢解离”型抑制剂,而奈玛特韦则为“快结合、快解离”型抑制剂,其解离速度较奈玛特韦下降近91%,因此靶标停留时间(residence time)远远高于奈玛特韦(tres, 104 VS 9 min),提示具有长效作用。在细胞水平的抗病毒活性评价中,来瑞特韦对新冠病毒多种VOCs毒株以及奈玛特韦耐药3CLpro突变体L50F/E166V均具有很好的抑制活性,EC50均为纳摩尔级别。在小鼠模型中,来瑞特韦可有效提高小鼠生存率,显著降低小鼠肺部病毒滴度,减轻肺部病理损伤,且其安全性和有效性在临床试验中得到进一步验证[45, 46]。此外,药代动力学研究表明,来瑞特韦在多种动物模型体内的半衰期均高于奈玛特韦,尤其是在食蟹猴(t1/2, 14.9 VS 1.4 h)中,显示出优良的药代属性,使其能够实现单药给药。来瑞特韦的成功研发和快速上市,突破了3CLpro拟肽类抑制剂需要与利托那韦联用的局限,为新冠治疗提供了多样化和差异化的选择。
阿泰特韦(atilotrelvir, GST-HG171)是继来瑞特韦后广州医科大学附属第一医院联合广生中霖等单位开发的另外一款口服3CLpro抑制剂。研发团队根据奈玛特韦与新冠病毒3CLpro的结合模式,对奈玛特韦P1~P3基团进行设计与优化,经过大量构效关系研究,最终获得候选化合物阿泰特韦[47]。临床前的体外研究结果显示,该化合物对新冠病毒Beta、Delta以及Omicron等多种变体毒株展现出广谱抗病毒活性,且抗病毒活性优于奈玛特韦5~10倍。阿泰特韦与利托那韦联用的体内药效研究表明,其在减少小鼠肺部组织病毒载量上优于奈玛特韦,并且在大鼠体内具有更显著的肺部组织分布偏好,肺部和血浆中的血药暴露较奈玛特韦高4~5倍[47]。临床研究结果显示,阿泰特韦与利托那韦联用可显著缩短患者临床症状的持续时间,且无明显安全性问题,该疗法在感染XBB毒株的试验中也展现了一致的有效性和安全性[48]。
2.2 临床在研的3CL蛋白酶抑制剂在以奈玛特韦、恩赛特韦为代表的口服抗新冠药物获批上市前,国内外针对3CLpro抑制剂的研究主要聚焦于拟肽类抑制剂的开发,但受限于该类分子固有的药代动力学缺陷,多数候选药物需与利托那韦联用,抑或选用不同给药方式,以提高药物疗效。为此,多个研发团队通过结构导向的分子设计策略,开发具有优良药代属性的单药制剂。此外,随着第一代3CLpro抑制剂的临床应用,新冠病毒逐渐进化产生的耐药突变,促使研究人员加速开发具有广谱抗突变活性的新一代抑制剂。目前,超过20个具有结构或作用模式差异化的在研药物进入临床研究阶段,其中五款处于临床研究后期阶段。以下对已公布化学结构的临床候选药物进行系统介绍(图 8)。
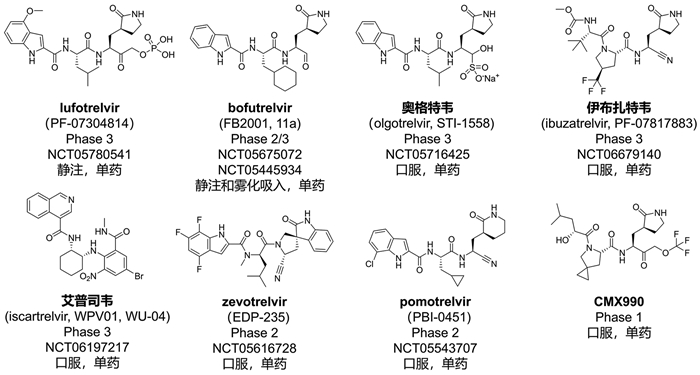
|
图 8 临床在研的新冠病毒3CLpro抑制剂 |
lufotrelvir (PF-07304814)是辉瑞公司开发的另外一款3CLpro抑制剂,它是PF-00835231的前药,静脉给药后,可在体内快速转为活性母体,抑制3CLpro活性,进而阻断病毒复制[49]。该化合物最后进入临床3期试验(NCT05780541),但由于静脉给药的繁琐以及疗效不及口服制剂,辉瑞公司终止了该分子的临床开发。
bofutrelvir (FB2001, 11a)是上海药物研究所、武汉病毒研究所联合前沿生物共同开发的一款注射、雾化吸入双剂型3CLpro抑制剂,分别用于治疗新冠病毒感染的住院患者和普通人群。疫情暴发初期,研发团队基于前期积累的化合物开展抗病毒活性筛选和复合物晶体结构解析,快速确定候选化合物[10]。目前该化合物已进入临床2/3期试验(NCT05675072、NCT05675072)。
奥格特韦(olgotrelvir, STI-1558)是深圳市第三人民医院联合艾森药业开发的双靶点口服抗新冠药物,对3CLpro和人体组织蛋白酶L (cathepsin L, CTSL)具有双重抑制作用,目前已进入临床3期试验(NCT05716425)。该化合物为AC1115的前药,其活性形式AC1115与FB2001结构极为相似,仅P2基团存在差异。研究表明[50],奥格特韦强效抑制3CLpro和CTSL活性,同时对新冠病毒及多种VOCs具有抑制活性,并且能有效抑制奈玛特韦耐药的3CLpro突变体(E166V),因此具有良好的广谱抗病毒活性。此外,药代动力学研究结果表明,奥格特韦在无须联用利托那韦的情况下,于动物模型和人体内均展现出显著提升的口服生物利用度及可观的血浆暴露水平。
伊布扎特韦(ibuzatrelvir, PF-07817883)是辉瑞公司基于奈玛特韦开发的第二代口服3CLpro抑制剂,目前已进入临床3期试验(NCT06679140)。为改善奈玛特韦的代谢稳定性,同时不影响化合物被动渗透性和细胞内抗病毒活性,研发团队对奈玛特韦P2及P4位点基团进行优化设计,最终获得该候选分子。与奈玛特韦相比,伊布扎特韦在人体内的口服药代动力学特性得到改善,因此可以进行口服单药给药,从而降低药物-药物相互作用风险[51]。
艾普司韦(iscartrelvir, WPV01, WU-04)是西湖大学研发的一款具有全新骨架的非拟肽、非共价3CLpro抑制剂,目前已完成临床3期试验(NCT06197217)。该候选分子源自DNA编码化合物库(DNA encoded compound Library, DEL)筛选得到的活性分子[52]。临床前研究显示,艾普司韦在多种人源细胞系中强效抑制新冠病毒及其变异株复制,EC50在10 nM范围内。此外,艾普司韦对SARS-CoV和MERS-CoV也有强效抑制活性,表明其具有广谱抗病毒活性。小鼠体内药效研究发现,艾普司韦单药口服给药具有与Paxlovid®相当的抗病毒疗效,因而被认为是具有潜力的抗新冠临床候选药物[53]。
zevotrelvir (EDP-235)是美国Enanta制药公司开发的一款口服3CLpro抑制剂,目前已进入临床2期试验(NCT05616728)。据报道[54],该分子对迄今为止的所有新冠病毒毒株以及临床相关的人类和人畜共患冠状病毒具有纳摩尔级的抑制活性,且对奈玛特韦耐药相关突变体具有与奈玛特韦相当或更优的抑制活性。单药口服给药可有效抑制叙利亚仓鼠体内新冠病毒的复制;预防性给药时,能阻断病毒在雪貂间的传播。这些结果表明zevotrelvir是一种广谱抗冠状病毒分子,可有效防控冠状病毒的原发性感染与传播。
pomotrelvir (PBI-0451)是美国Pardes Biosciences公司开发的一款口服3CLpro抑制剂,目前已进入临床2期试验(NCT05543707)。其具体研发历程未公布,但已有报道表明该化合物对多种冠状病毒具有强效抑制作用,包括新冠病毒和多种VOCs(如Delta和部分Omicron谱系),以及其他具有大流行潜力的人类冠状病毒(SARS-CoV和MERS-CoV)和普通感冒相关人类冠状病毒(HCoV-OC43和HCoV-229E)[55]。
CMX990是Scripps研究所研发的一款口服3CLpro抑制剂,目前已进入临床1期试验。该分子是在PF-00835231基础上,通过大量构效关系研究而得[56]。该化合物采用全新的三氟甲氧基甲基酮共价弹头,P2和P3位基团分别为4-螺环丙基脯氨酸和α-羟基戊基。研究结果表明,CMX990对3CLpro具有与奈玛特韦相当的抑制活性,但口服生物利用度并未改善。由于具有更高的体外清除率(肝微粒体清除率提高4倍,肝细胞清除率提高10倍),该化合物能够单药给药,从而降低药物-药物相互作用风险。
3 总结与展望本文系统阐述了新冠病毒3CLpro的生物学特性,包括其活性口袋三维结构特征、底物识别位点序列的保守性,以及该靶点在病毒适应性进化过程中产生的耐药突变谱系,为开发广谱抗冠状病毒的3CLpro抑制剂提供理论依据。在此基础上,文章针对该靶点药物研发的最新进展,重点剖析蛋白质晶体结构解析与基于结构的药物设计在代表性药物奈玛特韦、恩赛特韦和先诺特韦等研发过程中的关键作用。尽管目前已有多款3CLpro抑制剂获批上市,但以奈玛特韦为代表的第一代药物仍面临两大挑战:(1)与利托那韦联用带来的潜在药物-药物相互作用风险;(2)药物选择压力下病毒进化逐步积累的耐药突变。这提示新一代3CLpro抑制剂研发须同步优化药代属性与抗耐药屏障。近期获批的来瑞特韦及处于临床阶段的奥格特韦等拟肽类候选药物,通过分子骨架优化显著改善了口服生物利用度及代谢稳定性,实现无须利托那韦联用而单药给药。同时,3CLpro抑制剂研发已逐渐突破模拟底物的拟肽类抑制剂设计理念,通过整合DNA编码化合物库筛选、基于深度学习的虚拟筛选和共价抑制剂理性设计等多种技术手段,开发大环化合物[57, 58]及非拟肽抑制剂[59, 60]等新型分子实体,从而实现抑制剂骨架及其与3CLpro结合模式的多样化,以应对病毒持续进化导致的逃逸突变累积和潜在的耐药突变。
值得关注的是,除了流行的新冠病毒变异株和耐药突变株外,新发跨物种传播冠状病毒(如PDCoV、CCoV-HuPn-2018和MRCoV)的频现[61-63],对现有3CLpro抑制剂的“广谱”程度提出严峻挑战。研究发现,部分针对SARS-CoV-2 3CLpro开发的抑制剂对上述跨物种毒株的抑制活性呈现数量级下降,甚至丧失活性[64, 65]。这提示后疫情时代亟须建立更具前瞻性的研发策略,通过整合耐药及跨物种冠状病毒3CLpro的结构数据、3CLpro与抑制剂构效关系的系统研究以及大规模临床用药监测,构建包含快速药代动力学优化、耐药突变实时追踪及超广谱抑制剂设计的战略储备体系,从而在新型人畜共患病冠状病毒暴发时,迅速获得具有临床转化潜力的候选分子,为全球公共卫生安全构筑长效防线。
| [1] |
Hu B, Guo H, Zhou P, et al. Characteristics of SARS-CoV-2 and COVID-19. Nat Rev Microbiol, 2021, 19: 141-54. |
| [2] |
Peiris JS, Lai ST, Poon LL, et al. Coronavirus as a possible cause of severe acute respiratory syndrome. Lancet, 2003, 361: 1319-25. |
| [3] |
Zaki AM, Van Boheemen S, Bestebroer TM, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med, 2012, 367: 1814-20. |
| [4] |
Zhou P, Yang XL, Wang XG, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature, 2020, 579: 270-3. |
| [5] |
Li G, Hilgenfeld R, Whitley R, et al. Therapeutic strategies for COVID-19: progress and lessons learned. Nat Rev Drug Discov, 2023, 22: 449-75. |
| [6] |
De Clercq E, Li G. Approved antiviral drugs over the past 50 years. Clin Microbiol Rev, 2016, 29: 695-747. |
| [7] |
Ghosh AK, Osswald HL, Prato G. Recent progress in the development of HIV-1 protease inhibitors for the treatment of HIV/AIDS. J Med Chem, 2016, 59: 5172-208. |
| [8] |
Jin Z, Du X, Xu Y, et al. Structure of Mpro from SARS-CoV-2 and discovery of its inhibitors. Nature, 2020, 582: 289-93. |
| [9] |
Dai W, Zhang B, Jiang XM, et al. Structure-based design of antiviral drug candidates targeting the SARS-CoV-2 main protease. Science, 2020, 368: 1331-5. |
| [10] |
Zhang L, Lin D, Sun X, et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. Science, 2020, 368: 409-12. |
| [11] |
Qiao J, Li YS, Zeng R, et al. SARS-CoV-2 Mpro inhibitors with antiviral activity in a transgenic mouse model. Science, 2021, 371: 1374-8. |
| [12] |
Owen DR, Allerton CMN, Anderson AS, et al. An oral SARS-CoV-2 Mpro inhibitor clinical candidate for the treatment of COVID-19. Science, 2021, 374: 1586-93. |
| [13] |
Unoh Y, Uehara S, Nakahara K, et al. Discovery of S-217622, a noncovalent oral SARS-CoV-2 3CL protease inhibitor clinical candidate for treating COVID-19. J Med Chem, 2022, 65: 6499-512. |
| [14] |
Ma C, Xia Z, Sacco MD, et al. Discovery of di- and trihaloacetamides as covalent SARS-CoV-2 main protease inhibitors with high target specificity. J Am Chem Soc, 2021, 143: 20697-709. |
| [15] |
Quan BX, Shuai H, Xia AJ, et al. An orally available Mpro inhibitor is effective against wild-type SARS-CoV-2 and variants including Omicron. Nat Microbiol, 2022, 7: 716-25. |
| [16] |
Jiang X, Su H, Shang W, et al. Structure-based development and preclinical evaluation of the SARS-CoV-2 3C-like protease inhibitor simnotrelvir. Nat Commun, 2023, 14: 6463. |
| [17] |
V′kovski P, Kratzel A, Steiner S, et al. Coronavirus biology and replication: implications for SARS-CoV-2. Nat Rev Microbiol, 2021, 19: 155-70. |
| [18] |
Ramos-Guzmán CA, Ruiz-Pernía JJ, Tuñón I. Unraveling the SARS-CoV-2 main protease mechanism using multiscale methods. ACS Catal, 2020, 10: 12544-54. DOI:10.1021/acscatal.0c03420 |
| [19] |
Wang F, Chen C, Wang Z, et al. The structure of the porcine deltacoronavirus main protease reveals a conserved target for the design of antivirals. Viruses, 2022, 14: 486. |
| [20] |
Xiong M, Su H, Zhao W, et al. What coronavirus 3C-like protease tells us: from structure, substrate selectivity, to inhibitor design. Med Res Rev, 2021, 41: 1965-98. |
| [21] |
Markov PV, Ghafari M, Beer M, et al. The evolution of SARS-CoV-2. Nat Rev Microbiol, 2023, 21: 361-79. |
| [22] |
Volz E, Hill V, Mccrone JT, et al. Evaluating the effects of SARS-CoV-2 spike mutation D614G on transmissibility and pathogenicity. Cell, 2021, 184: 64-75. |
| [23] |
Davies NG, Abbott S, Barnard RC, et al. Estimated transmissibility and impact of SARS-CoV-2 lineage B.1.1.7 in England. Science, 2021, 372: eabg3055. |
| [24] |
Konings F, Perkins MD, Kuhn JH, et al. SARS-CoV-2 variants of interest and concern naming scheme conducive for global discourse. Nat Microbiol, 2021, 6: 821-3. |
| [25] |
Tegally H, Wilkinson E, Giovanetti M, et al. Detection of a SARS-CoV-2 variant of concern in South Africa. Nature, 2021, 592: 438-43. |
| [26] |
Faria NR, Mellan TA, Whittaker C, et al. Genomics and epidemiology of the P.1 SARS-CoV-2 lineage in Manaus, Brazil. Science, 2021, 372: 815-21. |
| [27] |
Dhar MS, Marwal R, Vs R, et al. Genomic characterization and epidemiology of an emerging SARS-CoV-2 variant in Delhi, India. Science, 2021, 374: 995-9. |
| [28] |
Bolze A, Luo S, White S, et al. SARS-CoV-2 variant Delta rapidly displaced variant Alpha in the United States and led to higher viral loads. Cell Rep Med, 2022, 3: 100564. |
| [29] |
Viana R, Moyo S, Amoako DG, et al. Rapid epidemic expansion of the SARS-CoV-2 Omicron variant in southern Africa. Nature, 2022, 603: 679-86. |
| [30] |
Ullrich S, Ekanayake KB, Otting G, et al. Main protease mutants of SARS-CoV-2 variants remain susceptible to nirmatrelvir. Bioorg Med Chem Lett, 2022, 62: 128629. |
| [31] |
Iketani S, Mohri H, Culbertson B, et al. Multiple pathways for SARS-CoV-2 resistance to nirmatrelvir. Nature, 2023, 613: 558-64. |
| [32] |
Zhu Y, Yurgelonis I, Noell S, et al. In vitro selection and analysis of SARS-CoV-2 nirmatrelvir resistance mutations contributing to clinical virus resistance surveillance. Sci Adv, 2024, 10: eadl4013. |
| [33] |
Moghadasi SA, Heilmann E, Khalil AM, et al. Transmissible SARS-CoV-2 variants with resistance to clinical protease inhibitors. Sci Adv, 2023, 9: eade8778. |
| [34] |
Hu Y, Lewandowski EM, Tan H, et al. Naturally occurring mutations of SARS-CoV-2 main protease confer drug resistance to nirmatrelvir. ACS Cent Sci, 2023, 9: 1658-69. |
| [35] |
Kiso M, Yamayoshi S, Iida S, et al. In vitro and in vivo characterization of SARS-CoV-2 resistance to ensitrelvir. Nat Commun, 2023, 14: 4231. |
| [36] |
Uehara T, Yotsuyanagi H, Ohmagari N, et al. Ensitrelvir treatment-emergent amino acid substitutions in SARS-CoV-2 3CLpro detected in the SCORPIO-SR phase 3 trial. Antiviral Res, 2025, 236: 106097. |
| [37] |
Tuttle JB, Allais C, Allerton CMN, et al. Discovery of nirmatrelvir (PF-07321332): a potent, orally active inhibitor of the severe acute respiratory syndrome coronavirus 2 (SARS CoV-2) main protease. J Med Chem, 2025, 68: 7003-30. |
| [38] |
Hammond J, Leister-Tebbe H, Gardner A, et al. Oral nirmatrelvir for high-risk, nonhospitalized adults with Covid-19. N Engl J Med, 2022, 386: 1397-408. |
| [39] |
Syed YY. Ensitrelvir fumaric acid: first approval. Drugs, 2024, 84: 721-8. |
| [40] |
Yang XM, Yang Y, Yao BF, et al. A first-in-human phase 1 study of simnotrelvir, a 3CL-like protease inhibitor for treatment of COVID-19, in healthy adult subjects. Eur J Pharm Sci, 2023, 191: 106598. |
| [41] |
Wang F, Xiao W, Tang Y, et al. Efficacy and safety of SIM0417 (SSD8432) plus ritonavir for COVID-19 treatment: a randomised, double-blind, placebo-controlled, phase 1b trial. Lancet Reg Health West Pac, 2023, 38: 100835. |
| [42] |
Cao B, Wang Y, Lu H, et al. Oral simnotrelvir for adult patients with mild-to-moderate Covid-19. N Engl J Med, 2024, 390: 230-41. |
| [43] |
Zhao L, Li C, Wang M, et al. Potent antiviral activity of simnotrelvir against key epidemic SARS-CoV-2 variants with a high resistance barrier. Antimicrob Agents Chemother, 2025, 69: e0155624. |
| [44] |
Chen X, Huang X, Ma Q, et al. Preclinical evaluation of the SARS-CoV-2 Mpro inhibitor RAY1216 shows improved pharmacokinetics compared with nirmatrelvir. Nat Microbiol, 2024, 9: 1075-88. |
| [45] |
Wang B, Li HJ, Cai MM, et al. Antiviral efficacy of RAY1216 monotherapy and combination therapy with ritonavir in patients with COVID-19: a phase 2, single centre, randomised, double-blind, placebo-controlled trial. EClinicalMedicine, 2023, 63: 102189. |
| [46] |
Zhan Y, Lin Z, Liang J, et al. Leritrelvir for the treatment of mild or moderate COVID-19 without co-administered ritonavir: a multicentre randomised, double-blind, placebo-controlled phase 3 trial. EClinicalMedicine, 2024, 67: 102359. |
| [47] |
Zhang G, Mao J, He H, et al. Discovery of GST-HG171, a potent and selective oral 3CL protease inhibitor for the treatment of COVID-19. SM J Infect Dis, 2023, 6: 1-9. |
| [48] |
Lu H, Zhang G, Mao J, et al. Efficacy and safety of GST-HG171 in adult patients with mild to moderate COVID-19: a randomised, double-blind, placebo-controlled phase 2/3 trial. EClinicalMedicine, 2024, 71: 102582. |
| [49] |
Cheruvu N, Van Duijn E, Spigt PA, et al. The metabolism of lufotrelvir, a prodrug investigated for the treatment of SARS-COV2 in humans following intravenous administration. Drug Metab Dispos, 2023, 51: 1419-27. |
| [50] |
Mao L, Shaabani N, Zhang X, et al. Olgotrelvir, a dual inhibitor of SARS-CoV-2 Mpro and cathepsin L, as a standalone antiviral oral intervention candidate for COVID-19. Med, 2024, 5: 42-61. |
| [51] |
Allerton CMN, Arcari JT, Aschenbrenner LM, et al. A second-generation oral SARS-CoV-2 main protease inhibitor clinical candidate for the treatment of COVID-19. J Med Chem, 2024, 67: 13550-71. |
| [52] |
Goodnow RA Jr, Dumelin CE, Keefe AD. DNA-encoded chemistry: enabling the deeper sampling of chemical space. Nat Rev Drug Discov, 2017, 16: 131-47. |
| [53] |
Hou N, Shuai L, Zhang L, et al. Development of Highly Potent Noncovalent Inhibitors of SARS-CoV-2 3CLpro. ACS Cent Sci, 2023, 9: 217-27. |
| [54] |
Rhodin MHJ, Reyes AC, Balakrishnan A, et al. The small molecule inhibitor of SARS-CoV-2 3CLpro EDP-235 prevents viral replication and transmission in vivo. Nat Commun, 2024, 15: 6503. |
| [55] |
Toussi SS, Hammond JL, Gerstenberger BS, et al. Therapeutics for COVID-19. Nat Microbiol, 2023, 8: 771-86. |
| [56] |
Dayan Elshan NGR, Wolff KC, Riva L, et al. Discovery of CMX990: a potent SARS-CoV-2 3CL protease inhibitor bearing a novel warhead. J Med Chem, 2024, 67: 2369-78. |
| [57] |
Wang X, Gotchev D, Fan KY, et al. Rational design of macrocyclic noncovalent inhibitors of SARS-CoV-2 Mpro from a DNA-encoded chemical library screening hit that demonstrate potent inhibition against pan-coronavirus homologues and nirmatrelvir-resistant variants. J Med Chem, 2024, 67: 19623-67. DOI:10.1021/acs.jmedchem.4c02009 |
| [58] |
Breidenbach J, Voget R, Si Y, et al. Macrocyclic azapeptide nitriles: structure-based discovery of potent SARS-CoV-2 main protease inhibitors as antiviral drugs. J Med Chem, 2024, 67: 8757-90. DOI:10.1021/acs.jmedchem.4c00053 |
| [59] |
Boby ML, Fearon D, Ferla M, et al. Open science discovery of potent noncovalent SARS-CoV-2 main protease inhibitors. Science, 2023, 382: eabo7201. |
| [60] |
Oneto A, Hamwi GA, Schäkel L, et al. Nonpeptidic irreversible inhibitors of SARS-CoV-2 main protease with potent antiviral activity. J Med Chem, 2024, 67: 14986-5011. |
| [61] |
Vlasova AN, Diaz A, Damtie D, et al. Novel canine coronavirus isolated from a hospitalized patient with pneumonia in East Malaysia. Clin Infect Dis, 2022, 74: 446-54. |
| [62] |
Lednicky JA, Tagliamonte MS, White SK, et al. Independent infections of porcine deltacoronavirus among Haitian children. Nature, 2021, 600: 133-7. |
| [63] |
Wang N, Ji W, Jiao H, et al. A MERS-CoV-like mink coronavirus uses ACE2 as entry receptor. Nature, 2025. DOI:10.1038/s41586-025-09007-w |
| [64] |
Yang Y, Cao L, Yan M, et al. Synthesis of deuterated S-217622 (Ensitrelvir) with antiviral activity against coronaviruses including SARS-CoV-2. Antiviral Res, 2023, 213: 105586. |
| [65] |
Jiang Z, Feng B, Chen L, et al. Discovery of novel nonpeptidic and noncovalent small molecule 3CLpro inhibitors as anti-SARS-CoV-2 drug candidate. J Med Chem, 2024, 67: 12760-83. |
 2025, Vol. 37
2025, Vol. 37 

 许叶春,中国科学院上海药物研究所研究员。2004年毕业于中国科学院上海药物研究所药物设计专业,获得理学博士学位;2005?2009年在以色列魏兹曼科学研究所从事博士后研究。2009年底通过中国科学院“百人计划”回到上海药物研究所开展独立工作,聚焦基于结构的理性药物设计与抗炎、抗病毒新药发现研究。作为主要发明人之一合作研发了已获批上市的抗新冠病毒创新药物“先诺欣”、进入临床研究的抗新冠病毒候选新药FB2001和抗银屑病候选新药KYHY2302,在Science、Nature、Nat Commun、PNAS、JACS、Angew Chem等期刊发表论文165篇,获得授权的中国及国际专利26项。主持了国家重点研发计划项目课题、国家自然科学基金优秀青年科学基金及国际合作项目、中国科学院先导专项子任务等。入选国家百千万人才工程“有突出贡献中青年专家”、国务院政府特殊津贴专家、国家“万人计划”科技创新领军人才等;曾荣获中国青年女科学家奖、树兰医学青年奖等
许叶春,中国科学院上海药物研究所研究员。2004年毕业于中国科学院上海药物研究所药物设计专业,获得理学博士学位;2005?2009年在以色列魏兹曼科学研究所从事博士后研究。2009年底通过中国科学院“百人计划”回到上海药物研究所开展独立工作,聚焦基于结构的理性药物设计与抗炎、抗病毒新药发现研究。作为主要发明人之一合作研发了已获批上市的抗新冠病毒创新药物“先诺欣”、进入临床研究的抗新冠病毒候选新药FB2001和抗银屑病候选新药KYHY2302,在Science、Nature、Nat Commun、PNAS、JACS、Angew Chem等期刊发表论文165篇,获得授权的中国及国际专利26项。主持了国家重点研发计划项目课题、国家自然科学基金优秀青年科学基金及国际合作项目、中国科学院先导专项子任务等。入选国家百千万人才工程“有突出贡献中青年专家”、国务院政府特殊津贴专家、国家“万人计划”科技创新领军人才等;曾荣获中国青年女科学家奖、树兰医学青年奖等