光合作用起源于细菌,通过基因在不同物种之间的水平转移而出现在藻类和高等植物中[1]。经过长期的演化,以保持高效能量转化为前提,各类光合生物呈现出多种多样的光系统[1-3]。植物、藻类和蓝细菌等放氧光合生物含有类囊体,利用光系统Ⅰ (photosystem Ⅰ, PSⅠ)和光系统Ⅱ (photosystem Ⅱ, PSⅡ)结合的叶绿素(chlorophyll, Chl)吸收可见光,裂解电子供体水而释放出O2,并通过碳同化(assimilation)过程将CO2转变为有机物[2]。
与此不同的是,不放氧光合细菌(anoxygenic phototrophic bacteria, APB)没有类囊体,利用质膜上捕光(light harvesting, LH)天线结合的细菌叶绿素(bacteriochlorophyll, BChl)吸收光能,并以H2S、有机物、氨等作为供氢体和碳源进行不放氧的光合作用[3]。捕光系统吸收的光能传递到反应中心(reaction center, RC),激发特殊细菌叶绿素对(special pair, P)而引起电荷分离,此光电转化过程被称为原初反应。原初反应释放的电子沿着质膜上分布的色素-蛋白质复合物进行传递,此过程中所产生的跨膜质子动力势驱动ATP的合成和NADP+的还原。光反应阶段产生的ATP和NAD(P)H进一步用于碳反应,通过碳固定(fixation)或同化将CO2、H2S等无机物转化为有机物(图 1)。
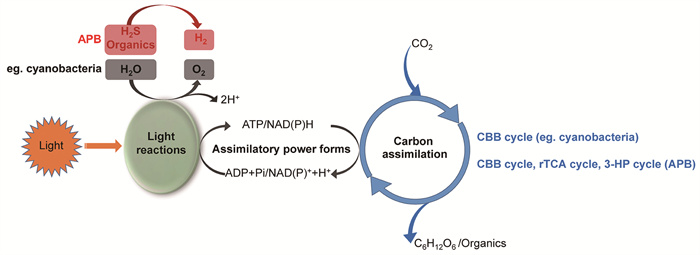
|
APB:不放氧光合细菌(anoxygenic phototrophic bacteria, APB);CBB cycle:卡尔文-本森-巴萨姆(Calvin-Benson-Bassham, CBB)循环;3-HP cycle:3-羟基丙酸(3-hydroxypropionate, 3-HP)循环;rTCA cycle:还原性三羧酸(reductive TCA, rTCA)循环。 图 1 放氧蓝细菌和APB的光合作用示意图 |
根据所含光合色素和电子供体的不同,APB主要分为紫细菌(purple bacteria)、绿硫细菌(green sulfur bacteria)、绿色非硫细菌(green non-sulfur bacteria)和太阳杆菌(heliobacteria)等[4]。紫细菌可进一步分为紫色硫细菌和紫色非硫细菌。其中,绿硫细菌和紫色硫细菌以H2S为电子供体,绿色非硫细菌利用包括氨基酸在内的多种有机酸,而紫色非硫细菌则以有机分子作为电子供体。各种APB所含有的光合色素种类和生长代谢方式也略有不同(表 1)[5-13]。在自身同化代谢过程中,APB可完成产氢、固氮、分解有机物三个自然界物质循环中极为重要的化学过程。这些独特的生理特性使它们在生态系统中占有非常重要的地位,并在环境治理、水产养殖、氢能利用和新能源开发方面具有广阔的应用前景[14-17]。
| 表 1 放氧蓝细菌和APB的区别 |
近年来,随着学科交叉和合成生物学的发展,APB的电子传递、能量和物质代谢系统逐渐被应用于开发新型光生物电化学电池、生物合成色素、小分子化合物等。基于此,本文依据APB光合作用的研究现状,概述捕光天线、光合电子传递链的重要元件,如RC、醌-细胞色素c氧化还原酶(quinol-cytochrome c oxidoreductase)和膜周质侧的电子受体;以及固碳途径,如卡尔文-本森-巴萨姆循环(Calvin-Benson-Bassham cycle, CBB cycle)、还原性三羧酸循环(reductive TCA cycle, rTCA cycle)和3-羟基丙酸循环(3-hydroxypropionate cycle, 3-HP cycle)的反应机制。
1 APB的光合电子传递链光合电子传递链由多个电子传递体组成。光合作用光反应所产生的电子,在多个电子传递体中按照它们氧化还原电位递增的顺序依次传递。在植物、藻类和蓝细菌的类囊体中,光合电子传递链由PSⅠ、PSⅡ、细胞色素(cytochrome, cyt) b6f复合物、质体醌(plastoquinone, PQ)和质体蓝素(platocyanin, PC)组成[18]。其中有两个逆电势梯度的电子传递,即P680至P680*以及P700至P700*[19]。根据电子传递至铁氧还蛋白(ferredoxin, Fd)后的去向不同,放氧光合生物的电子传递链分为非环式、环式以及假环式三种类型[20]。PSII反应中心的色素分子吸收光能后发生光化学反应即电荷分离,失去电子的色素分子是强氧化剂,夺取附近水分子的电子,导致水分子的裂解并释放O2和质子。该过程释放的电子经PQ、cyt b6f、PC、PSI和Fd组成的“Z链”依次传递,最终通过Fd-NADP+还原酶(ferredoxin-NADP+ reductase, FNR)还原NADP+为NADPH,形成非环式电子传递链。当NADP+不足或遭受环境胁迫时,PSI的电子经Fd、PQ、cyt b6f、PC等返回PSI,构成环式电子传递链,此过程不产生O2[21]。在假环式电子传递链中,O2替代NADP+成为最终的电子受体,在消耗O2的同时产生H2O2 (图 2)。
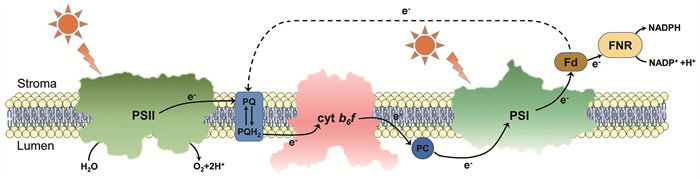
|
Cyt b6f、PQ和PC介导了PSII和PSI两个光系统之间的电子传递。黑色箭头代表电子传递路径。 图 2 放氧光合生物的光合电子传递链示意图 |
对于没有类囊体的APB,其细胞质膜内陷或出芽形成独立于质膜的片层状或者囊泡状载色体(chromatophore),该结构容纳光合电子传递链的关键组分(图 3)[22]。在电子传递链的构成上,拥有单一光系统的APB与放氧光合生物明显不同。以研究最为广泛的紫细菌为例,其载色体上含有两种LH,外周捕光天线LH2用于吸收光能,核心捕光天线LH1接收光能并传递至脱镁叶绿素-醌型(Ⅱ型)的RC[23]。RC接受光能后发生原初反应,释放出的电子将醌还原为氢醌。氢醌从RC-LH脱离进入质膜的醌池,继而被质膜上的醌-细胞色素c氧化还原酶氧化,释放的电子经膜周质侧游离的电子受体(如cyt c)传回RC,从而完成环式电子传递[24]。此电子传递过程产生的跨膜质子动力势驱动ATP合酶(ATP synthase)合成ATP。
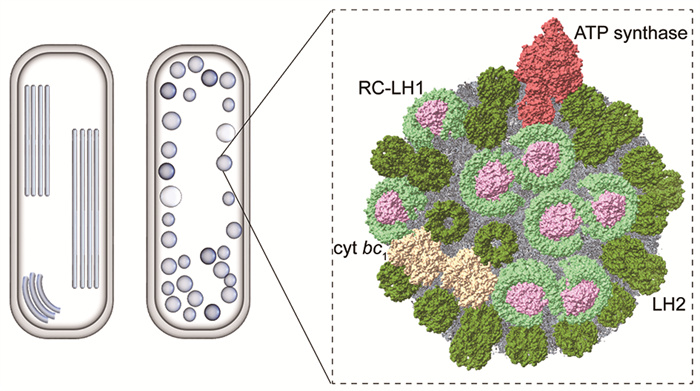
|
片层状或囊泡状载色体的示意图(左)。紫细菌独立于质膜的囊泡状载色体中光系统的结构模型(右)。LH2、LH1、RC、cyt bc1以及ATP合酶(ATP synthase)分别显示为深绿色、浅绿色、浅粉色、浅黄色和浅红色。 图 3 光合细菌载色体的结构示意图 |
与紫细菌显著不同的是,绿硫细菌和部分绿色非硫细菌具有一种独特的外周天线——绿色体(chlorosome),其特征吸收波长为740 nm。太阳杆菌则利用融合天线结构域的RC捕获光能并转化为电能[2]。在含有铁硫簇(Ⅰ型) RC的绿硫细菌和太阳杆菌中,铁硫簇所接受的电子传给Fd,最终经FNR生成NADPH。绿硫细菌Chlorobaculum (Cba.) tepidum的体外研究表明,在Fd和RC共同存在的情况下,NADP+和NAD+能够高效地被光还原[25]。因此,APB的光合电子传递不仅通过光合磷酸化有效合成ATP,为细胞代谢提供能量,还利用与RC相互作用的电子载体Fd,在FNR的介导下将NAD(P)+转化为稳定的还原剂NAD(P)H。
1.1 捕光系统 1.1.1 紫细菌的捕光系统早在1995年,研究人员解析了紫细菌Rhodoblastus (Rbl.) acidophilus LH2的晶体结构。在此结构中,9个脱辅基蛋白的异二聚体(LHαβ)组成环状,每个LHαβ中都结合3个BChl a和1个类胡萝卜素(carotenoid, Car)分子用于捕获光能。光谱分析显示,此LH2在804 nm和858 nm处有两个近红外的BChl a Qy吸收峰[26]。因此,Rbl. acidophilus的LH2也被称为B800-850复合物。在适应生存环境变化的过程中,紫细菌衍生出具有不同吸收光谱的LH2变体,如LH3 (B800-820复合物)和LH4 (B800复合物)[26-27]。
除LH2外,紫细菌质膜上还分布由多个脱辅基蛋白的异二聚体(LH1αβ)组成的环状LH1,将RC紧密包围在中间,每个LH1αβ都结合色素分子用于吸收光能。随着晶体学和冷冻电镜技术的发展,紫细菌RC-LH1复合物的结构陆续被解析。根据LH1环的封闭、开放和聚合状态,RC-LH1复合物呈现出多种组装模式和构象。例如,以单体形式存在于Blastochloris (Blc.) viridis、Thermochromatium (Tch.) tepidum、Allochromatium (Alc.) tepidum、Thiorhodovibrio (Trv.) strain 970、Rhodospirillum (Rsp.) rubrum和Rhodopila (Rpi.) globiformis中的闭环RC-LH1;以开放环状形式存在的Rhodobacter (Rba.) veldkampii、Rba. capsulatus RC-LH1;兼具开环和闭环两种构象的Rhodopseudomonas (Rps.) palustris RC-LH1,以及Rba. sphaeroides中天然存在的单体和二聚体形式的RC-LH1等[28-37]。
紫细菌Tch. tepidum拥有典型的闭环状RC-LH1,其LH1由16个LH1αβ组成,每个LH1αβ在膜周质侧结合两个BChl a,在LH1αβ亚基之间结合倾斜插入的Car分子用于吸收和传递光能。闭环RC-LH1中相邻LH1αβ接触面上的缝隙被认为是氢醌交换的通道[28]。在紫细菌的开放RC-LH1中,通常由PufX、PufY (或蛋白U)、PufW等蛋白形成1~2股跨膜螺旋,在LH1环上形成一个缺口而作为氢醌交换的通道[36-37]。对于Rba. sphaeroides的RC-LH1来说,二聚体形式比单体开环状具有更高的能量传递效率。然而,当从基因组中敲除PufX的编码基因时,天然提取的RC-LH1以单体闭环形式存在,此结构阻碍醌的扩散并降低RC-LH1的光合效率[38-39]。此外,Rps. palustris的RC-LH1在缺少PufW时也形成闭合的LH1环,且相较于含PufW的开环结构,其醌交换速率显著降低[36]。因此,RC-LH1的组装和醌穿梭通道的构象,以及醌交换的速率都受到PufX、PufW等跨膜蛋白的调节。
1.1.2 含有绿色体的捕光系统与紫细菌截然不同,绿硫细菌和部分绿色非硫细菌通过绿色体吸收光能。绿色体通常由单层脂质包裹而成,呈扁平椭圆状,长度约为100~200 nm,宽度约为40~60 nm,厚度约为10~40 nm[40]。对绿硫细菌Cba. tepidum的电镜研究显示,其绿色体的平均长度约为118~180 nm,平均宽度约为43~60 nm[41]。巨大的绿色体像“囊泡”一样附着在由CsmA (chlorosome protein A)蛋白和BChls组成的基底板上;而光能通过内周捕光天线FMO (Fenna-Matthews- Olson)的同源三聚体传递到RC[42]。研究表明,绿色体到FMO之间的能量传递效率接近100%,但FMO和RC之间的能量传递效率仅有35%~75%[43]。与绿硫细菌略有不同,绿色非硫细菌的代表菌株Chloroflexus (Cfl.) aurantiacus具有相对较小的绿色体。这些绿色体由数千个BChl c和Car分子自组装而成,可吸收波长为740 nm的光,并通过基底板上分布的BChl a将光能传递到LH[44]。适应低强度的光照后,Cfl. aurantiacus的BChl a和c浓度随着绿色体数量和体积的增加而显著提高[45]。而绿硫细菌Cba. tepidum则通过增加绿色体中BChl c的含量和甲基化水平来适应弱光环境[46]。
1.1.3 缺少绿色体的捕光系统2002年,研究人员在日本的温泉中发现一种新型的绿色非硫细菌Roseiflexus (Rfl.) castenholzii,因其菌体呈红色,被称为光合玫瑰菌[47]。在电子显微镜下,Rfl. castenholzii呈可移动的丝状,细胞直径0.8~1.0 µm,菌体含有BChl a和γ-胡萝卜素;其最佳生长条件为50 ℃,pH 7.5~8.0[47]。Rfl. castenholzii是为数不多的缺失绿色体的绿色非硫细菌,它没有外周捕光天线,仅有的LH可吸收波长为800 nm的远红外光。因而,缺失绿色体使Rfl. castenholzii能够更广谱地利用光能[48],还可避免色素对膜组分光谱吸收的影响[49]。Rfl. castenholzii的LH融合紫细菌LH1和LH2的优势,其组成和装配与LH1相似,而光谱学特征却与LH2相仿。LH由15对LHαβ异二聚体形成一个开放的椭圆环,每个LHαβ结合3个BChl a (2个B880和1个B800)和2个Car分子用于捕获光能[50]。在LH环的开口处,由cyt c亚基的跨膜螺旋和新发现的X亚基共同形成一个独特的醌穿梭通道[51]。近期研究结果表明,在Rfl. castenholzii的培养基中添加Car合成的抑制剂二苯胺,可显著减少LH环中结合的Car数目,并引起LH环开口处X亚基的缺失和RC-LH醌交换速率的提高[50]。因此,通过改变LH环上色素分子的排布,有望调控APB的光合效率。
1.2 RCRC是将光能转化为化学能的核心场所。植物、藻类和蓝细菌等放氧光合生物同时含有Ⅰ型(PSⅠ,P700)和Ⅱ型(PSⅡ,P680)的RC,而APB通常只含有一种类型的RC[52]。绿硫细菌和太阳杆菌的RC是铁硫型(Ⅰ型),以铁硫簇作为末端电子受体;而绿色非硫细菌和紫细菌的RC属于脱镁叶绿素-醌型(Ⅱ型),以醌分子作为电子受体[53]。
1.2.1 APB的Ⅰ型RC太阳杆菌和绿硫细菌的Ⅰ型RC往往形成同源二聚体。在典型的太阳杆菌Heliobacterium (Hbt.) modesticaldum中,RC的核心亚基PshA融合天线结构域和电子传递结构域,两者分别通过6个和5个跨膜螺旋锚定在细菌的质膜上[54]。天线结构域中54个BChl形成彼此靠近的两层,有助于激发能快速转移至电子传递结构域中。而电子传递结构域含有两对BChls (分别称为P800和ACC)、一对叶绿素(A0)和一个[4Fe-4S](FX)。其中,P800是原初电子供体,FX作为末端电子受体接受来自A0的电子[55]。除了PshA的核心二聚体外,在太阳杆菌Hbt. modesticaldum的RC中新发现一个PshX亚基,其通过组氨酸残基与4个天线BChl配位结合,所处的位置与细长聚球藻(Synechococcus elongates) PSⅠ中的PsaI和PsaJ亚基相似,但与BChl配位的氨基酸序列并不保守[55]。
不同于太阳杆菌,绿硫细菌Cba. tepidum的RC主要包括2个核心亚基PscA、2个PscC、1个PscB和1个PscD亚基。其中,PscB位于胞质侧,结合2个[4Fe-4S](FA和FB),与含有[4Fe-4S](FX)的PscA二聚体核心相互作用[9, 56-57];PscD同时与PscB和内周捕光天线FMO接触,起到稳定复合物的作用;PscC与PscA密切相互作用,并通过N端的三股跨膜螺旋锚定在膜上,其C端含有一个血红素结合结构域,接受电子并传回PscA的特殊细菌叶绿素对P840[56]。绿色体和FMO捕获的光能激发P840导致电荷分离,产生的电子沿着两个分支((B)Chls→FX→FA→FB)传递。此外,Cba. tepidum的RC中结合的30个(B)Chls以上、下两层的方式排列,中间缺少其他Ⅰ型RC中的连接色素,而是代之以2个Car分子[9],推测这些Car可能在稳定RC结构以及辅助捕光方面发挥作用。
1.2.2 APB的Ⅱ型RC典型的紫细菌RC由L、M和H三个亚基组成。高度保守的L和M亚基各自含有5个跨膜螺旋,紧密结合形成异二聚体。其中,L和M亚基结合2个辅助BChl (简称为BA和BB)、2个细菌脱镁叶绿素(bacteriopheophytin, Bphe;简称为HA和HB)、1个铁原子和2个醌分子(QA和QB),这些递电子体在RC中排列成两个分支:活性分支A和无活性分支B[58]。在原初反应中,特殊细菌叶绿素对(P)接受LH中BChl传递的光能而被激发(P*),释放的电子经一侧的BA、HA、QA传递给铁原子,后者再传递电子给QB。在连续接受两个电子后,QB−被还原为氢醌,并脱离RC进入质膜上的醌池以维持电子的循环传递。紫细菌的H亚基往往通过一个跨膜螺旋锚定在膜上,在胞质侧形成球状结构。H亚基可以保护RC的醌结合位点不受胞质侧离子的影响,并为QB的质子化提供通道,是维持RC结构稳定所必需的;但缺失H亚基并不影响电子传递[59-60]。在紫细菌Gemmatimonas (G.) phototrophica的RC中,H亚基被一个较小的跨膜多肽Ht和一个较大的胞质侧多肽Hc所替代[61]。更特别的是,G. phototrophica的RC在膜周质侧和胞质侧分别结合1个S亚基和1个U亚基,通过氢键相互作用维持RC-dLH复合物的稳定[61]。
紫细菌Blc. viridis、Tch. tepidum、Trv. strain 970等的RCs具有额外的cyt c亚基。经过翻译后加工,cyt c亚基通过N端半胱氨酸残基与脂肪酸之间的硫醚键锚定在L和M亚基的膜周质侧[28, 29, 31]。这些cyt c亚基都结合4个c型血红素,接受来自膜周质侧的电子并传回RC,还原失去电子的P+使其恢复到基态,从而完成环式电子传递(图 4A)。已有研究表明,绿色非硫细菌Rfl. castenholzii的RC-LH在膜周质侧结合含4个血红素的cyt c554亚基,可接受膜周质侧电子受体金色蓝素(auracyanin, Ac)或cyt c551传回的电子(图 4B)。在紫细菌Tch. tepidum的RC-LH1中,cyt c亚基还可以接受高电势铁蛋白(high-potential iron protein, HiPIP)传回的电子[62-63]。对于这些含有4个血红素cyt c的RC-LH,cyt c亚基在质膜上的构象也不相同。紫细菌Rpi. globiformis的cyt c在N端形成单跨膜螺旋,延伸至LH1环的内侧,并与M、H、LH1α亚基以及脂质产生极性和氢键相互作用[33];而Rfl. castenholzii的cyt c亚基N端跨膜螺旋则以相反的方向插入LH环的开口,与X亚基共同形成醌交换通道[50]。
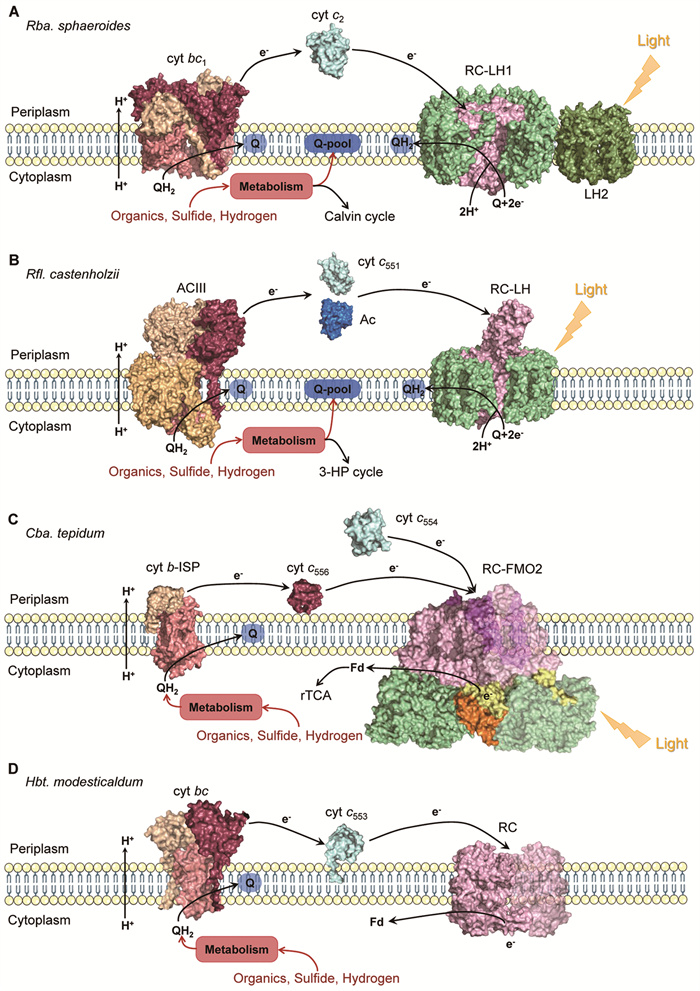
|
(A)紫细菌Rba. sphaeroides的光合电子传递链,RC-LH1结构(PDB:7VNY)、LH2结构(PDB:7PBW)、cyt bc1结构(PDB:2QJP)、cyt c2结构(PDB:1CXC)。(B)绿色非硫细菌Rfl. castenholzii的光合电子传递链,RC-LH1结构(PDB:5YQ7)、ACⅢ结构(PDB:6LOE)、cyt c551结构(PDB:8GTH)、Ac结构(PDB:6KOL)。(C)绿硫细菌Cba. tepidum的光合电子传递链,RC-FMO2结构(PDB:7Z6Q)、cyt b结构(AlphaFold预测的模型)、ISP (PDB:8HN2)、cyt c556结构(PDB:8HN3)、cyt c554结构(PDB:4J20)。(D)太阳杆菌Hbt. modesticaldum的光合电子传递链,RC结构(PDB:5V8K)、cyt bc和cyt c553结构(AlphaFold预测的模型)。除Rba. sphaeroides的LH2为深绿色外,其他捕光天线LH1、LH、FMO均显示为浅绿色。Cba. Tepidum RC的PscA、PscB、PscC和PscD亚基分别显示为浅粉色、黄色、粉红色和橙色,其他RC均为浅粉色。Cyt bc1/bc复合物中cyt b亚基为橘红色,ISP亚基为浅黄色,cyt c1/c亚基和cyt c556为红棕色,cyt c2、cyt c551、cyt c553、cyt c554均为浅青色。Ac为蓝色。Rfl. castenholzii的ACⅢ中,ActA和ActE亚基为红棕色,ActC亚基为橘红色,ActB亚基为浅黄色,ActD和ActF亚基为浅橙色。黑色箭头表示电子传递以及质子转运方向,红色箭头表示电子供体。 图 4 APB光合电子传递链的示意图 |
醌-细胞色素c氧化还原酶,也被称为复合物Ⅲ或cyt bc1,是大部分APB光合和呼吸电子传递链共有的组分,在耦联电子传递和质子跨膜转运中发挥不可或缺的作用。Cyt bc1的亚基组成因物种而异,但都包含3个高度保守的核心亚基:结合b型血红素的cyt b,结合c型血红素的cyt c1和1个Rieske铁硫蛋白(iron-sulfur protein, ISP)[64-65]。每个cyt b亚基含有8个跨膜螺旋,同时结合两个b型血红素,分别称为低电位血红素(bL)和高电位血红素(bH)。ISP通过斜插入膜中的单次跨膜螺旋固定在膜上,在膜周质侧含有一个高电势的[2Fe-2S]。位于同侧的cyt c1亚基也通过单次跨膜螺旋锚定在膜上,活性区域共价结合的c型血红素接受来自[2Fe-2S]的电子以还原游离的cyt c。Cyt bc1具有醌氧化(QP)和醌还原(QN)两个醌结合位点,通过氧化还原作用驱动的“醌循环”耦联电子传递和质子转运。每一个“醌循环”将两个电子从氢醌传递给两个cyt c,跨膜转运4个质子[66-68]。氧化还原反应中醌结合位点的变化引起ISP的移动,进而通过cyt b亚基的构象变化[69-71]驱动质子的跨膜净转移[72-73]。
近年,在非光合嗜热菌Rhodothermus. (R.) marinus的呼吸链[74-75]和绿色非硫细菌Cfl. aurantiacus[76-78]的电子传递链中发现一种全新的电子载体蛋白质复合物。这两种细菌都没有编码cyt bc1的基因[75-76],却通过一种相似的蛋白质复合物执行醌-细胞色素c氧化还原酶的功能[79-80]。从R. marinus分离的复合物中含有b/c型血红素,可还原HiPIP[74, 79];从Cfl. aurantiacus分离的复合物与铁硫钼酶复合物(complex iron-sulfur molybdoenzymes, CISM)同源,只含有c型血红素[78-80]。基因测序和组分鉴定最终确定两者具有相同的组成[79-80]。在Cfl. aurantiacus的光合和呼吸电子传递链中,此复合物都能氧化氢醌,并将电子传递给新发现的Ac[77, 80]。虽然这个复合物的组成和一级结构都与cyt bc1完全不相关,但其功能与复合物Ⅲ相似,因此被命名为替代复合物Ⅲ (alternative complex Ⅲ, ACⅢ)[81]。
目前已在66种微生物中发现了ACⅢ的编码基因,随着基因测序的发展,这一数据还在不断更新。值得关注的是,ACⅢ主要出现在不含cyt bc1编码基因的物种中;少数微生物中ACⅢ与cyt bc1的编码基因共存,其表达取决于代谢活动的需要[82]。来自Flavobacterium johnsoniae和R. marinus的呼吸链,以及绿色非硫细菌Rfl. castenholzii和Cfl. aurantiacus的光合电子传递链ACⅢ的电镜结构[83-85]显示,ACⅢ由6个核心亚基(ActA、ActB、ActC、ActD、ActE和ActF)组成。膜周质侧的ActA和ActE分别结合5个和1个c型血红素,ActB亚基结合4个铁硫簇(1个[3Fe-4S]和3个[4Fe-4S]),共同构成ACⅢ内部的电子传递链。ActC和ActF各自含有10股跨膜螺旋,ActC在膜周质一侧距离[3Fe-4S]约10~12 Å的范围内有一个甲基萘醌(menaquinol, MQ)结合口袋和一条质子转运通道。ActD有两个跨膜螺旋,其亲水结构域暴露在胞质侧;ActE是一个单血红素亚基,被认为是电子传递的最后一站,接受ActA的电子并传递给膜周质侧的电子受体。除6个核心亚基外,在Cfl. aurantiacus的ACⅢ结构中发现2个新的亚基ActG和I[86];而在R. marinus的ACⅢ电镜结构中还发现1个新的ActH亚基[84]。这些亚基可能在维持复合物的结构稳定方面发挥作用。
绿色非硫细菌Rfl. castenholzii和Cfl. aurantiacus的ACⅢ都具有还原态MQ(MQH2)-Ac氧化还原酶的活性,可氧化MQH2类似物MK-4和MK-7并传递电子给Ac[77]。多种谱学与物理化学研究结果显示,Rfl. castenholzii的ACⅢ氧化MQH2后释放的电子经内部结合的1个[3Fe-4S]和6个c型血红素传递给Ac。Cfl. aurantiacus ACⅢ结合抑制剂HQNO (2-heptyl-4-hydroxyquinoline-N-oxide)的冷冻电镜结构显示,HQNO头部的喹啉环与MQ结合口袋中的His141和Asp171通过氢键相互作用,在MQ氧化过程中发挥关键作用[86]。而ActC亚基质子转运通道中的三个关键氨基酸残基His99、His246和Arg394与HQNO结合口袋形成广泛的氢键网络,在耦联氢醌氧化和质子转运中发挥重要作用[86]。ActF亚基虽然也含有1条潜在的质子转运通道,但却没有醌结合口袋,很可能由于缺乏氢醌氧化所产生的驱动力而无法跨膜转运质子[85]。关于ACⅢ结构和功能的研究对于理解醌-细胞色素c氧化还原酶和电子传递机制的多样性,阐明APB能量代谢机制具有重要的意义。
1.4 膜周质侧的电子受体 1.4.1 Cyt cCyt c广泛分布于真核生物的线粒体、原核生物及其他各类光合生物中,是细胞内介导氧化还原反应的一个重要分子。根据结合血红素的数目、血红素铁的轴向配位类型及氧化还原电位,cyt c大体可分为4类[87]。第一类是含有组氨酸/甲硫氨酸轴向配位的单一低自旋血红素,其结合的保守氨基酸基序为Cys-Xaa-Xaa-Cys-His (CXXCH),在序列和结构上与线粒体cyt c高度同源[88]。第二类cyt c具有4股α-螺旋束的结构基序,也含有低自旋的血红素,如cyt c556。第三类cyt c则结合多个血红素,如结合4个血红素的cyt c3。第四类属于高分子量的cyt c,如cyt cd1、RC结合的cyt c亚基等。在不同的细胞定位和氧化还原环境中,cyt c参与多种生命活动和能量传递过程,如产生和清除活性氧、脂质过氧化、调节细胞凋亡等[89]。本文主要探讨cyt c在APB电子传递链中的作用。
作为膜周质侧的电子载体,cyt c以游离和膜结合两种方式参与APB的电子传递。膜结合的cyt c通常存在于RC和cyt bc1中。紫细菌RC的cyt c亚基结合两个低电位和两个高电位血红素,两者在空间上呈现垂直堆叠排列。根据氧化还原电位,cyt c亚基中的4个血红素和BChl形成了低电位-高电位-低电位-高电位-特殊细菌叶绿素对的电子传递路径[90]。与此相似,太阳杆菌使用膜结合的cyt c553作为还原RC的电子供体,绿硫细菌的cyt cz/cyt c551 (也称为PscC)也具有相同的功能[91]。在绿硫细菌Cba. tepidum的RC-FMO2超级复合物的结构中,PscC亚基可以接受来自膜周质侧cyt c556的电子(图 4C)[56]。特别的是,在太阳杆菌Hbt. Modesticaldum中cyt bc复合物取代紫细菌的cyt bc1,执行电子传递和质子转运的功能。这两种复合物的主要区别在于,cyt bc含有结合2个血红素的cyt c4亚基,可作为末端电子受体,接受电子后进一步还原cyt c553 (图 4D)[92]。
除了结合在膜上的cyt c,游离的可溶性cyt c对于膜蛋白复合物之间的电子传递也是必不可少的。在紫细菌的光合电子传递中,游离的cyt c2通过与RC和cyt bc1表面的静电相互作用,将电子从cyt bc1传回到RC,从而完成环式电子传递[93]。从Rfl. castenholzii中分离提取出的一种结合c型血红素的cyt c551[62],被证实是ACⅢ的另一种直接电子受体,可接受ACⅢ氧化MQH2后释放的电子并将其传回RC-LH,从而完成一个替代的环式电子传递(图 4B)。通过对Rfl. castenholzii的Ac和cyt c551基因表达水平的检测,发现cyt c551很可能在缺铜条件下取代Ac作为膜周质侧的电子受体,介导ACⅢ和RC-LH之间的环式电子传递[62]。此外,cyt c4、cyt c8和HiPIP也可作为RC的电子供体[63]。绿硫细菌含有一种游离的cyt c554,通过含有1个血红素的cyt cz亚基将电子传回RC。因此,cyt c554被认为是二级电子供体,也是硫代硫酸盐氧化的有效电子受体[94]。
1.4.2 Ac绿色非硫细菌Cfl. aurantiacus和Rfl. castenholzii主要利用Ac作为膜周质侧的电子受体。1988年,在Cfl. aurantiacus中发现第一个Ac,紧接着陆续发现3种不同的Ac。根据发现时间的先后,依次命名为Ac-A、Ac-B、Ac-C和Ac-D[95-96]。其中,Ac-A和Ac-B锚定在膜上并被糖基化,在体外实验中它们将电子传递给RC[97]。Ac-A和Ac-B都具有一个Ⅰ型的铜离子结构域,其中铜离子与2个组氨酸、1个半胱氨酸和1个甲硫氨酸配位结合[97-98]。然而,Ac-A和Ac-B的N端序列完全不同:Ac-A的N端含有信号肽,通过甘氨酸富集序列末端一个半胱氨酸残基的甘油脂肪酰化修饰,将铜离子结构域连接在细菌的质膜上[99];Ac-B则通过N端的一个单次跨膜螺旋将铜离子结构域锚定在膜上[99-100]。蛋白质表达水平的检测显示,Ac-A在厌氧光照条件下发挥作用,而Ac-B在呼吸和光合电子传递链中均可起作用[101]。然而,质谱研究则显示出完全相反的结果,光照条件下生长的Cfl. aurantiacus仅表达Ac-B,黑暗异养条件下Ac-A和Ac-B均有表达[102]。目前,关于Ac-A和Ac-B发挥作用的生理条件仍不明确。
与Cfl. aurantiacus不同的是,在Rfl. castenholzii的基因组中仅发现一个编码Ac的基因。Rfl. castenholzii的Ac (rcAc)具有和Ac-A相似的Ⅰ型铜离子结构域,其较低的OD465nm/OD603nm吸收比率又和Ac-B相似[97, 99, 103]。与Ac-A相似,rcAc的N端包含一个信号肽和富含甘氨酸的接头,保守的铜离子结构域通过半胱氨酸残基的甘油脂肪酰化修饰连接在质膜上[99]。铜离子由来自于同一段无规卷曲(loop)的Cys141、His146和Met151以及另一段loop的His77所锚定,其中His146的咪唑基团暴露的一侧可能与ACⅢ相互作用[104]。通过电子顺磁共振、光谱学、酶学和氧化还原电势测定等方法,目前已证实rcAc很可能在膜周质侧来回摆动铜离子结构域,通过His146侧的疏水作用和静电相互作用介导ACⅢ和RC-LH之间的电子传递[104]。
2 APB的固碳途径利用同化力形成阶段产生的ATP和NAD(P)H,光合生物通过多种酶促反应将CO2等无机物转变为有机物的过程称为CO2固定或同化。在适应不同生长环境的过程中,APB演化出多样化的固碳途径。大多数APB (如紫细菌等)通过CBB循环固定CO2,绿硫细菌主要通过rTCA循环固碳,而绿色非硫细菌则通过一种新的途径——3-HP循环进行碳同化[8]。此外,好氧的APB (如Roseobacter denitrificans OCh114)通过CO2的回补途径(anaplerotic pathway),即利用苹果酸酶(malic enzyme)固定CO2,产生10%~15%的含碳有机物[13]。下面主要介绍APB中普遍存在的三种固碳途径。
2.1 CBB循环紫细菌和部分绿色非硫细菌(主要是Oscillochloris等)以CBB循环进行碳同化[8]。1945年,Calvin等发现小球藻能固定CO2,转化1, 5-二磷酸核酮糖(ribulose bisphosphate, RuBP)为3-磷酸甘油醛(glyceraldehyde-3-phosphate, G3P)。经过十余年的系统研究,CO2同化的途径被成功阐明,即广为人知的CBB循环[105]。尽管APB在光合生物的演化历程中处于早期阶段,但对于这些细菌固碳机制的研究却相对滞后。直到1959年,Fuller和Gibbs[106]在Rsp. rubrum和Chromatium sp.的提取物中首次检测到CBB循环的关键酶——核酮糖-1, 5-二磷酸羧化酶/加氧酶(ribulose-1, 5-bisphosphate carboxylase/oxygenase, Rubisco),说明APB中也有CBB循环。随后,在紫细菌的其他物种中陆续发现Rubisco。例如,1970年Akazawa等[107]分离得到Rba. sphaeroides和Rsp. rubrum的Rubisco;1978年,Tabita等[108]发现这两种细菌在光合自养条件下通过CBB循环将CO2固定为有机碳。
与植物的CBB循环相似,APB的这个循环也包括羧化、还原和RuBP再生三个阶段,将3分子的CO2合成1分子的三碳化合物(G3P)。具体而言,在Rubisco的催化下,CO2首先被整合到RuBP,转化为3-磷酸甘油酸(3-phosphogylcerate, 3-PGA);经过ATP的磷酸化和NADPH的还原,3-PGA转化为G3P,后者通过一系列反应,最终转化为RuBP[8, 109](图 5)。
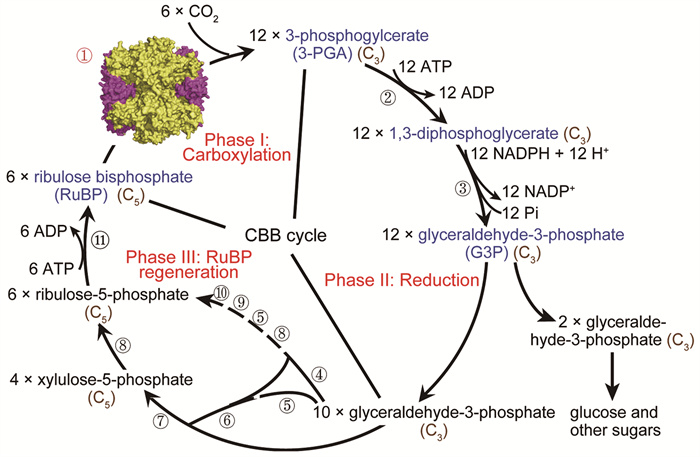
|
图中带圈的数字代表催化各步反应的酶:①核酮糖-1, 5-二磷酸羧化酶/加氧酶(ribulose-1, 5-bisphosphate carboxylase/oxygenase, Rubisco),② 3-磷酸甘油酸激酶(3-phosphoglycerate kinase),③甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase),④丙糖磷酸异构酶(triose phosphate isomerase),⑤醛缩酶(aldolase),⑥果糖-1, 6-双磷酸酶(fructose-1, 6-bisphosphoesterase),⑦转酮酶(transketolase),⑧景天庚酮糖-1, 7-二磷酸酶(sedoheptulose-1, 7-bisphosphatase),⑨差向异构酶(epimerase),⑩磷酸戊糖异构酶(phosphopentose isomerase),⑪磷酸核酮糖激酶(phosphoribulokinase)。图中Rubisco的电镜结构(PDB:7JN4)来源于莱茵衣藻(Chlamydomonas reinhardtii)。 图 5 CBB循环关键反应步骤示意图 |
由于Rubisco在CBB循环中的核心地位,目前的研究主要关注真核生物该酶的催化机制和底物选择性,以提高其羧化活性和对O2/CO2的特异性等[109]。关于APB中该酶结构和催化机制的研究还相对较少,不利于对其固碳机制的深入理解。根据结构和序列的相似性,该酶被分为6种类型:Ⅰ型、Ⅰ’型、Ⅱ型、Ⅲ型、Ⅱ/Ⅲ型和Ⅳ型[110]。其中,绿弯菌属(Chloroflexi)的APB含有Ⅰ’型Rubisco,但仅包含大亚基。与Ⅰ型相似,Ⅰ’型的大亚基N端包含1个α/β结构域,C端包含1个磷酸丙糖异构酶结构域和1个柔性的尾巴[111]。特别的是,大亚基C端loop 6的灵活运动对于酶活性位点的开闭是必需的[110, 112]。此外,绿硫细菌Cba. tepidum的Ⅳ型Rubisco缺少催化位点,并不具备催化功能[113]。
2.2 rTCA循环相较于CBB循环,rTCA循环(又称还原柠檬酸循环[114])消耗较少的能量。由于该循环包含一些对O2非常敏感的酶,因此主要存在于厌氧菌或O2含量极低环境下生存的需氧菌中,如APB中的少数紫细菌和绿硫细菌[8, 109]。通过建立生命树谱系,Kandler[115]发现rTCA循环广泛分布于原核生物域谱系,是演化早期出现的固碳途径;这也可能是早期地球上有机分子的来源[116]。
rTCA循环的发现起源于Fd参与的还原性羧基反应。1964年,Bachofen等[117]在紫细菌Chromatium vinosum中发现一种丙酮酸: 铁氧还蛋白氧化还原酶(pyruvate : ferredoxin oxidoreductase, PFOR),可催化乙酰CoA和CO2合成丙酮酸。1966—1967年,在绿硫细菌Chlorobium limicola forma thiosulfatophilum (缩写为Chlorobium)[114]和紫色非硫细菌Rsp. rubrum[118]中也发现该酶。1965—1967年,Buchanan等[119-120]在Chlorobium和Rsp. rubrum的细胞提取液中又发现另一种酶——α-酮戊二酸(又名2-氧戊二酸) : 铁氧还蛋白氧化还原酶(2-oxoglutarate : ferredoxin oxidoreductase, OGOR),这种酶的催化机制与PFOR相似,可以催化琥珀酰CoA和CO2合成α-酮戊二酸。由于OGOR催化的反应是TCA循环中不可逆转的反应,这为rTCA循环的发现提供强有力的证据。在此期间,该研究团队在Chlorobium中证明rTCA循环是一个独立且完整的固碳循环:Chlorobium中缺少CBB循环固定CO2的关键酶,而其细胞提取液中不仅检测到rTCA循环需要的所有酶,还鉴定到了该循环预期的代谢产物[114]。尽管后来关于rTCA循环的争论持续很长的时间,随着科学研究的深入,rTCA循环已作为一种新的碳同化方式逐渐被人们接受。
rTCA循环起始于草酰乙酸,通过TCA循环的逆反应,固定2分子CO2得到柠檬酸,柠檬酸再经过一系列反应重新生成草酰乙酸,从而完成一轮循环(图 6)。草酰乙酸转化为柠檬酸的过程主要涉及两步羧化反应,即琥珀酰CoA在OGOR催化下被还原羧化为α-酮戊二酸和CoA;α-酮戊二酸被还原羧化为异柠檬酸[109, 120]。由异柠檬酸生成的柠檬酸在ATP-柠檬酸裂解酶的作用下,直接裂解为乙酰CoA和草酰乙酸。而乙酰CoA在PFOR催化下,被还原羧化为丙酮酸;后者经磷酸烯醇式丙酮酸合成酶(phosphoenolpyruvate (PEP) synthetase)和PEP羧化酶的催化而生成草酰乙酸。最终,1分子柠檬酸可转化为2分子草酰乙酸(图 6)。OGOR、ATP-柠檬酸裂解酶和PFOR是该循环的关键酶[109]。
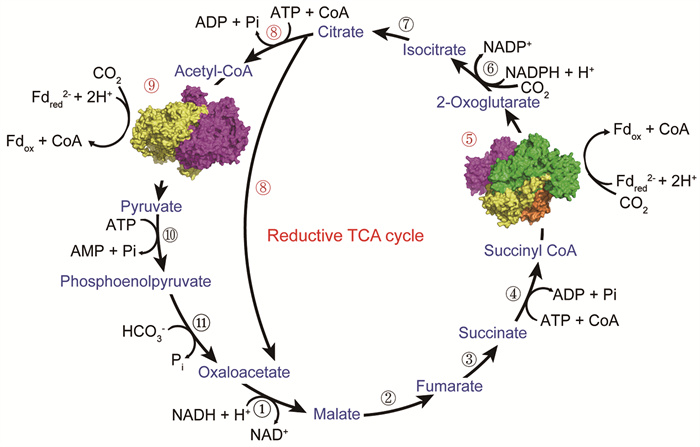
|
图中带圈的数字代表催化各步反应的酶:①苹果酸脱氢酶(malate dehydrogenase),②富马酸水化酶(fumarate hydratase),③富马酸还原酶(fumarate reductase),④琥珀酰CoA合成酶(succinyl-CoA synthetase),⑤ 2-氧戊二酸(又称α-酮戊二酸)﹕铁氧还蛋白氧化还原酶(2-oxoglutarate﹕ferredoxin oxidoreductase,OGOR),⑥异柠檬酸脱氢酶(isocitrate dehydrogenase),⑦乌头酸梅(aconitase),⑧ ATP-柠檬酸裂解酶(ATP citrate lyase),⑨丙酮酸﹕铁氧还蛋白氧化还原酶(pyruvate﹕ferredoxin oxidoreductase,PFOR),⑩磷酸烯醇式丙酮酸合成酶(phosphoenolpyruvate (PEP) synthetase),PEP羧化酶(phosphoenolpyruvate carboxylase)。图中OGOR的电镜结构(PDB:6N2N)来源于海洋单链趋磁球菌(Magnetococcus marinus MC-1);PFOR的晶体结构(PDB:1B0P)来源于非洲脱硫杆菌(Desulfocurvibacter africanus)。 图 6 rTCA循环反应步骤示意图 |
尽管人们对APB的rTCA循环催化步骤和关键酶都已有所了解,但对其关键酶的结构和催化机制研究仍相对滞后。目前PFOR和OGOR的结构在热乙酸穆尔氏菌(Moorella thermoacetica, Mt)和海洋单链趋磁球菌(Magnetococcus marinus MC-1, Mm)等物种中已有报告。MtPFOR的结构呈现为同源二聚体,每个亚基包含六个结构域(Ⅰ~Ⅳ)。其中,1个硫胺素焦磷酸(thiamine pyrophosphate, TPP)结合在结构域Ⅰ和Ⅵ的界面上,结构域Ⅴ和Ⅵ分别结合2个和1个[4Fe-4S]。MtPFOR可催化乙酰CoA、CO2与丙酮酸、CoA之间可逆的转变。在丙酮酸的氧化过程中,MtPFOR利用TPP激活丙酮酸,形成丙基-TPP共价中间体,进而发生脱羧反应;脱羧后的羟基乙叉-TPP阴离子中间体将一个电子转移至[4Fe-4S],形成羟基乙叉-TPP自由基中间体;该自由基中间体结合CoA后,加速其向[4Fe-4S]传递电子;通过[4Fe-4S]内部的电子转移,电子最终传递给外部的MtFd,使其转变为还原态MtFd,而结合CoA的自由基中间体转变为乙酰-TPP中间体;最后,CoA被乙酰化并释放,从而完成整个氧化反应[121]。
与MtPFOR同属2-氧酸: 铁氧还蛋白氧化还原酶(2-oxoacid : ferredoxin oxidoreductase)超家族,MmOGOR由两个αβ异二聚体组成四聚体。每个αβ形成一个催化单位,结合1个TPP和1个[4Fe-4S]。其中,α亚基的Glu45在固定CO2的催化反应中发挥重要作用。MmOGOR催化的反应也具有双向性。在正向反应中,还原态MmFd参与MmOGOR固定CO2和琥珀酰CoA转变为α-酮戊二酸,而氧化态MmFd参与其逆向反应。Mm体内存在3个Fd (MmFd1~3),其中MmFd1是MmOGOR催化的最佳电子供受体,具有最高的反应效率[122]。
2.3 3-HP循环绿色非硫细菌(主要是Chloroflexi)以3-HP循环取代CBB循环进行碳同化[8]。1986年,Holo和Sirevåg[123]发现Cfl. aurantiacus既不是通过CBB循环也不是通过rTCA循环来固定CO2;在该菌的细胞提取物中检测不到这两个途径的关键酶。1989年,Holo [124]通过同位素标记和核磁共振技术发现Cfl. aurantiacus在光合自养时分泌3-HP;自养状态下利用乙酸产生的中间代谢物中也有3-HP,而添加氰化钾会使3-HP进一步积累。因此,Cfl. aurantiacus中存在一种以CO2为底物生成乙酰CoA的新代谢途径,而3-HP是其自养固定CO2的中间产物[124]。1993年,Eisenreich等[125]对Cfl. aurantiacus的细胞抽提物进行活性测定,检测出催化反应涉及的酶,从而确定这种固定CO2的新途径。由于3-HP是该循环的标志代谢物,因此命名为3-HP循环。2009年,Zarzycki等[126]通过精巧的实验,鉴定了3-HP循环中所有的特征酶,最终确立了这种固碳途径所催化的反应。
3-HP循环是两个循环耦联的代谢过程,通过13种酶催化19步化学反应,最终吸收3分子HCO3−而生成1分子丙酮酸。第一个循环始于乙酰CoA,固定2分子HCO3−生成1分子乙醛酸。在这个循环中,初始产物乙酰CoA被乙酰CoA羧化酶(acetyl-CoA carboxylase, ACC)催化生成丙二酰CoA,后者被丙二酰CoA还原酶(malonyl-CoA reductase, MCR)还原生成3-HP。在第二个循环中,乙醛酸与第1个循环的中间产物——丙酰CoA发生歧化反应,生成乙酰CoA和丙酮酸[126](图 7)。3-HP循环的一些酶在其他碳代谢通路中也存在,例如ACC催化脂肪酸合成的初始限速反应;催化第4、5、6步反应的丙酰CoA羧化酶、甲基丙二酰CoA异构酶和变位酶都参与丙酸和奇数链脂肪酸的代谢;而催化第8、9步反应的琥珀酸脱氢酶(succinate dehydrogenase, SDH)和富马酸水合酶都属于TCA循环。其他关键酶的编码基因在Roseiflexus属的Rfl. castenholzii、Roseiflexus sp. RS-1及一些海洋变形菌中也存在,这些微生物利用全部或者部分3-HP循环的反应来吸收环境中的各种有机小分子[127],有助于解决日益恶化的环境污染问题[128]。

|
图中带圈的数字代表催化各步反应的酶:①乙酰CoA羧化酶(acetyl-CoA carboxylase, ACC),②丙二酰CoA还原酶(malonyl-CoA reductase, MCR),③丙酰CoA合酶(propionyl-CoA synthase),④丙酰CoA羧化酶(propionyl-CoA carboxylase),⑤甲基丙二酰CoA异构酶(methylmalonyl-CoA epimerase),⑥甲基丙二酰CoA变位酶(methylmalonyl-CoA mutase),⑦琥珀酰CoA﹕(S)-苹果酸CoA转移酶(succinyl-CoA﹕(S)-malate-CoA transferase),⑧琥珀酸脱氢酶(succinate dehydrogenase),⑨富马酸水合酶(fumarate hydratase),⑩ (S)-苹果酰CoA/β-甲基苹果酰CoA/(S)-柠苹酰CoA裂解酶((S)-malyl-CoA/β-methylmalyl-CoA/(S)-citramalyl-CoA lyase, MCL),C1-中康酰CoA水合酶(mesaconyl-C1-CoA hydratase),中康酰CoA C1-C4转移酶(mesaconyl-CoA C1-C4 transferase, MCT),C4-中康酰CoA水合酶(mesaconyl-C4-CoA hydratase)。图中,Cfl. aurantiacus生物素羧化酶(biotin carboxylase, BC1;PDB:8HZ4)以及Rfl. castenholzii MCR (PDB:8HI4)、MCT (PDB:7XKG)和MCL (PDB:4L7Z)的晶体结构已被解析。ACC的结构图展示的是以Cfl. aurantiacus BC1的晶体结构和AlphaFold预测的羧基转移酶(carboxyltransferase, CTα-CTβ)对接后的模型。 图 7 3-HP循环反应步骤示意图 |
然而,国内外对3-HP循环催化机制的研究仍处于起步阶段。ACC是该循环的限速酶,目前还没有关于异质型ACC完整复合物结构和催化机制的报道。绿色非硫细菌Cfl. aurantiacus的异质型ACC通过融合生物素羧化酶(biotin carboxylase, BC)和生物素羧基载体蛋白(biotin carboxyl carrier protein, BCCP)结构域,与羧基转移酶(carboxyltransferase, CTα-CTβ)直接结合,形成的ACC复合物可在体外和重组的大肠杆菌细胞中催化乙酰CoA羧化为丙二酰CoA[129]。对Rfl. castenholzii双功能酶MCR的结构研究发现,全长MCR由两个单体呈“X”交叉形成一个同源二聚体,每个单体包含4个串联的短链脱氢酶/还原酶(short-chain dehydrogenase/reductase, SDR)结构域,其中SDR3和SDR4之间多一个额外的结构域(extra domain, ED)[130]。尽管MCR含有4个SDR结构域,但发挥催化作用的只有SDR1和SDR3,SDR2和SDR4不具备结合底物和辅因子的结构特征,可能起到稳定MCR的构象和辅助催化的作用。以NADPH作为辅因子,MCR的C端结构域(SDR3-ED-4)催化丙二酰CoA还原为丙二酸半醛,而N端结构域(SDR1-2)则催化丙二酸半醛生成3-HP。以上两项工作为重构丙二酰CoA途径用于3-HP的生物合成奠定了基础。
对Rfl. castenholzii苹果酰CoA裂解酶(malyl-CoA lyase, MCL)的结构研究结果表明,MCL的C端结构域在活性口袋的开放与关闭中发挥关键作用,使其兼具(S)-苹果酰CoA/β-甲基苹果酰CoA/(S)-柠苹酰CoA裂解酶三种催化活性,催化乙醛酸的产生、同化和丙酮酸的生成[131]。关于3-HP循环中首次发现的分子内CoA转移酶——中康酰CoA C1~C4转移酶(mesaconyl-CoA C1~C4 transferase, MCT)的结构和催化机制研究,则揭示了MCT催化第12步分子内酰基转移反应的机制[132]。MCT中高度保守的Asp165和Arg47在催化可逆的分子内CoA转移反应中起到关键作用,通过质子化和去质子化推动CoA基团从底物分子的C1端转移到C4端,而Arg314侧链的构象变化有助于区分中康酰C1~C4 CoA。目前,还没有见到关于3-HP循环中其他关键酶结构和催化机制的报道。
2.4 APB固碳途径与生长环境的相关性在演化过程中,各种APB所选择的固碳方式与其生长环境中可利用的O2含量呈现相关性[133]。早期的地球环境被认为是严格厌氧的,随着蓝细菌的出现,大气环境中O2含量逐渐升高[134]。根据大气中O2含量的变化趋势,推测厌氧的固碳途径(如rTCA循环)在演化中首先出现,逐渐演化出耐氧(如3-HP循环)、有氧(如植物、藻类和蓝细菌的CBB循环)的固碳途径[133]。rTCA循环的特征反应是乙酰CoA和琥珀酰CoA的还原,这两步反应都需要对O2敏感的Fdred参与(图 6),说明rTCA循环可能是适应厌氧环境的原始固碳途径。3-HP循环是一种对O2不敏感的固碳途径,出现在兼性厌氧的绿色非硫细菌(Chloroflexi等)中[130]。以乙酰CoA为底物转变为琥珀酰CoA的代谢过程,在3-HP循环中是由依赖生物素的羧化酶(如ACC,丙酰CoA羧化酶等)催化的;但在泉古生菌的二羧酸/4-羟基丁酸(dicarboxylate/4-hydroxybutyrate, DC/4-HB)循环[133, 135]中,却由对O2敏感的PFOR催化[133, 135],说明3-HP循环可能比DC/4-HB循环更适合在耐氧条件下发生。而CBB循环较为特殊[8],Rubisco存在于自养、异养、厌氧和好氧的各种物种中。由于该酶对O2不敏感,也许为生物体从厌氧到有氧生活方式的转变提供了机会[133]。然而,CBB循环的催化效率受其副反应——RuBP加氧反应的影响。在演化过程中,这一副反应在生物体内被控制在可耐受的范围内,以适应大气环境中O2含量的增加和CO2浓度的降低。这种适应性某种程度上可以解释为何CBB循环能够在演化中被保留,并普遍存在于大部分光合细菌、藻类和高等植物中[136]。
生长环境中的电子供体、碳源和氮源等营养成分对APB选择固碳途径有重要影响。在微碱性水环境中,碳酸氢盐的浓度远高于溶解的CO2浓度,可能造成水环境中生活的APB使用碳酸氢盐而不是溶解的CO2进行碳固定[133]。rTCA循环的PEP羧化酶和3-HP循环的ACC、丙酰CoA羧化酶都参与碳酸氢盐的同化(图 6、7),推测可能是APB为适应早期水环境而演化出固碳反应。此外,很多自养微生物或古细菌可能在营养贫瘠条件下,演化出自养和异养混合的生长方式,以充分利用环境中已有的、其他微生物和共生藻类等排出的低量可用有机物进行碳同化,从而获得碳代谢的中间产物来满足生长需求[133]。例如,一些海洋变形菌利用全部或者部分3-HP循环的反应来吸收环境中的各种有机小分子[127];而3-HP循环的很多中间产物可从其他代谢途径获得,如通过二甲基巯基丙酸内盐(dimethylsulfoniopropionate)的裂解反应得到3-HP[137]。相较于rTCA和3-HP循环,CBB循环的中间产物更复杂,最难从其他代谢途径中获得[133]。综上所述,APB固碳途径的演化和选择离不开对生长环境的适应。
3 总结与展望近年来,国内外学者对APB光合电子传递和固碳途径的作用机制进行了深入研究。通过对捕光天线、光合电子传递链的重要元件,如RC、cyt bc1或ACⅢ,以及多种膜周质侧电子受体的结构和功能研究,揭示了紫细菌、绿色非硫细菌Rfl. castenholzii环式电子传递的机制;通过研究CBB、rTCA和3-HP循环等固碳途径的代谢过程,发现了APB丰富多样的固碳方式及其对环境的适应性。然而,由于缺乏有效的遗传操作体系,对绿色非硫细菌光合电子传递和固碳机制的研究仍欠缺生理水平的实验数据支持。同时,对绿硫细菌、绿色非硫细菌和太阳杆菌等APB光系统的研究仍不够深入,各种光合膜蛋白复合物在质膜上的空间排布、动态相互作用以及协同机制尚不清晰。其次,对APB固碳途径涉及的结构和功能各异的多种酶及其催化机制的研究还不够全面,限制对其物质和能量代谢机制的深入理解和应用。
此外,关于APB光合电子传递和固碳途径之间相互联系的了解相对较少。目前已知在APB光合电子传递和rTCA循环中同时发生Fd和醌分子的氧化还原反应[138]。在含有Ⅰ型RC的APB中,Fd和FNR介导光合电子传递的最后一环,将电子从RC传递给NAD(P)+,从而使电子流入NADPH所驱动的碳同化。在rTCA循环中,PFOR催化乙酰CoA和CO2合成丙酮酸;OGOR催化琥珀酰CoA和CO2合成α-酮戊二酸。这两个rTCA循环的特征催化步骤都依赖于Fd驱动的还原反应(图 6)。而在具有Ⅱ型RC的APB中,醌分子介导rTCA循环中间代谢物琥珀酸与富马酸之间的氧化还原反应,将光合电子传递与碳代谢相耦联(图 6)[138]。在绿色非硫细菌中,琥珀酸脱氢酶(SDH)催化3-HP循环的第8步反应,还原琥珀酸生成富马酸,释放的电子传给受体FAD。SDH是3-HP循环中唯一与细菌质膜结合的酶,也是连接氧化磷酸化与电子传递的重要枢纽。但是,目前仍缺乏对APB光合电子传递链、呼吸链和固碳途径相互作用网络和调控机制的深入研究。
针对上述科学问题,可通过建立APB的遗传转化体系,对光合电子传递和固碳途径相关的基因进行编辑。结合多种谱学、生物化学、生物物理学和分子生物学方法,在生理水平上探究光合电子传递、能量转化和固碳代谢的相互作用网络,实现对其光系统能量转化、电子传递和固碳代谢的有效调控和合理利用。其次,针对APB光合膜动态组织方式尚不明确的问题,可以借助原子力显微镜(atomic force microscopy, AFM)或冷冻电子断层扫描(cryo-electron tomography, Cryo-ET)成像,对APB的光合膜进行原位结构观测,揭示光合电子传递链各复合物的空间分布、相互联系和协同机制。再次,针对APB固碳途径中关键酶结构和催化机制的研究,可通过时间分辨的冷冻电镜技术等深入解析其催化过程中的动态构象变化和调控机制,结合酶工程和合成生物学方法,实现对APB固碳途径的高效利用。
植物或细菌的光合复合物能够以接近100%的量子效率进行光化学反应——电荷分离,其高效的光电转换能力使得以此类复合物制备的光生物电化学电池备受关注。目前,APB或其光合元件逐渐被用于开发光生物电化学电池[139-140],但其性能优化、推广和应用等方面仍有待深入而广泛的研究。其次,APB在电子传递过程中所产生的泛醌,也称辅酶Q (coenzyme Q, CoQ),在保护脂蛋白或脂质免受氧化损伤、硫化物氧化等方面发挥重要作用[141-142]。然而,化学合成CoQ的成本高、产量低、环境污染严重。关于生物合成CoQ有文献报告,在限制磷酸盐的条件下,Rba. sphaeroides HY01的CoQ10产量可提高至220 mg/L[143]。Yajima等[144]通过混合培养Rps. palustris JCM 2524和Rba. capsulatus SB 1003菌株获得生产还原性CoQ10的专利技术,且能应用于工业规模的简单生产。除了CoQ,APB在物质代谢过程中产生大量Car。这些Car分子不仅参与辅助捕光,还具有光破坏防御作用,可作为抗氧化剂抵御有害的氧自由基对细胞或者大分子物质的损伤,如紫细菌中的球形烯、螺菌黄质、番茄红素、视紫红质和绿色非硫细菌中的α-和β-胡萝卜素等[145-146]。通过基因工程方法,阻断紫细菌Rsp. rubrum中Car的生物合成途径,可诱导细胞中番茄红素的积累[147];对绿硫细菌Cba. tepidum的遗传改造,则可获得番茄红素和ζ-胡萝卜素,使大量生产Car成为可能[148]。因此,未来还可探索其他不同APB的物质代谢系统在CoQ、Car等保健品生产中的应用。
随着合成生物学和代谢工程技术的发展,APB的固碳途径有望被用于天然固碳途径的改造及人工固碳途径的构建和优化,以提升代谢反应发生的宿主细胞即底盘细胞(chassis cells)的固碳效率。Schwander等[149]在2016年设计出的人工CO2固定途径——巴豆酰CoA/乙基丙二酸CoA/羟基丁酰CoA循环,能够以CO2为底物,从头合成终产物乙醛酸。该循环是第一条完全人工设计的固碳途径,包含来自于古菌、细菌和植物等物种的17种酶。其中,甲基琥珀酰CoA氧化酶(methylsuccinyl-CoA oxidase)来自于紫细菌Rba. sphaeroides,催化琥珀酰CoA转变为中康酰CoA[149]。除此之外,科学家们还将催化3-HP循环前两步反应的酶——ACC和MCR重组,构建出丙二酰CoA途径,已在大肠杆菌、酵母等细胞中实现3-HP的生物合成[150-151]。然而,由于ACC、MCR在细胞中表达水平低,以及MCR的N端和C端结构域催化活性的不平衡[152],所有包含丙二酰CoA途径的细胞合成3-HP的产量都不高,限制了3-HP的生物合成及其工业应用[130, 152]。为了解决这一问题,未来可通过优化关键酶的活性和适配性,调控底盘细胞的代谢途径等,提高3-HP的生物合成效率。其次,通过合成生物学方法将3-HP循环引入其他物种,在不干扰其他需氧固碳过程的前提下,可同化和吸收其他代谢通路的副产品,从而改善物种原有的固碳能力。例如,通过代谢工程方法,将上述第2个循环的6个关键酶在蓝细菌中异源表达,可以同化光呼吸过程中产生的乙醛酸,额外固定1分子HCO3−并生成丙酮酸,降低光呼吸过程中碳和氮的损失[153]。
| [1] |
Olson JM, Blankenship RE. Thinking about the evolution of photosynthesis. Photosynth Res, 2004, 80: 373-86. |
| [2] |
Hohmann-Marriott MF, Blankenship RE. Evolution of photosynthesis. Annu Rev Plant Biol, 2011, 62: 515-48. |
| [3] |
Croce R, van Amerongen H. Natural strategies for photosynthetic light harvesting. Nat Chem Biol, 2014, 10: 492-501. |
| [4] |
Blankenship RE. Early evolution of photosynthesis. Plant Physiol, 2010, 154: 434-8. |
| [5] |
Singh H. Desiccation and radiation stress tolerance in cyanobacteria. J Basic Microbiol, 2018, 58: 813-26. |
| [6] |
鲜文东, 张潇橦, 李文均. 绿弯菌的研究现状及展望. 微生物学报, 2020, 60: 1801-20. |
| [7] |
杨素萍, 林志华, 崔小华, 等. 不产氧光合细菌的分类学进展. 微生物学报, 2008, 48: 1562-6. |
| [8] |
胡章, 李思东. 生物固碳途径及其进化. 广东化工, 2010, 37: 62-3+77. |
| [9] |
Chen JH, Wu H, Xu C, et al. Architecture of the photosynthetic complex from a green sulfur bacterium. Science, 2020, 370: eabb6350. |
| [10] |
卓民权, 赵春贵, 程茜茹, 等. 紫细菌光合色素指纹图谱的建立与色素分析. 微生物学报, 2012, 52: 760-8. |
| [11] |
Yang J, Yin L, Lessner FH, et al. Genes essential for phototrophic growth by a purple alphaproteobacterium. Environ Microbiol, 2017, 19: 3567-78. |
| [12] |
张海涵, 王燕, 刘凯文, 等. 水域生境好氧不产氧光合细菌(AAPB)研究进展. 湖泊科学, 2020, 32: 1585-98. |
| [13] |
Tang KH, Feng X, Tang YJ, et al. Carbohydrate metabolism and carbon fixation in Roseobacter denitrificans OCh114. PLoS One, 2009, 4: e7233. |
| [14] |
Jin H, Chen L, Wang J, et al. Engineering biofuel tolerance in non-native producing microorganisms. Biotechnol Adv, 2014, 32: 541-8. |
| [15] |
Lassen LM, Nielsen AZ, Ziersen B, et al. Redirecting photosynthetic electron flow into light-driven synthesis of alternative products including high-value bioactive natural compounds. ACS Synth Biol, 2014, 3: 1-12. |
| [16] |
Nguyen K, Bruce BD. Growing green electricity: progress and strategies for use of photosystem Ⅰ for sustainable photovoltaic energy conversion. Biochim Biophys Acta, 2014, 1837: 1553-66. |
| [17] |
Yu J. Bio-based products from solar energy and carbon dioxide. Trends Biotechnol, 2014, 32: 5-10. |
| [18] |
Rochaix JD. Regulation of photosynthetic electron transport. Biochim Biophys Acta, 2011, 1807: 375-83. |
| [19] |
Noy D, Moser CC, Dutton PL. Design and engineering of photosynthetic light-harvesting and electron transfer using length, time, and energy scales. Biochim Biophys Acta, 2006, 1757: 90-105. |
| [20] |
Allen JF. Cyclic, pseudocyclic and noncyclic photophosphorylation: new links in the chain. Trends Plant Sci, 2003, 8: 15-9. |
| [21] |
Govindjee, Shevela D, Björn LO. Evolution of the Z-scheme of photosynthesis: a perspective. Photosynth Res, 2017, 133: 5-15. |
| [22] |
Mullineaux CW, Liu LN. Membrane dynamics in phototrophic bacteria. Annu Rev Microbiol, 2020, 74: 633-54. |
| [23] |
Lokstein H, Renger G, Gotze JP. Photosynthetic light-harvesting (antenna) complexes-structures and functions. Molecules, 2021, 26: 3378. |
| [24] |
Hu X, Ritz T, Damjanovic A, et al. Photosynthetic apparatus of purple bacteria. Q Rev Biophys, 2002, 35: 1-62. |
| [25] |
Seo D, Sakurai H. Purification and characterization of ferredoxin-NAD(P)+ reductase from the green sulfur bacterium Chlorobium tepidum. Biochim Biophys Acta, 2002, 1597: 123-32. |
| [26] |
Gardiner AT, Cogdell RJ, Takaichi S. The effect of growth conditions on the light-harvesting apparatus in Rhodopseudomonas acidophila. Photosynth Res, 1993, 38: 159-67. |
| [27] |
McLuskey K, Prince SM, Cogdell RJ, et al. The crystallographic structure of the B800-820 LH3 light-harvesting complex from the purple bacteria Rhodopseu-domonas acidophila strain 7050. Biochemistry, 2001, 40: 8783-9. |
| [28] |
Yu LJ, Suga M, Wang-Otomo ZY, et al. Structure of photosynthetic LH1-RC supercomplex at 1.9 A resolution. Nature, 2018, 556: 209-13. |
| [29] |
Qian P, Siebert CA, Wang P, et al. Cryo-EM structure of the Blastochloris viridis LH1-RC complex at 2.9 A. Nature, 2018, 556: 203-8. |
| [30] |
Tani K, Kobayashi K, Hosogi N, et al. A Ca2+-binding motif underlies the unusual properties of certain photosynthetic bacterial core light-harvesting complexes. J Biol Chem, 2022, 298: 101967. |
| [31] |
Tani K, Kanno R, Makino Y, et al. Cryo-EM structure of a Ca2+-bound photosynthetic LH1-RC complex containing multiple αβ-polypeptides. Nat Commun, 2020, 11: 4955. |
| [32] |
Tani K, Kanno R, Ji XC, et al. Cryo-EM structure of the photosynthetic LH1-RC complex from Rhodospirillum rubrum. Biochemistry, 2021, 60: 2483-91. |
| [33] |
Tani K, Kanno R, Kurosawa K, et al. An LH1-RC photocomplex from an extremophilic phototroph provides insight into origins of two photosynthesis proteins. Commun Biol, 2022, 5: 1197. |
| [34] |
Bracun L, Yamagata A, Christianson BM, et al. Cryo-EM structure of the photosynthetic RC-LH1-PufX supercomplex at 2.8-A resolution. Sci Adv, 2021, 7: eabf8864. |
| [35] |
Bracun L, Yamagata A, Christianson BM, et al. Cryo-EM structure of a monomeric RC-LH1-PufX supercomplex with high-carotenoid content from Rhodobacter capsulatus. Structure, 2023, 31: 318-28.e3. |
| [36] |
Swainsbury DJK, Qian P, Jackson PJ, et al. Structures of Rhodopseudomonas palustris RC-LH1 complexes with open or closed quinone channels. Sci Adv, 2021, 7: eabe2631. |
| [37] |
Cao P, Bracun L, Yamagata A, et al. Structural basis for the assembly and quinone transport mechanisms of the dimeric photosynthetic RC-LH1 supercomplex. Nat Commun, 2022, 13: 1977. |
| [38] |
Chenchiliyan M, Timpmann K, Jalviste E, et al. Dimerization of core complexes as an efficient strategy for energy trapping in Rhodobacter sphaeroides. Biochim Biophys Acta, 2016, 1857: 634-42. |
| [39] |
Geyer T. On the effects of PufX on the absorption properties of the light-harvesting complexes of Rhodobacter sphaeroides. Biophys J, 2007, 93: 4374-81. |
| [40] |
Gunther LM, Jendrny M, Bloemsma EA, et al. Structure of light-harvesting aggregates in individual chlorosomes. J Phys Chem B, 2016, 120: 5367-76. |
| [41] |
Kouyianou K, De Bock PJ, Muller SA, et al. The chlorosome of Chlorobaculum tepidum: size, mass and protein composition revealed by electron microscopy, dynamic light scattering and mass spectrometry-driven proteomics. Proteomics, 2011, 11: 2867-80. |
| [42] |
Pedersen MO, Underhaug J, Dittmer J, et al. The three-dimensional structure of CsmA: a small antenna protein from the green sulfur bacterium Chlorobium tepidum. FEBS Lett, 2008, 582: 2869-74. |
| [43] |
Magdaong NCM, Niedzwiedzki DM, Saer RG, et al. Excitation energy transfer kinetics and efficiency in phototrophic green sulfur bacteria. Biochim Biophys Acta Bioenerg, 2018, 1859: 1180-90. |
| [44] |
Bina D, Gardian Z, Vacha F, et al. Supramolecular organization of photosynthetic membrane proteins in the chlorosome-containing bacterium Chloroflexus aurantiacus. Photosynth Res, 2014, 122: 13-21. |
| [45] |
Golecki J, Oelze JJAom. Quantitative relationship between bacteriochlorophyll content, cytoplasmic membrane structure and chlorosome size in Chloroflexus aurantiacus. Arch Microbiol, 1987, 148: 236-41. |
| [46] |
Gomez Maqueo Chew A, Frigaard NU, Bryant DA. Bacteriochlorophyllide c C-82 and C-121 methyltrans-ferases are essential for adaptation to low light in Chlorobaculum tepidum. J Bacteriol, 2007, 189: 6176-84. |
| [47] |
Hanada S, Takaichi S, Matsuura K, et al. Roseiflexus castenholzii gen. nov., sp. nov., a thermophilic, filamentous, photosynthetic bacterium that lacks chlorosomes. Int J Syst Evol Microbiol, 2002, 52: 187-93. |
| [48] |
Collins AM, Kirmaier C, Holten D, et al. Kinetics and energetics of electron transfer in reaction centers of the photosynthetic bacterium Roseiflexus castenholzii. Biochim Biophys Acta, 2011, 1807: 262-9. |
| [49] |
Xin Y, Pan J, Collins AM, et al. Excitation energy transfer and trapping dynamics in the core complex of the filamentous photosynthetic bacterium Roseiflexus castenholzii. Photosynth Res, 2012, 111: 149-56. |
| [50] |
Xin J, Shi Y, Zhang X, et al. Carotenoid assembly regulates quinone diffusion and the Roseiflexus castenholzii reaction center-light harvesting complex architecture. eLife, 2023, 12: e88951. |
| [51] |
Xin Y, Shi Y, Niu T, et al. Cryo-EM structure of the RC-LH core complex from an early branching photosynthetic prokaryote. Nat Commun, 2018, 9: 1568. |
| [52] |
Allen J, Williams JJFl. Photosynthetic reaction centers. FEBS Lett, 1998, 438: 5-9. |
| [53] |
Schubert WD, Klukas O, Saenger W, et al. A common ancestor for oxygenic and anoxygenic photosynthetic systems: a comparison based on the structural model of photosystem Ⅰ. J Mol Biol, 1998, 280: 297-314. |
| [54] |
Cardona T. A fresh look at the evolution and diversification of photochemical reaction centers. Photosynth Res, 2015, 126: 111-34. |
| [55] |
Gisriel C, Sarrou I, Ferlez B, et al. Structure of a symmetric photosynthetic reaction center-photosystem. Science, 2017, 357: 1021-5. |
| [56] |
Puskar R, Du Truong C, Swain K, et al. Molecular asymmetry of a photosynthetic supercomplex from green sulfur bacteria. Nat Commun, 2022, 13: 5824. |
| [57] |
Xie H, Lyratzakis A, Khera R, et al. Cryo-EM structure of the whole photosynthetic reaction center apparatus from the green sulfur bacterium Chlorobaculum tepidum. Proc Natl Acad Sci U S A, 2023, 120: e2216734120. |
| [58] |
Cardona T, Sedoud A, Cox N, et al. Charge separation in photosystem Ⅱ: a comparative and evolutionary overview. Biochim Biophys Acta, 2012, 1817: 26-43. |
| [59] |
Lupo D, Ghosh RJJob. The reaction center H subunit is not required for high levels of light-harvesting complex 1 in Rhodospirillum rubrum mutants. J Bacteriol, 2004, 186: 5585-95. |
| [60] |
Wei RJ, Zhang Y, Mao J, et al. Comparison of proton transfer paths to the QA and QB sites of the Rb. sphaeroides photosynthetic reaction centers. Photosynth Res, 2022, 152: 153-65. |
| [61] |
Qian P, Gardiner AT, Simova I, et al. 2.4-A structure of the double-ring Gemmatimonas phototrophica photosystem. Sci Adv, 2022, 8: eabk3139. |
| [62] |
Yu L, Min Z, Liu M, et al. A cytochrome c551 mediates the cyclic electron transport chain of the anoxygenic phototrophic bacterium Roseiflexus castenholzii. Plant Commun, 2024, 5: 100715. |
| [63] |
Kawakami T, Yu LJ, Liang T, et al. Crystal structure of a photosynthetic LH1-RC in complex with its electron donor HiPIP. Nat Commun, 2021, 12: 1104. |
| [64] |
Xia D, Yu CA, Kim H, et al. Crystal structure of the cytochrome bc1 complex from bovine heart mitochondria. Science, 1997, 277: 60-6. |
| [65] |
Iwata S, Lee JW, Okada K, et al. Complete structure of the 11-subunit bovine mitochondrial cytochrome bc1 complex. Science, 1998, 281: 64-71. |
| [66] |
Crofts AR, Berry EA. Structure and function of the cytochrome bc1 complex of mitochondria and photosynthetic bacteria. Curr Opin Struct Biol, 1998, 8: 501-9. |
| [67] |
Lange C, Hunte C. Crystal structure of the yeast cytochrome bc1 complex with its bound substrate cytochrome c. Proc Natl Acad Sci U S A, 2002, 99: 2800-5. |
| [68] |
Solmaz SR, Hunte C. Structure of complex Ⅲ with bound cytochrome c in reduced state and definition of a minimal core interface for electron transfer. J Biol Chem, 2008, 283: 17542-9. |
| [69] |
Yu CA, Xia D, Kim H, et al. Structural basis of functions of the mitochondrial cytochrome bc1 complex. Biochim Biophys Acta, 1998, 1365: 151-8. |
| [70] |
Zhang Z, Huang L, Shulmeister VM, et al. Electron transfer by domain movement in cytochrome bc1. Nature, 1998, 392: 677-84. |
| [71] |
Esser L, Gong X, Yang S, et al. Surface-modulated motion switch: capture and release of iron-sulfur protein in the cytochrome bc1 complex. Proc Natl Acad Sci U S A, 2006, 103: 13045-50. |
| [72] |
Gao X, Wen X, Esser L, et al. Structural basis for the quinone reduction in the bc1 complex: a comparative analysis of crystal structures of mitochondrial cytochrome bc1 with bound substrate and inhibitors at the Qi site. Biochemistry, 2003, 42: 9067-80. |
| [73] |
Palsdottir H, Lojero CG, Trumpower BL, et al. Structure of the yeast cytochrome bc1 complex with a hydroxyquinone anion Qo site inhibitor bound. J Biol Chem, 2003, 278: 31303-11. |
| [74] |
Pereira MM, Antunes AM, Nunes OC, et al. A membrane-bound HIPIP type center in the thermohalophile Rhodothermus marinus. FEBS Lett, 1994, 352: 327-30. |
| [75] |
Pereira MM, Carita JN, Teixeira M. Membrane-bound electron transfer chain of the thermohalophilic bacterium Rhodothermus marinus: a novel multihemic cytochrome bc, a new complex Ⅲ. Biochemistry, 1999, 38: 1268-75. |
| [76] |
Yanyushin MF. Fractionation of cytochromes of phototrophically grown Chloroflexus aurantiacus. Is there a cytochrome bc complex among them?. FEBS Lett, 2002, 512: 125-8. |
| [77] |
Majumder EL, King JD, Blankenship RE. Alternative complex Ⅲ from phototrophic bacteria and its electron acceptor auracyanin. Biochim Biophys Acta, 2013, 1827: 1383-91. |
| [78] |
Yanyushin MF, del Rosario MC, Brune DC, et al. New class of bacterial membrane oxidoreductases. Biochemistry, 2005, 44: 10037-45. |
| [79] |
Pereira MM, Refojo PN, Hreggvidsson GO, et al. The alternative complex Ⅲ from Rhodothermus marinus -- a prototype of a new family of quinol: electron acceptor oxidoreductases. FEBS Lett, 2007, 581: 4831-5. |
| [80] |
Gao X, Xin Y, Blankenship RE. Enzymatic activity of the alternative complex Ⅲ as a menaquinol: auracyanin oxidoreductase in the electron transfer chain of Chloroflexus aurantiacus. FEBS Lett, 2009, 583: 3275-9. |
| [81] |
Refojo PN, Sousa FL, Teixeira M, et al. The alternative complex Ⅲ: a different architecture using known building modules. Biochim Biophys Acta, 2010, 1797: 1869-76. |
| [82] |
Refojo PN, Teixeira M, Pereira MM. The alternative complex Ⅲ of Rhodothermus marinus and its structural and functional association with caa3 oxygen reductase. Biochim Biophys Acta, 2010, 1797: 1477-82. |
| [83] |
Sun C, Benlekbir S, Venkatakrishnan P, et al. Structure of the alternative complex Ⅲ in a supercomplex with cytochrome oxidase. Nature, 2018, 557: 123-6. |
| [84] |
Sousa JS, Calisto F, Langer JD, et al. Structural basis for energy transduction by respiratory alternative complex Ⅲ. Nat Commun, 2018, 9: 1728. |
| [85] |
Shi Y, Xin Y, Wang C, et al. Cryo-EM structures of the air-oxidized and dithionite-reduced photosynthetic alternative complex Ⅲ from Roseiflexus castenholzii. Sci Adv, 2020, 6: eaba2739. |
| [86] |
Xin J, Min Z, Yu L, et al. Cryo-EM structure of HQNO-bound alternative complex Ⅲ from the anoxygenic phototrophic bacterium Chloroflexus aurantiacus. Plant Cell, 2024, koae029. |
| [87] |
Bertini I, Cavallaro G, Rosato A. Cytochrome c: occurrence and functions. Chem Rev, 2006, 106: 90-115. |
| [88] |
Ambler RP. Sequence variability in bacterial cytochromes c. Biochim Biophys Acta, 1991, 1058: 42-7. |
| [89] |
Santucci R, Sinibaldi F, Cozza P, et al. Cytochrome c: an extreme multifunctional protein with a key role in cell fate. Int J Biol Macromol, 2019, 136: 1237-46. |
| [90] |
Dracheva SM, Drachev LA, Konstantinov AA, et al. Electrogenic steps in the redox reactions catalyzed by photosynthetic reaction-centre complex from Rhodopseu-domonas viridis. Eur J Biochem, 1988, 171: 253-64. |
| [91] |
Azai C, Tsukatani Y, Itoh S, et al. C-type cytochromes in the photosynthetic electron transfer pathways in green sulfur bacteria and heliobacteria. Photosynth Res, 2010, 104: 189-99. |
| [92] |
Baymann F, Nitschke W. Heliobacterial Rieske/cytb complex. Photosynth Res, 2010, 104: 177-87. |
| [93] |
Meyer TE, Cusanovich MA. Discovery and characterization of electron transfer proteins in the photosynthetic bacteria. Photosynth Res, 2003, 76: 111-26. |
| [94] |
Azai C, Tsukatani Y, Harada J, et al. Sulfur oxidation in mutants of the photosynthetic green sulfur bacterium Chlorobium tepidum devoid of cytochrome c-554 and SoxB. Photosynth Res, 2009, 100: 57-65. |
| [95] |
Pierson BK, Castenholz RW. A phototrophic gliding filamentous bacterium of hot springs, Chloroflexus aurantiacus, gen. and sp. nov. Arch Microbiol, 1974, 100: 5-24. |
| [96] |
Tang KH, Barry K, Chertkov O, et al. Complete genome sequence of the filamentous anoxygenic phototrophic bacterium Chloroflexus aurantiacus. BMC Genomics, 2011, 12: 334. |
| [97] |
McManus JD, Brune DC, Han J, et al. Isolation, characterization, and amino acid sequences of auracyanins, blue copper proteins from the green photosynthetic bacterium Chloroflexus aurantiacus. J Biol Chem, 1992, 267: 6531-40. |
| [98] |
Van Driessche G, Hu W, Van de Werken G, et al. Auracyanin A from the thermophilic green gliding photosynthetic bacterium Chloroflexus aurantiacus represents an unusual class of small blue copper proteins. Protein Sci, 1999, 8: 947-57. |
| [99] |
Tsukatani Y, Nakayama N, Shimada K, et al. Characterization of a blue-copper protein, auracyanin, of the filamentous anoxygenic phototrophic bacterium Roseiflexus castenholzii. Arch Biochem Biophys, 2009, 490: 57-62. |
| [100] |
Bond CS, Blankenship RE, Freeman HC, et al. Crystal structure of auracyanin, a "blue" copper protein from the green thermophilic photosynthetic bacterium Chloroflexus aurantiacus. J Mol Biol, 2001, 306: 47-67. |
| [101] |
Lee M, del Rosario MC, Harris HH, et al. The crystal structure of auracyanin A at 1.85 A resolution: the structures and functions of auracyanins A and B, two almost identical "blue" copper proteins, in the photosynthetic bacterium Chloroflexus aurantiacus. J Biol Inorg Chem, 2009, 14: 329-45. |
| [102] |
Cao L, Bryant DA, Schepmoes AA, et al. Comparison of Chloroflexus aurantiacus strain J-10-fl proteomes of cells grown chemoheterotrophically and photoheterotrophically. Photosynth Res, 2012, 110: 153-68. |
| [103] |
King JD, McIntosh CL, Halsey CM, et al. Metalloproteins diversified: the auracyanins are a family of cupredoxins that stretch the spectral and redox limits of blue copper proteins. Biochemistry, 2013, 52: 8267-75. |
| [104] |
Wang C, Xin Y, Min Z, et al. Structural basis underlying the electron transfer features of a blue copper protein auracyanin from the photosynthetic bacterium Roseiflexus castenholzii. Photosynth Res, 2020, 143: 301-14. |
| [105] |
Govindjee G, Nonomura A, Lichtenthaler HK. Remembering Melvin Calvin (1911–1997), a highly versatile scientist of the 20th century. Photosynth Res, 2019, 143: 1-11. |
| [106] |
Fuller R, Gibbs M. Intracellular and phylogenetic distribution of ribulose 1, 5-diphosphate carboxylase and D-glyceraldehyde-3-phosphate dehydrogenases. Plant Physiol, 1959, 34: 324. |
| [107] |
Akazawa T, Sugiyama T, Kataoka H. Further studies on ribulose-l, 5-diphosphate carboxylase from Rhodopseudomonas spheroides and Rhodospirillum rubrum. Plant Cell Physiol, 1970, 11: 541-50. |
| [108] |
Tabita FR, Caruso P, Whitman W. Facile assay of enzymes unique to the Calvin cycle in intact cells, with special reference to ribulose 1, 5-bisphosphate carboxylase. Anal Biochem, 1978, 84: 462-72. |
| [109] |
常青, 张展华, 刘雅琪, 等. 化能自养微生物的固碳功能及人工调控策略. 科学通报, 2023, 68: 3792-808. |
| [110] |
周昱婕, 李霞, 陈根云, 等. 植物RuBisCO研究进展. 中国科学: 生命科学, 2023, 53: 1213-29. |
| [111] |
West-Roberts JA, Matheus-Carnevali PB, Schoelmerich MC, et al. The Chloroflexi supergroup is metabolically diverse and representatives have novel genes for non-photosynthesis based CO2 fixation. BioRxiv, 2021. |
| [112] |
Okano Y, Mizohata E, Xie Y, et al. X-ray structure of Galdieria Rubisco complexed with one sulfate ion per active site. FEBS Lett, 2002, 527: 33-6. |
| [113] |
Hanson TE, Tabita FR. A ribulose-1, 5-bisphosphate carboxylase/oxygenase (RubisCO)-like protein from Chlorobium tepidum that is involved with sulfur metabolism and the response to oxidative stress. Proc Natl Acad Sci U S A, 2001, 98: 4397-402. |
| [114] |
Evans MC, Buchanan BB, Arnon DI. A new ferredoxin-dependent carbon reduction cycle in a photosynthetic bacterium. Proc Natl Acad Sci U S A, 1966, 55: 928-34. |
| [115] |
Kandler O. The position of microorganisms in the global phylogenetic system of three domains. Mycoses, 1994, 37: 13-27. |
| [116] |
Schönheit P, Schäfer T. Metabolism of hyperthermophiles. World J Microbiol Biotechnol, 1995, 11: 26-57. |
| [117] |
Bachofen R, Buchanan BB, Arnon DI. Ferredoxin as a reductant in pyruvate synthesis by a bacterial extract. Proc Natl Acad Sci U S A, 1964, 51: 690-4. |
| [118] |
Buchanan BB, Evans M, Arnon DI. Ferredoxin-dependent carbon assimilation in Rhodospirillum rubrum. Arch Mikrobiol, 1967, 59: 32-40. |
| [119] |
Buchanan BB, Evans M. The synthesis of α-ketoglutarate from succinate and carbon dioxide by a subcellular preparation of a photosynthetic bacterium. Proc Natl Acad Sci U S A, 1965, 54: 1212-8. |
| [120] |
Buchanan BB, Arnon DI. A reverse KREBS cycle in photosynthesis: consensus at last. Photosynth Res, 1990, 24: 47-53. |
| [121] |
Chen PYT, Aman H, Can M, et al. Binding site for coenzyme A revealed in the structure of pyruvate: ferredoxin oxidoreductase from Moorella thermoacetica. Proc Natl Acad Sci U S A, 2018, 115: 3846-51. |
| [122] |
Chen PYT, Li B, Drennan CL, et al. A reverse TCA cycle 2-oxoacid: ferredoxin oxidoreductase that makes CC bonds from CO2. Joule, 2019, 3: 595-611. |
| [123] |
Holo H, Sirevåg R. Autotrophic growth and CO2 fixation of Chloroflexus aurantiacus. Arch Microbiol, 1986, 145: 173-80. |
| [124] |
Holo H. Chloroflexus aurantiacus secretes 3-hydroxypropionate, a possible intermediate in the assimilation of CO2 and acetate. Arch Microbiol, 1989, 151: 252-6. |
| [125] |
Eisenreich W, Strauss G, Werz U, et al. Retrobiosynthetic analysis of carbon fixation in the phototrophic eubacterium Chloroflexus aurantiacus. Eur J Biochem, 1993, 215: 619-32. |
| [126] |
Zarzycki J, Brecht V, Muller M, et al. Identifying the missing steps of the autotrophic 3-hydroxypropionate CO2 fixation cycle in Chloroflexus aurantiacus. Proc Natl Acad Sci U S A, 2009, 106: 21317-22. |
| [127] |
Zarzycki J, Fuchs G. Coassimilation of organic substrates via the autotrophic 3-hydroxypropionate bi-cycle in Chloroflexus aurantiacus. Appl Environ Microbiol, 2011, 77: 6181-8. |
| [128] |
McKinlay JB, Harwood CS. Carbon dioxide fixation as a central redox cofactor recycling mechanism in bacteria. Proc Natl Acad Sci U S A, 2010, 107: 11669-75. |
| [129] |
Shen J, Wu W, Wang K, et al. Chloroflexus aurantiacus acetyl-CoA carboxylase evolves fused biotin carboxylase and biotin carboxyl carrier protein to complete carboxylation activity. mBio, 2024, 15: e0341423. |
| [130] |
Zhang X, Xin J, Wang Z, et al. Structural basis of a bi-functional malonyl-CoA reductase (MCR) from the photosynthetic green non-sulfur bacterium Roseiflexus castenholzii. mBio, 2023, 14: e0323322. |
| [131] |
Tang W, Wang Z, Zhang C, et al. The C-terminal domain conformational switch revealed by the crystal structure of malyl-CoA lyase from Roseiflexus castenholzii. Biochem Biophys Res Commun, 2019, 518: 72-9. |
| [132] |
Min Z, Zhang X, Wu W, et al. Crystal structure of an intramolecular mesaconyl-coenzyme A transferase from the 3-hydroxypropionic acid cycle of Roseiflexus castenholzii. Front Microbiol, 2022, 13: 923367. |
| [133] |
Fuchs G. Alternative pathways of carbon dioxide fixation: insights into the early evolution of life?. Annu Rev Microbiol, 2011, 65: 631-58. |
| [134] |
Demoulin CF, Lara YJ, Cornet L, et al. Cyanobacteria evolution: insight from the fossil record. Free Radic Biol Med, 2019, 140: 206-23. |
| [135] |
Huber H, Gallenberger M, Jahn U, et al. A dicarboxylate/ 4-hydroxybutyrate autotrophic carbon assimilation cycle in the hyperthermophilic Archaeum Ignicoccus hospitalis. Proc Natl Acad Sci U S A, 2008, 105: 7851-6. |
| [136] |
Tcherkez GGB, Farquhar GD, Andrews TJ. Despite slow catalysis and confused substrate specificity, all ribulose bisphosphate carboxylases may be nearly perfectly optimized. Proc Natl Acad Sci U S A, 2006, 103: 7246-51. |
| [137] |
Todd JD, Curson ARJ, Nikolaidou-Katsaraidou N, et al. Molecular dissection of bacterial acrylate catabolism -- unexpected links with dimethylsulfoniopropionate catabolism and dimethyl sulfide production. Environ Microbiol, 2010, 12: 327-43. |
| [138] |
Klamt S, Grammel H, Straube R, et al. Modeling the electron transport chain of purple non-sulfur bacteria. Mol Syst Biol, 2008, 4: 156. |
| [139] |
Qi X, Ren Y, Liang P, et al. New insights in photosynthetic microbial fuel cell using anoxygenic phototrophic bacteria. Bioresour Technol, 2018, 258: 310-7. |
| [140] |
Du J, Xin J, Liu M, et al. Preparation of photo-bioelectrochemical cells with the RC-LH complex from Roseiflexus castenholzii. Front Microbiol, 2022, 13: 928046. |
| [141] |
Lankin VZ, Tikhaze AK, Kapel'ko VI, et al. Mechanisms of oxidative modification of low density lipoproteins under conditions of oxidative and carbonyl stress. Biochemistry (Mosc), 2007, 72: 1081-90. |
| [142] |
Lagoutte E, Mimoun S, Andriamihaja M, et al. Oxidation of hydrogen sulfide remains a priority in mammalian cells and causes reverse electron transfer in colonocytes. Biochim Biophys Acta, 2010, 1797: 1500-11. |
| [143] |
Lee SQ, Tan TS, Kawamukai M, et al. Cellular factories for coenzyme Q10 production. Microb Cell Fact, 2017, 16: 39. |
| [144] |
Yajima K, Kato T, Kanda A, et al. Processes for producing coenzyme Q10: United States, US2011136191 [P/OL]. 2011-02-03. https://europepmc.org/article/PAT/US2011136191
|
| [145] |
Frank HA, Cogdell RJ. Carotenoids in photosynthesis. Photochem Photobiol, 1996, 63: 257-64. |
| [146] |
Frigaard NU. Biotechnology of anoxygenic phototrophic bacteria. Adv Biochem Eng Biotechnol, 2016, 156: 139-54. |
| [147] |
Wang GS, Grammel H, Abou-Aisha K, et al. High-level production of the industrial product lycopene by the photosynthetic bacterium Rhodospirillum rubrum. Appl Environ Microbiol, 2012, 78: 7205-15. |
| [148] |
Frigaard NU, Maresca JA, Yunker CE, et al. Genetic manipulation of carotenoid biosynthesis in the green sulfur bacterium Chlorobium tepidum. J Bacteriol, 2004, 186: 5210-20. |
| [149] |
Schwander T, Schada von Borzyskowski L, Burgener S, et al. A synthetic pathway for the fixation of carbon dioxide in vitro. Science, 2016, 354: 900-4. |
| [150] |
Chen Y, Bao J, Kim IK, et al. Coupled incremental precursor and co-factor supply improves 3-hydroxypropionic acid production in Saccharomyces cerevisiae. Metab Eng, 2014, 22: 104-9. |
| [151] |
Cheng Z, Jiang J, Wu H, et al. Enhanced production of 3-hydroxypropionic acid from glucose via malonyl-CoA pathway by engineered Escherichia coli. Bioresour Technol, 2016, 200: 897-904. |
| [152] |
Liu C, Ding Y, Zhang R, et al. Functional balance between enzymes in malonyl-CoA pathway for 3-hydroxypropionate biosynthesis. Metab Eng, 2016, 34: 104-11. |
| [153] |
Shih PM, Zarzycki J, Niyogi KK, et al. Introduction of a synthetic CO2-fixing photorespiratory bypass into a cyanobacterium. J Biol Chem, 2014, 289: 9493-500. |
 2024, Vol. 36
2024, Vol. 36 

 徐晓玲,杭州师范大学基础医学院教授。2008年毕业于清华大学生物科学与技术系,获理学博士学位;2009-2013年在美国Scripps研究所从事博士后研究,2013年任职于杭州师范大学,建立“蛋白质结构与分子设计”课题组。近年来,围绕“光合细菌的电子传递和能量代谢机制”,系统揭示了进化上非常古老的一种丝状不产氧光合细菌——光合玫瑰菌在光反应阶段通过环式电子传递链吸收和转化光能,在暗反应阶段通过3-羟基丙酸循环固定二氧化碳的分子机理,为研究此类细菌的物质和能量代谢机制及其合成生物学应用奠定了基础。在Science Advances、Nature Communications、The Plant Cell、eLife、mBio等期刊发表研究论文38篇,主持国家自然科学基金项目(4项),浙江省自然科学基金杰出青年项目等多项课题,作为参与者获得国家科技进步奖二等奖1项,入选浙江省高层次人才特殊支持计划青年拔尖人才等,现任Communications Biology期刊编委
徐晓玲,杭州师范大学基础医学院教授。2008年毕业于清华大学生物科学与技术系,获理学博士学位;2009-2013年在美国Scripps研究所从事博士后研究,2013年任职于杭州师范大学,建立“蛋白质结构与分子设计”课题组。近年来,围绕“光合细菌的电子传递和能量代谢机制”,系统揭示了进化上非常古老的一种丝状不产氧光合细菌——光合玫瑰菌在光反应阶段通过环式电子传递链吸收和转化光能,在暗反应阶段通过3-羟基丙酸循环固定二氧化碳的分子机理,为研究此类细菌的物质和能量代谢机制及其合成生物学应用奠定了基础。在Science Advances、Nature Communications、The Plant Cell、eLife、mBio等期刊发表研究论文38篇,主持国家自然科学基金项目(4项),浙江省自然科学基金杰出青年项目等多项课题,作为参与者获得国家科技进步奖二等奖1项,入选浙江省高层次人才特殊支持计划青年拔尖人才等,现任Communications Biology期刊编委